文章信息
- 安云鹤, 程小艳, 田彦捷, 马凯, 高丽娟, 武会娟.
- AN Yun-he, CHENG Xiao-yan, TIAN Yan-jie, MA Kai, GAO Li-juan, WU Hui-juan.
- DNA提取对微生物多样性测序分析的影响
- Influence of DNA Extraction Method on Microbial Diversity Analysis Through Next Generation Sequencing
- 中国生物工程杂志, 2017, 37(11): 12-18
- China Biotechnology, 2017, 37(11): 12-18
- http://dx.doi.org/DOI:10.13523/j.cb.20171103
-
文章历史
- 收稿日期: 2017-09-20
2. 北京市基因测序与工程技术研究中心 北京 100094
2. Beijing Engineering Technique Research Center for Gene Sequencing & Function Analysis, Beijing 100094, China
目前,能够在实验室条件下培养的微生物种类占自然界中微生物总数的不到1%,相当多的菌种因此尚未被人类所认识[1]。随着分子生物学技术的发展,PCR指纹技术[2-3],以及DNA文库[4-5]、RAPD[6-7]、ITS[8-9]、DGGE[10-11]等技术相继被广泛应用于微生物群落结构和基因资源的开发利用,从而摆脱了不可培养微生物的限制。尤其近几年,新一代测序技术以高通量、速度快、低成本等特点迅速发展,在微生物多样性研究领域,正在逐步取代传统的分子生物学方法[12-16],从而进一步丰富拓展了人们对于不可培养微生物的认识。
利用高通量测序技术研究微生物的多样性,对于样品的要求较为严格。首先,提取到合适的微生物基因组是进行微生物多样性测序的第一步,也是关键步骤之一,这直接影响后续样品目的片段扩增的效率和质量;其次,目的片段的扩增,不同的扩增循环数会直接影响最终数据分析中微生物种群的组成和丰度。由此可见,在微生物多样性测序中,环境样品的DNA提取和扩增是非常关键和重要的环节。
关于扩增循环数对微生物多样性分析的影响,本团队之前已经有过详细的报道[17],但关于DNA提取对微生物多样性分析的影响,目前尚没有系统的报道。尽管之前已有研究对土壤和羊瘤胃食糜样品的提取方法进行报道[18-20],但均止步于普通的PCR反应以验证DNA提取的质量,均未用于微生物的高通量测序分析。本研究以土壤和羊瘤胃食糜样品为研究对象,对最适DNA提取方法进行了调研。研究发现,MOBIO进口试剂盒提取的DNA较国产天根的提取试剂盒质量好,DNA更为完整,且杂质少,但二者均能成功扩增细菌16S V3区。但在相同DNA模板量和PCR条件下,前者提取的DNA能够获得更多的微生物种类。
1 材料与方法 1.1 主要试剂和仪器土壤样品、羊瘤胃食糜样品、土壤基因组DNA提取试剂盒分别购自天根生化科技(北京)有限公司和美国MOBIO公司;ExTaq DNA聚合酶购自大连宝生物工程有限公司;Truseq DNA library prep kit、Illumina 2×150bp测序试剂均购自美国Illumina公司。高速离心机购自德国Eppendorf公司;PCR仪购自美国Bio-Rad公司;Miseq测序仪购自美国Illumina公司。
1.2 DNA提取选取一个土壤样品SD10(采自山东)和一个羊瘤胃食糜样品W361,分别用天根土壤微生物基因组提取试剂盒和MOBIO土壤微生物基因组提取试剂盒进行总DNA的提取,分别命名为SD10-TG、SD10-MO、W361-TG和W361-MO。具体操作见试剂盒说明书。然后用Nanodrop测定DNA的浓度和质量,同时取500ng DNA进行琼脂糖凝胶电泳。
1.3 PCR扩增选取SD10-TG、SD10-MO、W361-TG和W361-MO各25ng,分别利用ExTaq酶对16S V3可变区进行PCR扩增,扩增循环数分别选取20cycles、25cycles和30cycles。扩增引物为:Forward: 5′-CCTACGGGAGGC AGCAG-3′;Reverse: 5′-ATTACCGCGGCTGCTGG-3′。每一条前向引物的5′端均加入10bp的一段barcode序列,用于区分不同条件的扩增产物。扩增产物分别命名为SD10-TG-20c、SD10-TG-25c、SD10-TG-30c、SD10-MO-20c、SD10-MO-25c、SD10-MO-30c、W361-TG-20c、W361-TG-25c、W361-TG-30c、W361-MO-20c、W361-MO-25c、W361-MO-30c。
1.4 文库构建和测序将12个扩增产物按照每个100ng的质量进行混合,然后对混合产物进行末端补平加A、连接接头和少量扩增进行富集。对构建好的文库进行Agilent 2100和qPCR质检,合格后用于Miseq 2×150bp的测序。
1.5 分析软件对测序得到的原始数据进行过滤,去掉接头和低质量的reads,得到可用于后续分析的clean data,并用fastqc软件对数据进行质量评估。然后使用FLASH软件对clean data进行组装,获得更多含barcode的reads。然后使用QIIME软件根据barcode序列将该组装结果回归样品,并对每个样品的序列数进行统计。然后使用uclust软件根据序列相似性(阈值为97%)进行聚类,得到OTU(操作分类单元)序列,并进行优化和分类。本研究采用菌群丰富度指数chao1、ACE,菌群多样性指数Simpson、Shannon和菌群覆盖度指数goods coverage、observed species对菌群进行多样性分析。最后使用greengenes数据库(201305版本)并根据分类学分析结果,分析样品在各分类水平上的物种组成比例情况。
2 结果首先将土壤样品的数据进行分组,比较SD10-TG-20c和SD10-MO-20c、SD10-TG-25c和SD10-MO-25c、SD10-TG-30c和SD10-MO-30c三组样品中,在扩增循环数相同的情况下,不同的提取试剂盒对测序数据的OTU数目、稀释曲线、微生物数目和种类的影响。然后将羊瘤胃食糜样品进行同样的分组和分析,即W361-TG-20c和W361-MO-20c、W361-TG-25c和W361-MO-25c、W361-TG-30c和W361-MO-30c,以验证土壤样品的分析结果。
2.1 不同提取试剂盒提取土壤和羊瘤胃食糜样品DNA称取0.25g土壤和羊瘤胃食糜样品,分别按照天根和MOBIO土壤微生物基因组提取试剂盒说明书提取基因组DNA。用Nanodrop测定DNA的浓度和质量(表 1),结果表明,天根提取的DNA总量较MOBIO提取得多,尤其是对于食糜样品,天根提取的DNA浓度为86.58ng/μl,而MOBIO提取的浓度仅为11.85ng/μl。但MOBIO提取的DNA质量较高,其提取的不论是土壤样品还是食糜样品,OD260/280的值均在1.7~2.0,而天根提取的样品OD260/280的值均大于2.0。琼脂糖凝胶电泳的结果如图 1所示,表明天根提取的样品质量较差,有较多RNA等杂质存在。
| Group | Sample | Concentration(ng/μl) | OD260/280 | OD260/230 |
| Soil | SD10-TG | 18.19 | 2.97 | 0.07 |
| SD10-MO | 12.25 | 1.78 | 0.95 | |
| Chyme | W361-TG | 86.58 | 2.06 | 0.3 |
| W361-MO | 11.85 | 1.8 | 0.84 |
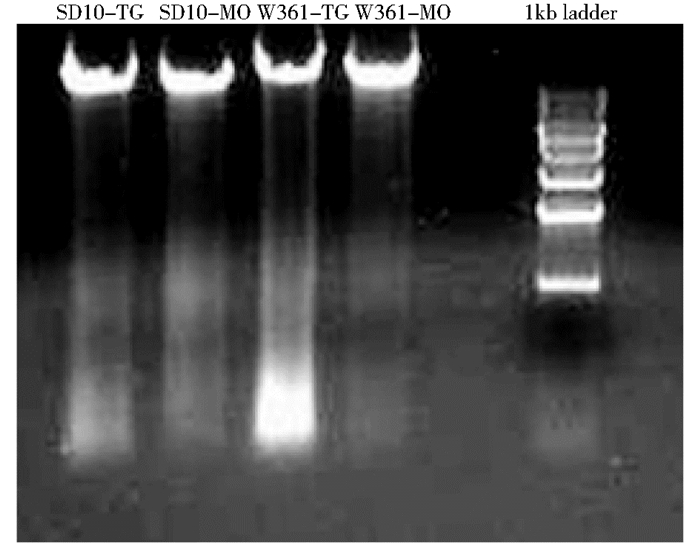
|
| 图 1 不同试剂盒提取土壤和食糜样品DNA胶图 Figure 1 Electrophoretogram of DNA extracted from soil and chyme samples |
首先以土壤样品为研究对象,研究天根和MOBIO不同提取试剂盒对土壤样品最终测序数据OTU和稀释曲线的影响。结果表明,当扩增循环数为20时,SD10-TG-20c的OTU数目是3 467,SD10-MO-20c的OTU数目是5 237;当扩增循环数为25时,SD10-TG-25c的OTU数目是5 277,SD10-MO-25c的OTU数目是12 937;当扩增循环数为30时,SD10-TG-30c的OTU数目是8 660,SD10-MO-30c的OTU数目是10 544。整体来看,用MOBIO提取的样品在其他条件一致的情况下,测序所得的OTU数目较天根提取的样品多(图 2a)。每个样品的稀释曲线基本都呈逐渐平缓的趋势(图 2b),说明测序数据基本能够覆盖目前状态下的微生物种类。
为了进一步验证试验,我们又选择微生物种类相对较少的羊瘤胃食糜样品为研究对象。同样地,在16S V3区扩增循环数分别为20、25和30的情况下,用MOBIO提取试剂盒提取的样品较天根试剂盒提取的样品,最终获得的OTU数目多(图 2c)。每个样品的稀释曲线也基本趋向平缓(图 2d)。
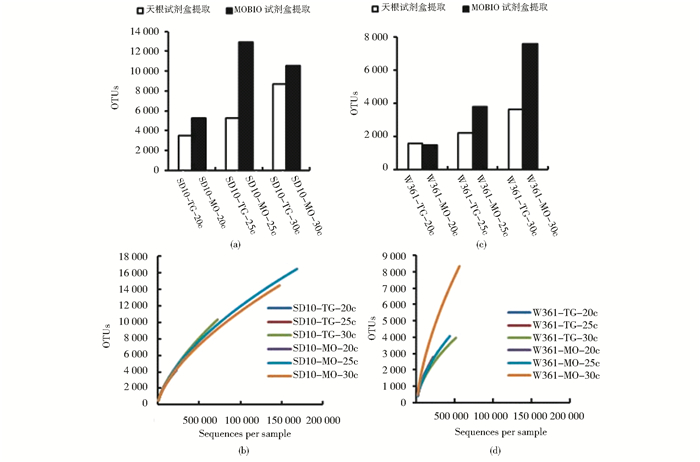
|
| 图 2 不同提取试剂盒对测序数据OTU和稀释曲线的影响 Figure 2 Effect of different DNA extraction method on OTU and Rarefaction curves (a) Effect of different DNA extraction method on OTU in soil samples (b) Effect of different DNA extraction method on Rarefaction curves in soil samples (c) Effect of different DNA extraction method on OTU in chyme samples (d) Effect of different DNA extraction method on Rarefaction curves in chyme samples |
两种不同类型样品的实验结果表明,MOBIO提取试剂盒提取的DNA样品较天根的提取试剂盒,对于后期样品的测序数据产出更为有利,不但能够产生更多的OTU,而且能够提高测序效率,容易达到测序数据的饱和状态。
2.3 不同提取试剂盒对微生物数量和种类的影响我们进一步分析了不同提取试剂盒对微生物数量和种类的影响。同样首先以土壤样品为研究对象。从数据分析的结果来看,用MOBIO试剂盒提取的样品最终产生的微生物数量较多。SD10-TG-20c/25c/30c产生的微生物数目分别是23 533、42 934和73 882;而SD10-MO-20c/25c/30c产生的微生物数目分别是49 475、169 257和147 895(图 3a)。
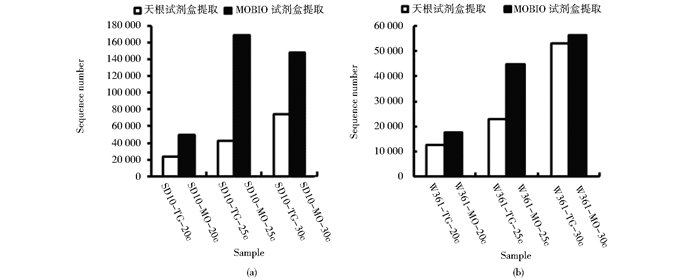
|
| 图 3 不同提取试剂盒对微生物数量的影响 Figure 3 Effect of different DNA extraction method on microbial population (a) Effect of different DNA extraction method on microbial population in soil samples (b) Effect of different DNA extraction method on microbial population in chyme samples |
从微生物多样性分析来看,MOBIO试剂盒获得的微生物种类数目更多(表 2)。在土壤样品中,MOBIO试剂盒在20、25和30三个不同的扩增循环数下得到的微生物种类分别是6 990、15 886和13 958;而天根提取试剂盒在相应的循环数下得到的微生物种类分别是4 056、6 605和10 118。
| Group | Sample | Shannon | PD_whole_tree | Chao1 | ACE | Simpson | Goods_coverage | Observed_species |
| Soil | SD10-TG-20c | 9.082 254 654 | 171.240 66 | 9 933.705 773 | 10 172.508 15 | 0.987 458 843 | 0.893 213 785 | 4 056 |
| SD10-MO-20c | 9.398 236 054 | 253.097 13 | 16 683.057 17 | 17 662.648 67 | 0.990 099 439 | 0.912 703 386 | 6 990 | |
| SD10-TG-25c | 9.321 629 098 | 286.768 49 | 17 100.866 67 | 18 335.250 84 | 0.987 340 375 | 0.901 313 644 | 6 605 | |
| SD10-MO-25c | 9.543 350 694 | 550.972 31 | 36 020.404 68 | 37 762.802 29 | 0.989 073 763 | 0.944 085 03 | 15 886 | |
| SD10-TG-30c | 9.178 013 711 | 385.932 63 | 26 693.114 78 | 29 232.774 37 | 0.981 174 917 | 0.910 018 678 | 10 118 | |
| SD10-MO-30c | 9.181 810 607 | 481.355 15 | 34 083.203 23 | 35 670.795 62 | 0.986 113 876 | 0.941 492 275 | 13 958 | |
| Chyme | W361-TG-20c | 8.273 031 398 | 74.301 17 | 4 180.231 076 | 4 438.593 746 | 0.988 144 245 | 0.913 952 936 | 1 833 |
| W361-MO-20c | 8.498 697 985 | 85.317 1 | 4 474.456 376 | 4 524.376 102 | 0.990 144 83 | 0.933 299 302 | 2 156 | |
| W361-TG-25c | 8.279 836 349 | 97.593 21 | 6 506.890 173 | 6 581.933 894 | 0.987 165 437 | 0.928 599 692 | 2 698 | |
| W361-MO-25c | 8.471 954 252 | 133.724 24 | 8 793.315 985 | 9 395.965 661 | 0.988 028 373 | 0.948 892 561 | 3 926 | |
| W361-TG-30c | 8.220 808 868 | 156.383 59 | 8 242.411 881 | 8 287.411 561 | 0.986 915 308 | 0.960 034 72 | 3 803 | |
| W361-MO-30c | 9.225 915 549 | 311.941 44 | 18 460.733 33 | 19 967.537 88 | 0.987 313 747 | 0.912 323 869 | 7 979 |
此外,两种试剂盒对微生物的群落组成也是有影响的。总体来说,在门水平上,对优势菌的捕捉,如变形菌门、拟杆菌门、厚壁菌门、绿弯菌门等,两种试剂盒的结果相差无几。但对一些相对丰度较低的菌门,MOBIO试剂盒在一定的扩增条件下,捕获效果会更好。图 4(a)~(c),对两种试剂盒捕获的低丰度差异菌门进行细分,发现当扩增循环数为20时,二者的在门上的种类差异不大,但MOBIO捕获到的差异菌门所占比例较大;当扩增循环数为25时,MOBIO捕获的差异菌门更多;当扩增循环数增加到30时,MOBIO在20和25扩增循环数下捕获到的菌门减少或消失,变化较大。以Poribacteria菌门为例,SD10-TG-20c中不含有Poribacteria菌门,而其在SD10-MO-20c中占到的比例是0.002 02%;当改变后续扩增条件后,SD10-TG-25c中Poribacteria菌门的比例达到0.002 33%,与SD10-MO-25c中Poribacteria菌门的比例相当;但在SD10-MO-30c中Poribacteria菌门却消失了。这可能是由于过度扩增导致某些菌门失衡所致。
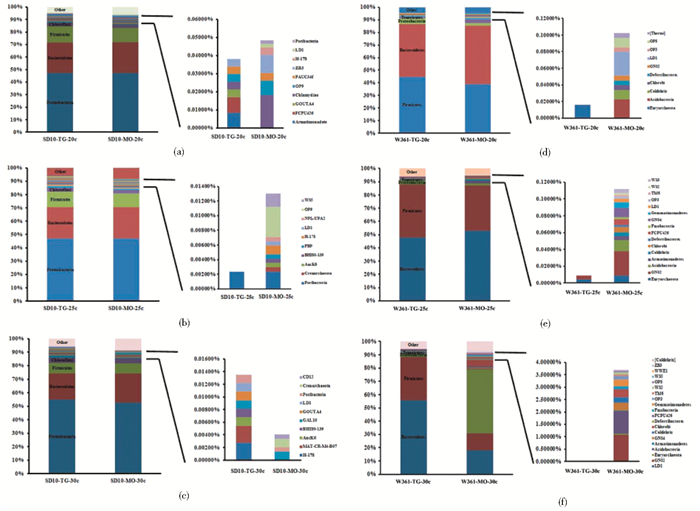
|
| 图 4 不同提取试剂盒对微生物门水平种类的影响 Figure 4 Effect of different DNA extraction method on microbial community composition (a)~(c) Effect of different DNA extraction method on microbial community composition in soil samples when amplification cycle numbers are 20, 25, 30 respectively (d)~(f)Effect of different DNA extraction method on microbial community composition in chyme samples when amplification cycle numbers are 20, 25, 30 respectively |
接下来,我们选择食糜样品对以上结果进行验证。从数据分析的结果来看,其趋势与土壤样品相同,均是MOBIO提取样品产生的微生物数量(图 3b)和种类(表 2)较多。在门水平上的群落组成分析结果同样表明,MOBIO试剂盒对捕获丰度较低的菌门更具优势,更能反映样本中微生物的群里组成情况(图 4d~f)。
3 讨论高通量测序技术通过对高质量样品的快速测序,以一种更为便捷经济的方式,可以获得样品中全面、准确、无偏的数据信息。也正因如此,利用高通量测序技术的微生物多样性测序[12-14, 16]和宏基因组测序[15, 21-22]越来越得到研究者的重视和青睐,其应用范围也越来越广。
高通量测序技术对样品的要求较高,不合格的样品质量将造成后续数据分析的巨大偏差。土壤中富含腐殖酸等化合物[23],因此提取的DNA纯度往往不能令人满意,同时影响后续PCR反应和DNA测序[24]。存在于动物和人体中的食糜样品,无论是取自胃部还是肠道,其中所含微生物种类对于健康[25]、养殖[13]、发酵[26]等领域均具有重要的指导意义。在此研究中,我们选取环境样品中较难处理的土壤样品和应用范围较广的食糜样品,旨在得到一种可用于指导高通量测序前期样品DNA提取的方法。
从DNA提取的结果来看,不论是土壤样品,还是食糜样品,进口试剂盒均比国产试剂盒的提取质量要高。尽管前者得到的DNA产率较后者低,但足以用于扩增模板。我们选取两种试剂盒提取的25ng DNA作为模板分别用于后续PCR扩增及测序,从数据分析的结果来看,进口试剂盒对于土壤和食糜样品的测序更为有利,利于产生更多的OTU、更多的微生物数量和种类。但在观察SD10-TG-30c和SD10-MO-30c样品中的群落组成时,发现后者比前者的微生物种类严重减少(图 4c),推测是扩增循环数的变化所造成的。我们在之前的工作中,即研究扩增循环数对群落组成的影响时,同样观察到SD10-MO-30c较SD10-MO-25c的种类组成严重减少[17],从而验证了我们的猜测。但SD10-TG-30c样品中微生物的种类仍然较SD10-MO-25c样品中的少。这些结果提示我们,对于土壤这样的复杂样品,我们需要用更为高效的试剂盒进行样品处理,并且扩增循环数并非越高越好。因为当扩增循环数使得优势菌的扩增达到饱和程度后,过度扩增会造成群落组成的比例失调。
| [1] |
Wommack K E, Nasko D J, Chopyk J, et al. Counts and sequences, observations that continue to change our understanding of viruses in nature. J Microbiol, 2015, 53(3): 181-192. DOI:10.1007/s12275-015-5068-6 |
| [2] |
Secundo de Souza A I, Freitas Neto O C, Batista D F, et al. ERIC-PCR genotyping of field isolates of Salmonella enterica subsp, Enterica serovar, Gallinarum biovars, Gallinarum and Pullorum. Avian Pathol, 2015, 44(6): 1-20. |
| [3] |
Mo Q H, Wang H B, Tan H, et al. Optimization and head-to-head comparison of MISSR-PCR, ERIC-PCR, RAPD and 16S rRNA evolutionary clock for the genotyping of Vibrio cholerae isolated in China. Indian J Med Microbiol, 2015, 33(4): 516-523. DOI:10.4103/0255-0857.167321 |
| [4] |
Dewhirst F E, Klein E A, Bennett M L, et al. The feline oral microbiome:a provisional 16S rRNA gene based taxonomy with full-length reference sequences. Vet Microbiol, 2015, 175(2-4): 294-303. DOI:10.1016/j.vetmic.2014.11.019 |
| [5] |
Hotta F, Eguchi H, Naito T, et al. Achromobacter buckle infection diagnosed by a 16S rDNA clone library analysis:a case report. BMC Ophthalmol, 2014, 14(1): 1-7. DOI:10.1186/1471-2415-14-1 |
| [6] |
Ren W, Zhao H, Shao W, et al. Identification of a novel phenamacril-resistance related gene by cDNA-RAPD method in Fusarium asiaticum. Pest Manag Sci, 2016, 72(8): 1558-1565. DOI:10.1002/ps.2016.72.issue-8 |
| [7] |
Sadiq F A, Li Y, Liu T, et al. A RAPD based study revealing a previously unreported wide range of mesophilic and thermophilic spore formers associated with milk powders in China. Int J of Food Microbiol, 2015, 217: 200-208. |
| [8] |
Choi I W, Kim H Y, Quan J H, et al. Monitoring of fasciola species contamination in water dropwort by cox1 mitochondrial and ITS-2 rDNA sequencing analysis. Korean J Parasitol, 2015, 53(5): 641-645. DOI:10.3347/kjp.2015.53.5.641 |
| [9] |
Marques S, Huss V A, Pfisterer K, et al. Internal transcribed spacer sequence-based rapid molecular identification of Prototheca zopfii and Prototheca blaschkeae directly from milk of infected cows. J Dairy Sci, 2015, 98(5): 3001-3009. DOI:10.3168/jds.2014-9271 |
| [10] |
Flórez A B, Mayo B. Diversity and dynamics of antibiotic-resistant bacteria in cheese as determined by PCR denaturing gradient gel electrophoresis. Int J of Food Microbiol, 2015, 214: 63-69. DOI:10.1016/j.ijfoodmicro.2015.07.027 |
| [11] |
El-Sayed W S, Ouf S A, Mohamed A A. Deterioration to extinction of wastewater bacteria by non-thermal atmospheric pressure air plasma as assessed by 16S rDNA-DGGE fingerprinting. Front in Microbiol, 2015, 6(3): 543-553. |
| [12] |
Xun W, Zhao J, Xue C, et al. Significant alteration of soil bacterial communities and organic carbon decomposition by different long-term fertilization management conditions of extremely low-productivity arable soil in South China. Environ Microbiol, 2016, 18(6): 1907-1917. DOI:10.1111/1462-2920.13098 |
| [13] |
Jami E, Israel A, Kotser A, et al. Exploring the bovine rumen bacterial community from birth to adulthood. ISME J, 2013, 7(6): 1069-1079. DOI:10.1038/ismej.2013.2 |
| [14] |
Kautz S, Rubin B E, Russell J A, et al. Surveying the microbiome of ants:comparing 454 pyrosequencing with traditional methods to uncover bacterial diversity. Appl Environ Microbiol, 2013, 79(2): 525-534. DOI:10.1128/AEM.03107-12 |
| [15] |
Bulgarelli D, Garrido-Oter R, Münch P C, et al. Structure and function of the bacterial root microbiota in wild and domesticated barley. Cell Host Microbe, 2015, 17(3): 392-403. DOI:10.1016/j.chom.2015.01.011 |
| [16] |
Dassi E, Ballarini A, Covello G, et al. Enhanced microbial diversity in the saliva microbiome induced by short-term probiotic intake revealed by 16S rRNA sequencing on the IonTorrent PGM platform. J Biotechnol, 2014, 190: 30-39. DOI:10.1016/j.jbiotec.2014.03.024 |
| [17] |
安云鹤, 高丽娟, 李俊博, 等. PCR扩增循环数对细菌群落多样性测序分析的影响. 生物工程学报, 2016, 32(8): 1115-1123. An Y H, Gao L J, Li J B, et al. Influence of PCR cycle number on microbial diversity analysis through next generation sequencing. Chinese Journal of Biotechnology, 2016, 32(8): 1115-1123. |
| [18] |
陈邦, 范代娣, 王琰, 等. 土壤微生物DNA提取方法的研究. 西北大学学报, 2009, 39(5): 785-788. Chen B, Fan D D, Wang Y, et al. Study on extraction method of soil microbial DNA. Journal of Northwestern University, 2009, 39(5): 785-788. |
| [19] |
王家昕, 谭晖, 李长影, 等. 几种土壤微生物总DNA提取方法的比较. 湖北农业科学, 2010, 49(11): 2651-2653. Wang J X, Tan H, Li C Y, et al. Comparison of several methods of total DNA extraction from soil microorganisms. Hubei Agricultural Sciences, 2010, 49(11): 2651-2653. DOI:10.3969/j.issn.0439-8114.2010.11.009 |
| [20] |
刘薇, 方国庆, 刘立威, 等. 奶牛瘤胃微生物DNA提取法的研究与优化. 动物生产, 2011, 47(13): 71-74. Liu W, Fang G Q, Liu L W, et al. Study and optimization of DNA extraction from rumen microorganisms of dairy cows. Animal Production, 2011, 47(13): 71-74. |
| [21] |
Bhatia S, Batra N, Pathak A, et al. Metagenomic analysis of bacterial and archaeal assemblages in the soil-mousse surrounding a geothermal spring. Genom Data, 2015, 5: 195-200. DOI:10.1016/j.gdata.2015.05.016 |
| [22] |
Karlsson F H, Tremaroli V, Nookaew I, et al. Gut metagenome in European women with normal, impaired and diabetic glucose control. Nature, 2013, 498(7452): 99-103. DOI:10.1038/nature12198 |
| [23] |
Courtois S, Frostegård A, Göransson P, et al. Quantification of bacterial subgroups in soil:comparison of DNA extracted directly from soil or from cells previously released by density gradient centrifugation. Environ Microbiol, 2001, 3(7): 431-439. DOI:10.1046/j.1462-2920.2001.00208.x |
| [24] |
Hurt R A, Qiu X, Wu L, et al. Simultaneous recovery of RNA and DNA from soils and sediments. Appl Environ Microbiol, 2001, 67(10): 4495-4503. DOI:10.1128/AEM.67.10.4495-4503.2001 |
| [25] |
Fei N, Zhao L. An opportunistic pathogen isolated from the gut of an obese human causes obesity in germfree mice. ISME J, 2013, 7(4): 880-884. DOI:10.1038/ismej.2012.153 |
| [26] |
Yin Y Y, Liu Y J, Zhu W Y, et al. Effects of acarbose addition on ruminal bacterial microbiota, lipopolysaccharide levels and fermentation characteristics in vitro. Asian-Australas J Anim Sci, 2014, 27(12): 1726-1735. DOI:10.5713/ajas.2014.14292 |
 2017, Vol. 37
2017, Vol. 37




