文章信息
- 任爽, 朱鸿亮.
- REN Shuang, ZHU Hong-liang.
- Taqman定量PCR技术检测基因编辑番茄中外源基因拷贝数体系的建立
- Establishment of Taqman Quantitative PCR System to Estimate Copy Numbers of Exogenous Transgene in Genome Edited Tomato
- 中国生物工程杂志, 2017, 37(10): 72-80
- China Biotechnology, 2017, 37(10): 72-80
- http://dx.doi.org/DOI:10.13523/j.cb.20171010
-
文章历史
- 收稿日期: 2017-04-13
- 修回日期: 2017-06-20
近些年基因编辑技术得到了飞速发展[1],成簇规律间隔的短回文重复序列相关核酸酶9系统(clustered regularly interspaced short palindromic repeats/CRISPR-associated 9, CAS9)[2]作为组装简便、高效精准的基因组编辑技术,已迅速应用于非转基因作物定向育种中,在运用该技术定点修饰获得有利的农业性状后,需要对大量T0代转基因植株进行高通量的转基因拷贝数鉴定,对筛选到的单拷贝杂合型转化子传代分离,经过分子选择,最终获得只含有靶位点编辑而不含转基因片段的精准遗传改良株系[3]。
目前外源基因整合拷贝数的检测主要采用Southern印迹杂交(Southern blot)[4],同时反义PCR(Inverse PCR)[5]、微滴式数字PCR(Droplet Digital PCR)[6]、多重连接探针扩增技术(Multiplex Ligation-dependent Probe Amplification, MLPA)[7]、微阵列(Micro array)[8]等方法也可用于拷贝数的分析,传统Southern blot存在操作复杂、价格昂贵、需要相对大量的实验材料和试剂仪器、甚至需要使用到放射性同位素等缺点[9-10],而且也有文献证实Southern blot对于发生重排的外源基因检测不够准确[11],其他新方法也都因价格昂贵、操作复杂难以广泛应用。
近年来实时荧光定量PCR技术(Real-time Fluorescent Quantitative Polymerase Chain Reaction, qPCR)为拷贝数检测提供了新方法[12],实时检测荧光量的变化,获得不同样品达到一定荧光信号时所需的循环次数CT值[13],进而通过计算对模板进行准确定量[14]。其优点有:可以对20pg~10ng的转基因成分进行有效监测[15]、花费试剂仪器少、节省劳力和时间[16];还可对外源基因的不同序列进行扩增,实现对基因重组的检测[17]
qPCR最常用的是非特异性荧光染料法(如SYBR Green)或特异性荧光探针法(如Taqman探针)[13],荧光染料法简便易行、成本低,但由于利用染料或特殊设计的引物来指示扩增的增加,其特异性受到质疑[18],而Taqman探针法是利用与靶序列特异杂交的探针进行指示,特异性高[19],反应结束后也不需要进行寡核苷酸融解曲线分析,缩短了实验时间[20-22]。
qPCR已成功应用于多种植物外源基因拷贝数的检测中,例如:小麦(Triticum aestivum)[23]、番茄(Solanum lycopersicum)SYBR Green I法[9]、大豆(Glycine max)[24-26]、棉花(Anemone vitifolia Buch)[27]、水稻(Oryza sativa)[11]等等。番茄作为重要的转基因受体植物,虽然Mason等[28]在2002年就采用了Taqman qPCR的方法来检测转基因番茄的外源基因整合拷贝数,但该方法计算较繁琐,有待改进,而且针对基因编辑番茄材料的检测研究报道较少。
本研究利用Taqman qPCR技术,以PDS基因CRISPR/Cas9编辑番茄植株作为实验材料,选择内源性基因APX为内参基因,外源性基因HPT为目的基因,根据CT值与起始模板量对数值的反比线性关系,对目的基因、内参基因模板数进行比较得出植株中HPT基因的拷贝数,初步建立一种检测基因编辑植株中外源基因的整合拷贝数的方法,为获得非转基因植株奠定一定的技术基础。
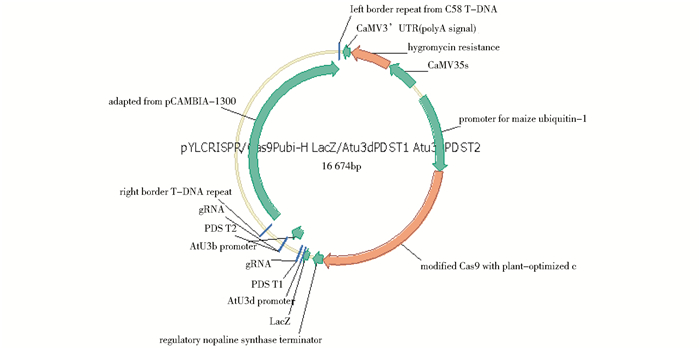
1 材料与方法 1.1 材料 1.1.1 生物材料CRISPR/Cas9基因编辑PDS系列番茄植株,培育于中国农业大学实验室,该系列是通过农杆菌介导的植物转化方法将植物表达载体pYLCRISPR/Cas9-PDS转入到番茄中,载体的构建过程如下:首先,通过第一轮PCR将每个靶序列连接到相应的sgRNA表达盒中,再进行二轮PCR扩增片段以及诱导靶向的BsaⅠ限制性酶切位点,然后通过Golden Gate ligation连接方法边切边连,在一轮克隆中将表达盒装配到pYLCRISPR/Cas9双元载体中[29]。其质粒载体图如图 1所示。
|
| 图 1 PDS基因编辑植物表达载体 Figure 1 The recombinant expression plasmid of genome editing of slyPDS |
CTAB手提法提取DNA所用的CTAB、酚、氯仿、异戊醇、乙醇、异丙醇等试剂均购于畅华志诚公司;实时荧光定量PCR的Mix为AQ401 Transtart Probe qPCR SuperMix购于Transgen公司;普通PCR试剂:2×EasyTaq PCR SuperMix、DL2000 DNA Marker购于Transgen公司;酶切试剂:EcoRⅠ-HF、HindⅢ-HF、CutSmart Buffer均购于New England Biolabs公司;引物和探针均由北京生工生物技术有限公司合成;PCR仪T100 Thermal Cycler购于Bio-Rad;实时定量PCR仪CFX96 Real-Time Syatem C1000 Thermal Cycler购于Bio-Rad。
1.2 方法 1.2.1 转基因番茄DNA的提取利用CTAB手提法提取转基因番茄基因组DNA,DNA提取情况由琼脂糖凝胶电泳及微量分光光度计检测确定,-20℃保存。
1.2.2 引物和探针的设计与合成APX的探针和引物[28]、HPT的探针[11]来自于标示的文献中,HPT基因的引物用PerlPrimer软件根据HPT基因序列进行设计,由北京生工生物技术有限公司合成,探针5′端标记FAM基团,3′端标记Elipse基团,其核苷酸序列、扩增长度见表 1和表 2。
| 名称 | 引物方向 | 引物序列(5′~3′) | 引物位置 | 扩增长度 |
| APXF | 正向引物 | TTTTACTTGTATATTCGAAGTGTGCCA | 2817~2843bp | 93bp |
| APXR | 反向引物 | ACAACTGCAAAATTAGAATCTAGTTGGTA | 2881~2909bp | |
| HPTF | 正向引物 | GTACACAAATCGCCCGCAG | 917~935bp | 111bp |
| HPTR | 反向引物 | TCTATTTCTTTGCCCTCGGAC | 1007~1027bp |
| 名称 | 探针序列(5′~3′) | 探针位置 |
| APXP | TTTCACCTATCTGGACCAAGTTTTGGCAAG | 2845~2874bp |
| HPTP | CGCCGATAGTGGAAACCGACGCCC | 975~998bp |
1.2.3 PDS系列转基因番茄植株APX和HPT基因检测
已优化的20 μl反应体系:10 μl 2×PCR Mix;1 μl正向引物;1 μl反向引物;适量DNA模板;灭菌水将体积补齐至20 μl。
已优化的扩增反应条件:94 ℃ 3 min;94 ℃ 30 s,58 ℃ 30 s,72 ℃ 20 s,30个循环。
1.2.4 标准品的制备选择基因编辑番茄植株DNA溶液作为标准品,根据其DNA浓度和长度计算该模板的copies/μl值后,依次进行系列梯度稀释,至少有4个梯度,以便更好地进行标准曲线的绘制。
1.2.5 Taqman qPCR检测HPT拷贝数20μl qPCR反应体系:10 μl Transtart Probe qPCR SuperMix;0.4 μl正向引物;0.4 μl反向引物;0.4 μl探针;1 μl DNA模板;灭菌水将体积补齐至20μl。
扩增反应条件(两步法):94℃ 30s,94℃ 5s,60℃ 30s,共45个循环。每个试样做3个平行,重复试验3次。
1.2.6 标准曲线的构建因目的基因qPCR反应后达到荧光阈值时的模板量为:
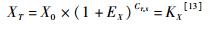 |
其中:XT是扩增目的基因后荧光达到阈值时的目的基因分子的量;
X0是起始目的基因分子的量;
EX是PCR反应效率;
CT, X是目的基因分子扩增使荧光信号到达阈值时的循环数;
KX在公式中表示一个常数。
而该公式两边同时取对数整理后得到:
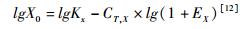 |
从该式可以看到CT, X与该模板的起始拷贝数X0的对数值存在线性关系,起始模板数越多,CT值越小,以此可以建立目的基因的标准曲线。
同理,内参基因的定量PCR反应的公式是:
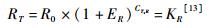 |
内参基因的标准曲线为:
 |
所以通过qPCR程序得到标准品的CT值和扩增曲线,根据不同拷贝数标准品的CT值为纵坐标,拷贝数的自然对数(Log starting quantity)为横坐标绘制标准曲线,分别得到目的基因和内参基因的标准曲线和线性方程。
1.2.7 转基因拷贝数的计算每一个被检测样本在达到阈值时都有一个循环数CT,将CT值代入标准曲线中得到该样本的起始模板量,目的基因与内源基因起始模板量的比值即是目的基因的拷贝数,即X0/R0。
标准品可以选择质粒溶液,但因番茄为双倍体植物,番茄单倍体基因组中APX是单拷贝的,所以以质粒作为标准品的时候,X0/R0值×2才为目的基因在该植株基因组中的拷贝数[11]。
2 结果与分析 2.1 基因编辑植株表型基因编辑番茄的植株表型结果见图 2,图 2A为野生型的正常植株,叶片翠绿,而图 2B为利用CRISPR/Cas9技术进行基因编辑后的植株,组织基本呈白色,通过植株的表型可以进一步确定实验样本均为表型明显的基因编辑植株。

|
| 图 2 基因编辑植株中的白化表型 Figure 2 Typical albino phenotypes of the genome edited AC lines regenerated from callus A: normal callus; B: typical albino callus |
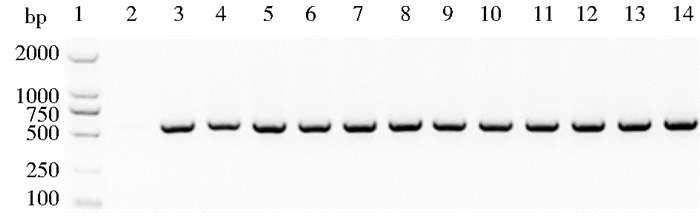
对基因编辑番茄DNA进行Cas9蛋白基因的检测,目的扩增基因片段长度为536bp,电泳结果如图 3,3~14泳道为PDS系列转基因实验材料,特异性条带单一且扩增条带大小一致,进一步确定所用的转基因植株为CRISPR/Cas9技术进行基因编辑后的植株。
|
| 图 3 Cas9基因PCR检测 Figure 3 PCR identification of Cas9 gene 1:2000bp DNA Marker; 2:Negative control, WT; 3~14:The PDS transgenic lines |
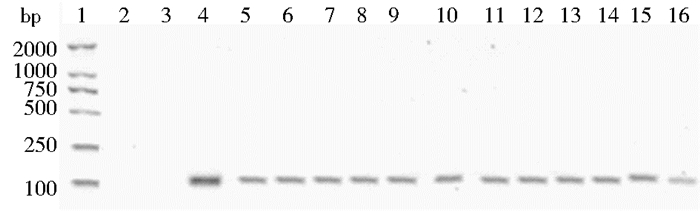
HPT基因PCR检测结果如图 4,5~16泳道为PDS系列转基因实验材料,结果显示实验材料DNA扩增均呈阳性,特异性条带单一,且扩增条带大小应为111bp,与电泳图中显示的大小相符,证明样本植株均为转基因植株,可以用于后续的实验。
|
| 图 4 HPT基因PCR检测 Figure 4 PCR identification of HPT gene 1:2000bp DNA Marker; 2:Blank control, water; 3:Negative control, WT; 4:Positive control, plasmid; 5~16:The PDS transgenic lines |
APX基因PCR检测结果如图 5,4~15泳道为PDS系列转基因实验材料,结果显示实验材料DNA扩增均呈阳性,特异性条带单一,且其扩增条带大小93bp,与电泳图中显示的大小相符,证明内源基因APX的可靠性,为后续实验奠定了基础。

|
| 图 5 APX基因PCR检测 Figure 5 PCR identification of APX gene 1:2000bp DNA Marker; 2:Blank control, water; 3:Positive control, WT; 4~15:The PDS transgenic lines |
标准品是得到标准曲线的前提,对于标准品,首先要求纯度高,其次因起始模板量越高,结果的重复性越差,所以起始模板量不宜太高,使其满足CT值在15~30之间,且CT值范围要覆盖所有待测样品所测量的CT值。基于标准品DNA所做的标准曲线是定量分析的关键,内参基因的标准品可以选择非转基因植株的DNA,也可以选择转基因植株的DNA,外源基因则可以选择转基因植株的DNA或质粒溶液,但Song等[30]认为,理想的标准品应是用Southern杂交已准确测得插入外源基因拷贝数的植物基因组DNA。
在通过线性方程计算外源基因拷贝数之前,需要通过线性相关系数R2和扩增效率E来验证标准曲线的正确性。R2大于0.98时才能表明标准曲线可靠可用,同时根据公式1+E=10-1/S(S是Slope,即标准曲线的斜率)计算出扩增效率E[12],扩增效率与很多因素有关,例如引物的好坏、标准品的纯度浓度等等,当扩增效率为100%时,斜率等于-3.32,实际上,-3.743至-3.008范围之间的斜率都是可以接受的[31],即扩增效率为0.85至1.15,从而保证qPCR实验的准确性。
HPT和APX基因扩增曲线与标准曲线示例如图 6、7,扩增曲线呈一组间距相等的平行曲线,扩增重复性良好;三个技术重复之间基本重合,实验重复性良好;标准曲线得到的R2在0.9882~0.9990之间,相关性良好;斜率在3.1044~-3.5184之间,扩增效率良好,可以进行拷贝数的计算。
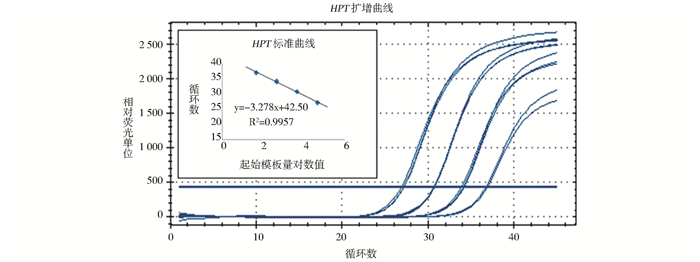
|
| 图 6 HPT标准曲线 Figure 6 Standard curve of HPT gene |

|
| 图 7 APX标准曲线 Figure 7 Standard curve of APX gene |
Taqman qPCR检测基因编辑番茄植株,重复实验3次,结果显示有12株番茄植株的拷贝数为1,通过实验筛选到了靶基因PDS编辑效果显著且单拷贝的转基因植株,可望将其用于非转基因遗传育种中,运用分离的方法得到只含有靶位点编辑而不含转基因片段的精准遗传改良株系(表 3)。
| 样本名称 | 拷贝数(重复1) | 拷贝数(重复2) | 拷贝数(重复3) | 拷贝数结果 |
| PDS-1 | 1.01 | 0.97 | 0.99 | 1 |
| PDS-2 | 1.05 | 1.15 | 0.87 | 1 |
| PDS-3 | 1.06 | 1.19 | 0.90 | 1 |
| PDS-4 | 0.95 | 1.04 | 0.82 | 1 |
| PDS-5 | 1.08 | 1.12 | 0.92 | 1 |
| PDS-6 | 1.20 | 1.19 | 1.01 | 1 |
| PDS-7 | 0.91 | 0.88 | 0.94 | 1 |
| PDS-8 | 1.03 | 1.09 | 1.01 | 1 |
| PDS-9 | 1.09 | 1.02 | 1.09 | 1 |
| PDS-10 | 1.02 | 0.98 | 0.97 | 1 |
| PDS-11 | 1.14 | 1.10 | 1.12 | 1 |
| PDS-12 | 1.08 | 1.05 | 1.08 | 1 |
3 讨论 3.1 Taqman qPCR技术建立可靠的拷贝数检测体系
本研究以基因编辑番茄为材料发现,利用Taqman qPCR技术检测的CRISPR/Cas9转基因番茄HPT拷贝数结果,与杨立桃、魏鹏程等人用Taqman方法检测的转基因水稻外源基因HPT拷贝数的结果十分接近。从DNA提取、引物和探针的设计、实时定量PCR体系及反应参数、平行实验之间的重复性、数据处理等方面环环控制实验,建立了一种利用Taqman实时荧光定量PCR检测基因编辑番茄植株外源基因拷贝数的方法。
实验证明这种方法可以在早期高通量进行有研究价值植株的筛选和鉴定,一般转基因植物的愈伤组织经过筛选分化后的苗细弱且生育期较长,不适宜大量取材来提取DNA进行Southern blot,以至于不能及早进行拷贝数检测,而Taqman实时荧光定量PCR解决了传统方法费时费力、浪费实验材料、所用仪器多的缺点,在早期筛选到遗传稳定的单拷贝基因编辑植株,而后对精准改良的转基因植株进行自交或回交,分离出只含有靶位点编辑而不含转基因片段的遗传改良株系。
众所周知,转基因作物的食品安全性和环境安全性问题,主要来自导入植物体内的外源目的基因和标记基因,而获得的品种中不含有人工分离和修饰过的外源DNA,该育种方法改变的是基因序列与传统自然诱变相同,得到的是非转基因作物,所以在定向改良作物的同时,还消除了转基因安全性的风险,在作物育种中具有良好的应用价值。在应用非转基因作物育种时很重要一个前提就是获得单拷贝的基因编辑植株,高通量的Taqman实时荧光定量PCR筛选方法推动了高效的非转基因作物育种工作。但实验设计也有一点不足的地方,如用Southern blot进行二次验证可以更有效地对该体系进行评估。
3.2 优化反应体系和反应条件影响反应重复性较为关键的因素就是其扩增效率,为了使反应体系达到最佳扩增效率,可以进行反应体系和条件的优化,即调整引物和探针的浓度、目的基因的初始浓度纯度等等。
若引物浓度太低,会导致反应不完全;引物浓度太高,则会发生错配而且产生非特异性产物的可能性大大增加;若探针浓度太低,荧光信号较弱;探针浓度太高,会影响信噪比[13]。其优化的过程一般是通过一系列梯度浓度的探针和引物的正交预实验来确定两者的相对量,而每一个体系中,可以产生最高荧光信号和最低CT值的引物和探针的最低浓度,即为最佳的引物和探针浓度。本研究中对杨立桃文献中的反应体系进行改进,将反应体积缩小为20 μl,引物浓度从500 nmol/L调整为200 nmol/L,将探针浓度从300 nmol/L调整为200 nmol/L,既节省了试剂又保证了反应的灵敏度以及较好的扩增效率;因为扩增片段较短,仍选择两步法扩增,扩增条件将循环数降低为30个,防止信号放大引起信噪比的增大和非特异性扩增。
对于样品,首先要保证其纯度,DNA提取过程中的氯仿:异戊醇的反复抽提有助于提高样品纯度,其次因初始拷贝数越高,结果的重复性越差,所以初始拷贝数不宜太高并使其满足CT值在20~35之间,若小于20需稀释选择更低的样品浓度,若大于35需浓缩选择更高的样品浓度。以本研究中的样品为例,实验结果发现起始拷贝数在106~103个/μl之间时,数据具有较好的线性关系以及扩增效率,超出这一范围时实时定量反应的灵敏度有所下降,这时如果标准样品按4个10倍梯度进行稀释而超过这一范围时,可以通过5个标准品5倍梯度稀释的方法来改进优化。
4 结论本研究以CRISPR/Cas9基因编辑番茄为材料,选择抗性基因HPT为外源目的基因,选择番茄单拷贝基因APX作为内源参照基因,利用Taqman实时定量PCR技术建立一种外源基因拷贝数的检测体系。结果表明该体系是一种快速可靠的方法,为检测基因编辑植株中外源基因的整合拷贝数提供了有效手段,避免了传统Southern杂交方法费时费力、需要样品多的缺点,可以借此高通量筛选出单拷贝的CRISPR/Cas9基因编辑植株,并更好地进行下一步非转基因遗传育种工作,分离得到无转基因片段的改良作物品种,在遗传育种领域十分有意义。
| [1] |
周想春, 邢永忠. 基因组编辑技术在植物基因功能鉴定及作物育种中的应用. 遗传, 2016, 38(3): 227-242. Zhou X C, Xing Y Z. The application of genome editing in identification of plant gene function and crop breeding. Hereditas, 2016, 38(3): 227-242. |
| [2] |
曾秀英, 侯学文. CRISPR/Cas9基因组编辑技术在植物基因功能研究及植物改良中的应用. 植物生理学报, 2015, 51(9): 1351-1358. Zeng X Y, Hou X W. Application of CRISPR/Cas9 genome editing technology in functional genomics and improvement of plants. Plant Physiology Journal, 2015, 51(9): 1351-1358. |
| [3] |
魏鹏程, 杨剑波, 李浩, 等. 一种主要农作物非转基因的基因组定向分子改良方法和应用. 安徽, CN103555711A, 2014. A Method and Application of Genome-oriented Molecular Modification for Non-transgenic Genes in a Main Crop. Anhui, CN103555711A, 2014. |
| [4] |
李敏, 汪洋, 张银萍, 等. TaqMan探针与SYBR Green实时定量PCR法检测转基因植物外源基因拷贝数的差异分析. 安徽农业大学学报, 2012(4): 568-570. LI M, Wang Y, Zhang Y P, et al. Variance analysis between TaqMan probe and SYBR Green real-time quantitative PCR for determining the copy number of transgene in transformed plants. Journal of Anhui Agricultural University, 2012(4): 568-570. |
| [5] |
Stefano B, Patrizia B, Matteo C, et al. Inverse PCR and quantitative PCR as alternative methods to southern blotting analysis to assess transgene copy number and characterize the integration site in transgenic woody plants. Biochemical Genet, 2016, 54(3): 1-15. |
| [6] |
Xu X, Peng C, Wang X, et al. Comparison of droplet digital PCR with quantitative real-time PCR for determination of zygosity in transgenic maize. Transgenic Res, 2016, 25(6): 855-864. DOI:10.1007/s11248-016-9982-0 |
| [7] |
Schouten J P, Mcelgunn C J, Waaijer R, et al. Relative quantification of 40 nucleic acid sequences by multiplex ligation-dependent probe amplification. Nucleic Acids Res, 2002, 30(12): e57. DOI:10.1093/nar/gnf056 |
| [8] |
Carter N P. Methods and strategies for analyzing copy number variation using DNA microarrays. Nat Genet, 2007, 39(7 suppl): S16-S21. |
| [9] |
苏慧慧, 李涛, 谢雯琦, 等. 基于实时荧光定量PCR对转基因樱桃番茄外源基因拷贝数的检测. 分子植物育种, 2015, 13(2): 345-354. Su H H, Li T, Xie W Q, et al. Detecting exogenous gene copy numbers of exogenous gene in transgenic tomato based on fluorescent quantitative real-time PCR. Molecular Plant Breeding, 2015, 13(2): 345-354. |
| [10] |
白国辉, 刘建国, 田源, 等. 转基因番茄防龋疫苗中外源目的基因拷贝数的检测. 上海口腔医学, 2013, 22(6): 613-617. Bai G H, Liu J G, Tian Y, et al. Detection of the exogenous gene copy number of the transgenic tomato anti-caries vaccine. Shanghai Journal of Stomatology, 2013, 22(6): 613-617. |
| [11] |
Yang L, Ding J, Zhang C, et al. Estimating the copy number of transgenes in transformed rice by real-time quantitative PCR. Plant Cell Rep, 2005, 23(10): 759-763. |
| [12] |
张贺, 李波, 周虚, 等. 实时荧光定量PCR技术研究进展及应用. 动物医学进展, 2006, 27(z1): 5-12. Zhang H, Li B, Zhou X, et al. Technology of capillary electrophoresis and its application in analysis of veterinary drug residues. Progress in Veterinary Medicine, 2006, 27(z1): 5-12. DOI:10.3969/j.issn.1007-5038.2006.z1.002 |
| [13] |
王爱民. 实时荧光定量PCR(TaqMan)法测定外源基因的拷贝数. 广西植物, 2009, 29(3): 408-412. Wang A M. Estimating copy number of transgenic gene by real-time fluorescent quantitative PCR(TaqMan). Guihaia, 2009, 29(3): 408-412. |
| [14] |
裘劼人, 许颖, 喻富根. 利用SYBR Green实时定量PCR法检测转基因植物外源基因的拷贝数. 安徽农业科学, 2011, 39(21): 12655-12657. Qiu J R, Xu Y, Yu F G. Estimating the copy number of transgenes in transformed Arabidopsis by SYBR green real-time quantitative PCR. Journal of Anhui Agricultural Sciences, 2011, 39(21): 12655-12657. DOI:10.3969/j.issn.0517-6611.2011.21.010 |
| [15] |
朱建楚, 胡银岗, 奚亚军, 等. 实时荧光定量PCR技术在检测外源基因拷贝数中的应用. 西北农业学报, 2005, 14(6): 78-82. Zhu J C, Hu Y G, Xi Y J, et al. The Application of quantitative real-time fluorescent PCR techniques in detecting the copy number of transgenic gene. Acta Agriculturae Boreali-occidentalis Sinica, 2005, 14(6): 78-82. |
| [16] |
Klein D. Quantification using real-time PCR technology:applications and limitations. Trends Mol Med, 2002, 8(6): 257-260. DOI:10.1016/S1471-4914(02)02355-9 |
| [17] |
梁彦君. 转基因番茄外源基因检测方法研究. 杭州: 浙江理工大学, 生命科学学院, 2013. Liang Y J. Research on the detection methods of exogenous genes in transgenic tomato. Hangzhou:Zhejiang Sci-Tech University, College of Life Sciences, 2013. |
| [18] |
赵焕英, 包金风. 实时荧光定量PCR技术的原理及其应用研究进展. 中国组织化学与细胞化学杂志, 2007, 16(4): 492-497. Zhao H Y, Bao J F. Research on the principle and application of Real-time Fluorescence Quantitative Polymerase Chain Reaction. Chinese Journal of Histochemistry and Cytochemistry, 2007, 16(4): 492-497. |
| [19] |
钟江华, 张光萍, 柳小英. 实时荧光定量PCR技术的研究进展与应用. 氨基酸和生物资源, 2011, 33(2): 68-72. Zhong J H, Zhang G P, Liu X Y. Development of Real-Time Fluorescent Quantitative PCR and its application. Amino Acids & Biotic Resources, 2011, 33(2): 68-72. |
| [20] |
洪云, 李津, 汪和睦, 等. 实时荧光定量PCR技术进展. 国外流行病学传染病学杂志, 2006, 33(3): 161-163. Hong Y, Li J, Wang H M, et al. Progress in real-time quantitative PCR technique. International Journal of Epidemiology and Infectious Disease, 2006, 33(3): 161-163. |
| [21] |
廉红霞, 高腾云, 傅彤, 等. 实时荧光定量PCR定量方法研究进展. 江西农业学报, 2010, 22(10): 132-136. Lian H X, Gao T Y, Fu T, et al. Research advance in method of Real-time Fluorescent Quantitative Polymerase Chain Reaction. Acta Agriculturae Jiangxi, 2010, 22(10): 132-136. |
| [22] |
陈旭, 齐凤坤, 康立功, 等. 实时荧光定量PCR技术研究进展及其应用. 东北农业大学学报, 2010, 41(8): 148-155. Chen X, Qi F K, Kang L G, et al. Advance and application of real-time fluorescent quantitative PCR. Journal of Northeast Agricultural University, 2010, 41(8): 148-155. |
| [23] |
李淑洁, 张正英. REAL-TIME PCR方法测定转基因小麦中外源基因拷贝数. 中国生物工程杂志, 2010, 30(3): 90-94. Li S J, Zhang Z Y. Using Real-time PCR to determine transgene copy number in wheat. China Biotechnology, 2010, 30(3): 90-94. |
| [24] |
冀志庚, 高学军, 敖金霞, 等. SYBR Green实时定量PCR检测转基因大豆中外源基因拷贝数. 东北农业大学学报, 2011, 42(10): 11-15. Ji Z G, Gao X J, Ao J X, et al. Establishment of SYBR Green-base quantitative real-time PCR assay for determining transgene copy number in transgenic soybean. Journal of Northeast Agricultural University, 2011, 42(10): 11-15. DOI:10.3969/j.issn.1005-9369.2011.10.003 |
| [25] |
韩强, 刘瑞芳, 陆玲鸿, 等. 实时荧光PCR检测转基因大豆外源基因的拷贝数. 核农学报, 2016, 30(4): 646-653. Han Q, Liu R F, Lu L H, et al. Detection of transgene copy number in transgenic soybean by Real-time Fluorescence Quantitative PCR. Journal of Nuclear Agricultural Sciences, 2016, 30(4): 646-653. DOI:10.11869/j.issn.100-8551.2016.04.0646 |
| [26] |
Qiu Y W, Gao X J, Qi B R, et al. Establishment of TaqMan Real-time Quantitative PCR Assay for foreign gene copy numbers in transgenic soybean. Journal of Northeast Agricultural University(English Edition), 2012, 19(4): 48-52. DOI:10.1016/S1006-8104(13)60050-1 |
| [27] |
Yi C X, Hong Y. Estimating the copy number of transgenes in transformed cotton by real-time quantitative PCR. Methods in Molecular Biology, 2013, 958(10-11): 109-130. |
| [28] |
Mason G, Provero P, Vaira A M, et al. Estimating the number of integrations in transformed plants by quantitative real-time PCR. BMC Biotechnol, 2002, 2(1): 20. DOI:10.1186/1472-6750-2-20 |
| [29] |
Ma X L, Zhang Q Y, Zhu Q L, et al. A robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in monocot and dicot plants. Mol Plant, 2015, 8(8): 1274-1284. DOI:10.1016/j.molp.2015.04.007 |
| [30] |
Song P, Cai C, Skokut M, et al. Quantitative real-tmie PCR as a screening tool for estimating transgene copy number in WHISKERSTM-derived transgenic maize. Plant Cell Reports, 2002, 20(10): 948-954. DOI:10.1007/s00299-001-0432-x |
| [31] |
文莉. 利用实时定量PCR技术检测柑橘外源基因的拷贝数. 武汉: 华中农业大学, 园艺林学学院, 2011. Wen L. Estimating transgene copy number in citrus by Real-Time Quantitative PCR. Wuhan:Huazhong Agricultural University, College of Horticulture & Forestry Sciences, 2011. |
 2017, Vol. 37
2017, Vol. 37




