文章信息
- 刘俊伟, 常瑞恒, 回鹏, 董士尚, 王金凤, 孙波, 杨诚.
- LIU Jun-wei, CHANG Rui-heng, HUI Peng, DONG Shi-shang, WANG Jin-feng, SUN Bo, YANG Cheng.
- 抗埃博拉病毒核蛋白抗体的制备与双抗夹心ELISA检测方法的建立
- Preparation of Anti-ebola Nucleoprotein Antibodies and Establishment of Sandwich ELISA Assay
- 中国生物工程杂志, 2017, 37(10): 53-59
- China Biotechnology, 2017, 37(10): 53-59
- http://dx.doi.org/DOI:10.13523/j.cb.20171007
-
文章历史
- 收稿日期: 2017-03-23
- 修回日期: 2017-06-01
2. 南开大学药学院 天津 300071
2. School of Pharmacy Nankai University, Tianjin 300071, China
出血热(Ebola hemorrhagic fever)是由埃博拉病毒(Ebola virus,EBOV)引发的人畜共患急性出血性传染病[1]。目前已鉴定的EBOV有5种亚型:本迪布焦型埃博拉病毒(Bundibugyo ebolavirus,BDBV),扎伊尔型埃博拉病毒(Zaire ebolavirus,ZEBOV),莱斯顿型埃博拉病毒(Reston ebolavirus,RESTV),苏丹型埃博拉病毒(Sudan ebolavirus,SUDV),塔伊森林型埃博拉病毒(TaïForest ebolavirus,TAFV)[2-3]。其中,扎伊尔型EBOV和苏丹型EBOV毒力最强[4]。
埃博拉病毒属丝状病毒科,为不分节段的单股负链RNA病毒,其分子量为4.2×106Da[5],于1976年首次分离[6-7]。整个基因组共编码7种蛋白质,分别为NP-VP35-VP40-GP-VP30-VP24-L,每种蛋白均由一条单独的mRNA所编码[8],其中核蛋白(nucleoprotein,NP)是核衣壳的主要组成部分。
埃博拉病毒核蛋白包括一个N端寡聚结构域,中间无序区以及C端结构域。其中N端寡聚结构域(EBOV NP,1~450氨基酸),包括参与病毒核蛋白寡聚的两端短臂以及负责与病毒RNA结合的中间核心结构域,在病毒转录复制过程中起着非常重要的作用[9]。此外,NP在埃博拉病毒颗粒中高度表达且较为保守,是可用于疾病诊断和监控的目标分子[10-12]。
1 材料与方法 1.1 材料 1.1.1 实验材料扎伊尔型埃博拉病毒核蛋白(EBOV NP)寡聚结构域的1~450位氨基酸重组蛋白(EBOV NP 1~450,Genebank No.911830)和36~450位氨基酸重组蛋白(EBOV NP 36~450)已由课题组前期表达制备[13]。经测定EBOV NP 1~450可与埃博拉病毒RNA结合,EBOV NP 36~450可与埃博拉病毒VP35蛋白结合,因此其生物学活性符合相关文献报道[14-16]。
1~35位氨基酸多肽(EBOV NP 1~35)为上海吉尔生化公司合成。弗氏完全佐剂、弗氏不完全佐剂、50%PEG1450溶液、HAT和HT选择性培养基均为Sigma产品;96孔酶标板购于丹麦Nunc公司;牛血清白蛋白(BSA)为上海生工公司产品;蛋白A-Sepharose 4B和蛋白G-Sepharose 4B亲和层析柱,购自GE公司;单抗亚型鉴定试剂盒,购自BD Biosciences公司;HRP偶联试剂盒购自北京博奥森公司。其它试剂均为国产分析纯试剂。
1.1.2 仪器酶标仪,美国Thermo;CO2培养箱,日本三洋;倒置显微镜,日本OLYMPUS;生物安全柜,海尔公司;PCR仪,塞维斯科技(北京)有限公司;高压细胞破碎仪,广州聚能生物科技有限公司;FPLC蛋白自动纯化系统,GE Healthcare公司。
1.1.3 实验动物Balb/c小白鼠,雌性,6~8周龄;大耳白兔,雄性,3个月龄,均购于天津实验动物中心。
1.2 方法 1.2.1 动物免疫(1) 将弗氏佐剂与EBOV NP 1~450混合后免疫大耳白兔,取血清并采用间接ELISA测定效价超过1:1×105后,取全血离心后获得抗血清。
(2) 将弗氏佐剂与EBOV NP 1~450混合免疫Balb/c小鼠。待血清效价达到1:1×104以上时,用常规剂量的抗原生理盐水稀释,尾静脉免疫小鼠,3天后取脾脏细胞备用。
1.2.2 细胞融合及杂交瘤细胞筛选将脾细胞与小鼠骨髓瘤细胞(NS-1)融合,再分别使用HAT和HT培养基进行培养初筛,并用有限稀释法筛选出2株可分泌单克隆抗体的杂交瘤细胞。亚型鉴定结果表明分泌出的单克隆抗体亚型均为IgG1, κ。
1.2.3 抗体纯化对小鼠进行体内接种杂交瘤细胞,1周后收集腹水,用蛋白G-Sepharose 4B亲和层析柱纯化得到单克隆抗体。将兔子抗血清用蛋白A-Sepharose 4B亲和层析柱纯化得到多克隆抗体,并对抗体浓度进行了测定。
1.2.4 抗体结合区域鉴定通过与不同长度片段的EBOV NP(1~450位氨基酸;1~35位氨基酸;36~450位氨基酸)作对照实验,利用Western blot与ELISA方法联合使用对兔多抗、鼠单抗的结合区域进行分析。
间接ELISA方法简述如下:(1)包被EBOV NP于酶标板上,4℃冰箱过夜;(2)用PBST洗板后,加入1% BSA进行封闭;(3)将抗体梯度稀释后加入到酶标板中;(4)洗板后加入酶标二抗,室温孵育;(5)洗板后加入TMB底物液,显色30min;(6)加入终止液。
1.2.5 双抗体夹心ELISA方法的优化与建立(1) 兔多抗与单抗配对优选:根据ELISA方法步骤,将5 μg/ml兔多抗包被到酶标板上,封闭后加入5 μg/ml EBOV NP 1~450,并加入0.25 μg/ml单克隆抗体及羊抗鼠-HRP后显色测定,根据A450值的高低选择可与多抗配对的单克隆抗体。
(2) 双抗夹心ELISA法操作流程:兔多抗包被聚苯乙烯酶标板(包被抗体),4℃过夜,封闭液封闭,室温孵育1h后加入抗原EBOV NP,室温温育1h后加入偶联HRP鼠单抗(酶标抗体),室温温育30 min后加入TMB底物溶液显色,室温孵育20 min后用2 mo1/L H2SO4终止反应,最后用酶标仪测定450 nm吸光度值。
(3) 包被抗体与酶标抗体最佳反应浓度的确定:本实验利用HRP偶联试剂盒将HRP偶联到鼠单抗上制备出酶标抗体,并利用方阵法对包被抗体和酶标抗体的反应浓度进行优化。包被抗体用PBS分别稀释为20μg/ml、10μg/ml、5μg/ml和2.5μg/ml;封闭后每个包被浓度分别加入阳性溶液和阴性溶液,其中阳性溶液为0.25 μg/ml EBOV NP;阴性溶液为PBS缓冲液溶液。酶标抗体用PBS梯度稀释1 000、2 000、4 000和8 000倍。显色后读数,根据阳性孔A450值(Positive,P)在l.0左右(A450吸光值为1.0左右时灵敏度最高),且阳性值与阴性值的比值较大所对应的抗体浓度为最佳反应浓度。
(4) 包被液的选择:分别以0.01 mo1/L PBS、0.05 mol/L Na2CO3-NaHCO3(NBS)和生理盐水作包被液,按双抗夹心ELISA步骤进行反应,比较不同包被液对结果的影响。
(5) 封闭液的选择:分别以1%的BSA-PBS,3%的BSA-PBS,3%的脱脂奶粉-PBS及酶标板稳定剂(湖州英创生物科技有限公司)为封闭液进行条件优化。
(6) TMB底物作用时间的选择:加入底物后,室温下分别作用10、20、30 min,最后根据A450值选择出最佳底物作用时间。
2 结果与分析 2.1 抗体纯化通过免疫,本实验获得了针对EBOV NP的抗血清,经亲和层析纯化得到兔源多克隆抗体,浓度为10 mg/ml(图 1)。
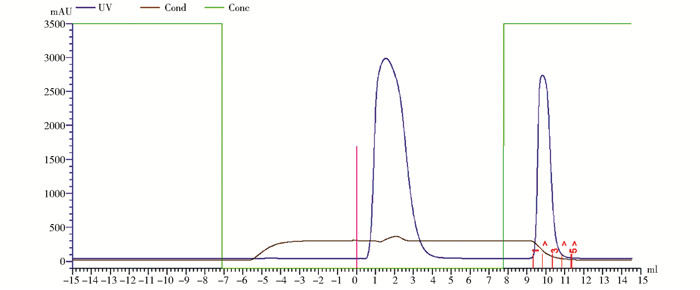
|
| 图 1 多克隆抗体纯化 Figure 1 Purification of polyclonal antibody |
利用细胞融合技术,本实验制备出2株可分泌单克隆抗体的杂交瘤细胞株,分别命名为1C3和4G3。经纯化小鼠腹水得到单克隆抗体1C3的浓度为3.4 mg/ml,4G3的浓度为2.1 mg/ml(图 2)。

|
| 图 2 单克隆抗体1C3的纯化 Figure 2 Purification of monoclonal antibody 1C3 |
为验证抗体纯化效果,本研究对单克隆抗体1C3进行了SDS-PAGE电泳实验,结果表明(图 3)样品在25~35 kDa和45~66kDa中间都出现了蛋白条带,其分子量大小与鼠源单克隆抗体轻链和重链的理论值(轻链26 kDa、重链56 kDa)基本一致,电泳条带没有拖尾现象及其他杂带,表明抗体纯度较好。

|
| 图 3 单抗1C3的SDS-PAGE Figure 3 The SDS-PAGE result of monoclonal 1C3 |
另外,本实验根据公式Kaff=(n-1)/{2(n[Ab′]t-[Ab]t)},计算出抗体的亲和力常数Kaff值[17],其中兔多抗的Kaff值为2.8×108 L/mol;4G3为4.3×108 L/mol,1C3为5.4×108 L/mol。
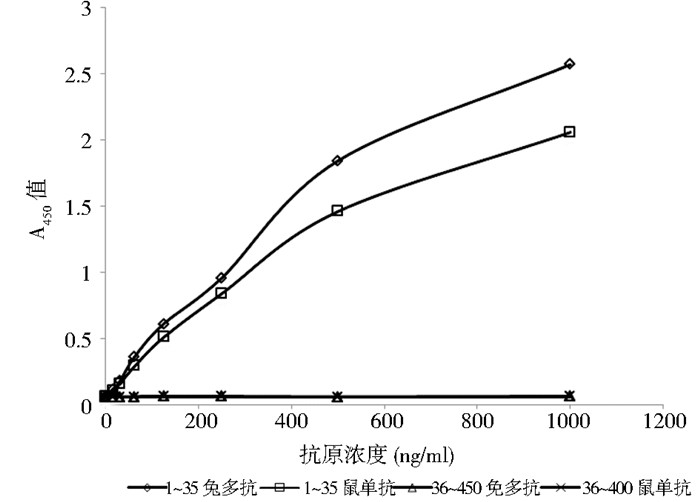
2.2 抗原结合区域鉴定Western blot实验结果表明(图 4),在EBOV NP 1~450(泳道1)有发光条带,而36~450(泳道2)没有发光条带,由此推测与抗体结合的抗原区域为EBOV NP 1~35。为进一步验证与抗体结合的抗原区域,本研究采用间接ELISA方法将多抗和单抗1C3分别与EBOV NP 1~35和36~450抗原进行结合实验。如图 5结果显示,以EBOV NP 1~35作为包被原进行ELISA实验,吸光值随着抗体浓度的增大而增大,说明1~35与抗体有特异性结合;而以36~450作包被原,ELISA的吸光值很低且基本不变。

|
| 图 4 EBOV NP Western blot结果 Figure 4 Western blot of EBOV NP left: made by monoclonal antibody 1C3, right: made by polyclonal antibodies |
|
| 图 5 ELISA方法测定抗原结合区域 Figure 5 Determination of antigen binding region by ELISA |
单抗4G3经Western blot和ELISA实验测定,其结果与1C3相似。说明本实验得到的两种单抗均与EBOV NP 1~35结合。
2.3 双抗体夹心ELISA法的建立 2.3.1 兔多抗与单抗配对的优选抗体配对实验结果表明,本实验获得的两株单抗均可以与兔多抗配对进行双抗夹心ELISA检测,其中单抗1C3的A450平均值为1.395(n=5),单抗4G3的A450平均值为1.105(n=5)。本实验选择了吸光值较高的单抗1C3与HRP偶联,制备酶标抗体。
2.3.2 包被抗体与酶标抗体最佳反应浓度的确定本研究采用方阵法同时对ELISA反应中的包被抗体与酶标抗体浓度进行优化(表 1)。选择P值在1.0左右且P/N值较大的包被抗体与酶标抗体的浓度。最终确定最佳包被抗体浓度为5 μg/ml,最佳酶标抗体稀释倍数为8 000倍。
| 酶标抗体 | 包被抗体 | |||
| 20μg/ml | 10μg/ml | 5μg/ml | 2.5μg/ml | |
| 1:8000(P) | 1.016 | 0.963 | 0.912 | 0.886 |
| (N) | 0.091 | 0.100 | 0.082 | 0.082 |
| P/N | 11.16 | 9.63 | 11.12 | 10.80 |
| 1:4000(P) | 1.879 | 1.796 | 1.7 | 1.622 |
| (N) | 0.146 | 0.149 | 0.116 | 0.118 |
| P/N | 12.86 | 12.05 | 14.66 | 13.74 |
| 1:2000(P) | 3.155 | 3.08 | 2.9 | 2.82 |
| (N) | 0.254 | 0.240 | 0.205 | 0.186 |
| P/N | 12.42 | 12.83 | 14.15 | 15.16 |
| 1:1000(P) | 3.989 | 3.872 | 3.794 | 3.565 |
| (N) | 0.404 | 0.356 | 0.291 | 0.247 |
| P/N | 9.87 | 10.88 | 13.04 | 14.43 |
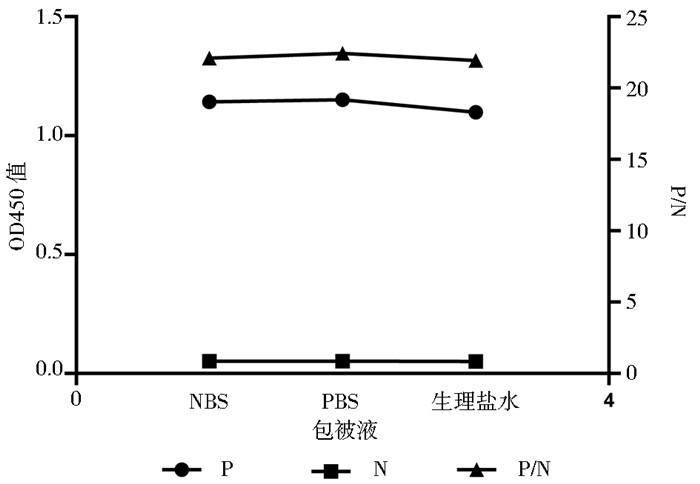
2.3.3 包被液的选择
利用3种包被液对实验进行测定,如图 6显示其效果差别不大,说明包被液对该检测方法的影响较小。因此选择了常用的磷酸盐缓冲液PBS作为包被液。
|
| 图 6 包被液的优化 Figure 6 Optimization of coating buffer |
以封闭液为单一变量进行优化,选择了产品化的酶标板稳定剂、3%脱脂奶粉、1%BSA和3%BSA分别对酶标板进行封闭。结果显示四种封闭液的封闭效果相近,由于1%BSA的P/N值略高于其他溶液,因此确定其作为最佳封闭液(图 7)。

|
| 图 7 封闭液的优化 Figure 7 Optimization of blocking buffer |
显色时间长短可能会对检测方法的灵敏度产生影响,因此本研究对底物作用时间进行了优化。根据P/N值的高低,选择了底物作用20min为最优条件(图 8)。

|
| 图 8 显色时间的优化 Figure 8 Optimization of color developing time |
根据以上优化结果,本研究建立了双抗夹心ELISA检测方法并绘制了标准曲线(图 9)。该检测方法在31.2 ~1 000 ng/ml之间线性范围较好(R2=0.9934)。
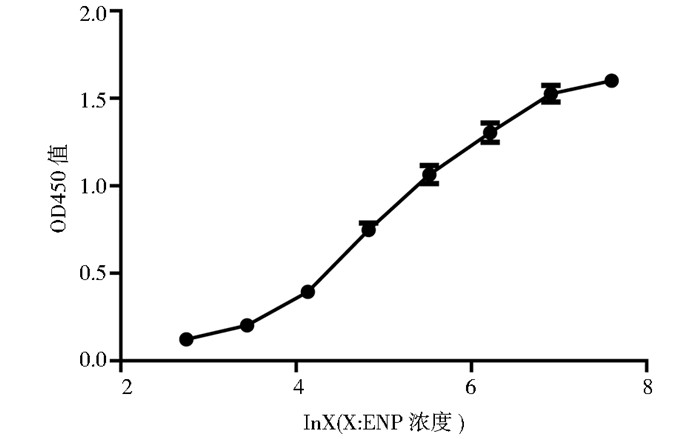
|
| 图 9 双抗夹心ELISA法标准曲线(n=5) Figure 9 Standard curve of sandwich ELISA assay(n=5) |
本研究测定出了该方法的最低检测限,即用PBS溶液作为空白样本进行检测,重复测定20次,计算A450的均数M和标准差SD,并将M+2SD值带入标准曲线,计算出对应的浓度值。经计算,最低检测限为2.6 ng/ml。
3 讨论2014年在西非多个国家爆发的埃博拉病毒疫情曾经触动了全世界的神经。虽然近两年人感染埃博拉病例鲜有报道,但对该病毒的防控工作仍不能松懈。在实验室检测方面,该病毒的核酸检测和免疫学检测方法可以在生物安全P3级实验室中进行,并且目前应用较为广泛[18]。PCR检测容易造成假阳性,恢复期患者在检测时也会有假阴性结果[19]。但由于该病疫情易爆发于非洲等不发达地区,因此非常有必要开发出操作简便、设备简单、能在现场快速检测EBOV的方法和试剂盒。
氨基酸序列高度保守的核蛋白是比较理想的检测目标物,而本实验选择的EBOV NP在不同亚型的埃博拉病毒之间较为保守。因此本研究以EBOV NP为检测目标物,瞄准对埃博拉病毒进行快速、定量检测这一目标,建立了针对EBOV NP的双抗夹心ELISA检测方法。本研究课题可作为我国防控埃博拉病毒的战略性技术储备,为今后开发出快速检测试剂盒及试纸条奠定了基础,并为埃博拉病毒病患者的治疗效果及预后监测提供工具。
4 结论本实验制备出了抗埃博拉核蛋白的单克隆和多克隆抗体,并对其结合区域进行了鉴定。在此基础上,通过对包被抗体与酶标抗体的浓度、包被液、封闭液以及TMB底物作用时间等条件的优化,建立了双抗夹心ELISA方法。该方法的最低检测限为2.6 ng/ml,可用于EBOV NP的定量检测。
| [1] |
林祥梅, 韩雪清, 王景林. 外来动物免疫. 北京: 科学出版社, 2014, 296-324. Lin X M, Han X Q, Wang J L. Exotic Animal Disease. Beijing: Science Press, 2014, 296-324. |
| [2] |
Pourrut X, Kumulungui B, Wittmann T, et al. The natural history of Ebola virus in Africa. Microbes and infection/Institut Pasteur, 2005, 7(7-8): 1005-1014. DOI:10.1016/j.micinf.2005.04.006 |
| [3] |
Li Y H, Chen S P. Evolutionary history of Ebola virus. Epidemiology and Infection, 2014, 142(6): 1138-1145. DOI:10.1017/S0950268813002215 |
| [4] |
Morikawa S, Saijo M, Kurane I. Current knowledge on lower virulence of Reston Ebola virus (in French:Connaissances actuelles sur la moindre virulence du virus Ebola Reston). Comparative Immunology, Microbiology and Infectious Diseases, 2007, 30(5-6): 391-398. DOI:10.1016/j.cimid.2007.05.005 |
| [5] |
程颖, 刘军, 李昱, 等. 埃博拉病毒病:病原学、致病机制、治疗与疫苗研究进展. 科学通报, 2014, 59(30): 2889-2899. Cheng Y, Liu J, Li Y, et al. Ebola virus disease:virology, pathogenesis, therapy, and vaccines. Chinese Science Bulletin, 2014, 59(30): 2889-2899. |
| [6] |
Knipe D M, Howley P M. Fields Virology. Philadelphia: Lippincott Williams & Wilkins, 2006.
|
| [7] |
Fauquet C, Mayo M A, Maniloff J. Virus Taxonomy. London: Elsevier/Academic Press, 2004.
|
| [8] |
Felomann H, Klenk H D. Marburg and Ebola virus. Advance in Virus Research, 1996, 47(2): 1-52. |
| [9] |
Shi W, Huang Y, Sutton-Smith M, et al. A filovirus-unique region of Ebola virus nucleoprotein confers aberrant migration and mediates its incorporation into virions. Journal of Virology, 2008, 82(13): 6190-6199. DOI:10.1128/JVI.02731-07 |
| [10] |
Watanabe S, Watanabe T, Noda T, et al. Production of novel Ebola virus-like particles from cDNAs:an alternative to Ebola virus generation by reverse genetics. Journal of Virology, 2004, 78(2): 999-1005. DOI:10.1128/JVI.78.2.999-1005.2004 |
| [11] |
Watanabe S, Noda T, Kawaoka Y. Functional mapping of the nucleoprotein of Ebola virus. Journal of Virology, 2006, 80(8): 3743-3751. DOI:10.1128/JVI.80.8.3743-3751.2006 |
| [12] |
Sullivan N, Yang Z Y, Nabel G J. Ebola virus pathogenesis:implications for vaccines and therapies. Journal of Virology, 2003, 77(18): 9733-9737. DOI:10.1128/JVI.77.18.9733-9737.2003 |
| [13] |
Dong S, Yang P, Li G, et al. Insight into the Ebola virus nucleocapsid assembly mechanism:crystal structure of Ebola virus nucleoprotein core domain at 1.8Å resolution. Protein & Cell, 2015, 6(5): 351-362. |
| [14] |
Kirchdoerfer R, Abelson D, Li S, et al. Assembly of the Ebola virus nucleoprotein from a chaperoned VP35 complex. Cell Reports, 2015, 12(1): 140-149. DOI:10.1016/j.celrep.2015.06.003 |
| [15] |
Dziubańska P J, Derewenda U, Ellena J F, et al. The structure of the C-terminal domain of the Zaire ebolavirus nucleoprotein. Acta Crystallographica, 2011, 70(Pt 9): 2420. |
| [16] |
Leung D W, Borek D, Luthra P, et al. An intrinsically disordered peptide from Ebola virus VP35 controls viral RNA synthesis by modulating nucleoprotein-RNA interactions. Cell Reports, 2015, 11(3): 376-389. |
| [17] |
Beatty J D, Beatty B G, Vlahos W G. Measurement of monoclonal antibody affinity by non-competitive enzyme immunoassay. J Immunol Methods, 1987, 100(1-2): 173-179. DOI:10.1016/0022-1759(87)90187-6 |
| [18] |
乔晋娟, 罗俊, 危宏平. 埃博拉病毒感染的实验室诊断方法研究进展. 科技导报, 2015, 33(1): 100-106. Qiao J J, Luo J, Wei H P. Research progress on laboratory diagnosis of Ebola virus infection. Science and Technology Review, 2015, 33(1): 100-106. DOI:10.3981/j.issn.1000-7857.2015.01.018 |
| [19] |
Towner J S, Rollin P E, Bausch D G, et al. Rapid diagnosis of Ebola hemorrhagic fever by reverse transcription-PCR in an outbreak setting and assessment of patient viral load as a predictor of outcome. Journal of Virology, 2004, 78(8): 4330-4341. DOI:10.1128/JVI.78.8.4330-4341.2004 |
 2017, Vol. 37
2017, Vol. 37




