文章信息
- 王世伟, 王敏, 王卿惠.
- WANG Shi-wei, WANG Min, WANG Qing-hui.
- Rhodococcus ruber CGMCC3090腈水合酶纯化、酶学性质及结晶研究
- Purification, Crystallization and Characterization of a Nitrile Hydratase from Rhodococcus ruber CGMCC3090 Strain
- 中国生物工程杂志, 2017, 37(10): 42-52
- China Biotechnology, 2017, 37(10): 42-52
- http://dx.doi.org/DOI:10.13523/j.cb.20171006
-
文章历史
- 收稿日期: 2017-06-27
- 修回日期: 2017-08-09
2. 工业微生物教育部重点实验室 天津科技大学生物工程学院 天津 300457
2. Key Laboratory of Fermentation Microbiology, Tianjin University of Science and Technology, Ministry of Education, Tianjin 300457, China
腈代谢微生物具有明显多态性,研究利用这些微生物资源,对生物转化(transformation)和生物修复(bioremediation)具有重要意义[1-2]。腈代谢微生物之所以能降解腈类化合物,是因为这些微生物能产生腈转化酶(nitrile converzymes)。腈转化酶包括腈水解酶(nitrilase, EC.3.5.5.1)、腈水合酶(NHase, EC4.2.1.84)和酰胺酶(amidase, EC3.5.1.4)[3]。不同菌株能产生的腈转化酶(腈水解酶、酰胺酶和腈水合酶)的种类不同。腈水解酶能将腈类化合物经酰胺转化为羧酸,酰胺酶只能将酰胺转化为相应的羧酸;而腈水合酶只能将腈转化为酰胺,不能继续将酰胺水解为羧酸[4]。因此,充分了解和利用不同微生物产生的不同的腈转化酶具有潜在价值。为了获得单一高附加值酰胺产品,人们希望获得只产生腈水合酶(NHase)的菌株,以避免产生副产物羧酸。然而遗憾的是,腈水合酶往往同酰胺酶共表达(co-expressed)[5],由此不可避免地会产生副产物羧酸。尽管有学者进行了各种尝试[6],例如降低培养温度抑制酰胺酶活性;在酰胺酶缺陷菌株中克隆和表达NHase基因;通过基因敲除技术阻断酰胺酶表达。但这些方法远未达到预期效果,无法获得所需要的高纯度的酰胺产品。
研究发现,使用酰胺酶和腈水解酶“双重”缺陷菌株是解决上述棘手问题最有效的方法。目前对此类“缺陷菌株”报道甚少,机理尚不清楚。例如,Ya-Jun等[7]分离到的Rhodococcus boritolerans FW815菌株表现出强烈的铁型水合酶活性,能将2, 2-二甲基环丙腈(DMCPCN)温和转化为2, 2-二甲基环丙甲酰胺(DMCPCA)。该菌株无酰胺酶和腈水解酶活性,属于酰胺酶和腈水解酶“双重”缺陷菌株。使用该菌株对腈类转化,能避免副产品2, 2-二甲基环丙甲酸的产生。我们课题组从天津近郊附近受腈类物污染的土壤中分离到一株产腈水合酶(NHase)的赤红球菌R.ruber CGMCC3090。研究发现该菌具有很高底物耐受性、宽泛底物特异性和二腈区域选择性,更重要的是该菌株为酰胺酶和腈水解酶双重缺陷型菌株[8-9]。经前期培养条件优化和菌种改良,目前菌体细胞酶活力已高达到4 296.8 U/ml[10-13]。使用该菌株能生产具商业价值的高纯度酰胺产品,如烟酰胺、5-氰基戊酰胺和肉桂酰胺[14-16]。笔者[17]在前期研究中,通过同源模建和底物对接,构建了该酶3D模型。研究发现,底物和酶的结合位点空穴为559Å3,与3QXE相比,具有更大催化空穴和结合位点空间。说明该菌株产生的腈水合酶具有很高的研究价值。为进一步研究该NHase结构和催化机制需要对其进行纯化和结晶。本实验对赤红球菌R.ruber CGMCC3090的纯化和酶学性质进行了研究,同时探索了酶蛋白的结晶方法,获得了NHase蛋白质单晶。经X射线衍射分析,分辨率达3.7Å,不仅为进一步研究酶结构和功能打下了基础,同时也为将来该腈水合酶在生物转化和生物修改中的实际应用提供重要科学数据。
1 材料 1.1 菌株及培养基 1.1.1 菌株Rhodococcus rube.CGMCC30菌株取自齐齐哈尔大学生命科学与农林学院分子实验室。该菌株分离于天津近郊受污染土壤。已在中国微生物菌种保藏管理委员会普通微生物中心(CGMCC)注册并保藏。斜面培养基、种子培养基和发酵培养基均使用本实验室常用方法配制。
1.1.2 仪器和药品烟腈和烟酰胺皆为色谱纯,购于美国Amresco公司;快速蛋白液相色谱(FPLC)仪、凝胶成像系统,以及电泳设备均为美国BIO-RAD产品;色谱柱购于日本JEOL公司;层析填料为北京(TransGen Biotech)产品;Agilent1100型高效液相色谱仪为美国Agilent产品;酶蛋白结晶试剂盒购于美国Hampton Research公司。结晶试剂盒共四套,分别是Crystal screen I,编号为HR2-110;Crystal screenII, 编号为HR2-112和Index screen,编号为HR-114。
2 方法 2.1 腈水合酶的提取按照Zhang等[18]报道的方法对R. ruber CGMCC3090菌株进行发酵培养96 h。收获菌体并测菌体酶活力。在4℃下,使用溶菌酶处理菌悬液40 h后,进行超声处理。超声破碎条件为400 W,60 min;超声分2次进行,每次30 min (60个循环,15 s超声,15 s间歇)。超声后用适量40 μmol/L磷酸缓冲液稀释,获得无细胞提取物,记作CFE。细胞提取物经40%~60%饱和度硫酸铵分级分离,沉淀用适量磷酸缓冲液收集,标记为ASF。
2.2 腈水合酶的纯化 2.2.1 疏水层析ASF液经真空冷干燥并浓缩10倍后,用0.45 μm滤膜过滤备用。采用Phenyl Sepharose 6 Fast Flow疏水层析方法,对浓缩的ASF液进行疏水柱层析(2.0×23)纯化。先用buffer B (1mol/L硫酸铵)平衡柱后,每次上样量为3 ml。上样后用64 ml buffer B,以4 ml/min流速平衡洗脱。平衡洗脱后,再用40%~0%硫酸铵(25 mmol/L PBS, pH7.4)溶液以4ml/min进行线性梯度洗脱。最后一步为等度洗脱(isocratic elution),使用25 mmol/L PBS buffer 120 ml以4.0 ml/min流速冲洗。将表现出最大酶活峰样品收集起来,标记为HIC,待做进一步层析。
2.2.2 离子交换层析HIC液经截留分子量为10 kDa透析膜透析过夜,然后用超滤管(Milipore Amicon Ultra-15,10kD)浓缩10倍。用25 mmol/L PBS buffer (pH 7.2)平衡柱后,将浓缩的HIC样品直接上样于DEAE-Sepharose FF(2.0×23cm)柱,每次进样量为3 ml。进样后用25 mmol/L PBS buffer (pH 7.2)以3.0 ml/min流速平衡洗脱。30 min后,用27 ml 80%~100%硫酸铵,以4.5 ml/min流速进行线性梯度洗脱(6 min)。最后用1.0 mol/L硫酸铵,以2.0 ml/min流速等梯度洗脱(isocratic flow),共用溶液8.0 ml。将表现最大酶活峰样品收集起来。样品经透析后,冻干、浓缩40倍,标记为IEC。
2.2.3 凝胶过滤层析取IEC浓缩样品,直接进样,进行凝胶过滤层析。采用SefinoseTMCL-6B (2.0×60 cm)进行凝胶过滤。每次进样5 ml。用25 mmol/L PBS buffer (pH 7.2)缓冲液平衡洗脱,流速4 ml/min。收集目的蛋白峰冷冻浓缩,标记为GFC。
2.3 分析方法 2.3.1 烟酰胺浓度测定参照相关文献[12]对烟酰胺浓度进行测定。
2.3.2 蛋白质浓度测定采用PIERCE公司生产的蛋白浓度测定试剂盒BCA kit方法,按说明操作,对所得酶液的蛋白质浓度进行测定。以BSA作为标准蛋白。
2.3.3 腈水合酶活性测定取1.5 ml EP管,加入8%烟腈溶液1.4 ml,摇匀。用微量移液枪吸取已经28℃预热粗酶液100 μl,与管中烟腈充分混合。28℃保温震荡5 min后,立即加10 μl 5 mol/L HCl终止反应。反应混合物放置在离心机内,12 000 r/min离心15 min。上清经0.22 μm水相滤膜过滤备用。取适当体积滤液进行稀释,20μl进样,经HPLC检验产物烟酰胺生成量,并计算腈水合酶活力。检测条件为:使用HPLC色谱仪(Agilent 1100 Series LC)、Kromasil 100-5 C18柱(4.6×250 mm)、检测器(G1322ADEGASSER G1314VWD),检测波长为260 nm,柱温为30℃,流动相为甲醇:冰乙酸:水=30:1:70,流速为1 ml/min,进样量为20 μl。活力单位定义(U):1个酶活力单位是指在特定条件(25℃,其它为最适条件)下,在1分钟内,经水合反应生成1 μmol/L产物(烟酰胺)所需要酶量。比活力定义为:酶毫克酶蛋白具有的酶活力(U/mg protein)。
2.3.4 电泳和分子量测定酶蛋白的电泳检验(Native-PAGE和SDS-PAGE)分别采用Davis[19]和Laemmli[20]的方法,并结合Okamoto等的方法[21]进行。
2.4 腈水合酶酶学性质研究 2.4.1 pH对腈水合酶的影响选用柠檬酸钠(pH 4, 4.5, 5和5.5)、磷酸钾(pH 6, 6.5,7, 和7.5)、Tris-HCl (pH 8 and 8.5)和甘氨酸-HCl (pH 9.0 and 10)不同pH缓冲体系,测定腈水合酶相对酶活力确定酶反应最佳pH值。
2.4.2 温度对腈水合酶的影响以产物烟酰胺峰面积百分比作为相对酶活。在最适pH下,考察了不同温度对腈水合酶活力的影响。所考察的温度范围在10℃~55℃之间,间隔5℃。
2.4.3 金属离子和螯合剂对腈水合酶的影响在最适pH和温度下,在0.25 mmol/L浓度下考察Cu2+, Zn2+, Fe3+, Fe2+, Ag+, Mg2+, Co2+, EDTA和碘乙酰胺(iodoacetamide)对腈水合酶的影响。
2.4.4 Vmax和Km的测定用25 mmol/L PBS buffer (pH 7.5)配制不同浓度(100 mmol/L,200 mmol/L,300 mmol/L,400 mmol/L)的底物烟腈溶液。取4个灭菌EP管,每管中加入不同浓度梯度的烟腈1.4 ml。30℃摇床预热10 min。然后迅速向各管中加入100 μl一定稀释倍数的酶液,立即在同样温度下反应5 min。反应后,加入10 μl 5 mol/L HCl终止反应。用HPLC测定产物烟酰胺生成量,计算不同底物浓度[S]下腈水合酶的反应初速度v。以1/v为纵坐标,以1/[S]为横坐标作Lineweaver-Burk图,建立直线回归方程。确定Vmax和Km。
2.4.5 试剂盒初筛采用坐滴和悬滴2种蛋白质结晶方法,对纯化的腈水合酶进行晶体初筛。
采用坐滴法时,向每个结晶池中分别加入试剂盒中不同的沉淀剂50 μl。向结晶版上的各加样孔中加入蛋白溶液(5 mg/ml)和沉淀剂各1 μl,混合。在温度为25℃,湿度55%的条件下结晶;每日显微镜下观察是否出现晶体。实验重复7次。
采用悬滴法时,由于板上有48池,点样必须使用硅化的盖玻片。池液加入量500 μl。蛋白溶液和沉淀剂各1 μl,混合后点样。结晶温度为20℃,湿度为55%,结晶时间为20 d。实验重复7次。
2.4.6 初筛样品染色使用考马斯亮兰G250(1%)对7次实验获得的所有坐滴板上的结晶样品进行染色。染色液需要经5 000 r/min离心5 min,弃沉淀,经无菌滤膜(0.22μm)过滤,除去染色剂杂质。染色时,染色液与各自样品池中的沉淀剂等量混合,每孔加1 μl混合液。重新封闭完好结晶板。次日观察,拍照记录。
2.4.7 结晶优化和蛋白质晶体的确定商业试剂盒(Hampton Research)以气相扩散法(Vapor diffusion)为基础。分别采用悬滴法(Hanging drop)和坐滴法(Sitting drop),对蛋白质结晶条件进行优化。优化参数包括蛋白质浓度和纯度(10、15 mg/ml)、温度为4℃、16℃和25℃、震动、湿度为55%。采用日本光机对盐晶体和蛋白质晶体分辨。
2.4.8 腈水合酶蛋白质单晶体的X衍射分析单晶体衍射在天津市国际生物医药联合研究院(Tianjin International Joint Academy of Biotechnology and Medicine, TJAB)进行。
3 结果与讨论 3.1 腈水合酶提取和纯化如图 1所示,菌体和底物烟腈反应后,稀释一定倍数(约10×),经HPLC得到层析图谱。前峰为烟酰胺峰,保留时间为2.540 min;后峰为烟腈峰,保留时间为4.408 min。从色谱图可以看出,没有副产物烟酸出现。说明该红球菌不产生腈水解酶和酰胺酶,只能产生腈水合酶,由此可见,Rhodococcus rube.CGMCC30菌株为腈水解酶和酰胺酶“双重缺陷”菌株。对该菌种的后续研究具有潜在应用价值。本次实验使用的菌体发酵液共计3.5 L,经计算总酶达1730240 U,发酵液比酶活约等于494 U/ml。研究发现,加一定溶菌酶进行二次超声,明显优于不加溶菌酶一次超声。采用40%~70%硫酸铵饱和度进行硫酸铵分集分离效果最佳。

|
| 图 1 高压液相色谱检验腈水合酶催化烟酰胺的生成 Figure 1 Determination of nicotinamide as a only product by using HPLC method |
如表 1所示,经超声的无细胞提取物(CFE)总酶活力达131641U。经计算菌体释放腈水合酶为13.1%。相比之下,Prasad等[22]所纯化的Rhodococcus rhodochrous PA-34菌株腈水合酶释放率仅8.7%。经3步层析,腈水合酶产率高达26.2%,纯化倍数为17.14倍。比酶活性高达1148.5U/mg。有学者[23]比较了近20种纯化后腈水合酶得比酶活,发现各种来源的腈水合酶的比酶活力处于5.9~1890 U/mg之间。大部分为1 000 U/mg。尽管个别高达6 290 U/mg[24],但属于重组腈水合酶。
| Purification steps | Total activity (U) | Total protein (mg) | Specific activity(U/mg) | Yield(%) | Fold purification |
| CFE | 131641 | 1964 | 67 | 100 | 0 |
| ASF | 123382 | 1560 | 79.1 | 93.7 | 1.18 |
| HIC | 86120 | 614 | 140 | 65.4 | 2.09 |
| IEC | 54234 | 180 | 301 | 41.2 | 4.49 |
| GFC | 34454 | 30 | 1148.5 | 26.2 | 17.14 |
研究发现,依次按HIC、IEC和GFC三种层析顺序,纯化腈水合酶,能节省大量时间和试剂。并能获得得率高和纯度优的腈水合酶。
图 2表示,纯化后的腈水合酶在4%浓缩胶和12%分离胶上获得SDS-PAGE电泳图。M为蛋白ladder(单位为kDa)由图 2可知,该酶由2个亚基组成,2个亚基在24 ~30 kDa之间。腈水合酶一般由2个亚基构成,其分子量在22 kDa~ 29 kDa之间,α和β亚基中较小者为α亚基,较大者为β亚基[4]。因此断定接近30 kDa条带是β亚基,接近24 kDa条带为α亚基。

|
| 图 2 纯酶SDS-PAGE电泳图 Figure 2 SDS-PAGE of purified NHase |
图 3为Native-PAGE电泳获得的电泳图谱,Native-PAGE电泳同SDS-PAGE电泳操作基本相同。腈水合酶属于酸性蛋白,通常在Native-PAGE电泳中采用pH8.8缓冲系统,蛋白带负电荷向阳极移动。图中的Native-PAGE Maker:Ferritin horse(450 kDa),Catalase bovine(240 kDa),Aldolase rabbit(160 kDa),Albumin bovine (BAS)(67 kDa)。根据Native-PAGE结合分子量标准对全酶的分子量进行计算发现该腈水合酶的全酶分子量为105 kDa。根据蛋白迁移距离和染料迁移距离的比值,可求出各蛋白相对迁移率mr。然后作出lgMr~mr关系图,确定两个亚基的分子量。计算结果表明,α亚基分子量约为24.3 kDa,β亚基分子量为28.0kDa。根据全酶分子量在105 kDa,可以断定腈水合酶全酶由α2β2四聚体组成。前面Prasad等已经报道[4],腈水合酶全酶分子量在54 kDa~530 kDa之间,NHase03090全酶分子量处于该范围内。

|
| 图 3 纯酶Native-PAGE电泳图 Figure 3 Nativve-PAGE of purified NHase |
图 4显示,不同pH值对腈水合酶相对酶活力影响。测试范围在pH4.0~10.0之间。在pH 7~8之间,其相对酶活力保持较高水平,酶最适pH值为7.5。在pH5和pH9.5时,仍保持50%左右的酶活。由此可见该腈水合酶的耐酸、耐碱能力较强。当pH7.5下降到5.5的过程中,酶活力缓慢下降,但仍然具80%以上的酶活力。然而,pH值从5.5下降到4.5时,该腈水合酶活力从83%陡然下降到22%。pH从7.5上升到10的过程中,酶活力快速下降一半以上。腈水合酶生理条件下,酸碱度处于pH6.5~8.5之间。pH值降低(如pH < 7)使酶活力迅速下降的原因可以从两个方面解释:一方面低pH能促使酶蛋白质变性,亚基解离;另一方面由于酶的氨基酸巯基的pKa正好为4.6[25],可能在酸性pH情况下酶活性位点关键氨基酸被离子化所致。

|
| 图 4 pH对腈水合酶的影响 Figure 4 Effect of pH on the relative activity of nitrile hydratase purified |
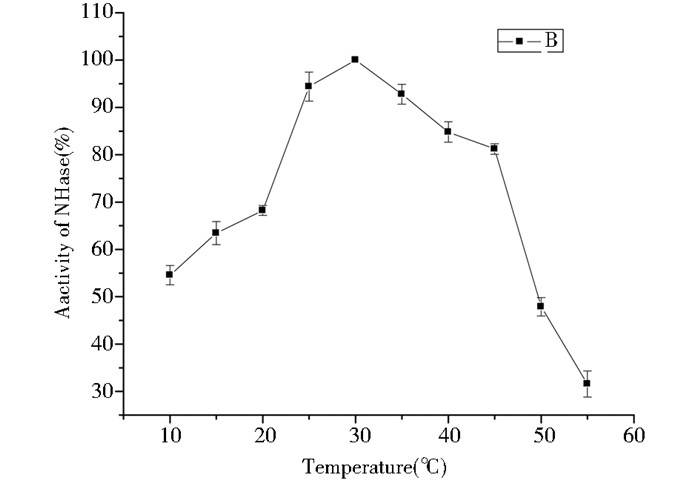
在最适pH7.5条件下,以烟腈为底物,研究了不同温度对腈水合酶的影响。
图 5所示腈水合酶最适温度为30℃,从30℃下降到20℃,活力下降了35%,表明该范围内温度变化对腈水合酶影响较大。然而,从20℃下降至10℃,活力仅下降了10%。另一方面,温度从30℃上升到45℃,腈水合酶仍然能保持80%;而温度从45℃继续上升到55℃,腈水合酶的酶活力迅速下降,从80%陡然下降到30%。同R. ruber CGMCC3090菌静息细胞相比[9]、,显然酶活性下降更快,其原因可能是由于菌体细胞相关成分对酶具有保护作用。尽管有些菌株的腈水合酶能耐受较高的温度,但大多数是不耐热的,最适温度一般20~35℃。一个例外是灵杆菌Serratia marcescens钴型腈水合酶[26]是嗜热的,最适温度可达为50℃,7小时内能有效将腈转化为相应的酰胺。然而R. ruber CGMCC3090腈水合酶在50℃时,酶活几乎损失一半。因此该腈水合酶耐热性是一个有待解决的问题。
|
| 图 5 温度对腈水合酶的影响 Figure 5 Effect of temperature on the relative activity of nitrile hydratase purified |
如表 2所示,不同金属盐离子、螯合剂和烷化剂对腈水合酶相对酶活力均具有影响。以不加上述金属离子的纯酶液为对照(control),酶活力记作100%。结果显示,Fe2+, Fe3+, Mg2+和Ca2+对腈水合酶相对活力有促进作用,但影响不大。然而,该睛水合酶明显受到Ag+、Cu2+和Mn2+抑制。Ag+几乎完全抑制腈水合酶相对酶活力,与对照相比,相对酶活仅存0.341%。Co2+对酶的促进作用最大,相对酶活力比对照(control)提高了36%。一般腈水合酶分铁型和钴型腈水合酶,Co2+能促进该腈水合酶,说明该腈水合酶属于后者。有意义的是Zn2+和Cd2+也能明显促进该腈水合酶活力。有报道Rhodococcus sp. RHA 1腈水合酶包括1个钴、2个铜和1个锌[27],R.ruber CGMCC3090腈水合酶活性中心是否存在其他的金属离子(例如Zn2+和Cd2+)尚有待进一步研究。
| 采用的各种试剂的浓度(0.25mmol/L) | 纯化的腈水合酶相对酶活(%) |
| CuS | 13 |
| ZnSO4 | 124 |
| FeCl3 | 105 |
| FeCl2 | 102 |
| AgNO3 | 0.341 |
| MgSO4 | 107 |
| MnCl2 | 69 |
| CoCl2 | 136 |
| CdSO4 | 129 |
| EDTA | 78 |
| Iodoacetamide | 0.214 |
Zn2+能明显促进酶活力,这一点与本实验室宋洋等研究的该菌体细胞受Zn2+的影响不同,推测不同浓度Zn2+会对腈水合酶有不同的影响,或者有可能在全细胞催化下,有些金属离子相互影响所致。Fe2+、Fe3+对酶活力有促进作用,与Song等[11]报道完全一致。0.25 mmol/L EDTA使腈水合酶相对酶活力下降22%;烷化剂碘乙酰胺(iodoacetamide)几乎使腈水合酶活力完全丧失,推测碘乙酰胺与腈水合酶半胱氨酸和组氨酸反应导致酶蛋白变性失活。
3.2.4 Km和Vmax根据双倒数作图法获得直线回归图,测得1/Vmax=4.7823,Km/Vmax=0.8551,可计算出Vmax=209.1μmol/L·min·mg,Km=178.8 mM。相比之下,R. rhodochrous J1的H-NHase的Vmax和Km值分别为370 μmol/L·min·mg和200 mM[28],说明该腈水合酶在动力学方面类似于H-NHase[29-30]。HNHase在工业生产酰胺上起了重要作用,可工业化生产包括烟酰胺在内的多种酰胺。作为第三代催化剂,已经取代了Pseudomonas chlororaphis B23和Rhodococcus sp. 774菌株。R. ruber CGMCC3090在动力学方面类似于HNHase,从某种意义来讲,该腈水合酶在工业上生物转化中具有潜在应用价值。
3.2.5 晶体筛选和优化(1) 腈水合酶的结晶的初步筛选采用坐滴板,使用Crystal screening I试剂盒,在温度25℃,湿度为55%条件下,结晶时间为10 d的条件下,从4套试剂盒中的194种不同试剂中,筛选出45种试剂可产生晶体(盐晶体或蛋白晶体)。在以后结晶实验中,使用初筛获得的这45种试剂进行实验以减轻工作量,节约腈水合酶蛋白。但在实验中存在棘手的问题是无法用肉眼直接分别盐晶和蛋白质晶体的区别。
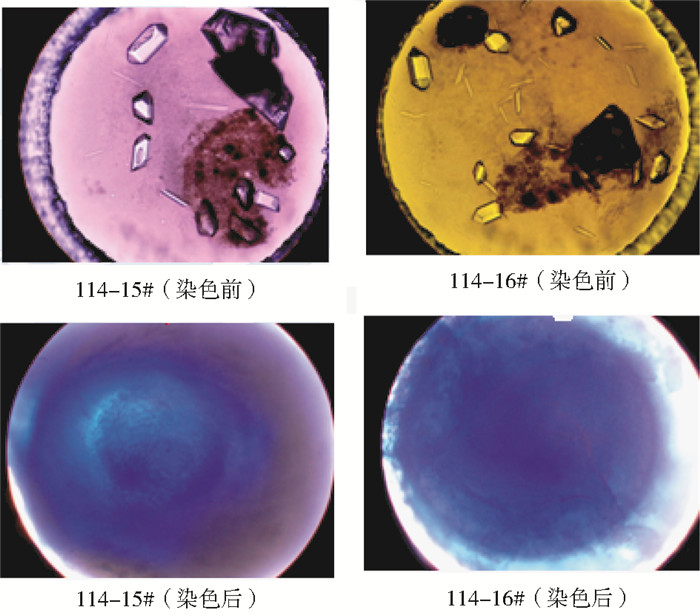
(2) 染色法对晶体的辨别晶体进行染色,主要目的是为了快速将盐晶和蛋白质晶体加以区分。一般蛋白晶体很漂亮,表面反光,没有盐晶体大,外表比较规则;但染色后,盐晶体不着色,蛋白晶体着色。这是染色法区别蛋白质晶体和盐晶体的主要依据。
如图 6所示,颜色对大部分‘晶体’不着色,说明不是蛋白晶体。图 6分别为采用114-15#和114-16#两种沉淀剂结晶出来的晶体。染色前晶体形状极佳,但染色后晶体消失,说明产生的为盐晶。也说明这两种试剂不易使该腈水合酶形成蛋白晶体。
|
| 图 6 染色前后结晶的对比 Figure 6 Hampton Research crystal by dyeing |
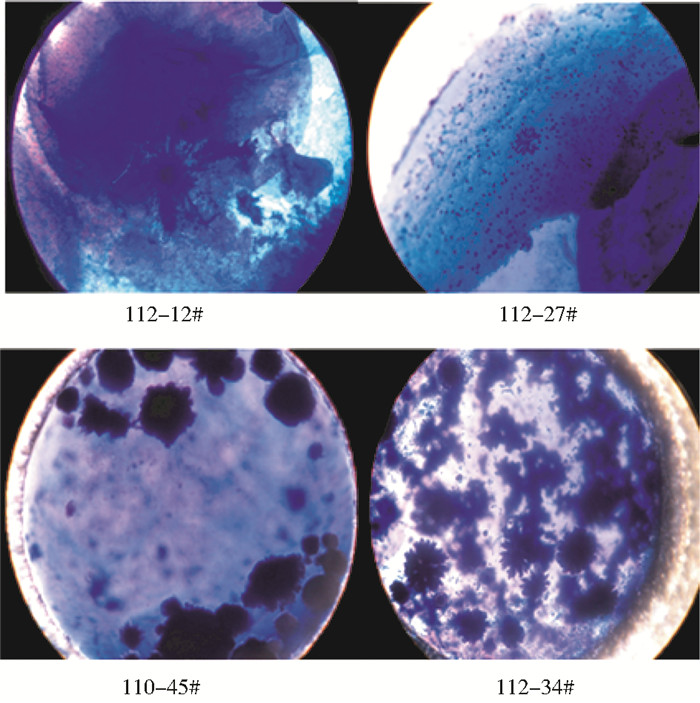
图 7为采用4种沉淀剂对酶蛋白进行结晶,结晶后经考马斯亮蓝染色获得的图片。结果发现,使用112-12#和112-27#试剂均有蛋白晶体出现;使用110-45#和112-34#沉淀剂能使酶蛋白出现着色的‘大花状’晶体。
|
| 图 7 染色后结晶形态 Figure 7 Hampton Research crystal by dyeing |
染色后从45种试剂中淘汰了大部分初筛结晶试剂。可以确定110-24#,110-45#,10-46#;1112-12#,112-27#;112-34#;114-60#,114-64#,114-93#沉淀剂能使酶蛋白高频率发生结晶,同时又能着色。说明经上述试剂结晶形成了蛋白质晶体。
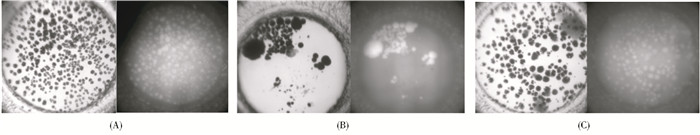
3.3 光机拍照和试剂盒对晶体的优化图 8所示为在蛋白质浓度为10 mg/ml、16℃情况下,经过21 d结晶获得的结果。
|
| 图 8 光机对110-45#(A)、112-12(B)和114-93(C)结晶的晶体进行拍照的结果 Figure 8 Complex drop imaged under visible (left) and UV (right) light using 110-45#(A), 112-12#(B) and 114-93#(C) |
图 8为使用日本理光(Desktop MinstrelTM)RIGAKU光机对形成的晶体的拍摄图。图中A、B和C均包括2张图片,左侧为光机的可见光拍摄,右侧为光机的紫外光拍摄。可以看出,相对可见光下,晶体是暗黑色的;而在紫外光下,晶体是光亮的。因此可以断定,这几种试剂均能使酶蛋白形成晶体。由此说明,使用日本理光光机(Desktop MinstrelTM)RIGAKU,进行盐晶和蛋白晶的辨别是十分有效的,能节省大量的颜色和观察时间。
实验发现其他一些试剂也能使酶蛋白形成晶体,但是效果不如上述这3种试剂。尽管上面的图中都有晶体出现,但质量很差,没有很规则的几何形状,基本是‘散花’状的、‘刺球’状的晶体或者是蛋白聚合物,不能进行X射线衍射。试验中也发现,冰箱(4℃)中放置或室温(25℃)放置21d,结晶板出现较多盐晶,因此确定16℃恒温是蛋白晶体形成的最佳温度。蛋白质单晶出现尽管和许多因素有关,但是在使用试剂盒情况下,蛋白质浓度起着至关重要作用。因此,实验又进行了一次优化。结果表明,当蛋白质浓度为10 mg/ml,pH 7.5,温度为16℃,湿度55%,结晶生长时间为21 d效果最佳。
图 9 (A和B)所示,后者(B)已经形成一定的小花状晶形,而且光机紫外拍照明显有花状亮点。而前者(A),尽管亮度很大,但仍然为球状亮点。图 9(C)为112-34#号试剂结晶的结果,从图中可见,晶体呈大花状,紫外光下,花状亮度明显。研究发现,使用不同的沉淀剂所产生的晶体有放射性的、圆饼状的、针状的和聚集成团的;而且晶体大小也各异。另一方面,使用紫外光机能辨别晶体与染色法基本吻合。实验还发现,许多试剂易使酶蛋白形成盐晶,其中试剂中大都含有Ca2+、Cu2+、Mg2+、Fe3+等离子;然而,含有Cd2+、Zn2+、Co2+等金属离子的试剂却易使该腈水合酶产生蛋白晶体。
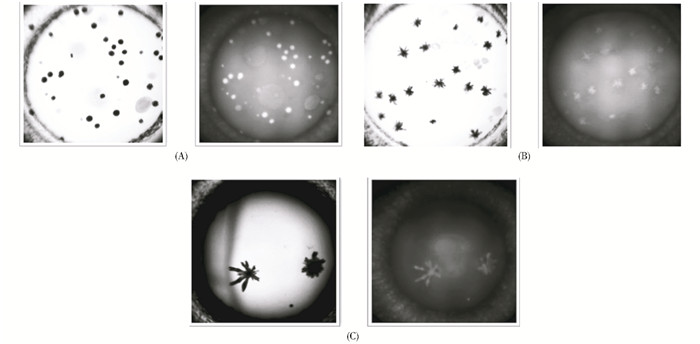
|
| 图 9 光机对优化的110-45#(A)、114-93#(B)和112-34#(C)拍照的结果 Figure 9 Optimization complex drop imaged under visible (left) and UV (right) light using 110-45#(A), 114-93#(B)and 112-34#(C) |
对110-45#结晶的蛋白初步进行X射线衍射结果。衍射结果表明,110-45#形成的晶体确实是蛋白晶体,不是盐晶体。同样也对其他几种晶体进行了衍射,结果和110-45#一样,也是蛋白晶体。它们分别是112-34#,114-93#,112-12#,112-25#和112-27#。
从表 3中可以看出,这6种试剂中的金属离子一共包括三种,它们分别是Zn2+, Co2+和Cd2+,为什么腈水合酶能在含这3种金属离子的试剂中培养时出现结晶值得研究。
| 110-45# | 112-12# | 112-25# | 112=27 | 112-34# | 114-93# |
| 0.2mol/L Zinc acetate dehydrate | 0.1 mol/L Cadmium chloride hydrate | 0.01mol/L Cobalt(II)chloride hexahydrate | 0.01mol/L Zinc sulfate heptahydrate | 0.05mol/L Cadmium sulfate hydrate | 0.05mol/L Zinc acetate dihydrate |
| 0.1mol/L Sodium cacodylate trihydrate pH6.5 | 0.1 mol/L Sodium acetate trihydrate pH4.6 | 0.1mol/L mol/MES monohydrate pH 6.5 | 0.1mol/L MES monohydrate pH 6.5 | 0.1mol/L HEPS pH7.5 | 20% w/v polyethylene glycol 3350 |
| 18% w/v Polyethylene glycol 8000 | 30% v/v Polyethylene glycol 400 | 1.8mol/L Ammonium sulfate | 25%v/v Polyethylene glycol monomethyl ether 550 | 1.0mol/L Sodium acetate trihydrate |
上述6种试剂尽管能产生蛋白晶体,但由于晶体质量差,衍射能力都不高。有必要进一步优化。由于112-34#号较好,因此选择112-34#号对结晶条件进行了进一步优化。保持蛋白质浓度不变,提高了结晶时间至30 d,其他条件不变。图 10所示为优化后的结果。

|
| 图 10 12-34#试剂盒优化的蛋白质晶体(10×10) Figure 10 The protein crystal optimization using 112-34#(10×10) |
研究发现,结晶初期根本看不见蛋白质晶体,有沉淀发生;但随后周边长出微小晶体,并形成“圆形部落圈”,在圈上小晶体逐渐长大。
图 11为腈水合酶用112-34#试剂优化的晶体(图左)和X射线衍射图谱(右图)。左图为30 d后拍照的结果。在100×s视野内形成了大小为100~120 μm不同的蛋白质晶体。右图为选取左图最大晶体进行X射线衍射。可见衍射图中有衍射点出现,说明蛋白质晶体为蛋白质单晶。在最佳结晶条件下,采用112-34#试剂(含有Cd2+)试剂,能使腈水合酶产生较好蛋白质单晶。X射线衍射结果表明,分辨率达到3.7Å(右图)。实验为进一步研究该腈水合酶分子结构和酶蛋白的功能奠定了基础。

|
| 图 11 对优化的112-34#结晶的蛋白进行X射线衍射结果 Figure 11 X ray diffraction of 112-34# |
| [1] |
Prasad S, Misra A, Jangir V P, et al. A propionitrile-induced nitrilase of Rhodococcus sp. NDB 1165 and its application in nicotinic acid synthesis. World Journal of Microbiology and Biotechnology, 2007, 23(3): 345-353. DOI:10.1007/s11274-006-9230-5 |
| [2] |
Cantarella L, Gallifuoco A, Malandra A, et al. High-yield continuous production of nicotinic acid via nitrile hydratase-amidase cascade reactions using cascade CSMRs. Enzyme and Microbial Technology, 2011, 48(4-5): 345-350. DOI:10.1016/j.enzmictec.2010.12.010 |
| [3] |
Pratush A, Seth A, Bhalla T C. Cloning, sequencing, and expression of nitrile hydratase gene of mutant 4D strain of Rhodococcus rhodochrous PA 34 in E. coli. Applied Biochemstry Biotechnology, 2012, 168(3): 465-486. DOI:10.1007/s12010-012-9790-9 |
| [4] |
Prasad S, Bhalla T. Nitrile hydratases (NHases):At the interface of academia and industry. Biotechnology Advances, 2010, 28(6): 725-741. DOI:10.1016/j.biotechadv.2010.05.020 |
| [5] |
Sharma M, Sharma N N, Bhalla TC. Amidases:versatile enzymes in nature. Rev Environ Sci Biotechnol, 2009, 8(4): 343-366. DOI:10.1007/s11157-009-9175-x |
| [6] |
Ma Y, Yu H, Pan W, et al. Identification of nitrile hydratase-producing Rhodococcus ruber TH and characterization of an amiE-negative mutant. Bioresource Technology, 2010, 101(1): 285-291. DOI:10.1016/j.biortech.2009.07.057 |
| [7] |
Jun Y, Wang Z Q, Liu R C, et al. Screening, cultivation, and biocatalytic performance of Rhodococcus boritolerans FW815 with strong 2, 2-dimethylcyclopropanecarbonitrile hydratase activity. Journal of Industrial Microbiology & Biotechnology, 2012, 39(3): 409-417. |
| [8] |
Zhang J L, Wang M, Sun H, et al. Isolation and characterization of Rhodococcus ruber CGMCC3090 that hydrolyzes aliphatic, aromatic and heterocyclic nitriles. African Journal of Biotechnology, 2009, 8(20): 5467-5486. |
| [9] |
Zhong L P, Zhang J L, Song Y, et al. Study on screening, identification and culture conditions of a strain with nitrile hydratase. Journal of Tianjin University of Science and Technology, 2008, 23(4): 35-39. |
| [10] |
Zhang J L, Wang M, Zhong L P, et al. Identification and study on enzymic activity of Rhodococcus sp.TCCC 28001 with nitrile hydratase activity. Journal of Shandong Agricultural University (Natural Science), 2010, 41(1): 11-16. |
| [11] |
Song Y, Zhong L P, Zhang J L, et al. Effect of metal ions on Rhrodococcus sp. nitrile hydratase activity. Journal of Tianjin University of Science and Technology, 2009, 24(1): 11-14, 19. |
| [12] |
Wang S W, Wang M, Wang Q H. Breeding of NHase hyper-producing Rhodococcus ruber strain LUV30-06 and verification of mutants by RAPD. American Journal of Molecular Biology, 2013, 3(2): 108-114. DOI:10.4236/ajmb.2013.32015 |
| [13] |
Wang S W, Wang Q H, Zhai Y, et al. Screening of NHase high-producing Rhodococcus ruber strain TQD-58 by both parents inactivated protoplast fusion. 2012 International Symposium on Information Technology in 279 Medicine and Education (ITME2012), Hokkaido, Japan.2012, 737-740.
|
| [14] |
Zhang J L, Wang M, Zhong L P, et al. Identification and study on enzymic activity of Rhodcoccus sp.TCCC28001 with nitrile hydratase activity. Journal of Shangdong Agricultural University (Natural science), 2010, 41(1): 11-16. |
| [15] |
Shen Y B, Wang M, Li X D, et al. Highly efficient synthesis of 5-cyanovaleramide by Rhodococcus ruber CGMCC3090 resting cells. Journal of Chemical Technology and Biotechnology, 2012, 87(10): 1396-1400. DOI:10.1002/jctb.v87.10 |
| [16] |
Zhai Y, Shen Y B, Tang R, et al. Biotransformation of cinnamonitrile into cinnamamide with Rhodococcus ruber CGMCC3090. Journal of Tianjin University of Science and Technology, 2014, 29(4): 11-15. |
| [17] |
Wang S W, Dai Y J, Wang J X, et al. Molecular insights into substrate specificity of Rhodococcus ruber CGMCC3090 by gene cloning and homology modeling. Enzyme and Microbial Technology, 2013, 52(2): 111-117. DOI:10.1016/j.enzmictec.2012.11.004 |
| [18] |
Wang S W, Wang M. Research process on microbial diversity of producing-nitrile hydratase and study on nitrile hydratase. China Biotechnology, 2011, 31(9): 117-123. |
| [19] |
Davis B, DISC J. Electrophoresis-Ⅱ. method and application to human serum proteins. Annals of the New York Academy of Sciences, 1964, 121(2): 404-427. |
| [20] |
Laemmli U K. Cleavage of structural protein during the assembly of the head of bacteriophage T4. Nature, 1970, 227: 680-685. DOI:10.1038/227680a0 |
| [21] |
Okamoto S, Eltis L D. Purification and characterization of a novel nitrile hydratase from Rhodococcus sp. RHA1. Molecular Microbiology, 2007, 65(3): 828-838. DOI:10.1111/mmi.2007.65.issue-3 |
| [22] |
Prasad S, Raj J, Bhalla T C. Purification of a hyperactive nitrile hydratase from resting cells of Rhodococcus rhodochrous PA-34. Indian J Microbiol, 2009, 49(3): 237-242. DOI:10.1007/s12088-009-0033-x |
| [23] |
Wang S W, Wang Q H, Wang W Y, et al. Research process on protein purification of nitrile hydratase. Journal of Science of Teachers College and University, 2013, 33(1): 50-53. |
| [24] |
Kamila Rzeznicka, Sebastian Schätzle, Dominique Böttcher, et al. Cloning and functional expression of a nitrile hydratase(NHase)from Rhodococcus equi TG328-2 in Escherichia coli, its purification and biochemical characterization. Appl Microbiol Biotechno, 2010, 85(5): 1417-1425. DOI:10.1007/s00253-009-2153-y |
| [25] |
Cramp R A, Cowan D A. Molecular characterization of a novel thermophilic nitrile hydratase. Biochem Biophys Acta, 1999, 1431(1): 249-260. |
| [26] |
Li Q J, Liu Z Q, Zhang Y G, et al. Identification and characterization of Serratia marcescens ZJB-09104, a nitrile-converting bacterium. World Journal of Microbiology and Biotechnology, 2010, 26(5): 817-823. DOI:10.1007/s11274-009-0238-5 |
| [27] |
Okamoto S, Eltis L D. Purification and characterization of a novel nitrile hydratase from Rhodococcus sp. RHA 1. Mol Microbiol, 2007, 65(3): 828-838. DOI:10.1111/mmi.2007.65.issue-3 |
| [28] |
Wieser M, Takeuchi K, Wada Y, et al. Low-molecular-mass nitrile hydratase from Rhodococcus rhodochrous J1:purification, substrate specificity and comparison with the analogous high-molecular-mass enzyme. FEMS Microbiology Letters, 1998, 169(1): 17-22. DOI:10.1111/fml.1998.169.issue-1 |
| [29] |
Mauger J, Nagasawa T, Yamada H. Synthesis of various aromatic amide derivatives using nitrile hydratase of Rhodococcus rhodochrous J1. Tetrahedron, 1989, 45(5): 1347-1354. DOI:10.1016/0040-4020(89)80133-4 |
| [30] |
Mauger J, Nagasawa T, Yamada H. Nitrile hydratase-catalyzed production of isonicotinamide, picolinamide and pyrazineamide from 4-cyanopyridine, 2-cyanopyridine and cyanopyrazine in Rhodococcus rhodochrous J1. Journal of Biotechnology, 1988, 8(1): 87-95. DOI:10.1016/0168-1656(88)90071-5 |
 2017, Vol. 37
2017, Vol. 37




