文章信息
- 刘宝利, 刘高冈, 林秋卉, 李炳志, 元英进.
- LIU Bao-li, LIU Gao-gang, LIN Qiu-hui, LI Bing-zhi, YUAN Ying-jin.
- 多质粒共转化组合筛选方法构建木糖利用酿酒酵母的研究
- Construction of Recombinant Xylose-utilizing Saccharomyces cerevisiae by Three-plasmid Co-transformation Combinatorial Screening Method
- 中国生物工程杂志, 2016, 36(12): 86-97
- China Biotechnology, 2016, 36(12): 86-97
- http://dx.doi.org/DOI:10.13523/j.cb.20161213
-
文章历史
- 收稿日期: 2016-05-03
- 修回日期: 2016-05-13
合成生物学和代谢工程领域的一个主要挑战,是实现底盘菌株内源和外源代谢模块之间的适配,获得高产目标产物的菌株。实现模块间的适配,主要是为了避免基因过表达带来的代谢负担、辅因子特异性造成的辅因子不平衡以及不稳定或有毒中间产物积累造成的生长抑制等[1-3]。以往的研究中,最常见的平衡代谢流的方法包括过表达关键基因、删除支路基因以及利用蛋白质工程改造关键酶的活性;改造元件通常包括基因(密码子)、启动子和基因调控核糖体结合位点(RBS)等[4-6]。随着合成生物学以及人工合成基因组的发展,对底盘细胞的改造已经由单点改造变成模块化、全局的改造,即“组合优化”的思想和方法[7]。
酿酒酵母的木糖利用是纤维素乙醇工业化生产的一个限制因素,木糖是木质纤维素原料中含量次高的单糖,其有效利用可以大幅度提高原料利用率。现有的木糖利用酿酒酵母工程菌株,普遍引入了自然界中天然存在的木糖代谢途径[8]。其中一条是广泛存在于树干毕赤酵母等真菌中的木糖还原酶-木糖醇脱氢酶途径(简称XR-XDH途径),在这条途径中木糖醇脱氢酶和木糖还原酶的辅因子偏好性不同,会导致细胞内氧化还原力的失衡,最终造成木糖醇的积累和乙醇得率的降低[9]。此外,有研究表明XK表达量过高或过低都会影响细胞生长,从而影响产物的得率[10]。因此,在XR-XDH途径中,优化编码XR、XDH和XK的三个基因的相对表达量对酿酒酵母的木糖利用有重要作用[11]。
2012年,Du等[12]通过易错PCR获得突变的启动子,以绿色荧光蛋白(GFP)作为报告基因筛选强度不等的启动子建立启动子文库。经启动子间的组合筛选得到的优势菌株最快木糖消耗速率可达0.92 g/L/h,证明了通过优化启动子改变代谢通量以优化代谢通路的可行性。2013年,Kim等[13]通过组合不同来源的XR、XDH和XK基因表达元件,筛选到一株木糖消耗速率达到0.62 g/L/h,乙醇得率为0.27 g/g的菌株。上述组合化的筛选方法可以经一轮组装得到理论上的所有组合,但是必须对所有的组合模块都进行组装、转化底盘菌株后才能进行筛菌,工作量大。此外,这种构建模式对组装也提出了要求,利用基于同源臂的酵母组装就必须提前构建含有同源臂的候选元件库,这不仅使片段的获得难度更大,还增加了实验研究成本。
本研究提出了三质粒共转化介导的基因表达模块组合筛选方法,并以木糖代谢路径(XR-XDH途径)在酿酒酵母中的构建为例对此方法进行阐释。如图 1所示,本方法由三部分组成:第一部分利用含有Yeast Golden Gate (yGG) [14]连接标准接口的元件库构建代谢路径基因单一表达盒(Transcription Unit, TU);第二部分组合转化单一表达盒,表达模块无需组装,直接利用三质粒共转化系统建立菌库,进行初步筛选,从而淘汰部分不良组合,筛选性状相对优良菌株,并对优良组合进行组装后再次进行转化,以确保更加稳定地表达;第三部分对转化后菌株进行进一步的发酵验证,研究各菌株性状,获得目标菌株。
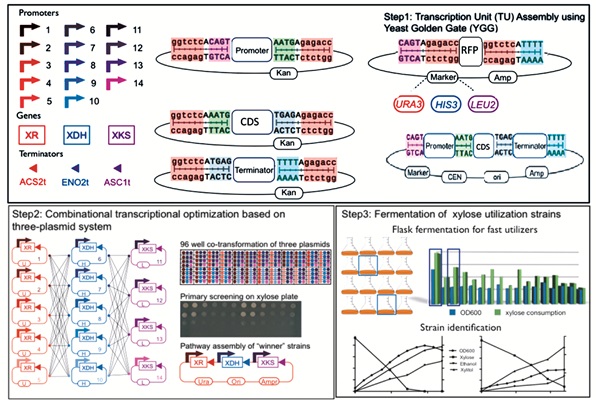
|
| 图 1 三质粒共转化介导的菌株组合筛选方法整体设计 Figure 1 Overall scheme of combinatorial screening method based on three-plasmid co-transformation system |
研究所用到的酵母菌株和质粒见表 1。大肠杆菌DH5α作为质粒克隆载体购自北京博迈德基因技术有限公司。
| 菌株与质粒 | 特征描述 | 来源 |
| 菌株 | ||
| BY4741 | MATa (Leu2Δ0 LYS2 met15 his3Δ1 ura3Δ0) | Research Genetics |
| Control | BY4741with plasmid pRS413, pRS415, pRS416 | 本研究构建 |
| G6 | BY4741with plasmid pRS426 | 本研究构建 |
| Sc-LQH35 | BY4741 with plasmid LQH35 | 本研究构建 |
| Sc-LQH36 | BY4741 with plasmid LQH36 | 本研究构建 |
| Sc-LQH37 | BY4741 with plasmid LQH37 | 本研究构建 |
| Sc-LQH38 | BY4741 with plasmid LQH38 | 本研究构建 |
| Sc-LQH39 | BY4741 with plasmid LQH39 | 本研究构建 |
| Sc-LQH40 | BY4741 with plasmid LQH40 | 本研究构建 |
| Sc-LQH41 | BY4741with plasmid LQH41 | 本研究构建 |
| Sc-LQH42 | BY4741 with plasmid LQH42 | 本研究构建 |
| Sc-LQH43 | BY4741 with plasmid LQH43 | 本研究构建 |
| Sc-LQH44 | BY4741 with plasmid LQH44 | 本研究构建 |
| Sc-LQH45 | BY4741 with plasmid LQH45 | 本研究构建 |
| Sc-LQH46 | BY4741 with plasmid LQH46 | 本研究构建 |
| Sc-LQH47 | BY4741 with plasmid LQH47 | 本研究构建 |
| Sc-LQH48 | BY4741 with plasmid LQH48 | 本研究构建 |
| Sc-LQH49 | BY4741 with plasmid LQH49 | 本研究构建 |
| Sc-LQH50 | BY4741 with plasmid LQH50 | 本研究构建 |
| 质粒 | ||
| pRS413 | HIS3, a centromeric plasmid | ATCC87518 |
| pRS415 | LEU2, a centromeric plasmid | ATCC87520 |
| pRS416 | URA3, a centromeric plasmid | ATCC87521 |
| pRS426 | URA3, a multiple copy plasmid | ATCC77107 |
| LQH1 | PMA1p-XR-ACS2t-pRS416 | 本研究构建 |
| LQH2 | ACT1p-XR-ACS2t-pRS416 | 本研究构建 |
| LQH3 | TDH3p-XR-ACS2t-pRS416 | 本研究构建 |
| LQH4 | POT1p-XR-ACS2t-pRS416 | 本研究构建 |
| LQH5 | APL5p-XR-ACS2t-pRS416 | 本研究构建 |
| LQH6 | FBA1p-XDH-ENO2t-pRS413 | 本研究构建 |
| LQH7 | PGK1p-XDH-ENO2t-pRS413 | 本研究构建 |
| LQH8 | RPS2p-XDH-ENO2t-pRS413 | 本研究构建 |
| LQH9 | AAC1p-XDH-ENO2t-pRS413 | 本研究构建 |
| LQH10 | CBS1p-XDH-ENO2t-pRS413 | 本研究构建 |
| LQH11 | PDC1p-XK-ASC1t-pRS415 | 本研究构建 |
| LQH12 | ZEO1p-XK-ASC1t-pRS415 | 本研究构建 |
| LQH13 | RVB2p-XK-ASC1t-pRS415 | 本研究构建 |
| LQH14 | ARC18p-XK-ASC1t-pRS415 | 本研究构建 |
| LQH35 | pRS426-TDH3p-XR-ACS2t-FBA1p-XDH-ENO2t-PDC1p-XK-ASC1t | 本研究构建 |
| LQH36 | pRS426-PMA1p-XR-ACS2t-FBA1p-XDH-ENO2t-PDC1p-XK-ASC1t | 本研究构建 |
| LQH37 | pRS426-ACT1p-XR-ACS2t-FBA1p-XDH-ENO2t-PDC1p-XK-ASC1t | 本研究构建 |
| LQH38 | pRS426-POT1p-XR-ACS2t-FBA1p-XDH-ENO2t-PDC1p-XK-ASC1t | 本研究构建 |
| LQH39 | pRS426-TDH3p-XR-ACS2t-FBA1p-XDH-ENO2t-ZEO1p-XK-ASC1t | 本研究构建 |
| LQH40 | pRS426-PMA1p-XR-ACS2t-FBA1p-XDH-ENO2t-ZEO1p-XK-ASC1t | 本研究构建 |
| LQH41 | pRS426-ACT1p-XR-ACS2t-FBA1p-XDH-ENO2t-ZEO1p-XK-ASC1t | 本研究构建 |
| LQH42 | pRS426-POT1p-XR-ACS2t-FBA1p-XDH-ENO2t-ZEO1p-XK-ASC1t | 本研究构建 |
| LQH43 | pRS426-TDH3p-XR-ACS2t-PGK1p-XDH-ENO2t-PDC1p-XK-ASC1t | 本研究构建 |
| LQH44 | pRS426-PMA1p-XR-ACS2t-PGK1p-XDH-ENO2t-PDC1p-XK-ASC1t | 本研究构建 |
| LQH45 | pRS426-ACT1p-XR-ACS2t-PGK1p-XDH-ENO2t-PDC1p-XK-ASC1t | 本研究构建 |
| LQH46 | pRS426-POT1p-XR-ACS2t-PGK1p-XDH-ENO2t-PDC1p-XK-ASC1t | 本研究构建 |
| LQH47 | pRS426-TDH3p-XR-ACS2t-PGK1p-XDH-ENO2t-ZEO1p-XK-ASC1t | 本研究构建 |
| LQH48 | pRS426-PMA1p-XR-ACS2t-PGK1p-XDH-ENO2t-ZEO1p-XK-ASC1t | 本研究构建 |
| LQH49 | pRS426-ACT1p-XR-ACS2t-PGKp-XDH-ENO2t-ZEO1p -XK-ASC1t | 本研究构建 |
| LQH50 | pRS426-POT1p-XR-ACS2t-PGKp-XDH-ENO2t-ZEO1p -XK-ASC1t | 本研究构建 |
1.2 培养基
本研究所用培养基配方如下:LB培养基:胰蛋白胨10g/L, 酵母提取物5g/L,NaCl 10g/L。添加工作浓度为100 mg/L的氨苄青霉素或者50 mg/L的卡那霉素。
YPD培养基:蛋白胨20 g/L,酵母浸粉10 g/L,葡萄糖20 g/L,固体YPD培养基加入琼脂粉20 g/L。
YPX培养基:蛋白胨20 g/L,酵母粉10 g/L,木糖20 g/L。
SC-Ura-His-Leu培养基和SC-Ura培养基:去氨基酸酵母氮源(YNB)6.7 g/L,葡萄糖20 g/L,氨基酸省却混合粉末2 g/L (缺少尿嘧啶、亮氨酸、组氨酸及色氨酸),pH调到6.0,加入相应氨基酸母液补齐缺陷型。固体培养基需调节pH到6.5,并加入琼脂粉20 g/L。
SX培养基:去氨基酸酵母氮源(YNB)6.7 g/L,氨基酸省却混合粉末2 g/L (缺少尿嘧啶、亮氨酸、组氨酸及色氨酸),木糖20 g/L,调节pH到6.0,加入相应氨基酸母液补齐缺陷型。固体培养基需调节pH到6.5,并加入琼脂粉20 g/L。
1.3 酵母转化用传统醋酸锂转化法进行酵母转化[15]。阳性转化子用SC-Ura-His-Leu或者含SC-Ura固体琼脂平板进行筛选。
1.4 分子操作和菌株构建 1.4.1 木糖代谢路径上各基因单一表达盒的构建根据2002年Wei等[16]的研究成果选取表达强度不同的14个启动子。利用yGG方法将所选14个启动子和酵母GFP分别连接,利用流式细胞仪测量14个启动子对应菌株的荧光强度,并按照强弱顺序交叉组合分成A、B、C三组,如图 2所示。
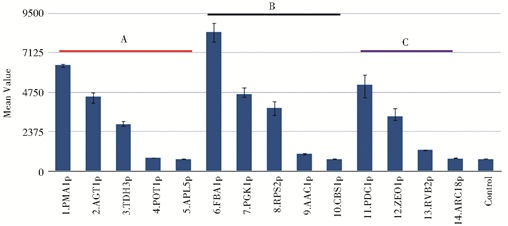
|
| 图 2 利用流式细胞仪测量不同启动子控制下的GFP荧光强度 Figure 2 GFP Fluorescence of 14 promoters using Flow cytometer |
设计单一表达盒时,为使调控木糖代谢路径上三个基因的启动子强度均匀分布,选取图 2中A组启动子调控XR基因,终止子为ACS2t,构建在载体pRS416上(筛选标记URA3);B组启动子调控XDH基因,终止子为ENO2t,构建在载体pRS413上(筛选标记HIS3);C组启动子调控XK基因,终止子为ASC1t,构建在载体pRS415上(筛选标记LEU2)。利用如图 3所示的yGG一步连接法将基因与对应的启动子终止子连接起来,构建出如表 2所示的14个单基因表达盒。
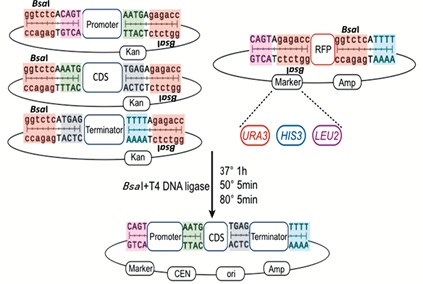
|
| 图 3 yGG一步连接法接口设计和连接过程示意 Figure 3 Design of yeast golden gate assembly |
| TU No. | Transcription Unit | Length (bp) | Marker | TU No. | Transcription Unit | Length (bp) | Marker |
| 1 | PMA1p-XR-ACS2t | 1684 | URA3 | 8 | RPS2p-XDH-ENO2t | 1816 | HIS3 |
| 2 | ACT1p-XR-ACS2t | 1684 | URA3 | 9 | AAC1p-XDH-ENO2t | 1816 | HIS3 |
| 3 | TDH3p-XR-ACS2t | 1684 | URA3 | 10 | CBS1p-XDH-ENO2t | 1723 | HIS3 |
| 4 | POT1p-XR-ACS2t | 1565 | URA3 | 11 | PDC1p-XK-ASC1t | 2773 | LEU2 |
| 5 | APL5p-XR-ACS2t | 1543 | URA3 | 12 | ZEO1p-XK-ASC1t | 2438 | LEU2 |
| 6 | FBA1p-XDH-ENO2t | 1945 | HIS3 | 13 | RVB2p-XK-ASC1t | 2530 | LEU2 |
| 7 | PGK1p-XDH-ENO2t | 1816 | HIS3 | 14 | ARC18p-XK-ASC1t | 2490 | LEU2 |
1.4.2 三质粒共转化筛选利用木糖启动子组合菌株
利用表 2中的含有不同筛选标记的基因表达盒,进行木糖代谢路径3个基因启动子强度组合设计,得到组合数5×5×4(5XR×5XDH×4XK)=100个。利用酵母醋酸锂转化法将上述100个组合中所对应的单基因表达盒进行共转化,共得到100个对应的含有3个质粒的酵母菌株。组合信息和对应菌株命名如表 3所示。
| Clone | TU No. | Clone | TU No. | Clone | TU No. | Clone | TU No. |
| 1 | 1, 6, 11 | 26 | 1, 6, 12 | 51 | 1, 6, 13 | 76 | 1, 6, 14 |
| 2 | 1, 7, 11 | 27 | 1, 7, 12 | 52 | 1, 7, 13 | 77 | 1, 7, 14 |
| 3 | 1, 8, 11 | 28 | 1, 8, 12 | 53 | 1, 8, 13 | 78 | 1, 8, 14 |
| 4 | 1, 9, 11 | 29 | 1, 9, 12 | 54 | 1, 9, 13 | 79 | 1, 9, 14 |
| 5 | 1, 10, 11 | 30 | 1, 10, 12 | 55 | 1, 10, 13 | 80 | 1, 10, 14 |
| 6 | 2, 6, 11 | 31 | 2, 6, 12 | 56 | 2, 6, 13 | 81 | 2, 6, 14 |
| 7 | 2, 7, 11 | 32 | 2, 7, 12 | 57 | 2, 7, 13 | 82 | 2, 7, 14 |
| 8 | 2, 8, 11 | 33 | 2, 8, 12 | 58 | 2, 8, 13 | 83 | 2, 8, 14 |
| 9 | 2, 9, 11 | 34 | 2, 9, 12 | 59 | 2, 9, 13 | 84 | 2, 9, 14 |
| 10 | 2, 10, 11 | 35 | 2, 10, 12 | 60 | 2, 10, 13 | 85 | 2, 10, 14 |
| 11 | 3, 6, 11 | 36 | 3, 6, 12 | 61 | 3, 6, 13 | 86 | 3, 6, 14 |
| 12 | 3, 7, 11 | 37 | 3, 7, 12 | 62 | 3, 7, 13 | 87 | 3, 7, 14 |
| 13 | 3, 8, 11 | 38 | 3, 8, 12 | 63 | 3, 8, 13 | 88 | 3, 8, 14 |
| 14 | 3, 9, 11 | 39 | 3, 9, 12 | 64 | 3, 9, 13 | 89 | 3, 9, 14 |
| 15 | 3, 10, 11 | 40 | 3, 10, 12 | 65 | 3, 10, 13 | 90 | 3, 10, 14 |
| 16 | 4, 6, 11 | 41 | 4, 6, 12 | 66 | 4, 6, 13 | 91 | 4, 6, 14 |
| 17 | 4, 7, 11 | 42 | 4, 7, 12 | 67 | 4, 7, 13 | 92 | 4, 7, 14 |
| 18 | 4, 8, 11 | 43 | 4, 8, 12 | 68 | 4, 8, 13 | 93 | 4, 8, 14 |
| 19 | 4, 9, 11 | 44 | 4, 9, 12 | 69 | 4, 9, 13 | 94 | 4, 9, 14 |
| 20 | 4, 10, 11 | 45 | 4, 10, 12 | 70 | 4, 10, 13 | 95 | 4, 10, 14 |
| 21 | 5, 6, 11 | 46 | 5, 6, 12 | 71 | 5, 6, 13 | 96 | 5, 6, 14 |
| 22 | 5, 7, 11 | 47 | 5, 7, 12 | 72 | 5, 7, 13 | 97 | 5, 7, 14 |
| 23 | 5, 8, 11 | 48 | 5, 8, 12 | 73 | 5, 8, 13 | 98 | 5, 8, 14 |
| 24 | 5, 9, 11 | 49 | 5, 9, 12 | 74 | 5, 9, 13 | 99 | 5, 9, 14 |
| 25 | 5, 10, 11 | 50 | 2, 10, 12 | 75 | 2, 10, 13 | 100 | 2, 10, 14 |
1.4.3 利用RADOM组装法将木糖代谢路径快速组装到单一表达载体
利用三质粒体系,在木糖平板上从100个组合中初筛得到了16个可以利用木糖的三基因启动子组合。为了使菌株的性能更加稳定,在进行发酵验证前将上述16个组合中XR、XDH、XK表达模块利用RADOM法[17]组装到含有URA3筛选标记的pRS426载体上,组装过程如图 4所示。

|
| 图 4 利用RADOM方法组装木糖代谢通路 Figure 4 Construction of xylose utilization pathway using RADOM method |
引物设计如图 4a所示,XR-F设计在启动子上游200 bp处,利用PCR得到与pRS426同源的序列。XR-R设计在ACS2t上,在引物的5′端加入40 bp的序列“Ha”(图 4a中XR-R斜线所示)作为XR和XDH表达盒的同源序列。XDH-F同样在5′端引入40 bp的序列“Ha” (图 4a中XDH-F斜线所示),XDH-R设计在ENO2t上,通过PCR在5′端引入40 bp的“Hb” (图 4a中XDH-R斜线所示)序列作为XDH和XK表达盒的同源序列。XK-F结合区在启动子序列之前的载体上,在5′端引入“Hb”序列(图 4a中XK-F斜线所示),XK-R在ASC1t下游约200 bp,通过PCR引入和pRS426同源的200 bp序列。其中,Ha和Hb序列是酵母染色体完全不同源的40 bp序列Ha:“CATCCTTCAGGTTTGCACGATCAAGTTCGTATGACAAGCC”, Hb:“ACCTCGACAAGATATGCCTTATTTACCAGATGGAACACCA”。具体引物序列见表 4。
| Primers | Sequence (5′-3′) |
| XR-F | TATTACGCCAGCTGGCGAAAG |
| XR-R | CATCCTTCAGGTTTGCACGATCAAGTTCGTATGACAAGCCTATATCTATACATATATATCACTAAGTGCA |
| XDH-F | GGCTTGTCATACGAACTTGATCGTGCAAACCTGAAGGATGGAATTCCTGCAGCCCCCAGT |
| XDH-R | TGGTGTTCCATCTGGTAAATAAGGCATATCTTGTCGAGGTAAAGCCACGCGTGTGCACCT |
| XK-F | ACCTCGACAAGATATGCCTTATTTACCAGATGGAACACCAGAATTCCTGCAGCCCCCAGT |
| XK-R | TCACTCATTAGGCACCCCAGG |
| Note:Homology arms were underlined | |
利用表 4中3对引物扩增16个组合中对应的4个XR表达盒、2个XDH表达盒和2个XK表达盒,胶回收目的片段,然后利用RADOM组装法对表达盒进行组装,组装在pRS426上的代谢路径如图 4b所示。利用表 4中三对通用引物对组装后质粒进行PCR验证,琼脂糖凝胶电泳结果见图 5。PCR验证结束后,再对组装的质粒进行测序,DNA测序由苏州金唯智生物科技有限公司完成。

|
| 图 5 PCR验证组装后质粒 Figure 5 PCR verification of assembled pathway |
将完成组装并测序正确的16个质粒分别转化到底盘菌株BY4741中,得到含有完整木糖代谢路径的单一表达载体的16株菌Sc-LQH35至Sc-LQH50。
1.5 菌株发酵培养摇瓶发酵用种子培养基为SC-Ura培养基,用接种环从平板上挑酵母单菌落,接种到5 ml SC-Ura液体培养基中,在30 ℃,200 r/min的条件下培养约24h作为一级种子。以初始OD600=0.2将细胞接种到含有50 ml SC-Ura液体培养基的摇瓶中,30 ℃,200 r/min有氧培养至指数后期,用作二级种子。取一定体积的二级种子培养物,4 000 r/min离心收集,用无菌水清洗细胞两次后,将细胞接种到含100 ml YPX的250 ml三角瓶中,发酵初始OD600=0.1。
限氧发酵在细口三角瓶中进行,用带有针头的胶塞塞住瓶口以释放发酵过程中产生的CO2,培养条件为30 ℃,150 r/min;有氧发酵在广口三角瓶中进行,瓶口用纱布密封,培养条件为30 ℃,200 r/min。
1.6 发酵液中残糖和产物的检测在设计的各取样点,取出1 ml发酵液,检测培养液的吸光度(OD600),同时10 000 r/min离心10 min收集上清液,经0.22 μm滤膜处理后用于残糖、发酵产物的检测。检测用的HPLC系统包括Waters1515 HPLC泵,美国伯乐HPX-87H色谱柱和Waters 2414示差检测器。操作过程中柱温控制在65℃,所用流动相为5mmol/L H2SO4,流速为0.6ml/min。
2 结果 2.1 平板初筛三质粒共转化构建的木糖利用酵母菌株利用深孔96孔细胞培养板将通过质粒共转化得到的100株菌以及只含有pRS413、pRS415和pRS416空载体的对照菌株Control培养在1 ml的SC-Ura-His-Leu培养基中,30℃培养至饱和。将培养液用无菌水洗一遍后,在合成型2%固体木糖培养基(SX)平板上进行梯度稀释,取5 μl原始浓度菌液和10-1、10-3、10-5稀释菌液点到SX培养基上,培养7天,结果如图 6所示。

|
| 图 6 2%合成型木糖培养基梯度稀释 Figure 6 Spot assay on 2% SX plate |
从图 6可以看出含有不同启动子组合的菌株在SX平板上生长有很大差异。有16个组合的菌株在SX平板上明显生长超过其他菌株,分别是表 3中的1,2,6,7,11,12,16,17,26,27,31,32,36,37,41,42号菌株。对比这16个菌株木糖代谢路径中各基因所用启动子,发现XDH基因的表达盒均为TU6或TU7,对应启动子为FBA1p和PGK1p,均为强启动子;XK基因的表达盒均为TU11或TU12,对应启动子为PDC1p和ZEO1p,为强启动子和中强启动子;而XR基因的表达盒则是除了含有最弱启动子APL5p的TU5之外的其余4个。
2.2 摇瓶限氧发酵复筛含有整合至同一载体上不同模块组合的菌株将Sc-LQH35至Sc-LQH50这16株菌及对照菌株G6在100 ml 2%木糖培养基YPX (木糖为唯一碳源)中进行摇瓶限氧发酵培养,发酵初始OD600为0.5。培养至96h,取样进行吸光度和残糖、发酵产物的检测,结果如图 7所示。
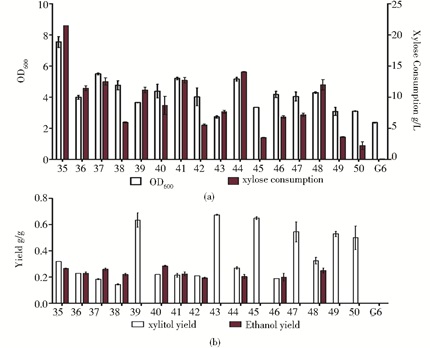
|
| 图 7 限氧条件下摇瓶复筛结果 Figure 7 Flask selection under oxgen-limited condition |
不同启动子控制下各基因表达模块间组合的不同会导致菌株表型的多样性,体现在木糖代谢速率和产物得率两方面。从图 7a可以看出,构建的16个菌株木糖代谢水平各不相同,但与对照菌株G6相比,菌株的生长都得到了提高。菌株的生长和木糖消耗的趋势基本一致。16个菌株中,在限氧条件下96h内只有菌株Sc-LQH35消耗了全部的木糖(21.5 g/L),木糖醇得率为0.32 g/g,乙醇得率为0.26 g/g。消耗木糖最少的菌株为Sc-LQH50,消耗木糖3.10 g/L,仅为总木糖含量的14%。
由图 7b可见16个菌株的产物生成也有很大的差异,其中乙醇得率范围为0~0.28 g/g,乙醇得率最高的是菌株Sc-LQH40,为0.28 g/g;木糖醇得率范围为0.15~0.67 g/g,最高的为Sc-LQH43,是得率最低的Sc-LQH38菌株的4.5倍。此外,还可以明显看出,不产乙醇的6株菌(Sc-LQH39、Sc-LQH43、Sc-LQH45、Sc-LQH47、Sc-LQH49、Sc-LQH50)木糖醇得率普遍较高,都超过了0.5 g/g。但在这6株菌中,除了Sc-LQH39,其他5株菌的木糖消耗均比较少,低于10 g/L。
2.3 不同条件重组菌株木糖发酵性能验证依据2.2中限氧条件下96h的发酵数据,选取在木糖利用、产物得率方面有着不同表型的重组酵母菌株Sc-LQH35、Sc-LQH39、Sc-LQH40、Sc-LQH48进行发酵研究,进一步验证菌株在木糖利用及产物生成方面的性能。在有氧条件下和限氧条件下发酵结果分别见图 8和图 9,并将产物得率总结于表 5。
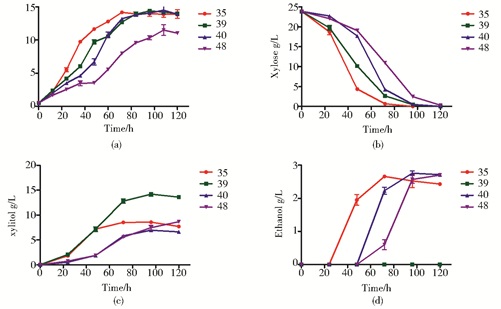
|
| 图 8 重组酵母在YPX培养基中有氧发酵结果 Figure 8 Comparison of (a) cell growth, (b) xylose consumption, (c) xylitol production and (d) ethanol production of the recombinant Saccharomyces cerevisiae in YPX medium under aerobic condition |
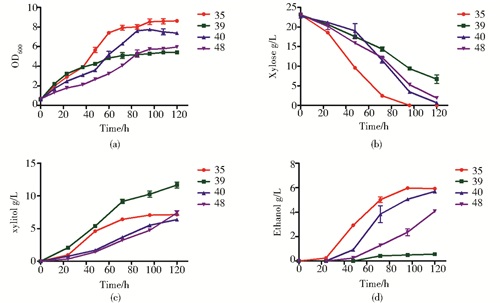
|
| 图 9 重组酵母在YPX培养基中限氧发酵结果(a)菌株生长、(b)木糖代谢、(c)木糖醇积累和(d)乙醇积累 Figure 9 Comparison of (a) cell growth, (b) xylose consumption, (c) xylitol production and (d) ethanol production of the recombinant Saccharomyces cerevisiae in YPX medium under oxygen-limited condition |
| 35 | 39 | 40 | 48 | |||||
| 有氧 | 限氧 | 有氧 | 限氧 | 有氧 | 限氧 | 有氧 | 限氧 | |
| 木糖醇得率(g/g) | 0.36±0.01 | 0.31±0.01 | 0.61±0.01 | 0.71±0.08 | 0.30±0.00 | 0.28±0.01 | 0.35±0.02 | 0.35±0.01 |
| 乙醇得率(g/g) | 0.11±0.01 | 0.26±0.01 | 0 | 0.03±0.00 | 0.12±0.00 | 0.25±0.01 | 0.12±0.01 | 0.19±0.00 |
根据图 8和图 9呈现的结果,可以看出在有氧和限氧条件下发酵,菌体生长速率与其对应的木糖代谢速率都表现出极大的正相关性。有氧条件下4株菌的木糖代谢速率均比限氧条件下快,而且菌体最终OD600值都几乎高出1倍左右。其中菌株Sc-LQH35在两种发酵条件下的生长和木糖代谢速率较其他3株菌均有明显的优势;在产物生成方面,从表 5中可发现,两种发酵条件下,菌株Sc-LQH35、Sc-LQH40和Sc-LQH48木糖醇得率相似,而Sc-LQH39在限氧条件下木糖醇得率有明显提高,达到0.71g/g。在限氧发酵条件下,4株菌的乙醇得率普遍有所提高,其中Sc-LQH35和Sc-LQH40提高幅度最为明显。乙醇得率的提高,一方面是由于限氧条件下酵母进行了无氧呼吸,生成了更多的乙醇;另一方面是在限氧条件产生的乙醇挥发量也明显减少的缘故。
3 讨论通过三质粒共转化得到的100种组合在木糖平板初筛时表型差异很大,这可能是由于不同模块组合和底盘菌株的适配情况不同而造成的。但是能够较好生长的16株菌具有某些共性,即控制它们路径中XDH基因对应的启动子是FBA1p和PGK1p,均为强启动子,而控制XK基因对应的启动子是PDC1p和ZEO1p,也是强启动子。因此推测XDH和XK的表达强度对木糖利用有重要的影响,这两个基因的表达需达到一定强度后菌株才可以利用木糖,关于这两个基因表达量对木糖利用的影响在前人的研究中也有类似发现[18]。但是对于控制XR基因的启动子并没有特殊要求,这可能是由于酵母有內源开放阅读框YHR104w (GRE3),其编码产物为醛糖还原酶,所以本身就具有一定的把木糖还原成木糖醇的能力[19];另外,XR反应底物直接为木糖,底物来源充足,再者其辅因子为NADPH,胞内含量和来源相对充足,也有利于木糖转化成木糖醇的反应顺利进行[20]。
对初筛出来的16种组合统一组装到单一质粒后再次转化底盘菌株,进一步摇瓶发酵复筛发现它们在菌体积累,木糖消耗以及产物生成方面有很大的差异。其中Sc-LQH39、Sc-LQH43、Sc-LQH45、Sc-LQH47、Sc-LQH49、Sc-LQH50这6株菌几乎不产乙醇,而产物全部为木糖醇,通过比较发现中这6株菌种除Sc-LQH39控制XDH基因的启动子为FBA1p外,其余5株均为相对较弱的PGK1p,而且这5株菌的木糖利用量也普遍偏低,这与Zha等[20-21]的研究结果相符。据此推测造成这一现象的原因是由于木糖醇脱氢酶(XDH)的表达量不够,从而导致木糖醇的积累,进而抑制木糖的利用,导致木糖利用量普遍较低,XDH的表达对具有XR-XDH木糖代谢路径的重组菌株的木糖利用能力影响较大。因此在一定程度上提高XR-XDH路径中XDH的表达量可能会带来菌株木糖利用能力的提升,并优化产物生成,减少副产物木糖醇的积累。
本文利用三质粒共转化组合筛选方法快速构建了含有100种木糖代谢路径上不同启动子控制下的基因组合的酿酒酵母菌株,实现了对不同组合间不同表达水平的木糖代谢路径的分步筛选。单基因表达盒构建完成后无需进行组装就能够通过共转化的方式将完整代谢路径引入酿酒酵母体内,经过初筛,进行组合间的性能评价,淘汰掉一些劣势组合,再对优势组合统一组装后做进一步的分析,并最终成功筛选出木糖利用性能优良的菌株Sc-LQH35(TDH3p-XR-ACS2t-FBA1p-XDH-ENO2t-PDC1p-XK-ASC1t)和Sc-LQH39(TDH3p-XR-ACS2t-FBA1p-XDH-ENO2t-ZEO1p-XK-ASC1t),而经过后续菌株保存与传代,以上两株重组酵母菌株木糖利用能力均未发生退化,进一步证实了该方法的可靠性。质粒共转化分步组合筛选方法,避免了部分由于筛菌过程中的盲目性而带来的工作量,节约了时间成本和资金投入。这种方法对酿酒酵母中其他外源代谢路径的构建也同样适用,对科研和生产过程中的菌株筛选工作有一定的指导作用。
| [1] | Jeppsson M, Björn J, Jensen P R, et al. The level of glucose-6-phosphate dehydrogenase activity strongly influences xylose fermentation and inhibitor sensitivity in recombinant Saccharomyces cerevisiae strains. Yeast , 2003, 20 (15) : 1263–1272. DOI:10.1002/yea.v20:15 |
| [2] | Jin H, Fan S, Wang C, et al. Fine-tuning of NADH oxidase decreases byproduct accumulation in respiration deficient xylose metabolic Saccharomyces cerevisiae. Bmc Biotechnology , 2014, 14 (1) : 1–10. DOI:10.1186/1472-6750-14-1 |
| [3] | Aguilera J, Prieto J. The Saccharomyces cerevisiae aldose reductase is implied in the metabolism of methylglyoxal in response to stress conditions. Current Genetics , 2001, 39 (39) : 273–283. |
| [4] | Bamba T, Hasunuma T, Kondo A. Disruption of PHO13 improves ethanol production via the xylose isomerase pathway. Amb Express , 2016, 6 (1) : 1–10. DOI:10.1186/s13568-015-0169-5 |
| [5] | Oh Y J, Lee T H, Lee S H, et al. Dual modulation of glucose 6-phosphate metabolism to increase NADPH-dependent xylitol production in recombinant Saccharomyces cerevisiae. Journal of Molecular Catalysis B Enzymatic , 2007, 47 (1) : 37–42. |
| [6] | Omotajo D, Tate T, Cho H, et al. Distribution and diversity of ribosome binding sites in prokaryotic genomes. Bmc Genomics , 2014, 16 (604) : 1–8. |
| [7] | Xin Q, Jian Z, Liu G G, et al. Heterologous xylose isomerase pathway and evolutionary engineering improve xylose utilization in Saccharomyces cerevisiae. Frontiers in Microbiology , 2015, 6 : 1165. |
| [8] | Kim J H, Block D E, Mills D A. Simultaneous consumption of pentose and hexose sugars:an optimal microbial phenotype for efficient fermentation of lignocellulosic biomass. Applied Microbiology & Biotechnology , 2010, 88 (5) : 1077–1085. |
| [9] | Chu B C, Lee H. Genetic improvement of Saccharomyces cerevisiae for xylose fermentation. Biotechnology Advances , 2007, 25 (5) : 425–441. DOI:10.1016/j.biotechadv.2007.04.001 |
| [10] | Jin Y S, Ni H J, Jeffries T W. Optimal growth and ethanol production from xylose by recombinant Saccharomyces cerevisiae require moderate D-xylulokinase activity. Applied & Environmental Microbiology , 2003, 69 (1) : 495–503. |
| [11] | Hahn-Hägerdal B, Karhumaa K, Fonseca C, et al. Towards industrial pentose-fermenting yeast strains. Applied Microbiology & Biotechnology , 2007, 74 (5) : 937–953. |
| [12] | Du J, Yuan Y, Zhao H. Customized optimization of metabolic pathways by combinatorial transcriptional engineering. Methods in Molecular Biology , 2013, 985 (18) : 177–209. |
| [13] | Kim B, Du J, Eriksen D T, et al. Combinatorial design of a highly efficient xylose-utilizing pathway in Saccharomyces cerevisiae for the production of cellulosic biofuels. Applied & Environmental Microbiology , 2013, 79 (3) : 931–941. |
| [14] | Agmon N, Mitchell L A, Cai Y, et al. Yeast Golden Gate (yGG) for the efficient assembly of S. cerevisiae transcription units. Acs Synthetic Biology , 2015, 4 (7) : 853–859. DOI:10.1021/sb500372z |
| [15] | Wang X, Bai X, Chen D F, et al. Increasing proline and myo-inositol improves tolerance of Saccharomyces cerevisiae to the mixture of multiple lignocellulose? derived inhibitors. Biotechnology for Biofuels , 2015, 8 : 142–154. DOI:10.1186/s13068-015-0329-5 |
| [16] | Wei W J, Michael C, David B, et al. A systematic approach to reconstructing transcription networks in Saccharomy cescerevisiae. Proceedings of the National Academy of Sciences , 2002, 99 (26) : 16893–16898. DOI:10.1073/pnas.252638199 |
| [17] | Lin Q, Jia B, Mitchell L A, et al. RADOM, an efficient in vivo method for assembling designed DNA fragments up to 10 kb long in Saccharomyces cerevisiae. Acs Synthetic Biology , 2015, 4 (3) : 213–220. DOI:10.1021/sb500241e |
| [18] | Kim S R, Ha S J, Kong I I, et al. High expression of XYL2 coding for xylitol dehydrogenase is necessary for efficient xylose fermentation by engineered Saccharomyces cerevisiae. Metabolic Engineering , 2012, 14 (4) : 336–343. DOI:10.1016/j.ymben.2012.04.001 |
| [19] | Toivari M H, Salusjärvi L, Ruohonen L, et al. Endogenous xylose pathway in Saccharomyces cerevisiae. Applied & Environmental Microbiology , 2004, 70 (6) : 3681–3686. |
| [20] | Zha J, Shen M, Hu M, et al. Enhanced expression of genes involved in initial xylose metabolism and the oxidative pentose phosphate pathway in the improved xylose-utilizing Saccharomyces cerevisiae through evolutionary engineering. Journal of Industrial Microbiology & Biotechnology , 2014, 41 (1) : 27–39. |
| [21] | Zha J, Hu M L, Shen M H, et al. Balance of XYL1 and XYL2 expression in different yeast chassis for improved xylose fermentation. Frontiers in Microbiology , 2012, 3 : 355. |
 2016, Vol. 36
2016, Vol. 36




