文章信息
- 李达, 代鹏, 王伟, 张文涛, 汪钦, 束毅, 祝春来, 纪奇峰, 梁平, 颜真.
- LI Da, DAI Peng, WANG Wei, ZHANG Wen-tao, WANG Qin, SHU Yi, ZHU Chun-lai, JI Qi-feng, LIANG Ping, YAN Zhen.
- PLCE1基因及rs2274223和rs3765524单体型的克隆与表达
- Cloning and Expression of PLCE1 Gene and Its Haplotypes of rs2274223 and rs3765524
- 中国生物工程杂志, 2016, 36(12): 1-7
- China Biotechnology, 2016, 36(12): 1-7
- http://dx.doi.org/DOI:10.13523/j.cb.20161201
-
文章历史
- 收稿日期: 2016-09-18
- 修回日期: 2016-10-11
磷脂酶C-ε1(phospholipase C epsilon-1,PLCE1)是磷脂酶C家族新成员,其编码基因位于染色体10q23,主要通过不同的剪接方式编码两种异构体蛋白,分别为PLCE1a和PLCE1b,它们分别含2302和1994个氨基酸[1]。近年来研究表明,PLCE1基因与皮肤肿瘤[2]、胃癌和食道癌[3-5]的发生和发展密切相关。本课题组前期采用病例-对照研究的方式,应用MassARRAY质谱芯片技术发现,PLCE1基因的单核苷酸多态性(single nucleotide polymorphism,SNP),尤其rs2274223和rs3765524两个SNP位点,与中国西北地区汉族人群的胃癌发病密切相关。虽然2010年2个国际团队通过全基因组关联分析(genome-wide association study,GWAS)报道了PLCE1基因多态性与胃、食道癌的关系[6-7],然而SNP与胃癌发生发展的分子机制尚待阐明。研究SNP与肿瘤遗传易感性的关系,对于揭示肿瘤的发生发展机制具有十分重要的作用。本课题拟构建PLCE1 rs2274223和rs3765524单体型真核表达重组载体,为研究PLCE1及其rs2274223和rs3765524多态性参与胃癌发生发展过程的相关机制奠定基础。
1 材料与方法 1.1 材料E.coli DH5α菌种、HEK-293T细胞由本实验室保存;真核表达载体为重组pcDNA3.0-Flag,由山东大学癌症中心馈赠;点突变试剂盒(Mut express fast mutagenesis kit V2)、反转录试剂盒[Hiscript qRT supermix for qPCR (+gDNA wiper)]、qPCR试剂盒(Chamq SYBR qPCR master mix)购自Vazyme公司;转染试剂DNAfectin购自Applied Biological Materials公司;In-Fusion HD Cloning试剂盒购自Clontech公司;PrimeSTAR GXL DNA聚合酶、DNA Marker购自TaKaRa公司;去内毒素质粒小提试剂盒、DNA纯化回收试剂盒、蛋白marker购自生工生物工程上海(股份)有限公司;SDS-page试剂盒、BSA封闭液、5×蛋白上样缓冲液购自上海康城生物技术有限公司;RNA提取试剂盒购自陕西先锋生物科技有限公司;硝酸纤维素(NC)膜、PLCE1抗体购自Millipore公司;Flag抗体、β-Actin抗体、山羊抗兔和山羊抗鼠二抗购自Abbkine公司;化学发光显色试剂盒购自ZETA公司;RPMI1640培养基购自Gibco公司;胎牛血清购自Biological Industries公司;青链霉素混合液(100×)购自Solarbio公司;6孔细胞培养板购自Corning公司。
1.2 方法 1.2.1 细胞培养用含10%胎牛血清且含1×青链霉素混合液的RPMI1640培养基将HEK-293T细胞培养于37℃和5%CO2的细胞培养箱。
1.2.2 PCR引物用Primer Premier 5.0软件设计PLCE1的分段扩增引物、实时定量检测引物,以及β-actin引物;CE Design V1.03软件设计点突变引物。委托生工生物工程上海(股份)有限公司合成引物。引物序列如表 1所示。
| 引物名称 | 引物序列(5′-3′) | 备注 |
| In-fusion-F | ATCACACTGGCGGCCGCGATGACTTCTGAAGAAATGAC | 融合克隆,构建表达载体 |
| In-fusion-R | AATAGGGCCCTCTAGATCACTGTCGGTAATCCATTGTGTC | |
| F-F | CACTGGCGGCCGCATGACTTCTGAAGAAATGAC | 克隆PLCE1基因第一段 |
| F-R | GCAGGAACTGGTGCACATCGGCTTGGATGGACAG | |
| S-F | CCGATGTGCACCAGTTCCTGC | 克隆PLCE1基因第二段 |
| S-R | CCTTCAAATGACATTAGTCCCTGATGAC | |
| T-F | GTCATCAGGGACTAATGTCATTTGAAGG | 克隆PLCE1基因第三段 |
| T-R | CAGAGGCATGCCCAGGACGTC | |
| F′-F | CCTGGGCATGCCTCTGGACAGC | 克隆PLCE1基因第四段 |
| F′-R | GTGCTCTAGATCACTGTCGGTAATCCATTGTGTC | |
| 5330-F | GAAACTGAcCCAGCACACCGCCTGTCA | rs3765524位点定点突变 |
| 5330-R | TGTGCTGGgTCAGTTTCTGAGAATACCTGCGAC | |
| 5780-F | TCTGTTCCaCGTTCACTTCGAAGATCTTGTATTTC | rs2274223位点定点突变 |
| 5780-R | AGTGAACGtGGAACAGAAACTGCTCGTTCCA | |
| F R |
CCAATCCAAGCGACTATGTGC TCCACTTGCTTTGGGCTTGA |
PLCE1实时定量引物 |
| Actin-F Actin-R |
CACGAAACTACATTCAATTCCATC CAGAGCAGTGATCTCCTTCT |
β-actin实时定量引物 |
1.2.3 RNA提取及反转录
用RNA提取试剂盒提取HEK-293T细胞的总RNA,并测浓度,-80℃保存备用;取500ng总RNA,按去gDNA反转录试剂盒说明书配制反应体系,合成cDNA。
1.2.4 PCR扩增按PrimeSTAR GXL DNA聚合酶说明书,配置PCR反应体系,进行如下反应程序:95℃ 5 min;95℃ 1 min,退火1 min, 72℃ 2~5 min (根据扩增片段长度设置),共30个循环;72℃ 10 min;4℃保存。琼脂糖凝胶电泳分析PCR扩增结果,DNA测序验证,DNA纯化回收试剂盒回收序列正确的DNA扩增片段,-20℃保存备用。
1.2.5 细胞转染取对数生长期的HEK-293T细胞,制成10万个/ml的单细胞悬液,每孔2 ml,铺于六孔板,培养过夜;用200 μl培养基稀释2μg质粒,并加入6 μl转染试剂,上下吹打混匀,室温孵育30 min,并将转染混合物逐滴加入六孔板内,培养24 h后检测PLCE1的表达。
1.2.6 实时定量PCR按实时定量PCR试剂盒说明书在冰上配制20 μl反应体系,进行如下反应程序:95℃ 3 min,95℃ 15 s,60℃ 30 s,反应40个循环。结果用2-△△CT表示。
1.2.7 Western blot (WB)吸弃旧培养基,加1 ml含蛋白酶抑制剂混合物的PBS,刮取细胞并300×g离心5 min,收取细胞沉淀,加RIPA裂解液适量,冰上裂解30 min,12 000 r/ min,离心5 min,收取上清,BCA法进行蛋白定量;取20 μg总蛋白经SDS-Page电泳分离(6%分离胶),转膜(100 V,3 h),BSA封闭液封闭1h,分别用抗PLCE1抗体(1:500)、Flag (1:3 000)标签抗体和β-actin抗体(1:3 000)杂交,4℃过夜,TBST洗膜3次后分别室温孵育相应二抗1 h,TBST洗膜3次后用化学发光试剂盒显色并检测。
1.2.8 统计学方法采用SPSS 18.0统计软件对实验数据行进行统计学分析,计量资料以均值±标准差表示,多组间均值比较采用One-way ANOVA,两两比较采用LSD-t检验,检验水准α=0.01。
2 结果 2.1 PLCE1基因扩增与真核表达载体构建因为PLCE1基因开放阅读框长达6 909 bp (参考序列NM_016341.3),为了增加克隆的成功率,我们采用了重叠延伸PCR (gene splicing by overlap extension PCR,SOE-PCR)技术分段扩增PLCE1基因全长,设计方案模式见图 1。通过PCR,我们从培养的HEK-293T细胞中分别扩增出了PLCE1的4个目的片段,并用DNA测序验证。利用SOE-PCR技术,我们分别连接第二、三段形成五段,加入第四段形成第六段,再加入第一段,从而成功获得全长目的基因PLCE1(图 2)。然后通过In-Fusion技术扩增PLCE1基因全长,Not I和Xba I酶切pcDNA3.0-Flag载体,同源重组的方式将线性化的空载体与PLCE1基因进行重组、DNA测序。Blast分析显示,我们扩增的PLCE1与参考序列PLCE1 transcript variant1(NM_016341)序列匹配程度达到99.7%,其中有2个同义点突变、3个错义点突变(分别为rs2274224,rs3765524和rs2274223),以及一段18bp的插入序列。并且证实我们所获基因为PLCE1 rs2274223G-rs3765524T单体型真核表达重组载体,因为GT基因型在亚洲人种的频率很低,我们将其命名为PLCE1 Minor,其编码的氨基酸序列如图 3中PLCE1 Minor.pro所示。

|
| 图 1 PLCE1基因分段扩增模式 Figure 1 Pattern diagram of PLCE1 gene segmented amplification |

|
| 图 2 PLCE1基因分段扩增电泳 Figure 2 Agarose gel electrophoresis of amplified segments of PLCE1 M: DNA marker; 1~7 represent the first to seventh segments of PLCE1 gene respectively |

|
| 图 3 PLCE1编码氨基酸序列及部分DNA序列比对 Figure 3 Alignments of PLCE1 encoded a mino acid and partial DNA sequences A:Alignments of partial encoded a mino acid sequences of PLCE1 mRNA transcript variant1 in NCBI database with PLCE1 Major and PLCE1 Minor. The a mino acid sequences in black are in complete agreement with the one encoded by PLCE1 mRNA transcript variant1.The gray box at 1185~1190bp represents 6 inserted a mino acid residues (RPCLCC); The differences at 1575, 1777 and 1927 represent the rs2274224R>P, rs3765524T>I and rs2274223H>R SNPs respectively. B: Alignments of the 18bp insert of PLCE1 Minor with PLCE1 DNA sequence (NG015799.1) in NCBI database and 3 PLCE1 mRNA transcript variants (NM_016341, NM_001165979, NM_001288989). It displays that the insert comes from different splicing of PLCE1 |
经过分析PLCE1基因序列的开放阅读框,我们发现所构建的PLCE1真核表达载体能够正确转录和翻译(图 3),与NCBI收录PLCE1转录本1的参考序列(NM_016341)进行比较,发现PLCE1各个结构域正确,rs3765524和rs2274223两个SNP位点所在密码子编码的氨基酸分别由野生型的苏氨酸(T)变为异亮氨酸(I)、组氨酸(H)变为精氨酸(R)。但与该参考序列不同的是,我们克隆的PLCE1的氨基酸序列第1 575位点由精氨酸(R)变为脯氨酸(P),该位点对应PLCE1基因的另一个SNP (rs2274224)。另外,我们所扩增的PLCE1比参考序列NM_016341在编码区3 554~3 572bp处有18bp插入序列编码的氨基酸RPCLCC。经查找分析,插入序列来源于PLCE1基因组DNA序列(NG_015799.1)第269 794~269 811bp处,位于PLCE1第11内含子,并与第12外显子紧邻。因此我们推断该转录本属于PLCE1的一种新的剪接异构体。
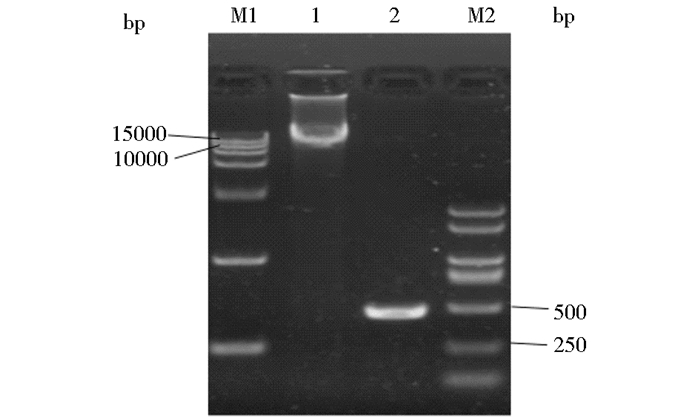
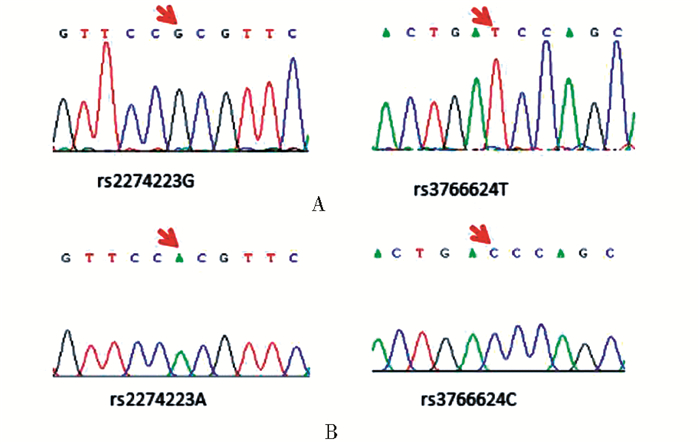
2.2 PLCE1 rs2274223A-rs3765524C (PLCE1 Major)单体型构建前期研究发现在胃癌组织样本中rs2274223和rs3765524两个SNP位点具有高度连锁的关系,因此我们拟研究PLCE1 rs2274223和rs3765524对PLCE1的表达和功能的影响,从而进一步揭示其在胃癌等肿瘤中的作用。我们在结果2.1的基础上,应用基因双点突变技术,以构建成功的PLCE1 rs2274223G-rs3765524T (PLCE1 Minor)为模板,用5330F-5780R和5780F-5330R两对引物分两段扩增PLCE1 Minor载体(图 4),关键位点的DNA序列如图 5;Dpn I内切酶去除甲基化模板,最后经同源重组成环的方法,获得了序列正确的单克隆PLCE1 rs2274223A-rs3765524C单体型重组真核表达载体,并命名为PLCE1 Major,其编码氨基酸序列如图 3中PLCE1 Major.pro所示。
|
| 图 4 PLCE1 Major重组表达载体构建的基因扩增电泳 Figure 4 Agarose gel electrophoresis of amplified segments of PLCE1 Major expression vector M1 and M2 represent DNA marker DL15000 and DL2000 respectively;1 and 2 represent the longer (11.9 kbp) and shorter (450 bp) amplified segments of PLCE1 Major respectively |
|
| 图 5 PLCE1 Minor (A)和PLCE1 Major (B)基因型鉴定序列图 Figure 5 DNA sequencing of PLCE1 Minor (A) and PLCE1 Major (B) |
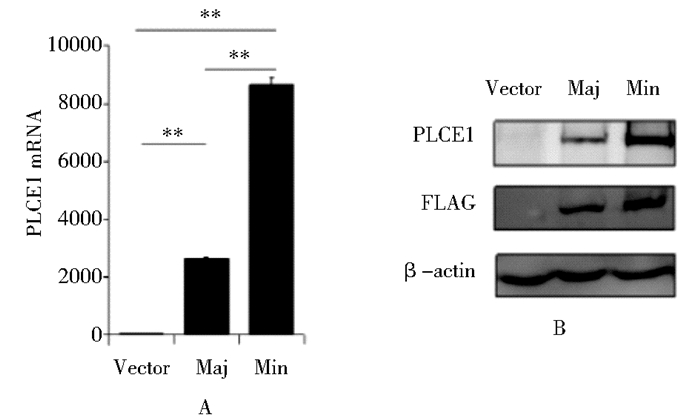
将构建的重组真核表达载体PLCE1 Minor (Min)和PLCE1 Major (Maj)分别转染HEK-293T细胞,检测PLCE1基因的表达,同时设立空载体对照组(Vector)。转染24 h后,分别提取细胞总RNA进行实时定量PCR分析mRNA转录水平,提取总蛋白进行WB分析蛋白质表达水平。如图 6所示,与Vector组比较,Min组和Maj组中PLCE1的mRNA和蛋白表达显著增加(P < 0.01)。不仅证明了我们构建的PLCE1重组表达载体都能够正确转录和翻译,还发现PLCE1基因表达水平在Maj组和Min组有显著差异(P < 0.01),Min组的表达明显高于Maj组,提示rs3765524T和rs2274223G多态性及其基因型能够影响PLCE1基因自身的表达水平。
|
| 图 6 PLCE1重组基因的转染与表达 Figure 6 Recombinant PLCE1 gene transfection and expression A:mRNA level detected by qPCR in HEK-293T cells 24h after transfection. n=3 for each group.** : P < 0.01 B:Protein expression detected by Western blotting in HEK-293T cells 24h after transfection. Vector: mock plasmlid; Maj: PLCE1 Major; Min:PLCE1 Minor |
与PLC其它家族成员相比,PLCE1具有独特的RA和CDC25结构域,能够被Ras家族成员激活,并持续水解PIP2等产生大量的第二信使,参与多种信号通路的调节[8]。已有的研究报道PLCE1基因多态性与多种肿瘤的易感性密切相关,特别是食道癌[9-10]、胃癌[11-13]等消化道癌症。有研究者通过Meta分析发现PLCE1 rs3765524和rs2274223多态性与胃癌易感性相关,且rs3765524T和rs2274223G都能独自增加胃癌易感性[14];又有人发现rs2274223G-rs3765524T-rs7922612C、rs2274223A-3765524C-7922612T和rs2274223G-3765524C-7922612T等单体型在克什米尔地区的胃癌人群中的频率较高[15],由此我们推断不同的SNPs连锁后可能发挥不同的作用,即使是保护性SNP连锁后也可能增加癌症的易感性;另外,虽然大多数学者发现PLCE1 rs2274223G能增加胃癌患病风险,但是Luo等发现PLCE1 rs2274223与胃癌患者的良好预后相关,即rs2274223G可以降低胃癌患者的死亡风险[16]。以上研究提示某些SNP的存在可能影响PLCE1的功能,并可能影响胃癌的发生。然而这种风险因素的作用机制尚未阐明。现阶段,相关研究对SNPs的认识尚有局限,缺乏功能研究。在前期研究中我们发现西北地区汉族人rs2274223和rs3765524在癌症样本中具有高度的连锁现象,主要可以形成4种单体型,其中rs2274223G-rs3765524T单体型与胃癌风险显著相关。因此我们构建了PLCE1 rs2274223G-rs3765524T (PLCE1 Minor)和PLCE1rs2274223A-rs3765524C (PLCE1 Major)单体型真核表达重组载体,为揭示基因多态性和单体型对基因表达和功能的影响奠定基础。
SOE-PCR通过末端部分互补引物产生重叠链,最后通过重叠链将不同片段连接起来,获得全长产物或者重组产物。这一技术获得成功的关键是引物设计,特别要考虑引物重叠长度是否利于扩增以及有效扩增。实验中,我们设计将PLCE1基因分割成容易获得的四段,每段引物之间给予充分的重叠序列(15 bp-30 bp)经过三次融合后成功获得全长基因。在构建PLCE1 Major单体型真核表达重组载体时需要创建2个突变位点,传统的分别突变法不仅费时,还容易导致随机突变产生。我们在实验中采用了限制性核酸内切酶DpnⅠ酶切后同源重组的方法,因为DpnⅠ酶能够特异性识别并切断腺嘌呤甲基化的GmATC序列,不能识别和切割未甲基化的GATC序列。根据这一特性,我们可以在需要突变的多个位点上同时设计突变引物,以PLCE1 Minor质粒为模板,用高保真DNA聚合酶进行PCR反应,最后用DpnⅠ酶切除原始的甲基化模板,获得未甲基化的质粒片段,并经同源重组后,直接用于转化感受态细菌,得到突变的质粒,整个过程操作简单。从实验过程来看,甲基化的模板是关键,一般使用从DH5α等dam+大肠杆菌中抽提的质粒用于定点突变的模板。
NCBI数据库(http://www.ncbi.nlm.nih.gov/)已经收录了3条人类PLCE1基因的转录序列(NM_016341、NM_001165979和NM_001288989)。将它们编码的氨基酸序列和本实验扩增的PLCE1 Minor基因编码的氨基酸序列比对分析,发现PLCE1 Minor基因编码的氨基酸序列与NM_016341和NM_001165979编码的序列存在6个连续氨基酸(RPCLCC)的差别,而与NM_001288989编码的序列存在22个连续氨基酸(RPVSSPVLSSSNKSPSRPCLCC)的差别,这种差别来源于该基因内含子的不同剪切方式。由此可见,我们扩增获得了一种新的PLCE1转录本,其所编码的氨基酸序列可折叠成一种新的PLCE1异构体存在于机体内,该异构体的生物学功能尚待进一步研究。对于PLCE1异构体研究的文献非常少,其中Sorli等[1]发现PLCE1异构体1和2(PLCE1a和PLCE1b)在不同的组织及不同的细胞系中有差异表达,但尚没有发现两种异构体在功能上的明显差异。
通过基因转染,我们发现PLCE1 Minor比PLCE1 Major能显著提高PLCE1基因的表达量,说明PLCE1 rs2274223 A/G和rs3765524 C/T多态性及单体型能对自身基因的表达产生影响。虽然Wang等[17]已经发现rs2274223位点等位基因G (AG或GG)能够提高食管癌组织和食管癌细胞系PLCE1 mRNA和蛋白质表达水平并提高PLCE1酶活性,但他们的研究结果基于不同的食管癌细胞系,并不能排除不同细胞系中PLCE1基因其他位点的突变,以及其他PLC家族成员对最终结果的影响。可见,PLCE1 SNP与基因表达及其功能的关系亟待阐明。
综上所述,我们从HEK-293T细胞中成功克隆了人PLCE1 cDNA,并证实其属于PLCE1一种新的转录本。成功构建了PLCE1 rs2274223和rs3765524两种单体型真核重组表达载体,发现PLCE1 Minor单体型能够促进自身mRNA和蛋白质表达。实验结果为进一步研究PLCE1SNP与癌症易感性的关系奠定了基础。
| [1] | Sorli S C, Bunney T D, Sugden P H, et al. Signaling properties and expression in normal and tumor tissues of two phospholipase C epsilon splice variants. Oncogene , 2005, 24 (1) : 90–100. DOI:10.1038/sj.onc.1208168 |
| [2] | Martins M, Mccarthy A, Baxendale R, et al. Tumor suppressor role of phospholipase C epsilon in Ras-triggered cancers. Proc Natl Acad Sci U S A , 2014, 111 (11) : 4239–4244. DOI:10.1073/pnas.1311500111 |
| [3] | Cui X B, Li S, Li T T, et al. Targeting oncogenic PLCE1 by miR-145 impairs tumor proliferation and metastasis of esophageal squamous cell carcinoma. Oncotarget , 2016, 7 (2) : 1777–1795. |
| [4] | Han N, Zhao W, Zhang Z, et al. MiR-328 suppresses the survival of esophageal cancer cells by targeting PLCE1. Biochem Biophys Res Commun , 2016, 470 (1) : 175–180. DOI:10.1016/j.bbrc.2016.01.020 |
| [5] | Cui X B, Pang X L, Li S, et al. Elevated expression patterns and tight correlation of the PLCE1 and NF-kappaB signaling in Kazakh patients with esophageal carcinoma. Med Oncol , 2014, 31 (1) : 791. DOI:10.1007/s12032-013-0791-5 |
| [6] | Wang L D, Zhou F Y, Li X M, et al. Genome-wide association study of esophageal squamous cell carcinoma in Chinese subjects identifies susceptibility loci at PLCE1 and C20orf54. Nat Genet , 2010, 42 (9) : 759–763. DOI:10.1038/ng.648 |
| [7] | Abnet C C, Freedman N D, Hu N, et al. A shared susceptibility locus in PLCE1 at 10q23 for gastric adenocarcinoma and esophageal squamous cell carcinoma. Nat Genet , 2010, 42 (9) : 764–767. DOI:10.1038/ng.649 |
| [8] | Dusaban S S, Brown J H. PLCepsilon mediated sustained signaling pathways. Adv Biol Regul , 2015, 57 : 17–23. DOI:10.1016/j.jbior.2014.09.014 |
| [9] | Qu Y, Zhang S, Cui L, et al. Two novel polymorphisms in PLCE1 are associated with the susceptibility to esophageal squamous cell carcinoma in Chinese population. Dis Esophagus , 2016 . |
| [10] | Guo L Y, Zhang S, Suo Z, et al. PLCE1 Gene in esophageal cancer and interaction with environmental factors. Asian Pac J Cancer Prev , 2015, 16 (7) : 2745–2749. DOI:10.7314/APJCP.2015.16.7.2745 |
| [11] | Yuan J, Li Y, Tian T, et al. Risk prediction for early-onset gastric carcinoma:a case-control study of polygenic gastric cancer in Han Chinese with hereditary background. Oncotarget , 2016, 7 (23) : 33608–33615. |
| [12] | Mou X, Li T, Wang J, et al. Genetic variation of BCL2(rs2279115), NEIL2(rs804270), LTA (rs909253), PSCA (rs2294008) and PLCE1(rs3765524, rs10509670) genes and their correlation to gastric cancer risk based on universal tagged arrays and Fe3O4 magnetic nanoparticles. J Biomed Nanotechnol , 2015, 11 (11) : 2057–2066. DOI:10.1166/jbn.2015.2113 |
| [13] | Sun H, Wu X, Wu F, et al. Associations of genetic variants in the PSCA, MUC1 and PLCE1 genes with stomach cancer susceptibility in a Chinese population. PLoS One , 2015, 10 (2) : e117576. |
| [14] | Mocellin S, Verdi D, Pooley K A, et al. Genetic variation and gastric cancer risk:a field synopsis and meta-analysis. Gut , 2015, 64 (8) : 1209–1219. DOI:10.1136/gutjnl-2015-309168 |
| [15] | Malik M A, Umar M, Gupta U, et al. Phospholipase C epsilon 1(PLCE1 rs2274223A>G, rs3765524C>T and rs7922612C>T) polymorphisms and esophageal cancer risk in the Kashmir Valley. Asian Pac J Cancer Prev , 2014, 15 (10) : 4319–4323. DOI:10.7314/APJCP.2014.15.10.4319 |
| [16] | Luo D, Gao Y, Wang S, et al. Genetic variation in PLCE1 is associated with gastric cancer survival in a Chinese population. J Gastroenterol , 2011, 46 (11) : 1260–1266. DOI:10.1007/s00535-011-0445-3 |
| [17] | Wang L D, Bi X, Song X, et al. A sequence variant in the phospholipase C epsilon C2 domain is associated with esophageal carcinoma and esophagitis. Mol Carcinog , 2013, 52 (Suppl 1) : e80–e86. |
 2016, Vol. 36
2016, Vol. 36




