文章信息
- 梁向南, 张鲲, 邹少兰, 王建军, 马媛媛, 洪解放.
- LIANG Xiang nan, ZHANG Kun, ZOU Shao lan, WANG Jian jun, MA Yuan yuan, HONG Jie fang.
- 鸡尾酒δ整合策略构建表达三类纤维素酶的酿酒酵母工程菌株及初步评价
- Construction and Preliminary Evaluation of Saccharomyces cerevisiae Strains Co-expressing Three Types of Cellulase Via Cocktail δ-integration
- 中国生物工程杂志, 2016, 36(11): 54-62
- China Biotechnology, 2016, 36(11): 54-62
- http://dx.doi.org/DOI:10.13523/j.cb.20161108
-
文章历史
- 收稿日期: 2016-04-13
- 修回日期: 2016-05-19
2. 天津大学石化中心 天津 300072
2. Tianjin R & D Center for Petrochemical Technology, Tianjin University, Tianjin 300072, China
石油、天然气和煤炭等化石能源是当前最主要的能源形式,由于它们不可再生,大量消耗必然导致其快速枯竭。因而,以化石能源为基础的现代化社会的发展模式是难以持续高速发展的。寻找能(部分)代替化石能源且污染较少的可再生的能源是未来发展的一个方向。
地球上每年光合作用产生的生物质高达1 500亿~2 000亿t[1],5%的生物质所含的能量与人类对石油和天然气的需求量相当。生物质中木质纤维素年产量约为1 000亿t[2],是自然界中最丰富并可再生的碳源,具有替代粮食并大规模转化成生物燃料而完全替代化石燃料的潜力。相比而言,以玉米等粮食作物生产生物燃料虽然工艺简单、技术成熟,但其生产潜能远达不到预期。2010年10月,美国国会研究部的报告指出,即使将2009年全美国的玉米都用于生产乙醇,其产量也只能替代美国汽油消费量的18%。因此,以木质纤维素为原料生产生物燃料已成为世界范围内生物燃料技术发展的趋势,其中木质纤维素燃料乙醇是发展力度最强的。
使用木质纤维素生物转化生产乙醇通常包括生物质预处理、纤维素酶生产、水解和发酵等多个步骤。纤维素酶的生产需要单独进行,虽然纤维素酶已商品化,但纤维素酶的用量巨大,如100kg预处理过的甘蔗渣需要超过2.8kg蛋白质的纤维素酶才能有效降解[3],这使得纤维素酶的成本成为纤维素乙醇进行商业化生产的主要制约因素之一。另外,将纤维素酶的生产、水解和发酵组合在一起的统合生物加工过程(consolidated bioprocessing,CBP)由于避免了纤维素酶生产过程中的设备投资、减少了原料和其它材料的使用,从而降低水解和发酵成本[4]而被认为是一种有应用前景的技术。该技术的关键是构建一种能有效降解纤维素类多聚糖成发酵糖并发酵产醇的微生物[5-6]。在酿酒酵母中表达纤维素酶,使之具有利用纤维素发酵产醇的能力即是其中的一种策略。
在酿酒酵母中表达纤维素酶已有许多报道,在相关综述中有详细介绍[7-9]。一些研究表明,能产纤维素酶的重组菌株能减少外加纤维素酶的用量,或是在同步糖化发酵(simultaneous saccharification and fermentation,SSF)过程中能提高外加纤维素酶的水解效率,从而提高乙醇产量。我们前期的工作也表明,在外加商业化纤维素酶的情况下表达纤维素酶的酿酒酵母菌株能提高发酵性能[6, 10]。在以往的研究中,酿酒酵母中一般仅表达一种外切葡聚糖酶(CBH)、一种内切葡聚糖酶(EG)和一种β-葡萄糖苷酶(BGL)。我们前期的研究中将Trichoderma reesei(瑞氏木霉)egl2、Aspergillus aculeatus(棘孢曲霉)bgl1和三种外切酶基因(A. aculeatus cbh1、T. reesei cbh1和cbh2)通过分步δ整合的方式整合至酿酒酵母染色体,得到的菌株W3与出发菌株相比能有效利用酸碱预处理的玉米芯[6]。另外,对纤维素的有效降解需要各类纤维素酶的协同作用,对于不同物料,微生物产生、分泌的纤维素酶不同酶组分的比例会发生相应变化,如T. reesei所分泌的各类纤维素酶之间的比例随环境条件变化,以此达到对纤维素底物的最大水解[11]。在酿酒酵母中表达各类纤维素酶基因,各基因之间的最优表达比例也受木质纤维素料的种类和特性的影响,而后者与木质纤维素的预处理方法紧密相关。故要在酿酒酵母中优化三类纤维素酶即EG、BGL和CBH的表达比例,一方面需要解决遗传操作技术手段,另一方面要建立行之有效的转化子筛选、鉴定方法。在遗传操作技术手段上,Yamada等[12]开发出了一种鸡尾酒δ整合的方法来实现三种纤维素酶的优化表达。纤维素的有效降解需要EG、BGL和CBH的协同作用,其中CBH的表达是构建CBP酿酒酵母菌株的一个制约因素[13],而EG的表达对纤维素料的快速降黏是至关重要的[14],为尽可能提高这两种酶的活性,本研究采用鸡尾酒δ整合的策略将三种外切葡聚糖酶基因(T. reesei cbh1、cbh2和A. aculeatus cbh1)、两种内切葡聚糖酶基因(T. reesei egl2和A. aculeatus egl1)和A. aculeatus bgl1同时整合至酿酒酵母W303-1A染色体上,经几轮整合后对筛选得到的菌株进行评价。
1 材料与方法 1.1 材料 1.1.1 菌株、质粒与引物本研究所用菌株见表 1。Escherichia coli Top10用于质粒构建和扩增,Saccharomyces cerevisiae W303-1A为纤维素酶表达的宿主,W3和AADY为对照菌株。
| Strains | Relevant features | Reference |
| Bacterial strain | ||
| E. coli TOP10 | F- mcrAΔ(mrr-hsd RMS-mcrBC) 80 lacZΔM15 ΔlacX74 recA1 araΔ139Δ(ara-leu)7697 galU galK rpsL (StrR) endA1 nupG | Invitrogen |
| S. cerevisiae strains | ||
| AADY | Industrial strain,Angel | Yichang,China |
| W3 | MATa ade2-1 can1-100 ura3-1 δ-Integration of eg2,cbh1,and cbh2 from T. reesei,bgl1 and cbh1 from A. aculeatus | [6] |
| W303-1A | MATa leu2-3,112 ura3-1 trp1-92 his3-11,15 ade2-1 can1-100 | In our lab |
| LA1 | MATa leu2-3,112 ura3-1 his3-11,15 ade2-1 can1-100 δ-Integration of eg2,cbh1,and cbh2 from T. reesei,eg1,bgl1 and cbh1 from A. aculeatus | This study |
| LA2 | MATa leu2-3,112 ura3-1 ade2-1 can1-100 δ-Integration of eg2,cbh1,and cbh2 from T. reesei,eg1,bgl1 and cbh1 from A. aculeatus | This study |
| LA3 | MATa ura3-1 ade2-1 can1-100 δ-Integration of eg2,cbh1,and cbh2 from T. reesei,eg1,bgl1 and cbh1 from A. aculeatus | This study |
| LA4 | MATa ade2-1 can1-100 δ-Integration of eg2,cbh1,and cbh2 from T. reesei,eg1,bgl1 and cbh1 from A. aculeatus | This study |
本研究所用部分质粒见表 2,这些质粒用于整合质粒的构建。
| Plasmid | Selection marker | Expressing cellulase gene or relevant features | Reference |
| pGEM-Ptpi-xyn2s-AaCBHI-TadhI | AmpR | Cellobiohydrolase I gene of Aspergillus aculeatus | [6] |
| pGEM-Ptpi-xyn2s-TrCBHI-TadhI | AmpR | Cellobiohydrolase I gene of Trichoderma reesei | [6] |
| pGEM-Ptpi-xyn2s-TrCBHII-TadhI | AmpR | Cellobiohydrolase II gene of Trichoderma reesei | [6] |
| pGEM-Ptpi-xyn2s-AaEGI-TadhI | AmpR | Endoglucanase I gene of Aspergillus aculeatus | [14] |
| YEplac195-Ptpi-xyn2s-TrEGII-TadhI | URA3,AmpR | Endoglucanase II gene of Trichoderma reesei | [15] |
| pGEM-Ptpi-xyn2s-AaBGLI-TadhI | AmpR | β-Glucosidase I gene of Aspergillus aculeatus | [16] |
| pGδL | LEU2,AmpR | Two δ sequences as the homologous arms | [17] |
| pGδT | TRP1,AmpR | Two δ sequences as the homologous arms | [17] |
| pGδH | HIS3,AmpR | Two δ sequences as the homologous arms | [17] |
| pGδU | URA3,AmpR | Two δ sequences as the homologous arms | [17] |
本研究所用引物见表 3,用于重组菌中整合的纤维素酶基因的鉴定。
| Primers | Sequence | Reference |
| ACBHI-u | GGAGAAGCGCCAGCAGGTCGGCACTT | This study |
| ACBHI-d | CTTGGAAGTGGTGGTGGTGGTCTTGGAGCT | This study |
| TCBHI-u | GCTGTGGAGAAGCGCCAGTCGGCCTGCACT | This study |
| TCBHI-d | CATGTTGGCGTAGTAATCATCCCACAGACT | This study |
| TCBHII-u | CAAGCTTGCTCAAGCGTCTGGGGCCAATGTGGTGGC | This study |
| TCBHII-d | TAAGAAATTCGCTTACAGGAACGATGGGT | This study |
| AEGI-u | GGAGAAGCGCCAACAGACTCAACTCT | This study |
| AEGI-d | AGAAATTCGCCTACTGTACGCTGGCA | This study |
| TEGII-u | GTACCACAGATGGCACTTGCGTTACC | This study |
| TEGII-d | AGAAATTCGCCTACTTTCTTGCGAGA | This study |
| ABGLI-u | AGCAGCGCCAGTCTCAGT | This study |
| ABGLI-d | CAAGATGCTGGTGTCGTG | This study |
1.1.2 培养基与培养条件
E. coli用LB培养基(蛋白胨1%、酵母粉0.5%、NaCl 1%,pH7.0~7.2,加入2%琼脂即为固体培养基)于37℃培养,必要时加入100μg/ml氨苄青霉素。
YPD培养基(2%蛋白胨、1%酵母粉、2%葡萄糖)和CMA培养基[6.7%YNB(yeast nitrogen base without amino acids),2%Avicel (PH101,Sigma-Aldrich Co.,Ireland),添加必要的氨基酸和核酸,2%琼脂]分别用于酵母菌株的培养和筛选;YPSC(2%蛋白胨、1%酵母粉、10%酸碱预处理的玉米芯,自然pH)和USC(10%酸碱预处理的玉米芯,0.033%尿素,pH5.0)用于酵母菌株的发酵评价。酵母菌培养温度为30℃。
1.2 方法 1.2.1 分子克隆操作、质粒构建及转化基因组DNA的提取纯化、DNA纯化、质粒提取、DNA酶切、连接与转化,以及E. coli转化等均参考文献[18];S. cerevisiae转化采用醋酸锂法[19]。
1.2.2 纤维素酶活性测定酿酒酵母菌株的β-葡萄糖苷酶活性(BGL activity)、羧甲基纤维素酶活性(CMCase activity)、滤纸酶活(FPase activity)和磷酸膨胀纤维素降解活性(PASC degradation activity)的测定参考Hong等[6]的方法。
1.2.3 酸碱预处理玉米芯发酵评价酸碱预处理玉米芯的制备参照Wang等[10]的方法。预处理之前原料纤维素含量(30.46±0.16)%(wt%),半纤维素含量(30.13±0.31)%(wt%);预处理之后纤维素和半纤维素含量分别为(83.01±2.73)%(wt%)和(3.07±0.49)%(wt%)。
酵母菌株在YPD液体培养基中好氧培养24h后离心收集菌体,用于接种用的种子,接种量为1g干重/kg料浆。纤维素料的发酵是在100ml螺旋口烧瓶中进行的,装有40g YPSC或USC培养基,添加5FPU/g干料的和氏璧纤维素酶。每隔24h取样,适当稀释后用0.22μm滤膜过滤,用HPLC(Agilent 1200)检测乙醇浓度。条件:带有保护柱(Cation-H Refill Cartridges)的Aminex HPX-87H(Bio-Rad)分离柱,柱温40℃;流动相为4mmol/L H2SO4,流速0.6ml/min;RID检测器,光学单元温度40℃。
2 结果 2.1 整合质粒的构建本研究中所用的纤维素酶基因表达盒如图 1(a)所示。表达盒元件:Ptpi和TadhI分别是酿酒酵母TPI1(磷酸丙糖异构酶)的启动子和ADHI(乙醇脱氢酶)的终止子;xyn2s为瑞氏木霉xylanase II(木聚糖酶)的信号肽;纤维素酶基因为来源于瑞氏木霉的cbh1、cbh2和egl2,来源于棘孢曲霉的cbh1、egl1和bgl1。
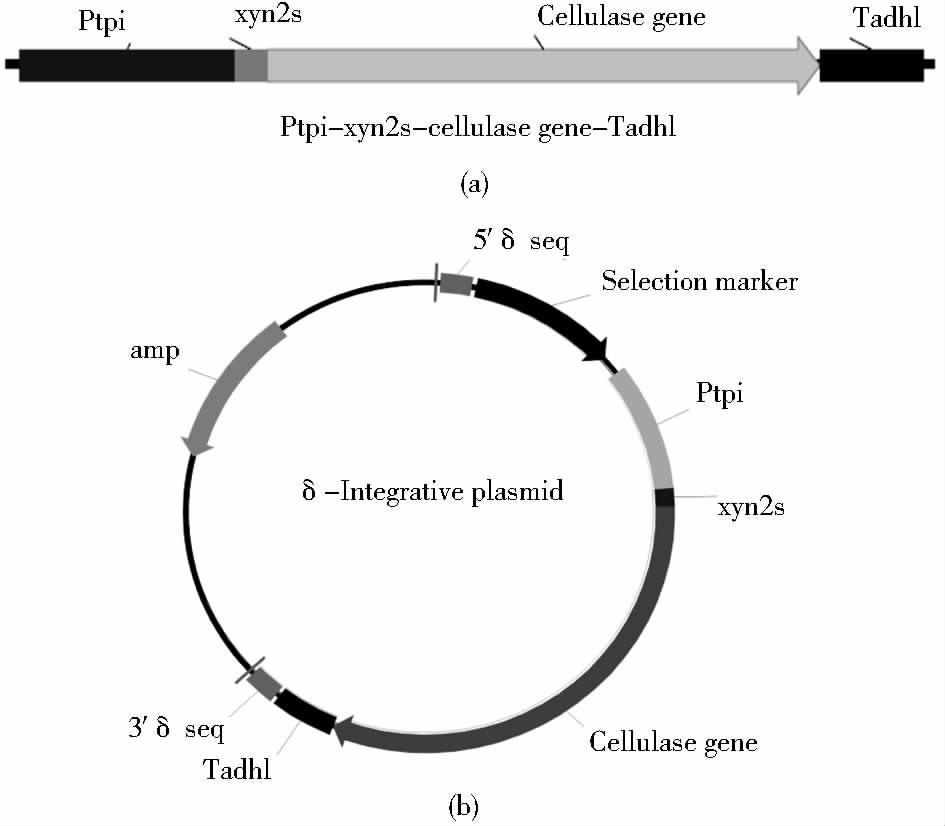
|
| 图 1 基因表达盒和δ-整合质粒示意图 Figure 1 Schematic drawing of cellulase genes expression cassette (a) and integrative plasmid (b) |
6种分别带有不同纤维素酶基因表达盒的质粒pGEM-Ptpi-xyn2s-AaCBHI- TadhI[6]、pGEM-Ptpi-xyn2s-TrCBHI-TadhI[6]、pGEM-Ptpi-xyn2s-TrCBHII-TadhI[6]、pGEM-Ptpi-xyn2s-AaEGI-TadhI[14]、YEplac195-Ptpi-xyn2s-TrEGII-TadhI[15]和pGEM-Ptpi-xyn2s-AaBGLI-TadhI[16](表 2)分别用合适的限制性内切酶消化后,琼脂糖凝胶电泳回收带有纤维素酶基因表达盒的片段,末端平滑化后,分别插入带有δ序列的质粒pGδL[17]、pGδT[17]、pGδH[17]和pGδU[17]中,构建得到24个整合型质粒,如图 1(b)所示,分别命名为L-AaCBHI、L-TrCBHI、L-TrCBHII、L-AaEGI、L-TrEGII、L-AaBGL1、T-AaCBHI、T-TrCBHI、T-TrCBHII、T-AaEGI、T-TrEGII、T-AaBGL1、H-AaCBHI、H-TrCBHI、H-TrCBHII、H-AaEGI、H-TrEGII、H-AaBGL1、U-AaCBHI、U-TrCBHI、U-TrCBHII、U-AaEGI、U-TrEGII和U-AaBGL1。这些整合型质粒涉及6种纤维素酶基因T. reesei cbh1、cbh2、egl2以及A. aculeatus cbh1、egl1和bgl1,分别带有4种筛选标记URA3、TRP1、HIS3和LEU2。这些质粒中的一些在我们的前期工作中已使用,在酿酒酵母中可实现纤维素酶基因的良好表达[6, 17]。
2.2 鸡尾酒δ整合及重组菌酶活分析本研究中构建得到的整合质粒,分别带有不同的营养缺陷型筛选标记(URA3、TRP1、HIS3和LEU2),而宿主酿酒酵母实验室菌株W303-1A具有包括这些标记在内的多种营养缺陷型筛选标记,因此三类纤维素酶基因可分步进行整合,每步整合采用不同的筛选标记进行筛选。
各取10μg质粒T-AaCBHI、T-TrCBHI、T-TrCBHII、T-AaEGI、T-TrEGII和T-AaBGL1,分别采用合适的限制性内切酶消化后纯化,混合后转化W303-1A,转化液涂布缺省L-色氨酸的CMA平板,结果仅长出一个单菌落,命名为LA1。该菌株用YPD液体培养后,提取基因组,用克隆纤维素酶基因的引物对ACBHI-u/ACBHI-d、TCBHI-u/TCBHI-d、TCBHII-u/TCBHII-d、AEGI-u/AEGI-d、TEGII-u/TEGII-d和ABGLI-u/ABGLI-d进行PCR验证,分别考察6种基因A. aculeatus cbh1、T. reesei cbh1、T. reesei cbh2、A. aculeatus egl1、T. reesei egl2和A. aculeatus bgl1的整合情况,目的片段大小分别为1 423bp、1 137bp、1 356bp、686bp、948bp和1 509bp,同时以菌株W303-1A染色体作为对照,结果见图 2,泳道1~6和泳道7~12为相应的PCR产物电泳结果。从图 2可以看出,与对照菌株相比,菌株LA1在相应泳道上均出现目的DNA条带,说明上述提及的6种纤维素酶基因均整合至菌株LA1染色体上。菌株LA1作为宿主用于后续整合。
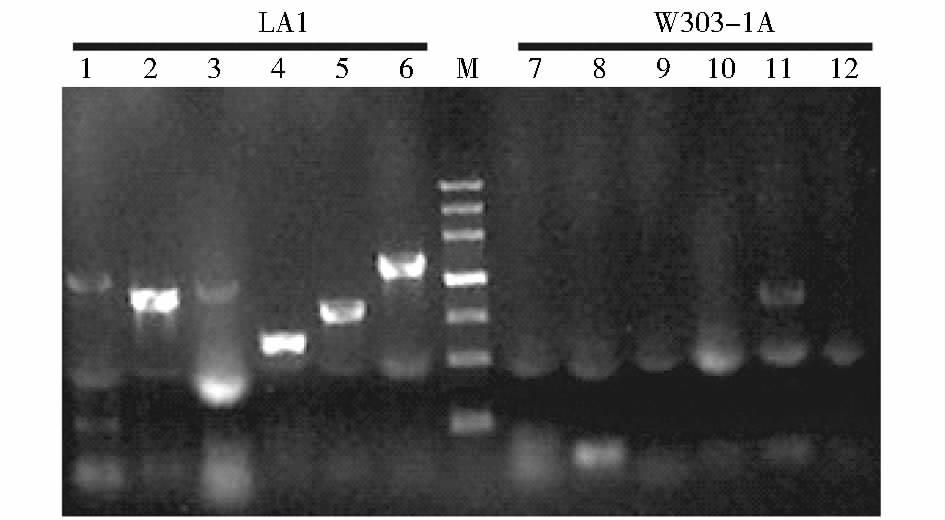
|
| 图 2 PCR鉴定菌株LA1 Figure 2 The identification of the strain LA1 by PCR analysis DNA marker included 7 lanes of 200bp,500bp,800bp,1 200bp,2 000bp,3 000bp,and 4 500bp |
各取10μg质粒H-AaCBHI、H-TrCBHI、H-TrCBHII、H-AaEGI、H-TrEGII和H-AaBGL1,分别采用合适的限制性内切酶线性化、纯化、混合后转化LA1, 转化液涂布缺省L-色氨酸和L-组氨酸的CMA平板。挑取20个大菌落用YPD液体培养基培养后,25%接种量接种YPSC液体培养基,和氏璧纤维素酶用量10FPU/g,发酵5天后用HPLC法测定发酵液乙醇浓度,选取其中乙醇浓度最高菌株作为后续试验,命名为LA2。
各取10μg质粒L-AaCBHI、L-TrCBHI、L-TrCBHII、L-AaEGI、L-TrEGII和L-AaBGL1,线性化后纯化、混合,转化LA2,转化液涂布缺省L-亮氨酸的CMA平板。挑取60个大菌落用YPD液体培养基培养后,25%接种量接种YPSC液体培养基,和氏璧纤维素酶用量10FPU/g,发酵5天后用HPLC法测定发酵液乙醇浓度,选取其中乙醇浓度最高菌株作为后续试验,命名为LA3。
各取10μg质粒U-AaCBHI、U-TrCBHI、U-TrCBHII、U-AaEGI、U-TrEGII和U-AaBGL1,线性化后纯化、混合,转化LA3,转化液涂布缺省尿嘧啶的CMA平板。挑取60个大菌落用YPD液体培养基培养后,25%接种量接种YPSC液体培养基,和氏璧纤维素酶用量10FPU/g,发酵5天后用HPLC法测定发酵液乙醇浓度,选取其中乙醇浓度最高菌株作为后续试验,命名为LA4。
经4轮鸡尾酒整合,得到4株整合有纤维素酶基因的工程菌株,首先对它们进行了纤维素酶活性分析,结果见图 3。由图 3可以看出:①前3轮整合中,菌株BGL、CMCase、FPase和PASC降解活性逐渐增加;②菌株LA4的4种酶活较菌株LA3的4种酶活均略有下降,说明第4轮整合未能更进一步提高菌株的纤维素酶活力。这与菌株LA4的发酵产醇性能与LA3相比未得到明显提升的结果相符(数据未给)。造成这一现象的可能原因:①前3轮中外源基因的整合已接近饱和;②与第4轮纤维素酶基因整合所采用的筛选标记URA3有关,根据我们前期的研究,使用该筛选标记所得的整合菌株具有最低的外源基因拷贝数和最低的纤维素酶活性。
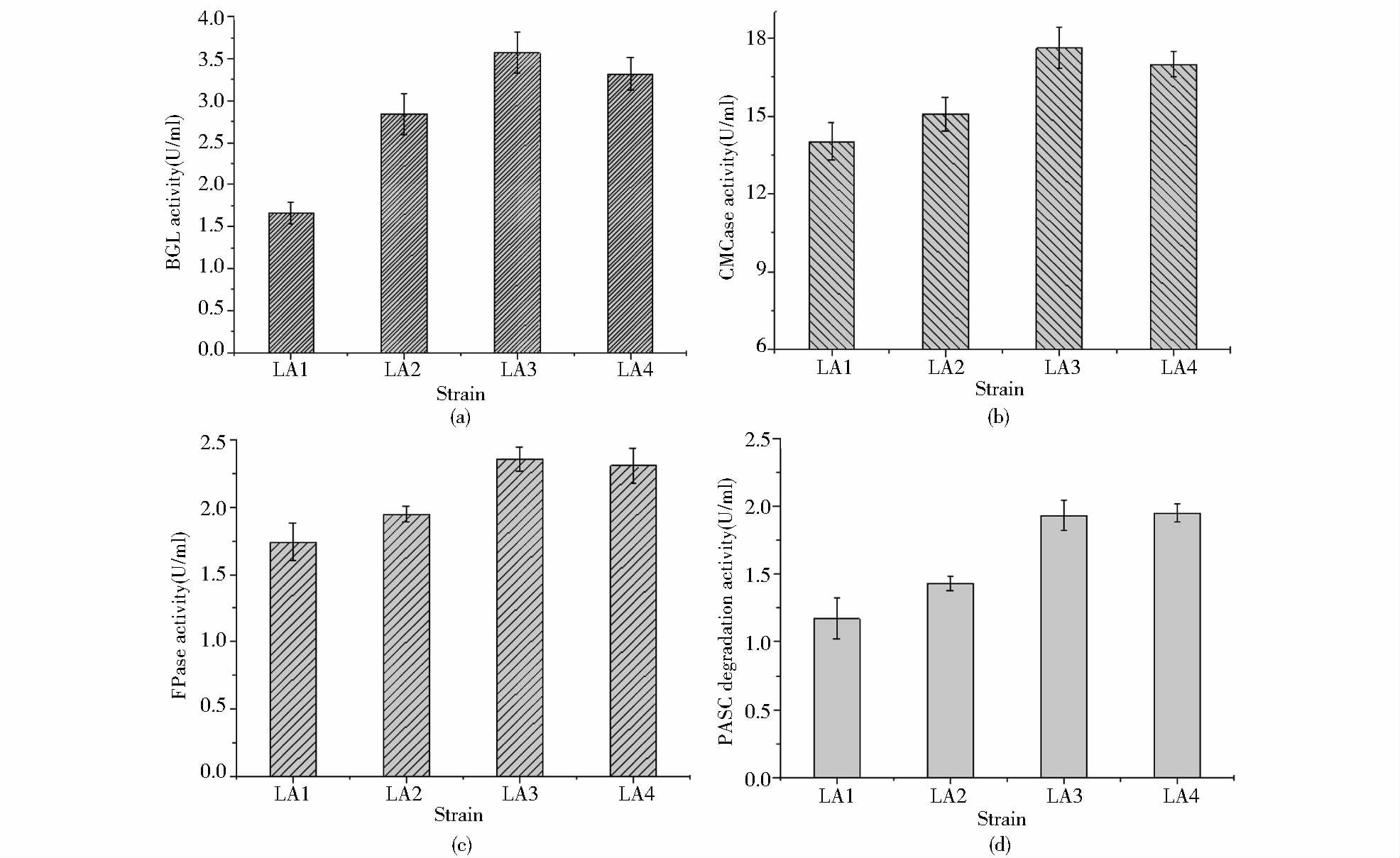
|
| 图 3 酿酒酵母菌株纤维素酶活力 Figure 3 The cellulases activity of S. cerevisiae strains (a) BGL (b) CMCase (c) FPase (d) PASC degradation activity |
选择菌株LA3进行纤维素料乙醇发酵评价,以菌株W303-1A、W3和AADY为对照菌株。已报道的大多数研究中,纤维素酶的用量一般在5~20FPU/g生物质,通常选择10FPU/g生物质。这里我们选择5FPU/g生物质,考察各酿酒酵母菌株在较低外加纤维素酶剂量下利用纤维素发酵产醇情况。而且,在试验中我们发现,培养基的类型也影响菌株的发酵性能,这里考察了各菌株在营养相对丰富的YPSC培养基和营养相对贫瘠的USC培养基中的发酵情况,结果如图 4所示。
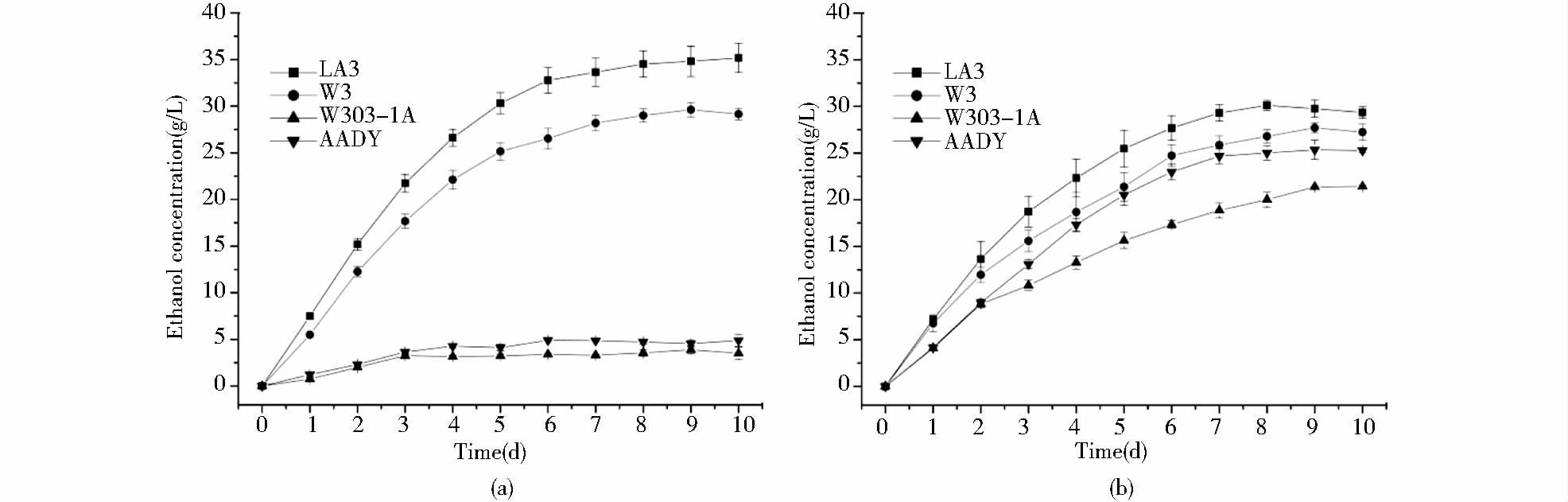
|
| 图 4 酿酒酵母菌株利用酸碱预处理玉米芯发酵产醇评价 Figure 4 The fermentation performance from acid- and alkali-pretreated corncob by S. cerevisiae strains (a) YPSC medium (b) USC medium |
在YPSC培养基中,从图 4(a)中可以看出,与菌株W303-1A和AADY相比,菌株LA3和W3均能有效利用酸碱预处理玉米芯发酵产醇,发酵10天最大乙醇浓度分别达到(35.18±1.56)g/L和(29.62±0.77)g/L,LA3发酵性能优于W3;在USC培养基中,从图 4(b)中可以看出,与W303-1A和AADY相比,菌株LA3和W3也有较好的发酵性能,10天发酵周期内最大乙醇浓度分别为(30.11±0.54)g/L和(27.70±0.53)g/L,LA3发酵性能也优于W3。
比较菌株在两种培养基的发酵情况,对菌株W303-1A和AADY,在营养相对丰富的YPSC培养基中发酵性能较在营养相对贫瘠的USC培养基中的差。在实验过程中我们测定了样品的pH(试纸法),结果发现在YPSC培养基中,两菌株发酵液在整个发酵周期内pH都在6以上,说明两菌株不能有效降低起始发酵液的pH,发酵液pH明显高于纤维素酶的最适pH(5.0),使得外加的纤维素酶不能充分降解纤维素,从而导致两菌株在YPSC培养基中的发酵性能较差。相反,在USC培养基中在整个发酵周期内,两菌株的发酵液pH在3.5~4.5,从结果看此pH范围内外加的纤维素酶活性还较好,从而使得两菌株在USC培养基中的发酵性能较佳。
对菌株LA3和W3则结果刚好相反。两菌株在YPSC中pH为3.5~5.5(发酵中后期均在3.5左右),在USC中pH为3.5~4.5。在营养相对丰富的YPSC培养基中发酵性能较在营养相对贫瘠的USC培养基中的较佳,推测营养丰富时能更好地促进重组菌分泌外源纤维素酶,从而能更好地提高菌株发酵性能。
从上面分析可以看出,在纤维素料发酵过程中pH的控制对不表达纤维素酶的酿酒酵母菌株发酵性能极为重要;发酵液中的营养盐对表达纤维素酶的酿酒酵母菌株发酵性能影响也较明显。
另外,纤维素酶的添加量会通过影响酶解速率和葡萄糖浓度进而影响酿酒酵母利用纤维素发酵产醇动力学和产率。外加纤维素酶用量增加,最大乙醇浓度增加,达到最大乙醇浓度的时间也变短(数据未给出),但高的外加纤维素酶用量使得构建CBP菌株失去意义。另外,LA3和W3的YPD培养物在离心收集菌体时,若带入一部分上清液,则能明显提高菌株的发酵性能(数据未给出),这也是我们在试验初期菌株发酵性能表现不稳定的原因。尽量不引入上清液,菌株发酵性能则较稳定。从反面也可看出,种子液带入的纤维素酶也会影响重组菌株的发酵性能。
3 讨论如同前文所述,构建CBP酿酒酵母菌株在纤维素乙醇生产中是一种有前景的策略。目前,虽然已有许多关于此方面的报道,但构建得到的菌株在无添加商品化纤维素酶的情况下基本还不能有效利用纤维素生产乙醇。其中一个原因可能是重组菌株仍然不能分泌足够多的纤维素酶,且比例也不太合适。因此,任何能提高重组菌纤维素酶表达和优化各组分比例的努力都是值得尝试的。
为提高外源基因整合拷贝数,本研究使用高拷贝δ整合的策略。δ整合是基于酿酒酵母高频率同源重组的特性,利用染色体上DNA重复序列δ序列作为整合位点[17]。δ序列为Ty元件的长末端重复序列,在酵母染色体中大约含有425个这样的位点[20],是纤维素酶基因整合的很好位点。另外,纤维素酶各组分比例的优化表达对重组菌发酵性能也是重要的,这在我们的综述[21]中已有介绍。在本研究中,通过鸡尾酒整合的策略6种纤维素酶基因表达盒整合至酿酒酵母实验室菌株W303-1A染色体,经多轮转化及筛选构建得到表达纤维素酶的菌株LA3,在外加纤维素酶的情况下与对照菌株W303-1A和AADY相比,能有效利用酸碱预处理玉米芯发酵产醇;与分步整合得到的重组菌W3[6]相比,菌株LA3发酵性能更优。这些结果说明鸡尾酒整合的策略较为有效。另外,在不添加商品化纤维素酶的情况下,菌株LA3在YPSC培养基中的最大乙醇浓度仅为(7.87±0.83)g/L。因此,继续提高重组菌的发酵性能仍然是未来工作的内容之一。
为达到这一目的,可以从以下几个方面进行尝试:①杂交,将不同方法构建得到的表达纤维素酶的重组菌进行杂交,通过合适的筛选方法有望构建得到发酵性能更优的重组菌株。②有性重组,通过酿酒酵母有性重组能提高其发酵性能[22]。δ整合结合同源三倍体生孢的方法也是一种有前景的方法来提高外源纤维素酶的表达从而提高菌株的发酵产醇性能[23]。③使用工业菌株作为宿主,与实验室菌株相比工业菌株更具有鲁棒性,使用工业菌株作为宿主有望能提高重组菌株的发酵性能。目前为止,构建得到的表达纤维素酶的重组菌最成功的即是以工业菌株衍生菌株为宿主的[24]。总之,这些方法结合起来使用可能会得到在纤维素培养基中具有优异发酵性能的表达纤维素酶的酿酒酵母重组菌株。
| [1] | 曲音波. 纤维素乙醇产业化. 化学进展 , 2007, 19 (7/8) : 1098–1108. Qu Y B. Industrialization of cellulosic ethanol. Progress in Chemistry , 2007, 19 (7/8) : 1098–1108. |
| [2] | Alvira P, Tomás-Pejó E, Ballesteros M, et al. Pretreatment technologies for an efficient bioethanol production process based on enzymatic hydrolysis:a review. Bioresour Technol , 2010, 101 (13) : 4851–4861. DOI:10.1016/j.biortech.2009.11.093 |
| [3] | Krishnan C, Sousa L D, Jin M, et al. Alkali-based APEX pretreatment for the conversion of sugarcane bagasse and cane leaf residues to ethanol. Biotechnol Bioeng , 2010, 107 (3) : 441–450. DOI:10.1002/bit.22824 |
| [4] | Lynd L R, van Zyl W H, McBride J E, et al. Consolidated bioprocessing of cellulosic biomass:an update. Curr Opin Biotechnol , 2005, 16 (5) : 577–583. DOI:10.1016/j.copbio.2005.08.009 |
| [5] | Hahn-Hägerdal B, Galbe M, Gorwa-Grauslund M F, et al. Bio-ethanol——the fuel of tomorrow from the residues of today. Trends Biotechnol , 2006, 24 (12) : 549–556. DOI:10.1016/j.tibtech.2006.10.004 |
| [6] | Hong J F, Yang H J, Zhang K, et al. Development of a cellulolytic Saccharomyces cerevisiae strain with enhanced cellobiohydrolase activity. World Journal of Microbiology & Biotechnology , 2014, 30 (11) : 2985–2993. |
| [7] | Hasunuma T, Kondo A. Development of yeast cell factories for consolidated bioprocessing of lignocellulose to bioethanol through cell surface engineering. Biotechnol Adv , 2012, 30 (6) : 1207–1218. DOI:10.1016/j.biotechadv.2011.10.011 |
| [8] | la Grange D C, de Haan R, van Zyl W H. Engineering cellulolytic ability into bioprocessing organisms. Appl Microbiol Biotechnol , 2010, 87 (4) : 1195–1208. DOI:10.1007/s00253-010-2660-x |
| [9] | van Zyl W H, Lynd L R, den Haan R, et al. Consolidated bioprocessing for bioethanol production using Saccharomyces cerevisiae. Adv Biochem Eng Biotechnol , 2007, 108 : 205–235. |
| [10] | Wang G Q, Liu C, Hong J F, et al. Comparison of process configurations for ethanol production from acid-and alkali-pretreated corncob by Saccharomyces cerevisiae strains with and without β-glucosidase expression. Bioresource Technology , 2013, 142 : 154–161. DOI:10.1016/j.biortech.2013.05.033 |
| [11] | Sticker A R, Mach R L, de Graaff L H. Regulation of transcription of cellulases-and hemicellulases-encoding genes in Aspergillus niger and Hypocrea jecorina (Trichoderma reesei). Appl Microbiol Biotechnol , 2008, 78 (2) : 211–220. DOI:10.1007/s00253-007-1322-0 |
| [12] | Yamada R, Taniguchi N, Tanaka T, et al. Cocktail delta-integration:a novel method to construct cellulolytic enzyme expression ratio-optimized yeast strains. Microb Cell Fact , 2010, 9 (1) : 32. DOI:10.1186/1475-2859-9-32 |
| [13] | Lynd L R, van Zyl W H, McBride J E, et al. Consolidated bioprocessing of cellulosic biomass:an update. Curr Opin Biotechnol , 2005, 16 (5) : 577–583. DOI:10.1016/j.copbio.2005.08.009 |
| [14] | 张韦娜. 表达纤维素酶基因的酿酒酵母菌株构建及其底物利用评价.天津: 天津大学, 化工学院, 2012. Zhang W N. The Construction and Evaluation of Recombinant Saccharomyces cerevisiae strains expressing cellulase genes. Tianjin: Tianjin University, School of Chemical Engineering and Technology,2012. |
| [15] | Zhang W N, Liu C, Wang G C, et al. Comparison of the expression in Saccharomyces cerevisiae of endoglucanase II from Trichoderma reesei and Endoglucanase I from Aspergillus aculeatus. BioResources , 2012, 7 (3) : 4031–4045. |
| [16] | 邹少兰. 利用木质纤维素水解液产醇工程菌株构建及其产醇研究.天津: 天津大学, 化工学院, 2011. Zou S L. Construction and Evaluation of Recombinant Strains for Ethanol Production from Lignocellulosic Hydrolysate.Tianjin: Tianjin University, School of Chemical Engineering and Technology,2011. |
| [17] | Liu L, Liu C, Zou S, et al. Expression of cellulase genes in Saccharomyces cerevisiae via δ-integration subject to auxotrophic markers. Biotechnol Lett , 2013, 35 (8) : 1303–1307. DOI:10.1007/s10529-013-1205-7 |
| [18] | 奥斯伯 F, 布伦特R, 金斯顿R E, 等. 精编分子生物学实验指南.颜子颖, 王海林译. 北京: 科学出版社, 1999. Ausubel F M, Brent R, Kingston R E, et al. Short Protocol in Molecular Biology.Yan Z Y, Wang H L. Beijing: Science Press, 1999. |
| [19] | Gietz R D, Sugino A. New yeast-Escherichia coli shuttle vectors constructed with in vitro mutagenized yeast genes lacking six-base pair restriction sites. Gene , 1988, 74 (2) : 527–534. DOI:10.1016/0378-1119(88)90185-0 |
| [20] | Dujon B. The yeast genome project: what did we learn?. Trends Genet , 1996, 12 (7) : 263–270. DOI:10.1016/0168-9525(96)10027-5 |
| [21] | 杨华军, 邹少兰, 洪解放, 等. 纤维素酶在酿酒酵母中的表达研究. 中国生物工程杂志 , 2014, 34 (6) : 75–83. Yang H J, Zou S L, Hong J F, et al. Advance in research on cellulase expression in Saccharomyces cerevisiae. China Biotechnology , 2014, 34 (6) : 75–83. |
| [22] | Hou L H. Improved production of ethanol by novel genome shuffling in Saccharomyces cerevisiae. Appl Biochem Biotechnol , 2010, 160 (4) : 1084–1093. DOI:10.1007/s12010-009-8552-9 |
| [23] | Yang H, Liu C, Zou S, et al. Improving bgl1 gene expression in Saccharomyces cerevisiae through meiosis in an isogenic triploid. Biotechnol Lett , 2014, 36 (6) : 1279–1285. DOI:10.1007/s10529-014-1471-z |
| [24] | Khramtsov N, McDade L, Amerik A, et al. Industrial yeast strain engineered to ferment ethanol from ligocellulosic biomass. Bioresource Technology , 2011, 102 (17) : 8310–8313. DOI:10.1016/j.biortech.2011.05.075 |
 2016, Vol. 36
2016, Vol. 36




