文章信息
- 张健, 吴昊, 李艳妮, 乔建军.
- ZHANG Jian, WU Hao, LI Yan-ni, QIAO Jian-jun.
- 革兰氏阳性菌非编码小RNA的研究进展
- Advances in Research on Small Non-coding RNAs of Gram-positive Bacteria
- 中国生物工程杂志, 2016, 36(10): 106-114
- CHINA BIOTECHNOLOGY, 2016, 36(10): 106-114
- http://dx.doi.org/DOI:10.13523/j.cb.20161015
-
文章历史
- 收稿日期: 2016-03-21
- 修回日期: 2016-04-06
非编码小RNA(small non-coding RNA)的长度在50~500 nt之间,作为细菌中的重要的调节因子,其调节功能在近十几年被逐渐报道[1]。它们主要由基因组编码,但是绝大多数不编码蛋白产物。sRNA在细胞对环境胁迫的应答过程中发挥作用,通过与mRNA分子互补配对作用或者与相应蛋白分子相互作用来发挥调控作用[2]。同时sRNA受到全局调节因子的调控,参与许多重要的细菌生理途径,另外还是许多信号通路中的重要节点[3-4]。而且sRNA可以在转录后水平上行使调控功能,影响细胞内mRNA的翻译,在环境发生变化的情况下,细胞内某些基因的转录可能已经停止,但之前完成转录的转录本依然存在于细胞中。此时,sRNA能够迅速调节这些转录本的翻译,实现细胞对环境变化的及时响应,进而达到sRNA介导分级调控和靶标mRNA高效周转的效果。sRNA这种转录后调控的方式在生物体内广泛存在并扮演重要角色,1967年,通过标记大肠杆菌E.coli的总RNA,Hindley[5]在聚丙烯酰胺凝胶上发现了第一个sRNA-6SRNA。第一个发现的反式编码的反义RNA是E.coli的MicF[6]。其可以被不同的环境应激(渗透性、温度和其他变化)诱导[7-8]。但是到目前为止,有关sRNA的研究大多集中于革兰氏阴性菌(如大肠杆菌、沙门氏菌等),仅大肠杆菌就已经有超过80个sRNA得到了实验验证[9]。随着生物信息学和RNA深度测序技术的发展,近几年在一些革兰氏阳性菌(枯草芽孢杆菌Bacillus subtilis[10],单核细胞增多性李斯特菌Listeria monocytogenes[11],肺炎链球菌Streptococcus pneumoniae[12],酿脓链球菌Streptococcus pyogenes[13],金黄葡萄球菌Staphylococcus aureus[14]等)中也发现了sRNA的存在,其生物学功能也逐渐揭示出来。本文从革兰氏阳性菌sRNA的发现,作用机制以及参与的生理活动等方面介绍sRNA的研究进展。
1 sRNA的特征和分类大多已知的细菌sRNA由基因组(少部分位于质粒、噬菌体或转座子上)转录形成但并不编码蛋白质,以RNA形式发挥调节作用的分子,由位于开放阅读框(Open reading frame,ORF)之间的基因间隔区(Intergenic regions)转录生成[15],或由编码基因的5′和3′非编码区(Untranslated regions,UTR)修饰处理形成[16]。sRNA基因的转录一般由一段可以形成稳定茎环结构的序列起始,止于Rho非依赖型终止子,形成独立的转录单元[17]。大多已知功能的sRNA以反义RNA(antisense RNA)形式特异性地与靶mRNA结合,调节基因表达,其中以与靶mRNA通过碱基互补配对方式相互作用最为普遍,根据其作用机制又分为顺式编码(cis-encoded)和反式编码(trans-encoded)两种情况[18]。革兰氏阳性菌sRNA与靶mRNA结合位点一般是富含CU的保守序列,如FasX与ska和cpa[19],RNAIII与coa[20],LhrC与lapB等[21]。
2 sRNA的作用机制通常情况下,sRNA主要参与基因转录后水平的调控,因而可以迅速而有效地发挥功能。与革兰氏阴性菌不同,由于缺乏类似于CsrB的调控蛋白螯合sRNA,革兰氏阳性菌的sRNA一般通过RNA杂交的方式实现调控mRNA翻译的功能。然而不同的sRNA所发挥的基因调节功能和作用机制也不尽相同。根据目前发现的革兰氏阳性菌sRNA的作用机制,可以将其分为以下三类:
2.1 顺式编码反义sRNA(cis-encoded antisense sRNA)顺式编码的sRNA根据其链特异性可分为顺式编码反义sRNA和顺式编码5′端元件sRNA。顺式编码反义sRNA存在于所调控的靶标基因的内部,由互补链转录而来,通过与其靶mRNA形成严格的互补配对,对其进行表达抑制或基因沉默[22]。所以,顺式编码反义sRNA一般只有唯一的靶标mRNA。与互补链基因相对位置的差异也会导致sRNA的作用机制有所区别,顺式编码反义sRNA一方面可以在转录后水平抑制基因的表达或调节mRNA降解过程,另一方面也能在转录水平发挥转录衰减或干扰的作用,甚至通过改变正向RNA和反向RNA的转录比例达到调控基因表达的目的[22]。
2.2 顺式编码5′端元件sRNA(cis-encoded 5′ element sRNA)顺式编码5′端元件sRNA一般位于受其调控的mRNA的5′端,在与代谢物结合或者温度、pH发生变化时,其结构也会相应改变以影响下游基因或同一多顺反子下基因的转录或翻译,甚至可能直接影响mRNA的降解(作用于转录终止因子如Rho和RNase E),进而达到调控的效果[22]。一般情况下,核糖开关主要负责编码蛋白基因的调控,但研究发现其中一些会同时行使核糖开关和sRNA的双重功能[23]。在E. faecalis中发现的sRNA EutX自身就包含一个核糖开关:通常情况下,EutX与EutV自然折叠形成发卡结构;当腺苷氰钴胺素(AdoCbl)存在时,与mRNA发生蛋白螯合作用,EutX不再结合EutV,激活下游多个eut基因转录后翻译过程[24]。
2.3 反式编码sRNA(trans-encoded sRNA)反式编码sRNA通常由基因组上与其靶标基因不相邻的基因间区序列转录而来。这类sRNA主要结合到临近核糖体结合位点(Ribosome bind site,RBS)的位置,通过与靶标mRNA不严格匹配的双链结构,控制靶标mRNA的翻译或介导mRNA降解,进而影响靶标基因的表达。一般来讲,反式编码sRNA可以与多个不同的靶mRNA发生相互作用,细菌中多以这种反式编码sRNA为主。以S. aureus的RNAIII为例,RNAIII通过碱基配对或降解的方式抑制rot,SA1000,spa,lytM和coa的表达[20, 25-27],同时以解开环状结构释放RBS的方式激活hla和map的表达[28-29]。革兰氏阳性菌反式编码sRNA的作用机制如图 1。
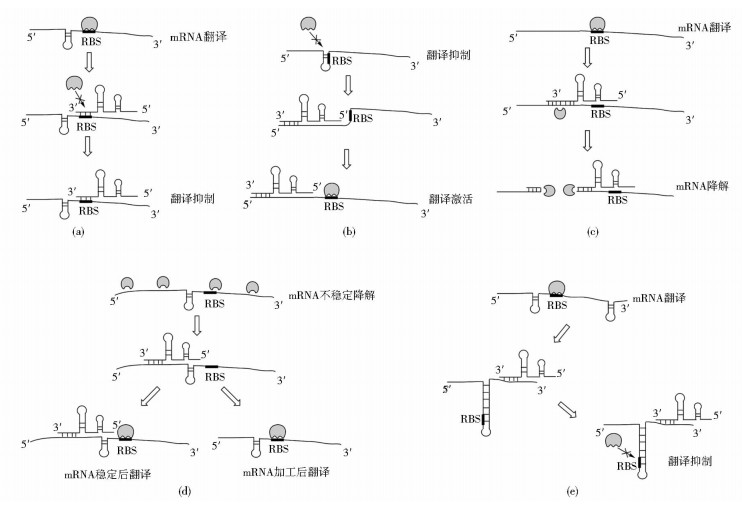
|
| 图 1 革兰氏阳性菌反式编码sRNA作用机制 Figure 1 Mechanisms of trans-encoded sRNA in Gram-positive bacteria (a) Translation inhibition by blocking of RBS (b) Translation activation by mediating mRNA structure change (c) mRNA degradation by forming cleavage sites (d) mRNA stabilization and processing (e) Translation inhibition by mediating mRNA structure change |
近几年,随着生物信息学和RNA测序技术的发展, 许多革兰氏阳性菌的sRNA被发现,其预测的相应靶基因也得到了验证[30-33]。目前细菌sRNA及其靶基因的识别方法主要有:生物信息学预测方法和实验分析方法。
3.1 生物信息学预测方法尽管sRNA及其靶标的确认一般需要通过实验分析方法来证实,但生物信息学预测仍然是一种相对简便快捷的筛选方法。常见的基于基因组预测sRNA的方法包括:比较基因组学、RNA二级结构相似性和碱基构成统计分析[34]。比较基因组学方法主要是利用亲缘相近的物种之间的基因间隔区(IGRs)序列相似性进行预测。利用QRNA,RNAz,sRNAPredict2等预测工具在B. subtilis,S. pneumoniae和S. mutans等革兰氏阳性菌的基因组中均发现了新的sRNA[35-37]。同一菌属的基因组序列通常具备一定的相似性,对不同菌种的基因组进行blast分析可以找到种间广泛存在的sRNA。此外,利用已知sRNA的二级结构(启动子、终止子或完整的转录本)信息,可以通过预测工具如RNAMotif来识别新的sRNA。将RNA二级结构保守性与最小自由能相结合,通过打分系统进行评估也是筛选基因间新sRNA的一种方法。一些低GC含量革兰氏阳性菌(如S.aureus)的二核苷酸组成可以揭示基因组序列存在的结构限制,这些分析结果能帮助发现sRNA[38]。
一个sRNA可能有多个靶标基因存在,其调控作用常常会引起多重效应。靶标基因是指与sRNA直接作用的mRNA,而sRNA还可以通过这些靶标基因的介导作用间接调控下游基因[39]。细菌基因组测序的完成为生物信息学预测sRNA的靶标mRNA提供了可能。目前常用的sRNA靶标mRNA预测的软件有:RNAPredator[40]、sRNATarBase[41]、sRNATarget[42]、IntaRNA[43]、TargetRNA[44]和RNAhybrid[45]等。靶标RNA预测的生物信息学算法主要基于序列长度、RNA二级结构、碱基匹配、保守性及其位置等特征。对于每一个sRNA,预测软件根据基因组中的比对情况或运用RNA二级结构自由能的计算规则对候选mRNA进行打分排序,其中得分高或P值小的序列被看作是可能的靶标基因[46-47]。
3.2 实验分析方法在特定的条件(如高温、低pH、体内等)下,高表达的sRNA可以通过变性PAGE、萃取、标记和RNA测序等处理过程分离纯化得以验证[38]。另外,鸟枪法(Shotgun cloning)是从总RNA分离一定长度(50~500nt)的RNA进行逆转录,然后构建cDNA文库对sRNA进行测序[48]。微阵列分析利用高丰度寡核苷酸探针与基因及其间隔区产生的杂交信号进行高通量分析、检测和记录,有效地鉴定不同实验条件或应激情况下转录水平发生变化的sRNA[49]。免疫共沉淀利用蛋白质(如Hfq)的抗体来调取与之相互作用的sRNA,Dambach等[50]在B. subtilis内源hfq基因表位添加FLAG标签,利用免疫共沉淀获得了22个与Hfq蛋白相互作用的sRNA。近年来,RNA深度测序(RNA deep sequencing)技术因其较好的灵敏度和精确度现已成为大批量筛选sRNA的重要研究手段[51]。此外,以上这些方法筛选得到的sRNA还需要应用如Northern印迹(利用寡核苷酸链与RNA相互作用获得杂交信号)和实时荧光定量PCR(qRT-PCR)等辅助方法加以验证。sRNA识别的流程如图 2所示。
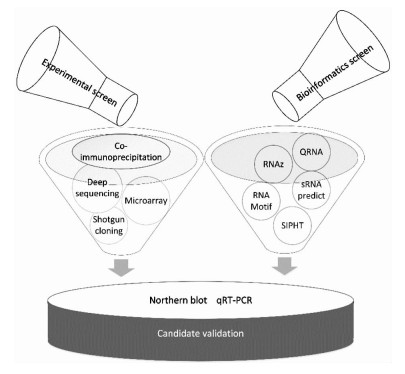
|
| 图 2 sRNA的识别流程 Figure 2 Schematic of the flow for sRNA identification |
传统的遗传学方法也可以用于sRNA靶标的识别,如利用lacZ和gfp报告基因融合表达的方法:sRNA在被诱导或过表达时,通过改变与候选基因融合的LacZ或GFP蛋白的表达来筛选sRNA的靶标[52-53]。利用His/flag标记的Hfq亲和纯化的方法来调取sRNA的靶标,该方法是基于Hfq蛋白与sRNA结合后能协助后者与靶标mRNA相互作用的原理。如果sRNA只是调控靶标mRNA的翻译而影响蛋白表达水平,就可以利用蛋白质组学分析识别sRNA的靶标基因,即在sRNA过表达或缺失的菌株中对不同丰度的蛋白质进行荧光标记来检测筛选靶标基因[54]。有关sRNA靶标识别方法的优缺点如表 1所示。
| 方法 | 优点 | 缺点 |
| 生物信息学 | 快速高通量,可用于不同菌种间的比较 | 需要合适的算法,已注释的基因或ORF可能被识别 |
| 蛋白亲和技术 | 识别sRNA与靶标的相互作用 | 取决于亲和力的强弱及靶标的特异性和丰度 |
| 报告基因融合方法 | 直接识别作用位点 | 工作量较大,与RNA结合的实际情况有一定差别 |
| 蛋白质组学 | 不需要清楚sRNA的调控模式,准确性高 | 部分小蛋白和难溶解的蛋白质难以检测 |
4 sRNA的生物学功能
sRNA的功能主要是调节翻译起始、RNA的稳定性和蛋白质的活性,其中最主要的是扮演翻译调控的角色:sRNA直接结合到RBS序列位置抑制mRNA翻译过程;sRNA结合诱导RBS序列发生结构变化,达到抑制或促进翻译的效果;sRNA封闭核糖体备用位点,保证mRNA的高效翻译。正是这些不同的调控方式,使得sRNA在细菌体内的诸多生理活动中发挥着重要的生物学功能。
4.1 sRNA与毒力基因的表达毒力调控对病原体的生存起着至关重要的作用,许多sRNA参与到这个过程中,通过研究sRNA的调控作用可以帮助更好地了解革兰氏阳性菌的发病机理。菌毛是S. pyogenes细菌荚膜外侧用于附着宿主细胞以及繁殖的重要菌体结构,而Cpa作为菌毛合成操纵子编码胶原结合蛋白,在菌毛荚膜之间起到连接与黏附的作用。FasX通过与cpa mRNA碱基配对作用,阻碍核糖体与后者RBS的结合,减少相关菌毛蛋白的表达[55]。L. monocytogenes中,S-腺苷甲硫氨酸(S-adenosly methionine,SAM)核糖开关SreA和SreB行使反式编码sRNA的功能,SreA上调agrD的表达而抑制prfA的翻译,SreB负责prfA和lmo2230两个基因的调控,这些调控机制揭示了细菌毒力与养分有效性之间的某种联系[56]。在较低温度下,prfA mRNA的5′UTR会形成稳定的茎环结构封闭SD序列导致核糖体无法进入;当进入宿主细胞后,温度上升至37℃刺激mRNA的结构发生变化,核糖体得以与其结合启动翻译,进一步诱导毒力基因的表达[57]。在RNAIII缺失的S. aureus菌种中,α毒素的产量相对于胞内hla mRNA含量会下降90%;RNAIII与hla mRNA3的5′UTR互补结合形成复合体,起到抑制mRNA自身形成二级结构的作用,从而释放RBS,激活Hla的翻译过程[28]。同时,ArtR与sarT mRNA的5′端非编码区直接结合以促进RNase III降解mRNA,进而介导激活α毒素的表达,参与毒力调节过程[58]。Rot蛋白是一种参与毒力基因表达的转录调控因子,RNAIII自身富含C的发卡结构与rot mRNA翻译起始区富含G的茎环结构发生互补作用,致使SD序列被封闭进而抑制了Rot的翻译过程[59]。由于Rot可以上调葡萄球菌蛋白A和凝固酶Coa的表达,所以RNAIII对这些细胞表面蛋白的抑制作用也可能是间接调控的结果。LhrC序列在Listeria菌属中高度保守,其靶基因lapB编码细胞壁锚定粘附蛋白,LhrC上富含CU的位点直接与lapB mRNA结合从而阻碍毒力因子的翻译,进而影响细胞的毒力作用[21]。在C. perfringens中,VirX被证明参与多种毒素相关基因(plc,col,pfoA等)的表达调控过程[60]。
4.2 sRNA与群体感应细菌通过向环境中分泌自诱导分子(AI)相互交流有关细胞密度的信息,临近的细胞通过监测周围AI浓度来感知种群规模的变化,进而调节自身生理活动(如生物被膜,生物发光或毒力因子的分泌等),发挥多细胞生物学功能,这种现象叫做群体感应。sRNA在群体感应信号传导方面有着重要的调控作用。Agr群体感应系统由两个反向的转录单元组成:RNAII和RNAIII;其中,RNAII编码AgrA(响应调节因子),AgrB(外排蛋白),AgrC(组氨酸激酶感应器)以及AgrD(AI前体)等RNAIII激活蛋白(RNAIII-activating protein,RAP) [31]。当AI与Agr结合,被激活的AgrA与agr启动子促进包括PSMα和PSMβ在内的一系列酚可溶性调控蛋白(phenol-soluble modulin,PSM)的表达,发挥群体感应的调节作用[61]。AgrA通过与psmα启动子结合的方式促进PSMα的表达,但psm-mec RNA与agrA mRNA相互作用会使AgrA的翻译受到抑制,从而达到间接下调表达PSMα的效果,影响PSM的产量[62-63]。在L. monocytogenes中,SreA通过调节AgrD的表达量来控制群体感应系统[56]。
4.3 sRNA与铁离子代谢铁是细菌代谢的最重要营养元素之一,在胞内的许多酶促反应里起着辅助因子的作用,同时还会影响细菌的感染能力。大多数细菌中,铁吸收调节蛋白(Ferric uptake regulator) Fur负责调控铁的吸收和利用,调控超过20个操纵子来保证胞内铁稳态[64]。B. subtilis的铁元素代谢与储存依赖于sRNA FsrA和Fur的相互作用[65]。与大肠杆菌中RyhB不同,FsrA的调控不需要Hfq蛋白的参与,而是与三个铁调控碱性蛋白(FbpA,FbpB和FbpC)一起阻遏靶标基因的翻译,主要涉及三羧酸循环中的一些含铁蛋白如琥珀酸脱氢酶(succinate dehydrogenase,SdhABC)和顺乌头酸酶(aconitase,CitB)以及相关代谢酶如谷氨酸合成酶(glutamate synthase,GltAB)和二羧酸阴离子通透酶(dicarboxylate permease,DctP)等[65-66]。在低铁浓度环境中,Fur不能与铁离子结合,处于失活状态,解除了对FsrA的抑制作用,此时,FsrA在RNA分子伴侣FbpB协助下,与编码铁硫蛋白氧化酶的lutABC mRNA结合,使其稳定性和翻译效率下降,进而维持细菌胞内的铁平衡[67]。而在调控异丙基苹果酸脱氢酶(isopropyl malate dehydrogenase,LeuCD)的表达时,FsrA则需要FbpC蛋白完成对leuCD mRNA翻译的抑制作用[68]。
4.4 sRNA与双组分系统双组分系统(Two-component system,TCS)是细胞感知和响应环境变化的一种基本机制。TCS一般由胞膜上的组氨酸激酶和胞质间的响应调节因子组成,前者负责监测环境状况,后者负责细胞应激反应[69]。一些革兰氏阳性菌的sRNA,如RivX(S. pyogenes)、RoxS(B. subtilis)、VR-RNA(C. perfringens)和CiaRH (S. pneumoniae)等,都受到TCS的调节。CovRS双组分系统由响应调控因子CovR和感应器激酶CovS组成,参与调控众多毒力基因包括sRNA RivX,再由RivX正向调控Mga调节子,RivX将CovRS双组分系统和Mga调控网络联系起来,起到了传递环境信号的作用[70]。ResDE双组分系统首先通过控制生物信号分子NO的表达来调节RoxS的转录,再由RoxS调控下游NAD+/NADH激酶(NAD+/NADH kinase,PpnKB)和琥珀酰辅酶A合成酶(succinyl-CoA synthase,SucCD)的表达,在一定程度上拓宽了细胞呼吸链和电子传递的调控途径。VirRS双组分系统负责多种生物酶,毒力因子和细胞代谢基因的调控,VR-RNA作为VirRS调节元件的一部分,同时参与编码磷脂酶C(Phospholipase C,Plc)和胶原酶(Collagenase,ColA)两个蛋白的表达[71]。VR-RNA还可以通过直接结合mRNA的方式控制pfoA和ccp等多个基因的表达,同时激活负责毒素基因转录调控的蛋白(CPE1447和CPE1446)翻译过程[72]。在Streptococus菌属中,CiaRH双组分系统包含多个(2到6个不等)sRNA,长度在50~200nt左右,这些高度相似的sRNA被称为cia依赖性sRNA(csRNA),csRNA在5′和3′末端形成两个茎环结构,茎环间未配对的部分与靶mRNA的SD序列和起始密码子AUG、GUG和UUG互补,参与多种生理活动的调节[73]。
4.5 sRNA与Hfq分子伴侣Hfq蛋白是一类高度保守的转录后调控因子,革兰氏阴性菌中大部分反式编码sRNA都需要在Hfq蛋白的协助下与靶mRNA结合从而调控翻译过程[74]。研究表明,Hfq可能参与抑制RNA二聚体的形成[75],与富含AU的RNase裂解位点结合还可以增强sRNA的稳定性,保护其免受降解[76]。L. monocytogenes中的LhrA是目前革兰氏阳性菌中唯一被证实的Hfq依赖型sRNA,以反义结合的形式直接调控包括lmo0850和lmo0302在内的多个靶标基因[77-78]。LhrA可以调控接近300个基因,显示出全局调控的功能,体外结合实验表明在Hfq协助下LhrA可以干扰核糖体与chiA mRNA的结合抑制翻译并参与降解过程,影响几丁质酶A(chitinase A,ChiA)的表达,从而调控L. monocytogenes的几丁质分解活动[79]。但大多数革兰氏阳性菌中的sRNA调节过程并不需要Hfq的参与[80-81],而且像链球菌属Streptococci和乳酸菌属Lactobacilli中没有Hfq的同源序列存在,目前也没有发现其它具有分子伴侣功能的sRNA结合蛋白。
5 总结与展望sRNA因其在细菌体内通过影响基因表达,进而起到一定生物学功能的特性,受到国内外学者的广泛关注。以E. coli为代表的革兰氏阴性菌sRNA被大量报道,有关革兰氏阳性菌sRNA的研究仍需完善。随着sRNA筛选、鉴定以及功能表征技术的不断成熟,越来越多的革兰氏阳性菌sRNA会被逐渐挖掘出来。sRNA作为一类调节因子,在革兰氏阳性菌错综复杂调控网络中发挥着重要作用,但sRNA具体调控过程以及与靶标基因结合方式尚不清楚,有待进一步探究。
利用人工合成sRNA改变细菌生理特性、调控代谢途径的尝试为细菌进化以及菌种改造手段的创新带来了新的可能。比如构建sRNA介导的耐酸网络提高菌株耐受性、利用sRNA的调控功能提高发酵效率。虽然现有的sRNA报道已经取得一定成果,但还有许多问题亟待解决,相关基础研究工作还需要进一步深入:sRNA上游调控机制尚未清楚;sRNA之间是否有相互作用以及调节关系;sRNA所调控下游不同路径是否存在相互联系;能否形成调控网络等等。这些问题的解决可以帮助我们更好地了解细菌体内复杂的调控机制,为合成生物学、基因工程以及工业微生物育种技术的发展奠定良好的基础。
| [1] | Hershberg R, Altuvia S, Margalit H. A survey of small RNA-encoding genes in Escherichia coli. Nucleic Acids Research , 2003, 31 (7) : 1813–1820. DOI:10.1093/nar/gkg297 |
| [2] | Wassarman K M. Small RNAs in bacteria: diverse regulators of gene expression in response to environmental changes. Cell , 2002, 109 (2) : 141–144. DOI:10.1016/S0092-8674(02)00717-1 |
| [3] | Livny J, Waldor M K. Identification of small RNAs in diverse bacterial species. Current Opinion in Microbiology , 2007, 10 (2) : 96–101. DOI:10.1016/j.mib.2007.03.005 |
| [4] | Storz G. An expanding universe of noncoding RNAs. Science , 2002, 296 (5571) : 1260–1263. DOI:10.1126/science.1072249 |
| [5] | Hindley J T. Fractionation of 32 P-labelled ribonucleic acids on polyacrylamide gels and their characterization by fingerprinting. Journal of Molecular Biology , 1967, 30 (1) : 125–136. DOI:10.1016/0022-2836(67)90248-3 |
| [6] | Andersen J, Delihas N, Ikenaka K, et al. The isolation and characterization of RNA coded by the micF gene in Escherichia coli. Nucleic Acids Research , 1987, 15 (5) : 2089–2101. DOI:10.1093/nar/15.5.2089 |
| [7] | Andersen J, Forst S, Zhao K, et al. The function of micF RNA. micF RNA is a major factor in the thermal regulation of OmpF protein in Escherichia coli. Journal of Biological Chemistry , 1989, 264 (30) : 17961–17970. |
| [8] | Delihas N, Forst S. MicF: an antisense RNA gene involved in response of Escherichia coli to global stress factors. Journal of Molecular Biology , 2001, 313 (1) : 1–12. DOI:10.1006/jmbi.2001.5029 |
| [9] | Kang Z, Zhang C, Zhang J, et al. Small RNA regulators in bacteria: powerful tools for metabolic engineering and synthetic biology. Applied Microbiology and Biotechnology , 2014, 98 (8) : 3413–3424. DOI:10.1007/s00253-014-5569-y |
| [10] | Rasmussen S, Nielsen H B, Jarmer H. The transcriptionally active regions in the genome of Bacillus subtilis. Molecular Microbiology , 2009, 73 (6) : 1043–1057. DOI:10.1111/mmi.2009.73.issue-6 |
| [11] | Wurtzel O, Sesto N, Mellin J R, et al. Comparative transcriptomics of pathogenic and non-pathogenic Listeria species. Molecular Systems Biology , 2012, 8 (1) : 583. |
| [12] | Acebo P, Martin-Galiano A J, Navarro S, et al. Identification of 88 regulatory small RNAs in the TIGR4 strain of the human pathogen Streptococcus pneumoniae. RNA , 2012, 18 (3) : 530–546. DOI:10.1261/rna.027359.111 |
| [13] | Perez N, Treviño J, Liu Z, et al. A genome-wide analysis of small regulatory RNAs in the human pathogen group A Streptococcus. PloS One , 2009, 4 (11) : e7668. DOI:10.1371/journal.pone.0007668 |
| [14] | Abu-Qatouseh L F, Chinni S V, Seggewiβ J, et al. Identification of differentially expressed small non-protein-coding RNAs in Staphylococcus aureus displaying both the normal and the small-colony variant phenotype. Journal of Molecular Medicine , 2010, 88 (6) : 565–575. DOI:10.1007/s00109-010-0597-2 |
| [15] | Waters L S, Storz G. Regulatory RNAs in bacteria. Cell , 2009, 136 (4) : 615–628. DOI:10.1016/j.cell.2009.01.043 |
| [16] | Kawano M, Reynolds A A, Miranda-Rios J, et al. Detection of 5'-and 3'-UTR-derived small RNAs and cis-encoded antisense RNAs in Escherichia coli. Nucleic Acids Research , 2005, 33 (3) : 1040–1050. DOI:10.1093/nar/gki256 |
| [17] | Li W, Ying X, Lu Q, et al. Predicting sRNAs and their targets in bacteria. Genomics, Proteomics & Bioinformatics , 2012, 10 (5) : 276–284. |
| [18] | Brantl S. Bacterial chromosome-encoded small regulatory RNAs. Future Microbiology , 2009, 4 (1) : 85–103. DOI:10.2217/17460913.4.1.85 |
| [19] | Miller E W, Cao T N, Pflughoeft K J, et al. RNA-mediated regulation in Gram-positive pathogens: an overview punctuated with examples from the group A Streptococcus. Molecular Microbiology , 2014, 94 (1) : 9–20. DOI:10.1111/mmi.2014.94.issue-1 |
| [20] | Chevalier C, Boisset S, Romilly C, et al. Staphylococcus aureus RNAIII binds to two distant regions of coa mRNA to arrest translation and promote mRNA degradation. PLoS Pathog , 2010, 6 (3) : e1000809. DOI:10.1371/journal.ppat.1000809 |
| [21] | Sievers S, Lillebæk E M, Jacobsen K, et al. A multicopy sRNA of Listeria monocytogenes regulates expression of the virulence adhesin LapB. Nucleic Acids Research , 2014, 42 (14) : 9383–9398. DOI:10.1093/nar/gku630 |
| [22] | Cho K H, Kim J H. Cis-encoded non-coding antisense RNAs in Streptococci and other low GC Gram (+) bacterial pathogens. Frontiers in Genetics , 2015, 6 : 110. |
| [23] | Mellin J, Cossart P. Unexpected versatility in bacterial riboswitches. Trends in Genetics , 2015, 31 (3) : 150–156. DOI:10.1016/j.tig.2015.01.005 |
| [24] | DebRoy S, Gebbie M, Ramesh A, et al. A riboswitch-containing sRNA controls gene expression by sequestration of a response regulator. Science , 2014, 345 (6199) : 937–940. DOI:10.1126/science.1255091 |
| [25] | Boisset S, Geissmann T, Huntzinger E, et al. Staphylococcus aureus RNAIII coordinately represses the synthesis of virulence factors and the transcription regulator Rot by an antisense mechanism. Genes & Development , 2007, 21 (11) : 1353–1366. |
| [26] | Chunhua M, Yu L, Yaping G, et al. The expression of LytM is down-regulated by RNAIII in Staphylococcus aureus. Journal of Basic Microbiology , 2012, 52 (6) : 636–641. DOI:10.1002/jobm.v52.6 |
| [27] | Huntzinger E, Boisset S, Saveanu C, et al. Staphylococcus aureus RNAIII and the endoribonuclease III coordinately regulate spa gene expression. The EMBO Journal , 2005, 24 (4) : 824–835. DOI:10.1038/sj.emboj.7600572 |
| [28] | Morfeldt E, Taylor D, Von Gabain A, et al. Activation of alpha-toxin translation in Staphylococcus aureus by the trans-encoded antisense RNA, RNAIII. The EMBO Journal , 1995, 14 (18) : 4569. |
| [29] | Liu Y, Mu C, Ying X, et al. RNAIII activates map expression by forming an RNA-RNA complex in Staphylococcus aureus. FEBS Letters , 2011, 585 (6) : 899–905. DOI:10.1016/j.febslet.2011.02.021 |
| [30] | Zheng H, Liu E, Shi T, et al. Strand-specific RNA-seq analysis of the Lactobacillus delbrueckii subsp. bulgaricus transcriptome. Molecular BioSystems , 2016, 12 (2) : 508–519. DOI:10.1039/C5MB00547G |
| [31] | Brantl S, Brückner R. Small regulatory RNAs from low-GC Gram-positive bacteria. RNA Biology , 2014, 11 (5) : 443–456. DOI:10.4161/rna.28036 |
| [32] | Mraheil M A, Billion A, Kuenne C, et al. Comparative genome-wide analysis of small RNAs of major Gram-positive pathogens: from identification to application. Microbial Biotechnology , 2010, 3 (6) : 658–676. DOI:10.1111/mbt.2010.3.issue-6 |
| [33] | Forsyth R, Haselbeck R J, Ohlsen K L, et al. A genome-wide strategy for the identification of essential genes in Staphylococcus aureus. Molecular Microbiology , 2002, 43 (6) : 1387–1400. DOI:10.1046/j.1365-2958.2002.02832.x |
| [34] | Pichon C, Felden B. Small RNA gene identification and mRNA target predictions in bacteria. Bioinformatics , 2008, 24 (24) : 2807–2813. DOI:10.1093/bioinformatics/btn560 |
| [35] | Silvaggi J M, Perkins J B, Losick R. Genes for small, noncoding RNAs under sporulation control in Bacillus subtilis. Journal of Bacteriology , 2006, 188 (2) : 532–541. DOI:10.1128/JB.188.2.532-541.2006 |
| [36] | Livny J, Brencic A, Lory S, et al. Identification of 17 Pseudomonas aeruginosa sRNAs and prediction of sRNA-encoding genes in 10 diverse pathogens using the bioinformatic tool sRNAPredict2. Nucleic Acids Research , 2006, 34 (12) : 3484–3493. DOI:10.1093/nar/gkl453 |
| [37] | Livny J, Teonadi H, Livny M, et al. High-throughput, kingdom-wide prediction and annotation of bacterial non-coding RNAs. PloS One , 2008, 3 (9) : e3197. DOI:10.1371/journal.pone.0003197 |
| [38] | Pichon C, Felden B. Small RNA genes expressed from Staphylococcus aureus genomic and pathogenicity islands with specific expression among pathogenic strains. Proceedings of the National Academy of Sciences of the United States of America , 2005, 102 (40) : 14249–14254. DOI:10.1073/pnas.0503838102 |
| [39] | Vogel J, Wagner E G. Target identification of small noncoding RNAs in bacteria. Current Opinion in Microbiology , 2007, 10 (3) : 262–270. DOI:10.1016/j.mib.2007.06.001 |
| [40] | Eggenhofer F, Tafer H, Stadler P F, et al. RNApredator: fast accessibility-based prediction of sRNA targets. Nucleic Acids Research , 2011, 39 (suppl 2) : W149–W154. |
| [41] | Cao Y, Wu J, Liu Q, et al. sRNATarBase: a comprehensive database of bacterial sRNA targets verified by experiments. RNA , 2010, 16 (11) : 2051–2057. DOI:10.1261/rna.2193110 |
| [42] | Cao Y, Zhao Y, Cha L, et al. sRNATarget: a web server for prediction of bacterial sRNA targets. Bioinformation , 2009, 3 (8) : 364–366. DOI:10.6026/bioinformation |
| [43] | Busch A, Richter A S, Backofen R. IntaRNA: efficient prediction of bacterial sRNA targets incorporating target site accessibility and seed regions. Bioinformatics , 2008, 24 (24) : 2849–2856. DOI:10.1093/bioinformatics/btn544 |
| [44] | Tjaden B. TargetRNA: a tool for predicting targets of small RNA action in bacteria. Nucleic Acids Research , 2008, 36 (suppl 2) : W109–W113. |
| [45] | Krüger J, Rehmsmeier M. RNAhybrid: microRNA target prediction easy, fast and flexible. Nucleic Acids Research , 2006, 34 (suppl 2) : W451–W454. |
| [46] | Zhang Y, Sun S, Wu T, et al. Identifying Hfq-binding small RNA targets in Escherichia coli. Biochemical and Biophysical Research Communications , 2006, 343 (3) : 950–955. DOI:10.1016/j.bbrc.2006.02.196 |
| [47] | Tjaden B, Goodwin S S, Opdyke J A, et al. Target prediction for small, noncoding RNAs in bacteria. Nucleic Acids Research , 2006, 34 (9) : 2791–2802. DOI:10.1093/nar/gkl356 |
| [48] | Abu-Qatouseh L F, Chinni S V, Seggewiss J, et al. Identification of differentially expressed small non-protein-coding RNAs in Staphylococcus aureus displaying both the normal and the small-colony variant phenotype. Journal of molecular medicine (Berlin, Germany) , 2010, 88 (6) : 565–575. DOI:10.1007/s00109-010-0597-2 |
| [49] | Tjaden B, Saxena R M, Stolyar S, et al. Transcriptome analysis of Escherichia coli using high-density oligonucleotide probe arrays. Nucleic Acids Research , 2002, 30 (17) : 3732–3738. DOI:10.1093/nar/gkf505 |
| [50] | Dambach M, Irnov I, Winkler W C. Association of RNAs with Bacillus subtilis Hfq. PLoS One , 2013, 8 (2) : e55156. DOI:10.1371/journal.pone.0055156 |
| [51] | Irnov I, Sharma C M, Vogel J, et al. Identification of regulatory RNAs in Bacillus subtilis. Nucleic Acids Research , 2010, 38 (19) : 6637–6651. DOI:10.1093/nar/gkq454 |
| [52] | Wanner B, Wieder S, McSharry R. Use of bacteriophage transposon Mu d1 to determine the orientation for three proC-linked phosphate-starvation-inducible (psi) genes in Escherichia coli K-12. Journal of Bacteriology , 1981, 146 (1) : 93–101. |
| [53] | Urban J H, Vogel J. Translational control and target recognition by Escherichia coli small RNAs in vivo. Nucleic Acids Research , 2007, 35 (3) : 1018–1037. DOI:10.1093/nar/gkl1040 |
| [54] | Ramachandran R, Stevens A M. Proteomic analysis of the quorum-sensing regulon in Pantoea stewartii and identification of direct targets of EsaR. Applied and Environmental Microbiology , 2013, 79 (20) : 6244–6252. DOI:10.1128/AEM.01744-13 |
| [55] | Quigley B R, Zähner D, Hatkoff M, et al. Linkage of T3 and Cpa pilins in the Streptococcus pyogenes M3 pilus. Molecular Microbiology , 2009, 72 (6) : 1379–1394. DOI:10.1111/mmi.2009.72.issue-6 |
| [56] | Loh E, Dussurget O, Gripenland J, et al. A trans-acting riboswitch controls expression of the virulence regulator PrfA in Listeria monocytogenes. Cell , 2009, 139 (4) : 770–779. DOI:10.1016/j.cell.2009.08.046 |
| [57] | Oliva G, Sahr T, Buchrieser C. Small RNAs, 5' UTR elements and RNA-binding proteins in intracellular bacteria: impact on metabolism and virulence. FEMS Microbiology Reviews , 2015, 39 (3) : 331–349. DOI:10.1093/femsre/fuv022 |
| [58] | Xue T, Zhang X, Sun H, et al. ArtR, a novel sRNA of Staphylococcus aureus, regulates alpha-toxin expression by targeting the 5' UTR of sarT mRNA. Medical Microbiology and Immunology , 2014, 203 (1) : 1–12. DOI:10.1007/s00430-013-0307-0 |
| [59] | Geisinger E, Adhikari R P, Jin R, et al. Inhibition of rot translation by RNAIII, a key feature of agr function. Molecular Microbiology , 2006, 61 (4) : 1038–1048. DOI:10.1111/mmi.2006.61.issue-4 |
| [60] | Ohtani K, Bhowmik S K, Hayashi H, et al. Identification of a novel locus that regulates expression of toxin genes in Clostridium perfringens. FEMS Microbiology Letters , 2002, 209 (1) : 113–118. DOI:10.1111/fml.2002.209.issue-1 |
| [61] | Queck S Y, Jameson-Lee M, Villaruz A E, et al. RNAIII-independent target gene control by the agr quorum-sensing system: insight into the evolution of virulence regulation in Staphylococcus aureus. Molecular Cell , 2008, 32 (1) : 150–158. DOI:10.1016/j.molcel.2008.08.005 |
| [62] | Ikuo M, Nagano G, Saito Y, et al. Inhibition of exotoxin production by mobile genetic element SCCmec-encoded psm-mec RNA is conserved in staphylococcal species. PLoS One , 2014, 9 (6) : e100260. DOI:10.1371/journal.pone.0100260 |
| [63] | Kaito C, Saito Y, Ikuo M, et al. Mobile genetic element SCCmec-encoded psm-mec RNA suppresses translation of agrA and attenuates MRSA virulence. PLoS Pathogens , 2013, 9 (4) : e1003269. DOI:10.1371/journal.ppat.1003269 |
| [64] | Semsey S, Andersson A M, Krishna S, et al. Genetic regulation of fluxes: iron homeostasis of Escherichia coli. Nucleic Acids Research , 2006, 34 (17) : 4960–4967. DOI:10.1093/nar/gkl627 |
| [65] | Gaballa A, Antelmann H, Aguilar C, et al. The Bacillus subtilis iron-sparing response is mediated by a Fur-regulated small RNA and three small, basic proteins. Proceedings of the National Academy of Sciences , 2008, 105 (33) : 11927–11932. DOI:10.1073/pnas.0711752105 |
| [66] | Smaldone G T, Revelles O, Gaballa A, et al. A global investigation of the Bacillus subtilis iron-sparing response identifies major changes in metabolism. J Bacteriol , 2012, 194 (10) : 2594–2605. DOI:10.1128/JB.05990-11 |
| [67] | Smaldone G T, Antelmann H, Gaballa A, et al. The FsrA sRNA and FbpB protein mediate the iron-dependent induction of the Bacillus subtilis lutABC iron-sulfur-containing oxidases. Journal of Bacteriology , 2012, 194 (10) : 2586–2593. DOI:10.1128/JB.05567-11 |
| [68] | Oglesby-Sherrouse A G, Murphy E R. Iron-responsive bacterial small RNAs: variations on a theme. Metallomics: Integrated Biometal Science , 2013, 5 (4) : 276–286. DOI:10.1039/c3mt20224k |
| [69] | Cotter P D, Emerson N, Gahan C G, et al. Identification and disruption of lisRK, a genetic locus encoding a two-component signal transduction system involved in stress tolerance and virulence in Listeria monocytogenes. Journal of Bacteriology , 1999, 181 (21) : 6840–6843. |
| [70] | Roberts S A, Scott J R. RivR and the small RNA RivX: the missing links between the CovR regulatory cascade and the Mga regulon. Molecular Microbiology , 2007, 66 (6) : 1506–1522. |
| [71] | Ohtani K, Hirakawa H, Tashiro K, et al. Identification of a two-component VirR/VirS regulon in Clostridium perfringens. Anaerobe , 2010, 16 (3) : 258–264. DOI:10.1016/j.anaerobe.2009.10.003 |
| [72] | Obana N, Nakamura K. A novel toxin regulator, the CPE1446-CPE1447 protein heteromeric complex, controls toxin genes in Clostridium perfringens. Journal of Bacteriology , 2011, 193 (17) : 4417–4424. DOI:10.1128/JB.00262-11 |
| [73] | Marx P, Nuhn M, Kovács M, et al. Identification of genes for small non-coding RNAs that belong to the regulon of the two-component regulatory system CiaRH in Streptococcus. BMC Genomics , 2010, 11 (1) : 661. DOI:10.1186/1471-2164-11-661 |
| [74] | Valentin-Hansen P, Eriksen M, Udesen C. MicroReview: The bacterial Sm-like protein Hfq: a key player in RNA transactions. Molecular Microbiology , 2004, 51 (6) : 1525–1533. DOI:10.1111/j.1365-2958.2003.03935.x |
| [75] | Vogel J, Luisi B F. Hfq and its constellation of RNA. Nature Reviews Microbiology , 2011, 9 (8) : 578–589. DOI:10.1038/nrmicro2615 |
| [76] | Faner M, Feig A. Identifying and characterizing Hfq-RNA interactions. Methods , 2013, 63 (2) : 144–159. DOI:10.1016/j.ymeth.2013.04.023 |
| [77] | Christiansen J K, Nielsen J S, Ebersbach T, et al. Identification of small Hfq-binding RNAs in Listeria monocytogenes. RNA , 2006, 12 (7) : 1383–1396. DOI:10.1261/rna.49706 |
| [78] | Nielsen J S, Lei L K, Ebersbach T, et al. Defining a role for Hfq in Gram-positive bacteria: evidence for Hfq-dependent antisense regulation in Listeria monocytogenes. Nucleic Acids Research , 2009, 38 (3) : 907–919. |
| [79] | Nielsen J S, Larsen M H, Lillebaek E M, et al. A small RNA controls expression of the chitinase ChiA in Listeria monocytogenes. PLoS One , 2011, 6 (4) : e19019. DOI:10.1371/journal.pone.0019019 |
| [80] | Bohn C, Rigoulay C, Bouloc P. No detectable effect of RNA-binding protein Hfq absence in Staphylococcus Aureus. BMC Microbiology , 2007, 7 (1) : 10. DOI:10.1186/1471-2180-7-10 |
| [81] | Rochat T, Bouloc P, Yang Q, et al. Lack of interchangeability of Hfq-like proteins. Biochimie , 2012, 94 (7) : 1554–1559. DOI:10.1016/j.biochi.2012.01.016 |
 2016, Vol. 36
2016, Vol. 36




