文章信息
- 杜红燕, 李天明, 刘金雷, 冯惠勇.
- DU Hong-yan, LI Tian-ming, LIU Jin-lei, FENG Hui-yong.
- 构建尿嘧啶磷酸核糖转移酶基因缺失菌株实现Gluconobacter suboxydans基因组无痕修饰
- Construct the Uracil Phosphoribosyl Transferase Gene Mutant Strain in Gluconobacter suboxydans for Seamless Genome Editing
- 中国生物工程杂志, 2016, 36(7): 64-71
- CHINA BIOTECHNOLOGY, 2016, 36(7): 64-71
- http://dx.doi.org/DOI:10.13523/j.cb.20160710
-
文章历史
- 收稿日期: 2016-01-18
- 修回日期: 2016-03-07
Gluconobacter suboxydans是一种严格好氧的革兰氏阴性菌[1],具有丰富的周质空间氧化还原酶类,可高效不完全氧化多种糖类和醇类等多羟基化合物,生成相应的醛类、酮类和有机酸类等产物释放到培养基中。Gluconobacter suboxydans对底物的广泛性及不完全氧化底物的特性使它成为应用于微生物发酵工业生产的理想菌[2]。例如,用于D-山梨醇生产L-山梨糖,后者是维生素C生产中重要的中间底物[3];1-氨基-D-山梨醇生产6-氨基-L-山梨糖,后者是糖尿病药物米格列醇的前体物质[4];还用于生产D-葡萄糖酸、二羟基丙酮(DHA)等重要的工业产品[5]。
利用代谢工程技术对菌种进行改造是工业育种的重要手段,基因敲除、基因敲入及点突变修饰等基因组改造技术中,大多采用抗性筛选标记。但是抗性本身并不是宿主菌生长所必需,抗性基因的引入存在诸多缺点和隐患[6]。例如,被改造菌株可被使用的抗生素数量有限,改造宿主菌会因抗性标记的叠加使用受到限制;多抗性选择压力可能会影响目的菌的正常生长,等等[7-9]。
为了获得高产优良菌种,迫切需要优化改进目前的基因组改造技术。Gluconobacter suboxydans中的upp基因可编码催化尿嘧啶生成尿嘧啶磷酸核糖转移酶(uracil phosphoribosyl transferase,UPRT), 使细胞可利用胞外尿嘧啶。5-氟尿嘧啶(5-fluorouracil, 5-FU)是胸腺嘧啶类似物,其也能作为UPRT的底物被转化成胸苷酸合成酶的强烈抑制剂,即5-氟单磷酸脱氧尿嘧啶(5-F-dUMP),5-F-dUMP的存在会导致宿主细胞死亡[10-11]。本研究以对5-氟尿嘧啶敏感的upp基因作为负向筛选标记,并与四环素抗性标记基因结合,构建用于upp敲除的自杀质粒pMD18-Jqupp,电转化入实验室保存的工业野生型菌株Gluconobacter suboxydans J12中,利用两次同源重组策略[12-14],敲入时四环素抗性标记正向筛选、抗性去除时upp负向标记筛选。对野生型菌株进行染色体修饰,利用upp编码基因对5-FU的生长敏感性筛选出缺失upp基因的突变株G.suboxydans-upp,获得了可进行无痕基因组修饰改造的底盘细胞。
1 材料与方法 1.1 材料 1.1.1 试剂DNA聚合酶购自北京全式金生物技术有限公司;PCR引物由上海英俊生物技术有限公司合成;T4 DNA连接酶、DNA分子质量标准购自大连宝生物工程公司(TaKaRa);质粒提取试剂盒、DNA回收试剂盒购自天根生化科技(北京)有限公司;其他试剂均为分析级产品,购自生工生物(上海)股份有限公司。
1.1.2 仪器与设备PCR扩增仪,Analytikjena PowerCycle;全自动凝胶成像仪,Bio-Rad Molecular Imager Gel DOC XR; 台式高速离心机,Eppendorf Mini Spin;高速冷冻离心机;电转仪,Bio-Rad Gene Pulser XcellTM。
1.1.3 菌株与质粒本实验研究所用菌株、质粒如表 1所示。
| Strain and plasmid | Properties | Source |
| E.coli DH5α | F-, φ80dlacZ△M15, △(lacZYA-argF)U169, deoR, recA1, endA1, hsdR17(rk-, mk+), phoA, supE44, λ-, thi-1, gyrA96, relA1 | TransGen Biotech |
| Gluconobacter suboxydans J12 | Wild stain producing sorbitol | Stored in lab |
| pBBR1MCS-5 | Gmr broad host vector | Stored in lab |
| pMD18-T | Cloning vector with multiple cloning sites; Ampr | TransGen Biotech |
| pMD18-Jqupp | Cloning ter cassette into pMD18-t | This study |
| G.suboxydans-ter | Cloning ter cassette into G.suboxydans | This study |
| G.suboxydans-upp | Knockout upp from G.suboxydans | This study |
| pBBR1MCS-3 | Tmr broad host vector | Stored in lab |
| pBBR1MCS-5-upp | Cloning SldAB promoter and upp gene into pBBR1MCS-5 | This study |
| G.suboxydans-uppHB | Express upp in G.suboxydans-upp | This study |
1.1.4 培养基
LB培养基:1%蛋白胨,0.5%酵母提取物,1%氯化钠,需要时加入氨苄霉素至终浓度100μg/ml,庆大霉素至终浓度70μg/ml。
G.suboxydans J12培养基:1%山梨醇,0.5%玉米浆,0.2%(NH4)2SO4, 0.05% KH2PO4, 0.05% NH4H2PO4, 0.025% MgSO4, 0.01%CaCO3, pH调至6.5。四环素至终浓度35μg/ml。
1.2 方法 1.2.1 G.suboxydansJ12对5-氟尿嘧啶的敏感性研究同时在液体培养基和固体培养基中测定G.suboxydans J12对5-FU的敏感性。固体平板选用0mg/ml、0.25mg/ml、0.5mg/ml、0.75mg/ml、1mg/ml 5个浓度梯度,30℃恒温培养,48h后观察记录菌的生长情况。液体摇瓶中5-FU浓度也选用0mg/ml、0.25mg/ml、0.5mg/ml、0.75mg/ml、1mg/ml 5个浓度梯度,种子培养基OD600等于2时接种于5-FU浓度梯度摇瓶中,30℃、180r/min振荡培养,间隔6h取样测定记录菌液浓度。
1.2.2 upp基因敲除打靶质粒的构建以G.suboxydans J12为模板,通过上游引物JUPPSF、JUPPSR和下游引物JUPPXF、JUPPXR扩增出upp基因的上游同源臂uppsy基因片段和下游同源臂uppxy基因片段;以pBBR1MCS-3[15]质粒为模板,由引物JTEUPF和JTEUPR扩增出四环素抗性基因片段ter。通过融合PCR将uppsy、uppxy、ter搭接,获得打靶片段JQUPP。纯化后的JQUPP片段和pMD18-T载体进行T克隆,得到自杀质粒pMD18-Jqupp。所用引物如表 2所示。
| Primers | Sequences(5′~3′) |
| JUPPSF | TCTCATAATGCACGTAAGCTTGAGGAG |
| JUPPSR | AGTGCCATACTGCACAATCCGTCTGCAA |
| JUPPXF | CGACCATTACCGGCAGTCTACCACCAAG |
| JUPPXR | CGATTTGGCTAAACGAGCCTTCAG |
| JTEUPF | CCGTCAGTGTTGGAGTAGTCAGTA |
| JTEUPR | AGGCTAAGGGATATCCTGCTATGCGT |
| SLDHPF | CGGGATCCACAAATCATACTGGCGGCG |
| SLDHPR | ATAATATACCAAATTGTGGCATAACCGAATGCCTCCCAAACTG |
| UPPF | GTACTCTCAGAGGAGCCGTCTGATGAGCATTCCATCCCGGC |
| UPPR | TCACTTTTGATCGGGCAGGGC |
| JQUPPYZF | GCCGCCCTATACCTTGTCTGCC |
| JQUPPYZR | GGACAGAGATGCCACAATCCTTGG |
| JQUPPDYZF | GAGTTCCAGCAACGATGCCACAATT |
| JQUPPDYZR | GTGTCCAAGCTTAAGCGATGTCCTGT |
| GENF | CGGGATCCGACGCACACCGTGGAAAC |
| GENR | ATAATATACCAAATTGTGGCATCGTTGCTGCTCCATAACATC |
1.2.3 转化G.suboxydans J12野生型敲除upp基因
G.suboxydans J12染色体上upp基因的敲除是通过两步同源重组策略实现的。G.suboxydans J12染色体上的upp基因被敲除,获得的重组菌株无抗生素标记基因。将获得的自杀质粒pMD18-Jqupp电转化至野生型G.suboxydans J12感受态细胞中,第一步同源重组中四环素抗性基因整合到G.suboxydans J12染色中,四环素抗性平板筛选并对转化子进行PCR验证,获得突变株G.suboxydans-ter。突变株G.suboxydans-ter在G.suboxydans J12培养基中无抗性培养使其发生第二次重组,G.suboxydans-ter染色体中upp基因和四环素抗性基因被删除,缺失upp基因的重组菌株不能利用5-FU产生5-F-αUMP,故不能对细胞产生毒害,重组菌株可在含5-FU的培养基中生长。四环素基因被删除则重组菌株在四环素抗性平板上不能生长。将G.suboxydans-ter在G.suboxydans J12液体摇瓶培养基中30℃、180r/min孵育48h后进行梯度稀释并涂布在5-FU平板上初筛,30℃培养,将长出的转化子同时影印在5-FU平板和四环素抗性平板上,四环素平板上不生长而5-FU平板上生长的即为二次重组的转化子。构建策略见图 1。
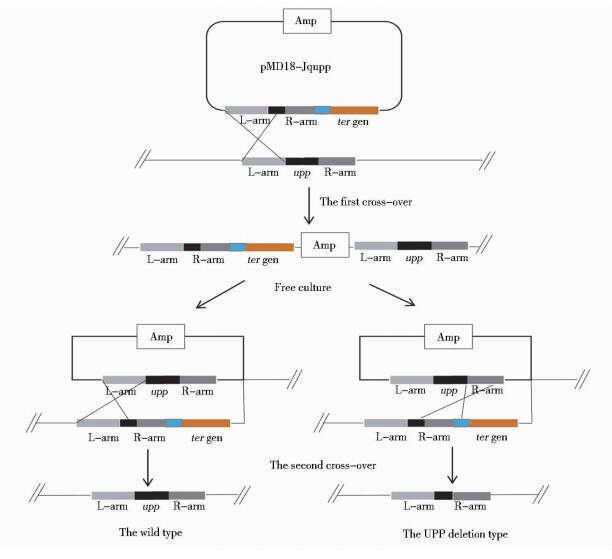
|
| 图 1 同源双交换原理示意图 Figure 1 Homologous crossing-over in this study |
依据Gluconobacter suboxydans 621H[16]基因组信息设计Gluconobacter suboxydans中山梨醇脱氢酶(SldAB)启动子引物SLDHPF、SLDHPR和upp基因引物UPPF、UPPR,以G.suboxydans J12为模版,PCR扩增出SldAB的启动子片段和upp基因片段,两个片段融合后纯化,并和pBBR1MCS-5[15]质粒经HindⅢ和SalⅠ双酶切,T4连接酶16℃过夜连接,转化入大肠杆菌DH5α,用庆大LB平板筛选转化子,经PCR验证庆大抗性基因,获得阳性转化子pBBR1MCS-5-upp。将穿梭表达质粒pBBR1MCS-5-upp电转化入缺失upp基因的突变株G.suboxydans-upp中,庆大抗性平板筛选出阳性转化子,PCR验证获得upp基因回补菌株G.suboxydans-uppHB,反向验证G.suboxydans-upp菌株的upp负向筛选功能。
2 结果与讨论 2.1 5-FU对G.suboxydans J12抑制浓度的确定5-FU对野生型G.suboxydans J12有一定毒性,会抑制其生长,当5-FU浓度达到一定时致死;而upp基因缺失后菌体不能利用5-FU产生毒素,可在含5-FU的培养基中生长,从而使突变株得以筛选。为了确定抑菌浓度,选用0mg/ml、0.25mg/ml、0.5mg/ml、0.75mg/ml、1mg/ml 5个浓度梯度, 将G.suboxydans J12野生型菌株固体平板30℃恒温培养48h,记录生长情况(图 2),同时对野生型菌株进行液体培养,5-FU浓度梯度同固体平板浓度,30℃、180r/min记录菌体生长OD值(图 3)。

|
| 图 2 野生型菌株在梯度浓度5-FU平板上的生长情况 Figure 2 Plate culture of wildtype in the different 5-FU concentration |
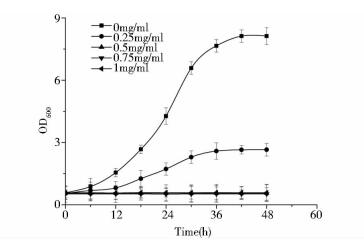
|
| 图 3 野生型菌株在梯度浓度5-FU摇瓶中的生长曲线 Figure 3 Growth curve of wildtype in the different 5-FU concentration |
由图 2可知野生型菌株在0.25mg/ml的平板上有少量菌体生长,从5-FU浓度为0.5mg/ml的平板上开始无菌体生长;从图 3中的菌株生长曲线可知野生型菌株在5-FU浓度为0mg/ml的摇瓶中正常生长,在0.25mg/ml的摇瓶中生长缓慢,而从0.5mg/ml开始,菌体没有生长趋势。故本实验设定5-FU工作浓度为0.5mg/ml。
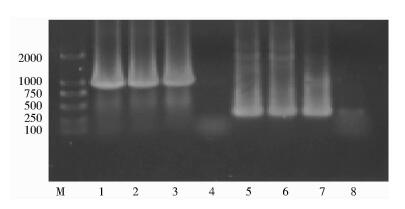
2.2 upp基因敲除打靶质粒的构建根据G.suboxydans J12染色体中upp基因序列设计引物,PCR扩增出upp基因的上同源臂与下同源臂,从pBBR1MCS-5质粒中扩增出四环素抗性标记基因(图 4),条带大小符合预期。将三个片段融合(原理见图 1),纯化T克隆到pMD18载体上,对阳性转化子进行PCR验证(图 5),分别得到三段基因,大小与阳性对照一致。选取正确的转化子pMD18-Jqupp,提取质粒。
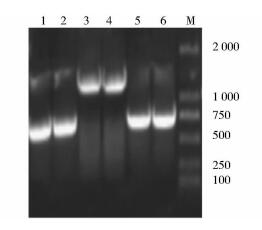
|
| 图 4 PCR扩增上下同源臂、四环素抗性基因 Figure 4 PCR verification of uppsy, uppxy, ter M: DNA maker; 1, 2:Up homologous arm of upp; 3, 4: Tetracycline; 5, 6: Down homologous arm of up |
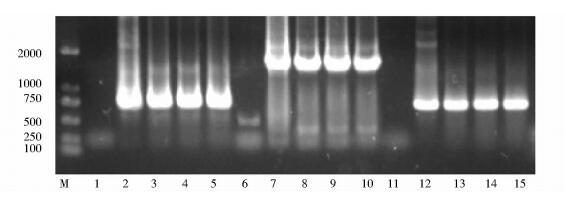
|
| 图 5 T克隆转化子PCR验证 Figure 5 PCR verification from T-clone M: DNA maker; 3-5:Up homologous arm of upp; 8-10: Tetracycline; 13-15: Down homologous arm of upp; 1, 6, 11:Negative control; 2, 7, 12:Positive control |
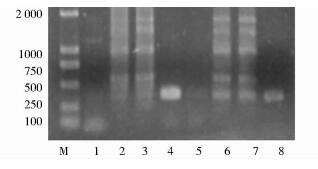
G.suboxydans J12染色体中的upp基因编码的尿嘧啶磷酸核糖转移酶可利用体外胸腺嘧啶类似物5-FU生成使宿主细胞死亡的5-dUMP。缺失upp基因突变株的筛选依据两步同源重组策略分为两次。第一次同源重组后四环素抗性基因整合到染色体中,故用四环素抗性平板筛选出重组菌株G.suboxydans-ter;第二次同源重组后突变株中upp基因和四环素抗性基因被删除,稀释涂布菌液于5-FU平板上初筛,长出的转化子在5-FU和四环素抗性平板上影印复筛,四环素平板上不生长而5-FU平板上生长的转化子进行PCR验证,upp敲除验证和定位验证, 得到重组菌株G.suboxydans-upp,PCR结果见图 6。
|
| 图 6 敲除upp基因PCR验证 Figure 6 PCR verification of upp gene in strains M:DNA Maker; 1:Negative control of upp; 2, 3:PCR amplified upp gene from G.suboxydans-upp strain1;4:Positive control of upp; 5:Negative control of orientation gene; 6, 7:PCR amplified orientation gene from G.suboxydans-upp strain; 8:Positive control of orientation gene |
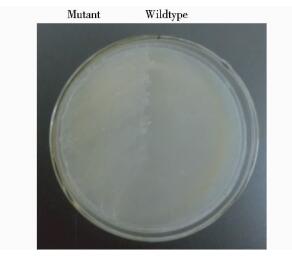
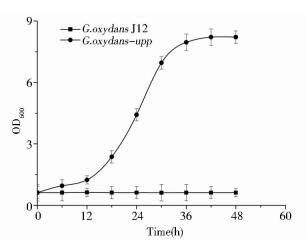
本实验确定野生型G.suboxydans J12菌株对5-FU的敏感浓度为0.5mg/ml,将获得upp基因缺失的突变株G.suboxydans-upp和野生型G.suboxydans J12在同一个含有0.5mg/ml的5-FU固体平板上画线培养,同时将两株菌分别接种于0.5mg/ml的5-FU液体摇瓶中培养,30℃、48h后结果见图 7、图 8。
|
| 图 7 野生型和突变株在5-FU平板上的生长情况 Figure 7 5-FU plate culture of wildtype and mutant |
|
| 图 8 野生型和突变株在5-FU摇瓶中的生长曲线 Figure 8 Growth curve of wildtype and mutant in the 5-FU fluid medium |
由图 7可以看到野生型G.suboxydans J12在0.5mg/ml的5-FU固体培养基上不能生长,突变株G.suboxydans-upp在0.5mg/ml的5-FU固体培养基上可以生长,证明突变株中由于upp基因缺失菌体不能利用体外胸腺嘧啶类似物5-FU产生毒害菌体的毒素5-dUMP,而使菌体可以得到生长。图 8所示液体摇瓶中野生型G.suboxydans J12和突变株G.suboxydans-upp的生长曲线亦说明突变株中upp基因已缺失。
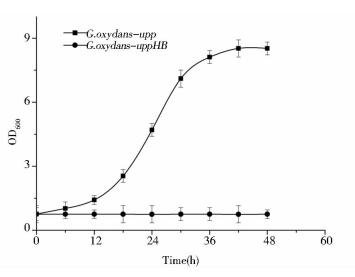
2.5 通过回补upp基因验证upp基因负向筛选功能穿梭表达质粒pBBR1MCS-5-upp电转化入G.suboxydans-upp底盘细胞后在山梨醇脱氢酶启动子作用下进行自我复制并表达upp基因。经upp基因回补的G.suboxydans-upp底盘细胞可利用培养基中的5-FU,生成5-dUMF而导致细胞死亡,由此反向验证了upp基因的负向筛选功能。G.suboxydans-uppHB菌株的验证见图 9,在5-FU平板和液体摇瓶中的结果见图 10、图 11。
|
| 图 9 G.suboxydans-uppHB PCR验证 Figure 9 PCR verification of gentamycin gene and promoter M:DNAmaker; 1:Positive control of gentamycin gene; 2, 3:Gentamycin gene from G.suboxydans-uppHB; 4:Negative control of gentamycin gene; 5:Positive control of promoter; 6, 7:Promoter from G.suboxydans-uppHB; 8:Negative control of promoter |

|
| 图 10 G.suboxydans-uppHB和G.suboxydans-upp在5-FU平板上的生长情况 Figure 10 5-FU plate culture of G.suboxydans-uppHB and G.suboxydans-upp |
|
| 图 11 G.suboxydans-uppHB和G.suboxydans-upp在5-FU摇瓶中的生长曲线 Figure 11 Growth curve of G.suboxydans-uppHB and G.suboxydans-upp in the 5-FU fluid medium |
图 10、图 11中upp基因回补的突变株G.suboxydans-uppHB在5-FU平板上不生长,在摇瓶中也没有生长趋势,与野生型的表型有显著性差异,证明upp基因可以作为负向筛选标记基因使用。
3 结论目前一些模式菌株的染色体无痕修饰改造的技术方法已经非常成熟,并在菌种的遗传改造及合成生物学研究方面取得了令人瞩目的成就。例如,E.coli利用Red重组系统[17-18]、酵母利用Cre重组酶系统[19-20]及谷氨酸棒杆菌利用sacB反向筛选标记等[21-23],都成功实现了对基因组高效准确的无痕修饰改造,而Gluconobacter suboxydans在这方面的研究还基本处于空白。
本研究利用Gluconobacter suboxydans染色体中upp基因编码尿嘧啶生成尿嘧啶磷酸核糖转移酶可利用体外胸腺嘧啶类似物5-FU产生对菌体有毒害作用的5-dUMP的原理,将upp基因作为负向筛选标记,与四环素抗性基因结合作用,构建Gluconobacter suboxydans无痕修饰改造的底盘工程菌。自杀质粒pMD18-Jqupp电转化至野生型菌株Gluconobacter suboxydans J12中,通过四环素抗性筛选得到重组菌株Gluconobacter suboxydans-ter,并将其在丰富无抗性培养基中自由培养发生第二次重组,四环素和5-FU平板同时筛选获得四环素抗性基因和upp基因被删除的期望菌株Gluconobacter suboxydans-upp。构建含山梨醇启动子和upp基因的穿梭表达质粒pBBR1MCS-5-upp,并电转化入Gluconobacter suboxydans-upp中,得到upp基因回补的突变株Gluconobacter suboxydans-uppHB,验证了upp基因可作为反向筛选标记基因的功能性。本实验所得突变株中无抗性基因残留,为后续Gluconobacter suboxydans的遗传操作提供了upp基因作为负向筛选标记的无痕修饰底盘细胞。
| [1] | De Ley J, Swings J, Gossele F. The Genus Gluconobacter.In:Krieg N R, Holt J G.Bergey's Manual of Systematic Bacteriology. Baltimore: Williams & Wilkins Co, 1984 : 267 -278. |
| [2] | Deppenmeier U, Hoffmeister M, Prust C. Biochemistry and biotechnological applications of Gluconobacter strains. Applied and Molecular Biotechnology , 2002, 60 (3) : 233–242. DOI:10.1007/s00253-002-1114-5 |
| [3] | Reichstein T, Grü ssner A. Eine ergiebige synthese der 1-ascorbinsäure (C-vitamin). Helvetica Chimica Acta , 1934, 17 (1) : 311–328. DOI:10.1002/hlca.19340170136 |
| [4] | Michael S. Regioselective oxidation of aminosorbitol with Gluconobacter oxydans, key reaction in the industrial synthesis of 1-deoxynojirimycin syntheis. Journal of Psychiatry & Neuroscience Jpn , 2004, 29 (5) : 364–382. |
| [5] | Adachi O, Moonmangmee D, Toyama H, et al. New developments in oxidative fermentation. Applied and Molecular Biotechnology , 2003, 60 (6) : 643–653. DOI:10.1007/s00253-002-1155-9 |
| [6] | Liu P, Jenkins N A, Copeland N G. A highly efficient recombineering-based method for generating conditional knockout mutations. Genome Research , 2003, 13 (3) : 476–484. DOI:10.1101/gr.749203 |
| [7] | 杨奇. 大肠杆菌DH5αupp基因的敲除及其应用研究. 南京: 南京理工大学, 环境与生物工程学院, 2013 . Yang Q. Knockout of the upp gene in Escherichia coli DH5αand its application research. Nanjing: Nanjing University of Science & Technology, Environmental and Biological Engineering, 2013 . |
| [8] | Bailey J E. Toward a science of metabolic engineering. Science , 1991, 252 (5013) : 1668–1675. DOI:10.1126/science.2047876 |
| [9] | 胡逢雪, 丁锐, 崔震海, 等. 大肠杆菌基因无痕敲除技术及策略. 生物技术通讯 , 2013, 24 (4) : 552–557. Hu F X, Ding R, Cui Z H, et al. Approaches and strategies of gene scarless knockout in the Escherichia coli genome. Letters In Biotechnology , 2013, 24 (4) : 552–557. |
| [10] | Hasegawa N, Abeil M K, Yokoyama K, et al. Cyclophosphamide enhances antitumor efficacy of oncolytic adenovirus expressing uracil phosphoribosyltransferase (UPRT) in immunocompetent Syrian hamsters. International Journal of Cancer , 2013, 133 (6) : 1479–1489. DOI:10.1002/ijc.v133.6 |
| [11] | Andersen P S, Smith J M, Mygind B. Characterization of the upp gene encoding uracil phosphoribosyltransferase of Escherichia coli K12. Federation of European Biochemical Societies , 1992, 204 (1) : 51–56. |
| [12] | Tan Z G, Zhu X N, Chen J, et al. Activating phosphoenolpyruvate carboxylase and phosphoenolpyruvate carboxykinase in combination for improvement of succinate production. Applied and Environmental Microbiology , 2013, 79 (16) : 4838–4844. DOI:10.1128/AEM.00826-13 |
| [13] | Jantama K, Zhang X L, Moore J C, et al. Eliminating side products and increasing succinate yields in engineered strains of Escherichia coli C. Biotechnology and Bioengineering , 2008, 101 (5) : 881–893. DOI:10.1002/bit.v101:5 |
| [14] | Zhang X L, Jantama K, Moore J C, et al. Production of L-alanine by metabolically engineered Escherichia coli. Applied Genetics And Molecular Biotechnology , 2007, 77 (2) : 355–366. |
| [15] | Michael E K, Philip H E, Steve D H, et al. Four new derivatives of the broad-host-range cloning vector pBBR1MCS, carring different antibiotic-resistance cassettes. Gene , 1995, 166 (1) : 175–176. DOI:10.1016/0378-1119(95)00584-1 |
| [16] | Prust C, Hoffmeister M, Liesegang H, et al. Complete genome sequence of the acetic acid bacterium Gluconobacter oxydans. Nature Biotechnology , 2005, 23 (2) : 195–200. DOI:10.1038/nbt1062 |
| [17] | 余华, 熊浚智, 何晓梅, 等. 采用Red重组系统敲除铜绿假单胞菌弹性蛋白酶基因. 中国人兽共患病学报 , 2013, 2 : 129–132. Yu H, Xiong J Z, He X M, et al. Generation of a Pseudomonas aeruginosa elastase gene targeted deletion mutant by Red recombination system. Chinese Journal of Zoonoses , 2013, 2 : 129–132. |
| [18] | Lim J H, Seo S W, Kim S Y, et al. Refactoring redox cofactor regeneration for high-yield biocatalysis of glucose to butyric acid in Escherichia coli. Bioresource Technology , 2013, 135 : 568–573. DOI:10.1016/j.biortech.2012.09.091 |
| [19] | Koresawa Y K, Miyagawa S J, Ikawa M H, et al. Synthesis of a new cre recombinase gene based on optimal codon usage for mammalian system. The Journal of Biochemistry , 2000, 127 (3) : 367–372. DOI:10.1093/oxfordjournals.jbchem.a022617 |
| [20] | Zhu D L, Zhao K, Xu H J, et al. Construction of thyA deficient Lactococcus lactis using the Cre-loxP recombination system. Annals of Microbiology , 2015, 66 (3) : 1659–1665. |
| [21] | Jager W, Schafer A, Puhler A, et al. Expression of the Bacillus subtilis SacB gene leads to sucrose sensitivity in the gram-positive bacterium Corynebacterium glutamicum but not in Streptomyces lividans. Journal of Bacteriology , 1992, 174 (16) : 5462–5465. |
| [22] | Hu F, Jiang X, Zhang J J, et al. Construction of an engineered strain capable of degrading two isomeric nitrophenols via a sacB-and gfp-based markerless integration system. Applied and Molecular Biotechnology , 2014, 98 (10) : 4749–4756. DOI:10.1007/s00253-014-5567-0 |
| [23] | Tan Y Z, Xu D Q, Li Y, et al. Construction of a novel sacB-based system for marker-free gene deletion in Corynebacterium glutamicum. Plasmid , 2012, 67 (1) : 44–52. DOI:10.1016/j.plasmid.2011.11.001 |
 2016, Vol. 36
2016, Vol. 36




