文章信息
- 陈娟, 杨慧林, 黄运红, 龙中儿.
- CHEN Juan, YANG Hui-lin, HUANG Yun-hong, LONG Zhong-er.
- 炭样小单孢菌中抗生素生物合成相关蛋白的筛选
- Screening of the Protein Related to Antibiotics Biosynthesis in Micromonospora carbonacea JXNU-1
- 中国生物工程杂志, 2016, 36(7): 55-63
- CHINA BIOTECHNOLOGY, 2016, 36(7): 55-63
- http://dx.doi.org/DOI:10.13523/j.cb.20160709
-
文章历史
- 收稿日期: 2016-01-27
- 修回日期: 2016-04-04
核苷类抗生素是一类主要由微生物产生的,具有生物活性的次级代谢产物,通常是由核苷或核苷酸经过一系列后修饰所形成的结构比较精细复杂的分子,具有广谱生物活性,如抗真菌、抗细菌、杀虫、除草、抗肿瘤、抗病毒及免疫抑制和激活作用等[1]。生物活性与结构密切相关,结构简单的核苷类似物通常抑制核酸的生物合成,结构复杂的核苷类似物则对蛋白质或多糖的生物合成有抑制作用,因此核苷类抗生素自发现以来便得到广泛应用[2]。
实验室在前期研究中获得一株具有广谱抗菌活性的炭样小单孢菌JXNU-1(Micromonospora carbonacea JXNU-1),其发酵产物对金黄色葡萄球菌、苏云金芽孢杆菌、枯草芽孢杆菌等革兰氏阳性菌,以及伤寒沙门氏菌、福氏志贺氏菌、铜绿假单孢菌等革兰氏阴性菌都有很强的抗菌活性[3-4]。朱跃进等[5]系统研究了该菌发酵产抗生素的工艺过程,确定了该菌发酵产抗生素的影响因素及其作用规律,建立了从该菌发酵液中分离纯化抗生素的方法,并从中获得了层析纯的单组分、粉末状抗生素产品,该抗生素经初步分析确定为一核苷类抗生素JX[6]。江云等[7]对炭样小单孢菌JXNU-1的全基因组序列进行了测定和注释,相关序列已提交至GenBank数据库(JXSX00000000)。但该抗生素的生物合成相关基因和蛋白质并不清楚。
本研究通过iTRAQ定量蛋白质组学技术对JXNU-1菌体生长期(36h)和产物合成期(108h)的差异蛋白进行鉴定和功能分析,筛选出与抗生素生物合成相关蛋白,为阐明抗生素JX的生物合成与分子调控机制奠定基础,为进一步提高抗生素JX产量和新型药物的开发提供理论依据。
1 材料与方法 1.1 菌株及培养基炭样小单孢菌JXNU-1(M.carbonacea JXNU-1)由本实验室从土壤中分离得到。培养基:高氏I号培养基、种子培养基和发酵培养基[6](实验过程中所用试剂均为生化试剂或分析纯化学试剂,下同)。
1.2 炭样小单孢菌培养M.carbonacea JXNU-1发酵培养[6],分别于36h和108h离心收集菌体(用于蛋白质组学分析)和上清液(用于抗菌活性测定),菌体用PBS缓冲液洗涤3次。
1.3 发酵液的抗菌活性测定采用杯碟法测定上清液的抗菌活性,以金黄色葡萄球菌(Staphylococcus aureas)为靶菌,抑菌圈直径大小表示抗菌活性的强弱。
1.4 菌体蛋白质提取称取适量菌体样品,加入1ml裂解液悬浮,超声破碎菌体。95℃水浴5min抽提蛋白质后于4℃下12 000r/min离心30min收集上清液。加入终浓度为10mmol/L的DTT在56℃下反应30min后加入终浓度为20mmol/L的IAA室温下避光反应30min。加入适量预冷的丙酮(丙酮:样品体积比=5∶1),于-20℃沉淀2h,然后在4℃下12 000r/min离心20min收集沉淀,加入含1mol/L尿素的TEAB溶解液20μl混悬,充分溶解样品。
1.5 炭样小单孢菌蛋白质组学测定及分析按照质量比1∶50(酶∶蛋白质)的比例加入胰蛋白酶,37℃下酶解15h。胰蛋白酶消化后,用真空离心泵抽干肽段,用0.5mol/L TEAB复溶肽段进行iTRAQ标记,每一组肽段用不同的iTRAQ标签标记,室温培养2h,将标记后的各组肽段混合。先进行高pH RPLC第一维分离,复溶后进行第二维分析,再采用Triple TOF 5600质谱仪器进行液相色谱-质谱检测。质谱扫描完毕,得到质谱信号图,经Data Analysis 4.0软件自动分析标峰得到mgf文件,利用Mascot软件搜索M.carbonacea蛋白数据库(本实验室构建)进行蛋白质鉴定和差异表达蛋白分析(FDR<0.05、FC<0.8或FC>1.2)。利用Blast2GO软件对鉴定出的所有蛋白质进行GO注释分析,并对差异表达蛋白进行GO富集、KEGG pathway等分析。此部分工作委托上海美吉生物医药科技有限公司完成。
2 结果与分析 2.1 发酵液的抗菌活性测定分别选取培养36h和108h的M.carbonacea JXNU-1发酵液测定抗菌活性(图 1),发现培养36h的菌体还未分泌抗生素(未见抑菌圈),而培养108h的发酵液具有明显的抑菌圈(直径约为27.60mm)。因此,选取上述两个阶段的菌体用于后续定量蛋白质组学分析抗生素生物合成相关蛋白是可行的。

|
| 图 1 发酵液的抗菌活性 Figure 1 The antimicrobial activity of broth |
取大约20μg蛋白质样本进行SDS-PAGE电泳检测,如图 2所示,两组样品之间平行度较好,相应的蛋白质条带清晰,初步表明蛋白质提取效果理想,样品纯度适合进行下一步实验。使用iTRAQ技术进行蛋白质相对定量,所用质谱仪器是Triple TOF 5600。在本次质谱实验中共得到谱图98 714张,通过Mascot软件进行分析后,匹配到的谱图数量是64 420张,共鉴定到23 621个肽段,匹配到2 390个蛋白质。

|
| 图 2 炭样小单孢菌JXNU-1蛋白样品SDS-PAGE电泳分析 Figure 2 SDS-PAGE analysis of proteins extracted from M.carbonacea JXNU-1 |
为了解M.carbonacea JXNU-1蛋白质组的功能,将鉴定到的所有蛋白质进行GO注释分析,共有1 502个蛋白质成功获得GO数据库的注释。通过GO二级分类统计结果表明,注释到的1 502个蛋白质被归类到40个主要的功能分组。其中,生物过程(biological process)相关蛋白主要集中于细胞过程(cellular process)、单有机体过程(single-organism process)、代谢过程(metabolic process);细胞组分(cellular component)相关蛋白主要集中于细胞器(organelle)、膜结构(membrane);分子功能(molecular function)相关蛋白主要是结合(binding)蛋白和催化(catalytic)蛋白(图 3)。
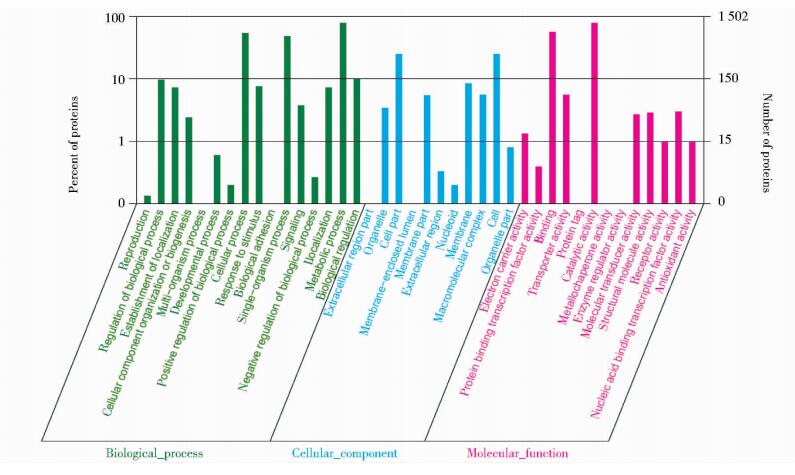
|
| 图 3 菌体蛋白的GO注释 Figure 3 GO annotation of bacterial proteins |
通过COG数据库注释分析,被鉴定到的蛋白质功能主要涉及到氨基酸运输和代谢(amino acid transport and metabolism),翻译、核糖体结构和生物转化(translation,ribosomal structure and biogenesis),能源生产和转换(energy production and conversion),碳水化合物的运输和代谢(carbohydrate transport and metabolism),转录(transcription),一般功能预测(General function prediction only),等等(图 4)。
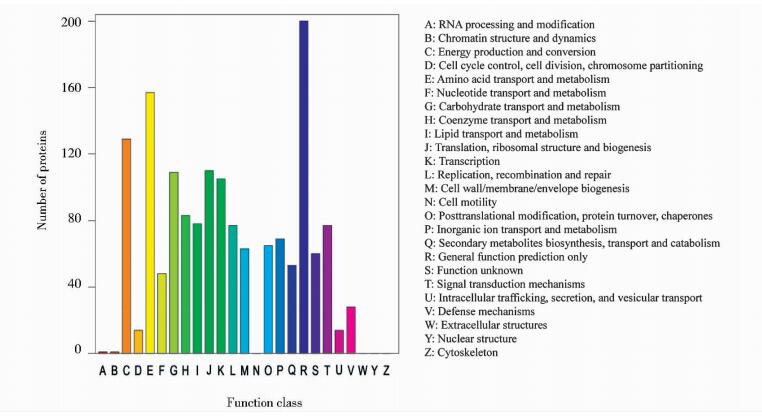
|
| 图 4 菌体蛋白的COG注释 Figure 4 COG annotation of bacterial proteins |
用iTRAQ技术对蛋白质进行相对定量,使用R语言中t.test函数计算样本间差异显著性P值,p.adjust函数进行多重检验FDR矫正,差异蛋白筛选标准为:FDR<0.05、FC<0.8或FC>1.2(FC为蛋白质在两样本间的差异倍数)。用火山图(volcano plot)描述差异表达蛋白的整体分布情况(图 5),与发酵36h相比,发酵108h的M.carbonacea JXNU-1的蛋白质表达呈现出显著差异,得到差异表达蛋白质共172个(FDR<0.05),其中,表达上调蛋白质76个(上调倍数>1.20,FDR<0.05),表达下调蛋白质96个(上调倍数<0.80,FDR<0.05)。
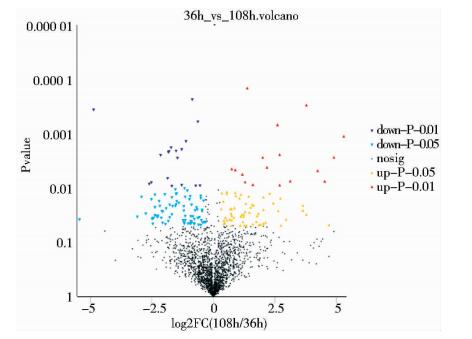
|
| 图 5 差异表达蛋白的火山图 Figure 5 Volcano plot of differentially expressed proteins |
对差异蛋白进行GO功能显著性富集分析,可以说明差异蛋白的功能富集情况,从而给出差异蛋白与哪些生物学功能显著相关,在功能水平阐明样本间的差异。生物过程分析发现,差异表达蛋白显著富集于细胞大分子生物合成过程(cellular macromolecule biosynthetic process)、细胞脂质代谢过程(cellular lipid metabolic process)和翻译(translation)过程;作为细胞组分的差异蛋白大多富集在细胞器(organelle);与分子功能相关的差异蛋白主要富集的GO功能是结构分子活性(structural molecule activity)(图 6)。
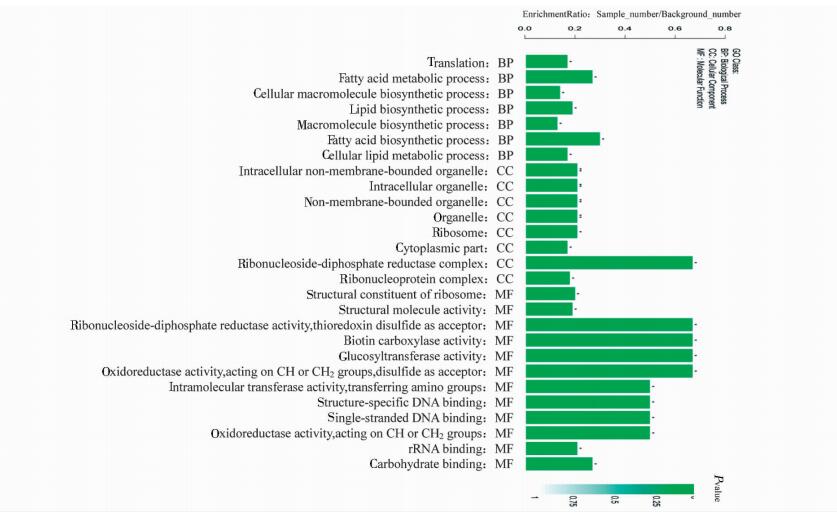
|
| 图 6 差异蛋白的GO富集分析 Figure 6 GO annotation of differentially expressed proteins |
通过对差异蛋白的功能分析,在上调蛋白中筛选到12个与核苷类抗生素生物合成密切相关的蛋白质(表 1),包括ABC转运器底物结合蛋白(ABC transporter substrate-binding protein, SBP)、ABC转运器ATP结合蛋白(ABC transporter ATP-binding protein)、糖基转移酶(glycosyltransferase, GT)、P450单加氧酶(P450 monooxygenase,CYP)、S-腺苷甲硫氨酸依赖的甲基转移酶(SAM-dependent methyltransferase, SAM-MT)、甲基转移酶(methyltransferase, MT)、丝氨酸羟甲基转移酶(serine hydroxymethyltransferase, SHMT)等。
| ID-Gene | NCBI accsssion No. | Most similar in NR database | Protein description | Fold change(108h/36h) |
| MC.JXNU.1GM002940 | WP_043964558 | WP_007071527 | ABC transporter substrate-binding protein | 30.0 |
| MC.JXNU.1GM002567 | WP_043962927 | gb|ACB37750 | glycosyltransferase | 26.0 |
| MC.JXNU.1GM005198 | WP_043962927 | gb|ACB37730 | glycosyltransferase | 23.2 |
| MC.JXNU.1GM002566 | WP_043962926 | gb|ACB46465 | p450 monooxygenase | 6.6 |
| MC.JXNU.1GM003157 | WP_052503802 | WP_010034411 | glycosyltransferase gacs | 4.3 |
| MC.JXNU.1GM004503 | WP_043966877 | WP_019072651 | ABC transporter substrate-binding protein | 4.1 |
| MC.JXNU.1GM004430 | WP_043966801 | WP_007072461 | ABC transporter ATP-binding protein | 3.6 |
| MC.JXNU.1GM005190 | WP_043969377 | gb|ACB37738 | SAM-dependent methyltransferase | 3.5 |
| MC.JXNU.1GM006116 | WP_043968955 | WP_007071263 | glycosyltransferase WbuB | 3.1 |
| MC.JXNU.1GM001043 | WP_043963881 | WP_010056154 | methyltransferase | 2.8 |
| MC.JXNU.1GM000080 | WP_043960791 | WP_008122103 | serine hydroxymethyltransferase | 1.9 |
| MC.JXNU.1GM005862 | WP_043968713 | WP_007076367 | glycosyl transferase family 1 | 1.3 |
ABC转运器ATP结合蛋白是ABC转运蛋白的结构蛋白,负责结合ATP并通过偶联它的水解来推动转运过程,ABC转运蛋白包括内运蛋白(importer)和外排蛋白(exporter),ABC内运蛋白主要是负责营养物质的摄入,一般需要一个底物结合蛋白(substrate-bindingprotein,SBP)来帮助亲水性底物的摄入[8]。ABC内运蛋白可以用来提高次级代谢产物的产量,Li等[9]在阿维链霉菌中超量表达malEFG,改善了淀粉的利用率,可以将阿维菌素的产量提高3倍左右。许多抗生素是放线菌产生的次级代谢产物,对自身具有毒性,ABC转运蛋白可以跨膜排出这些次级代谢产物。在放线菌中,许多抗生素的排出是由ABC外排蛋白介导的,在螺旋霉素、纺锤菌素等抗生素的生物合成基因簇中,也都存在ABC转运蛋白的编码基因[10-11]。
在抗生素生物合成过程中糖基化现象十分普遍,对抗生素的生物活性发挥起着举足轻重的作用。糖基转移酶是糖基化过程的关键酶,这类酶通常负责将活性供体的糖单体部分转移到特异的受体(糖苷配基,可以是脂类、蛋白质、核酸、各种杂环化合物和其它糖类)上,从而形成特异的糖苷键,而在核苷类抗生素中也常具有脱氧糖基结构[12]。友菌素是一类具有抗病毒和抗病菌功能的二糖核苷类抗生素,在友菌素生物合成基因簇中有2个糖基转移酶基因(AmiG、AmiJ)[13]。
细胞色素P450(cytochorme P450,CYP)是广泛存在于生物体内的一类含血红素和硫羟基的蛋白质,参与天然化合物及信号分子生物合成途径中的氧化过程,能够催化多种反应类型,包括烃类的羟基化、稀烃环氧化、N-羟化、N-氧化、氧化脱氨基、脱氢反应、脱水反应、异构反应、N-氧化还原等,在合成各种各样的抗生素,尤其是在解决广谱细菌抗生素的耐药性问题上具有潜在的应用价值[14]。经研究显示CYP基因在链霉菌抗生素生物合成过程中发挥着重要的作用,它们多存在于抗生素生物合成基因簇内,参与抗生素的生物合成。例如,CYP129A1和CYP131A12是道诺霉素生物合成基因簇的组分[15],来自S.tendae的CYP162A1(NikQ)是核苷类抗生素尼可霉素生物合成基因簇的组分[16]。
甲基转移酶催化甲基化反应,通常以S-腺苷甲硫氨酸(S-adenosyl-L-methionine,SAM)作为甲基的供体,能催化多种生理过程中间产物的甲基化进而合成或降解生理活性物质[17]。在一些核苷类抗生素的生物合成中有甲基转移酶的参与,如嘌呤霉素生物合成中氮甲基转移酶Pur5负责己酰化的去三甲基嘌呤霉素氮位上两个甲基的转移,氧甲基转移酶dmpM负责氧位上甲基的转移[18]。推测出的杀稻瘟菌素的生物合成基因簇中含有编码SAM-dependent methyltransferase的blsA基因,编码N-methyltransferase的blsL基因[19]。
丝氨酸羟甲基转移酶可逆性催化丝氨酸和甘氨酸之间的相互转化,反应中会产生N5,N10-亚甲基四氢叶酸,可以为合成甲硫氨酸等甲基供体提供重要底物——碳单位[20],抗生素的生物合成中经常有甲基转移酶的参与,而甲基转移酶通常以SAM作为甲基的供体,丝氨酸羟甲基转移酶可以间接推动抗生素生物合成中的甲基化反应。
2.4.4 抗生素合成相关基因簇筛选在放线菌中,次级代谢产物的生物合成基因常常是成簇排列的,为揭示M.carbonacea JXNU-1发酵产抗生素机制,基于筛选到的抗生素合成密切相关蛋白,将表达这些蛋白质的基因定位到可能与抗生素合成相关的5个基因簇中(表 2),经NCBI数据比对分析发现,Cluster 4与四制癌素A基因簇(tetrocarcin A gene cluster)同源性很高(99%)。四制癌素A是RNA合成抑制剂,其化学结构也属于核苷类抗生素,这与本研究的产物JX属同一种类型,表明Cluster 4极有可能是JXNU-1菌株生物合成抗生素JX的基因簇。
| Cluster name | Gene ID | Gene function | NCBI accsssion No. |
| Cluster 1 | MC.JXNU.1GM001040 | hypothetical protein | WP_052503624 |
| MC.JXNU.1GM001041 | dipeptide/oligopeptide/nickel ABC transporter ATP-binding protein | WP_010056152 | |
| MC.JXNU.1GM001042 | argininosuccinate lyase | WP_043961693 | |
| MC.JXNU.1GM001043 | methyltransferase | WP_043963881 | |
| MC.JXNU.1GM001044 | hypothetical protein | WP_052503625 | |
| MC.JXNU.1GM001045 | methyltransferase | WP_043961694 | |
| MC.JXNU.1GM001046 | MFS transporter permease | WP_043961695 | |
| MC.JXNU.1GM001047 | hypothetical protein | WP_043961696 | |
| Cluster 2 | MC.JXNU.1GM002560 | cytochrome p450 | WP_043964381 |
| MC.JXNU.1GM002561 | hypothetical protein | WP_052503708 | |
| MC.JXNU.1GM002562 | LPS biosynthesis protein | WP_043962924 | |
| MC.JXNU.1GM002563 | hypothetical protein | WP_043962925 | |
| MC.JXNU.1GM002564 | ferredoxin | WP_043964383 | |
| MC.JXNU.1GM002565 | hypothetical protein | WP_043964384 | |
| MC.JXNU.1GM002566 | p450 monooxygenase | WP_043962926 | |
| MC.JXNU.1GM002567 | glycosyltransferase | WP_043962927 | |
| Cluster 3 | MC.JXNU.1GM004430 | ABC transporter ATP-binding protein | WP_043966801 |
| MC.JXNU.1GM004431 | membrane protein | WP_043966802 | |
| MC.JXNU.1GM004432 | membrane protein | WP_043969230 | |
| MC.JXNU.1GM004433 | membrane protein | WP_043966803 | |
| MC.JXNU.1GM004434 | haloalkane dehalogenase | WP_043969231 | |
| MC.JXNU.1GM004435 | AAC(3) family N-acetyltransferase | WP_007072457 | |
| MC.JXNU.1GM004436 | sugar isomerase | WP_043966804 | |
| MC.JXNU.1GM004437 | amine oxidase | WP_043966805 | |
| Cluster 4 | MC.JXNU.1GM005190 | SAM-dependent methyltransferase | WP_043969377 |
| MC.JXNU.1GM005191 | daunorubicin biosynthesis sensory transduction protein DnrJ | WP_043967839 | |
| MC.JXNU.1GM005192 | acyl-CoA dehydrogenase | WP_043967841 | |
| MC.JXNU.1GM005193 | dTDP-glucose 4, 6-dehydratase | WP_043967844 | |
| MC.JXNU.1GM005194 | glucose-1-phosphate thymidylyltransferase | WP_043967845 | |
| MC.JXNU.1GM005195 | hypothetical protein TK50_25775 | KIR61157 | |
| MC.JXNU.1GM005196 | glutamine--scyllo-inositol aminotransferase | WP_043967847 | |
| MC.JXNU.1GM005197 | hypothetical protein TK50_25785 | KIR62137 | |
| MC.JXNU.1GM005198 | glycosyltransferase | WP_043962927 | |
| Cluster 5 | MC.JXNU.1GM005859 | cytochrome ubiquinol oxidase subunit I | WP_043968710 |
| MC.JXNU.1GM005860 | ATP-binding protein | WP_043968711 | |
| MC.JXNU.1GM005861 | membrane protein | WP_043969536 | |
| MC.JXNU.1GM005862 | glycosyl transferase family 1 | WP_043968713 | |
| MC.JXNU.1GM005863 | glucosyl-3-phosphoglycerate synthase | WP_043968715 | |
| MC.JXNU.1GM005864 | oxidoreductase | WP_043969537 | |
| MC.JXNU.1GM005865 | zinc-binding alcohol dehydrogenase | WP_043968716 | |
| MC.JXNU.1GM005866 | accessory Sec system translocase SecA2 | WP_043968718 |
3 讨论
随着微生物来源抗生素的深入研究,一些新型核苷类抗生素被开发出来,如嘌呤霉素、杀稻瘟菌素、多氧霉素、中生菌素等,因其具有广谱的生物活性与巨大应用潜力,自发现以来便得到广泛应用。另外,嘌呤霉素、杀稻瘟菌素和多氧霉素等抗生素生物合成途径[17-18, 21-22]相继被成功推测,相关合成基因簇也被成功克隆[23],为后续合成生物学和代谢调控奠定了良好基础。
蛋白质组学是从整体水平上来认识蛋白质的存在及活动方式(表达、修饰功能、相互作用等),从而更好的阐明生命科学本质[24]。相对和绝对定量同位素标记(iTRAQ技术)是近年来最新开发的一种新的蛋白质组学定量研究技术,具有较好的定量效果和良好的重复性[25]。Ma等[26]基于iTRAQ技术对产前21天和产后1天的奶牛的血清蛋白质进行分析,筛选到19个与应激反应相关的差异表达蛋白,且其中有3个上调蛋白质(V因子、抗纤维蛋白溶酶和凝血素)参与凝血途径,为阐明奶牛调节哺乳和分娩压力的机制奠定了基础。Poonam等[27]采用iTRAQ技术对恶性胶质瘤患者和正常人的血清蛋白质进行研究,筛选到296个差异表达蛋白,其中在患者血清中上调表达蛋白质61个。王雅娟等[28]采用iTRAQ技术对空间环境诱导的褪色沙雷菌LCT-SM166和地面对照组LCT-SM213进行蛋白质组质谱检测,共鉴定到1 713个蛋白质,111个差异表达蛋白中29个表达上调、91个表达下调,分析显示大多数差异蛋白主要与能量代谢有关,该研究为后续贯穿组学的研究奠定了基础。
本文利用iTRAQ技术对菌体生长期和产物合成期的M.carbonacea JXNU-1的蛋白质组进行分析,共鉴定出炭样小单孢菌体蛋白2 390个,有1 502个蛋白质得到GO数据库的注释。获得172个差异表达蛋白,其中上调蛋白质76个、下调蛋白质96个。通过对差异蛋白进行分析,发现ABC转运器底物结合蛋白、ABC转运器ATP结合蛋白、糖基转移酶、P450单加氧酶、S-腺苷甲硫氨酸依赖的甲基转移酶、甲基转移酶和丝氨酸羟甲基转移酶在抗生素分泌期均表达上调,经分析表明它们很可能在抗生素的生物合成过程中扮演重要角色。为更好的研究抗生素生物合成机制,基于上述筛选到的抗生素合成密切相关蛋白,将表达这些蛋白质的基因定位到可能与抗生素合成相关的5个基因簇中,经NCBI数据比对分析发现,Cluster 4与核苷类抗生素四制癌素A基因簇同源性较高,表明Cluster 4极有可能是JXNU-1菌株生物合成JX的基因簇。本研究将为阐明抗生素JX的生物合成与分子调控机制奠定基础,为进一步提高抗生素JX产量和新型药物的开发提供实验依据。
4 结论本文采用iTRAQ技术对M.carbonacea JXNU-1分泌抗生素前后期的差异表达蛋白进行分析,筛选出12个与抗生素合成密切相关的蛋白和5个生物合成相关基因簇,其中Cluster 4极有可能是JXNU-1菌株生物合成JX的基因簇,为阐明该菌的抗生素合成机制提供了实验依据,但是对于其具体生物合成途径还有待于进一步的研究。
| [1] | Chen W, Qi J, Wu P, et al. Natural and engineered biosynthesis of nucleoside antibiotics in Actinomycetes. Journal of Industrial Microbiology & Biotechnology , 2015, 43 (2) : 1–17. |
| [2] | Niu G, Tan H. Nucleoside antibiotics:biosynthesis, regulation, and biotechnology. Trends in Microbiology , 2015, 23 (2) : 110–119. DOI:10.1016/j.tim.2014.10.007 |
| [3] | 龙中儿, 朱跃进, 黄运红, 等. 一株具有广谱抗菌活性小单孢菌的分离和鉴定. 微生物学通报 , 2008, 35 (3) : 378–383. Long Z E, Zhu Y J, Huang Y H, et al. Isolation and identification of a strain of Micromonospora with broad-spectrum antimicrobial activity. Microbiology , 2008, 35 (3) : 378–383. |
| [4] | 黄运红, 高兴强, 龙中儿. 炭样小单孢菌抗生素抗植物病原真菌作用. 农药 , 2009, 48 (11) : 843–845. Huang Y H, Gao X Q, Long Z E. The antibacterial activities of antibiotics produced by Micromonospora carbonacea to plant pathogenic fungi. Agrochemicals , 2009, 48 (11) : 843–845. |
| [5] | 朱跃进, 龙中儿, 黄运红, 等. 一株稀有放线菌发酵产抗生素的工艺研究. 化学与生物工程. 2006 : 39 -42. Zhu Y J, Long Z E, Huang Y H, et al. Studies on fermentation technology for a strain of rare Actinomycete producing antibiotics. Chemistry & Bioengineering. 2006 : 39 -42. |
| [6] | 龙中儿, 朱跃进, 黄运红, 等. 炭样小单孢菌JXNU-1广谱抗生素产物的分离及其理化性质. 微生物学通报 , 2008, 35 (9) : 1450–1454. Long Z E, Zhu Y J, Huang Y H, et al. Isolation and purification of antibiotic from the fermentation broth of Micromonospora carbonacea JXNU-1 and its physical-chemical properties. Microbiology , 2008, 35 (9) : 1450–1454. |
| [7] | 江云, 黄运红, 李非, 等. 一株具有广谱抗菌活性炭样小单孢菌的全基因组序列测定. 微生物学通报 , 2015, 42 (11) : 2178–2188. Jiang Y, Huang Y H, Li F, et al. Whole genome sequencing of a Micromonospora carbonacea strain with broad-spectrum antimicrobial activity. Microbiology China , 2015, 42 (11) : 2178–2188. |
| [8] | Zoghbi M E, Altenberg G A. ATP binding to two sites is necessary for dimerization of nucleotide-binding domains of ABC proteins. Biochemical and Biophysical Research Communications , 2014, 443 (1) : 97–102. DOI:10.1016/j.bbrc.2013.11.050 |
| [9] | Li M, Chen Z, Zhang X, et al. Enhancement of avermectin and ivermectin production by overexpression of the maltose ATP-binding cassette transporter in Streptomyces avermitilis. Bioresource Technol , 2010, 101 (23) : 9228–9235. DOI:10.1016/j.biortech.2010.06.132 |
| [10] | Aigle B, Lautru S, Spiteller D, et al. Genome mining of Streptomyces ambofaciens. Journal of Industrial Microbiology & Biotechnology , 2014, 41 (2) : 251–263. |
| [11] | Vingadassalon A, Lorieux F, Juguet M, et al. Natural combinatorial biosynthesis involving two clusters for the synthesis of three pyrrolamides in Streptomyces netropsis. ACS Chemical Biology , 2014, 10 (2) : 601–610. |
| [12] | Gloster T M. Advances in understanding glycosyltransferases from a structural perspective. Current Opinion in Structural Biology , 2014, 28 : 131–141. DOI:10.1016/j.sbi.2014.08.012 |
| [13] | Liu X, Xu Z, Xue S, et al. Characterization of the amicetin biosynthesis gene cluster from Streptomyces vinaceusdrappus NRRL 2363 implicates two alternative strategies for amide bond formation. Applied & Environmental Microbiology , 2012, 78 (7) : 2393–2401. |
| [14] | Peter F, Guengerich, Andrew W, et al. Unusual cytochrome p450 enzymes and reactions. Journal of Biological Chemistry , 2013, 288 (24) : 17065–17073. DOI:10.1074/jbc.R113.462275 |
| [15] | Dickens M L, Priestley N D, Strohl W R. In vivo and in vitro bioconversion ofε-rhodomycinone glycoside to doxorubicin:functions of DauP, DauK, and DoxA. Journal of Bacteriology , 1997, 179 (8) : 2641–2650. |
| [16] | Liao G, Li J, Li L, et al. Cloning, reassembling and integration of the entire nikkomycin biosynthetic gene cluster into Streptomyces ansochromogenes lead to an improved nikkomycin production. Microbial Cell Factories , 2010, 9 (4) : 1–7. |
| [17] | Park S C, Song W S, Yoon S. Structural analysis of a putative SAM-dependent methyltransferase, YtqB, from Bacillus subtilis. Biochemical and Biophysical Research Communications , 2014, 446 (4) : 921–926. DOI:10.1016/j.bbrc.2014.03.026 |
| [18] | Tercero J A, Espinosa J C, Lacalle R A, et al. The biosynthetic pathway of the aminonucleoside antibiotic puromycin, as deduced from the molecular analysis of the pur cluster of Streptomyces alboniger. Journal of Biological Chemistry , 1996, 271 (3) : 1579–1590. DOI:10.1074/jbc.271.3.1579 |
| [19] | 杜爱芹, 吴俊, 邓子新, 等. 杀稻瘟菌素生物合成基因簇的边界确定. 微生物学通报 , 2014, 41 (7) : 1318–1325. Du A Q, Wu J, Deng Z X, et al. Determination of the boundary of blasticidin S biosynthetic gene cluster. Microbiology , 2014, 41 (7) : 1318–1325. |
| [20] | Yin K. Positive correlation between expression level of mitochondrial serine hydroxymethyltransferase and breast cancer grade. OncoTargets and Therapy , 2015, 8 : 1069–1074. |
| [21] | Feng J, Wu J, Dai N, et al. Discovery and characterization of BlsE, a radical S-adenosyl-L-methionine decarboxylase involved in the blasticidin S biosynthetic pathway. PloS One , 2013, 8 (7) : e68545. DOI:10.1371/journal.pone.0068545 |
| [22] | Chen W, Huang T, He X, et al. Characterization of the polyoxin biosynthetic gene cluster from Streptomyces cacaoi and engineered production of polyoxin H. Journal of Biological Chemistry , 2009, 284 (16) : 10627–10638. DOI:10.1074/jbc.M807534200 |
| [23] | 田云龙. 中生菌素生物合成基因簇克隆及分析. 北京: 中国农业科学院, 农业环境与可持续发展研究所研究生院, 2010 . Tian Y L. Cloning and analysis of Zhongshengmycin Biosynthetic Gene Cluster. Beijing: Chinese Academy of Agricultural Sciences, Graduate School of Agricultural Environment and Sustainable Development, 2010 . |
| [24] | Lambert D G. Proteomics and metabolomics. Anaesthesia & Intensive Care Medicine , 2013, 14 (4) : 169–170. |
| [25] | 赵静, 王宏伟, 田二杰, 等. 蛋白质组学实验技术及其应用. 动物医学进展 , 2015, 36 (1) : 116–120. Zhao J, Wang H W, Tian E J, et al. Proteomics experiment technologies and their applications. Progress in Veterinary Medicine , 2015, 36 (1) : 116–120. |
| [26] | Ma L, Bu D P, Yang Y X, et al. iTRAQ quantitative analysis of plasma proteome changes of cow from pregnancy to lactation. Journal of Integrative Agriculture , 2015, 14 (7) : 1407–1413. DOI:10.1016/S2095-3119(14)60916-3 |
| [27] | Poonam G, Sudha C, Na ir, et al. Proteins with altered levels in plasma from Glioblastoma patients as revealed by iTRAQ-based quantitative proteomic analysis. PLoS One , 2012, 7 (9) : e46153–e46153. DOI:10.1371/journal.pone.0046153 |
| [28] | 王雅娟, 刘进文, 方向群, 等. 空间环境诱导褪色沙雷菌LCT-SM166的蛋白质组学分析. 解放军医学院学报 , 2013, 34 (1) : 10–13. Wang Y J, Liu J W, Fang X Q, et al. Proteomics of space Serratia marcescens LCT-SM166 strain. Academic Journal of Pla Postgraduate Medical School , 2013, 34 (1) : 10–13. |
 2016, Vol. 36
2016, Vol. 36




