文章信息
- 张心愿, 邓佳, 刘馨, 陈琳, 林峻, 蔡伟文.
- ZHANG Xin-yuan, DENG Jia, LIU Xin, CHEN Lin, LIN Jun, CAI Wei-wen.
- BstDNA聚合酶大片段在全基因组扩增中的应用
- Bst DNA Polymerase Large Fragment for Whole Genome Amplification
- 中国生物工程杂志, 2016, 36(7): 48-54
- CHINA BIOTECHNOLOGY, 2016, 36(7): 48-54
- http://dx.doi.org/DOI:10.13523/j.cb.20160708
-
文章历史
- 收稿日期: 2015-12-28
- 修回日期: 2016-02-28
2. 福州大学生物科学与工程学院 福州 350108
2. College of Biological Science and Engineering, Fuzhou University, Fuzhou 350108, China
近年来人们对多种肿瘤基因特性以及基因组检测的有效性日益关注,然而基因组分析的广泛应用却受到从生物样本中得到的基因组DNA总量的限制。利用甲醛固定和石蜡包埋的方法可以保存人类组织样本的绝大部分形态,这一方法已经被沿用了数十年。因此需要建立一种针对从石蜡包埋组织中提取的DNA进行全基因组扩增的方法。目前已经发展并逐渐成熟和完善的方法主要有两种:聚合酶链反应(polymerase chain reaction,PCR)和等温扩增技术(isothermal amplification)[1]。
PCR主要包括简并寡核苷酸引物PCR(degenerate oligonucleotide primed-polymerase chain reaction,DOP-PCR)[2];引物延伸预扩增(primer extension preamplification,PEP)[3];标记PCR(tagged-PCR,T-PCR)[4];单细胞比较基因组杂交(single cell comparative genomic hybridization,SCOMP),即衔接头PCR(linker adaptor-PCR)[5-6]。这些PCR方法都依赖于高质量和高精度的热循环仪,而且使用随机引物以增加与模板的结合位点,区别在于不同的引物设计和温度变化。这些方法可将模板扩增几百倍,产物大小为200~3 000bp。但是由于易发生非特异性扩增,位点覆盖不完全,DNA产物片段小于1kb等缺点(如DOP-PCR更倾向于扩增短的等位基因[2],SCOMP的扩增倍数和精确度也各不相同),限制了这些方法的应用[7]。
等温扩增技术即超支化链置换扩增技术(hyperbranched strand displacement amplification,HSDA),也就是人们熟知的多重置换扩增(multiple displacement amplification,MDA)[5, 8]。MDA基于两个要素[5, 9-10]:引发链置换反应的酶的能力和复制过程中随机引物产生的随机起始位点。某些DNA聚合酶在延伸新链的过程中如果遇到下游DNA链,可以继续延伸反应并同时将下游双链剥离而产生游离的单链,这种反应称为链置换反应[11]。目前发现三种酶能够在MDA中起到催化作用:Ф29 DNA聚合酶、嗜热脂肪芽孢杆菌(Bacillus stearothermophilus)DNA聚合酶大片段(Bst DNA polymerase, large fragment)和Klenow片段。反应启动时,基因组模板变性成为单链,体系中大量的随机引物在多个位点与其进行退火结合,然后在Bst DNA聚合酶大片段的作用下进行延伸;新合成出的DNA链可将下游的模板互补链置换掉,被置换的单链又作为新的模板与随机引物退火结合并进行新的DNA合成。随着越来越多的DNA新链产生,越来越多的随机引物与之退火结合,逐渐形成呈分支放射状的网络样DNA产物,包含模板DNA的数千乃至数百万拷贝[9],如图 1[12]所示。它是近几年迅速发展起来的一种全基因组扩增(whole genome amplification,WGA)技术。一些研究者[8-10]的工作表明,使用Bst DNA聚合酶大片段对完整DNA(从新鲜或者冷冻组织中提取的)进行MDA,能够使其产生稳定、均匀的扩增,且扩增后不会失真。

|
| 图 1 多重置换扩增 Figure 1 Multiple displacement amplification |
Bst DNA聚合酶大片段是嗜热脂肪芽孢杆菌(Bacillus stearothermophilus)DNA聚合酶Ⅰ的一部分,具有5′~3′DNA聚合酶活性,但不具有3′~5′外切核酸酶活性。因其本身无3′~5′外切核酸酶活性,在测序反应中不会对反应造成停顿,且兼具极好的条带均一性和高保真性,能够有效解开测序模板的二级结构,提高测序的准确度[13-14]。鉴于Bst DNA聚合酶大片段以上的特点和其所具有的链置换反应活性,本研究将其应用于癌症石蜡包埋组织中基因组DNA的扩增,并将扩增产物用于本实验室制备好的胃癌微阵列比较基因组杂交芯片(array comparative genomic hybridization,aCGH)研究中。但鉴于其高昂的价格不利于大量MAD和基因组分析研究的发展,本实验室独立克隆、表达和纯化该酶用于后续研究。
1 材料与方法 1.1 材料 1.1.1 菌种与载体本实验室所用主要菌种有Geobacillus stearothermophilus (Donk) (ATCC® 29609TM)及大肠杆菌(Escherichia coli)10β、E.coli ER2566等,所用主要质粒载体为pTWIN1(N6951),购自美国New England Biolabs(NEB)公司。
1.1.2 酶和化学试剂人类基因组由本实验室提供;限制性内切核酸酶、T4 DNA连接酶等购自Thermo Scientific公司;Bst DNA polymerase large fragment、几丁质亲和层析柱购自NEB公司;250bp DNA Ladder、λ-HindⅢdigest DNA Marker购自TaKaRa公司;广谱蛋白Marker购自北京康为世纪生物科技有限公司;其他试剂药品均采用国产分析纯。
1.2 仪器和设备实验所用主要仪器包括PCR仪(南京赛飞生物科技有限公司),电泳仪(Bio-Rad公司)全自动凝胶成像分析仪JS-680B(上海培清科技有限公司),分子杂交箱Mini Oven MKII(Thermo Scientific Hybaid,美国),分子杂交仪FYY-4(兴化市分析仪器厂),分光光度计Nanodrop 2000(Thermo Scientific,美国),荧光光度计TKO 100(Hoefer Scientific公司,美国),微阵列芯片扫读仪Gene Pix 4000A(Axon公司,美国)。
2 方法 2.1 Bst DNA聚合酶大片段的克隆表达与纯化以Bacillus stearothermophilus DNA polymerase I (polA) gene GenBank: U33536的基因组作为模板设计引物,长度为2 631bp。根据其DNA序列和所采用的表达载体pTWIN1(N6951)的具体情况设计引物。Bst DNA聚合酶大片段序列为868~2 631bp[15],故对此1 764bp左右的片段进行引物设计。以嗜热脂肪芽孢杆菌染色体DNA为模板,进行PCR反应,以此获得Bst DNA聚合酶大片段蛋白质的完整基因;将其重组到pTWIN1质粒中,形成表达质粒pTWIN1-LF(LF表示Bst DNA聚合酶大片段的基因片段),并将其转化到宿主菌E.coli ER2566中;然后将含有表达质粒pTWIN1-LF的E.coli ER2566接种于100ml含有100mg/L氨苄青霉素的LB液体培养基中,于37℃、200r/min振荡培养至A600为0.3~0.5。加入IPTG(1 mol/L)至终浓度为0.1mmol/L;再20℃、200r/min诱导培养12h。
收集经诱导表达的大肠杆菌菌体,洗涤后重悬于裂解液(20mmol/L Tris-HCl, pH 8.5, 500mmol/L NaCl, 20μmol/L PMSF)中用酶法将菌体裂解,离心所得上清上样于几丁质亲和层析柱,过夜诱导自切,经洗脱缓冲液洗脱、收集、浓缩,最终获得纯化了的Bst DNA聚合酶大片段溶液。使用超滤管对该蛋白质进行脱盐置换缓冲液,最终将蛋白保存于缓冲液(10mmol/L Tris-HCl, 50mmol/L KCl, 1mmol/L DTT, 0.1mmol/L EDTA, 0.1% Triton X-100, 50%甘油,pH7.1)后置于-20℃保存。
2.2 Bst DNA聚合酶大片段扩增活性及最适温度测定取0.2μg人类基因组(512ng/μl)DNA,加入ddH2O至60μl,将溶液置于100℃,5min;加入40μl 2.5×Bst Mix(随机引物dN9 600ng, 2.5mmol/L dNTP, 10×Bst Buffer),将混合溶液置于4℃,30min;加入5μl Bst DNA聚合酶大片段,45℃反应3h;检测DNA浓度。并在35~65℃设置温度梯度,探究其最适温度。
2.3 癌症石蜡包埋组织基因组DNA的提取方法步骤参考袁亚婷等[16]二甲苯脱蜡-酚氯仿法从胃癌样本1和2石蜡包埋组织中提取DNA,但略有改动。
2.4 Bst DNA聚合酶大片段应用于癌症石蜡包埋组织DNA基因组扩增用Bst DNA聚合酶大片段对胃癌样本1和2石蜡包埋组织DNA进行扩增,每组设置三个平行对照,扩增方法同2.2。
2.5 将扩增产物应用于胃癌aCGH芯片将2.4中的胃癌样本基因组扩增产物和正常人类基因组扩增产物用荧光染料Cy3和Cy5分别标记,并运用荧光交换标记法(dye-swap labeled),即用Cy5标记同样的胃癌样本基因组扩增产物,用Cy3标记同样的正常人类基因组扩增产物,充分混合后,两组标记好的产物与本实验室制备好的胃癌aCGH芯片进行杂交。用微阵列芯片扫读仪对杂交后的芯片进行扫读。
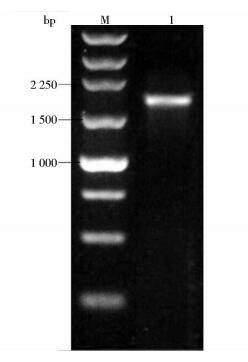
3 结果 3.1 PCR反应反应得到Bst DNA聚合酶大片段基因经1%琼脂糖凝胶电泳,结果如图 2所示,目的基因长度大约1 764bp,与电泳结果吻合。
|
| 图 2 DNA聚合酶大片段基因PCR产物 Figure 2 PCR products of Bst DNA polymerase large fragment gene M:250bp DNA ladder; 1:Bst DNA polymerase large fragment gene |
Bst DNA聚合酶大片段的蛋白质经几丁质亲和层析柱纯化后,进行12% SDS-PAGE凝胶电泳,结果如图 3所示:目的蛋白大小约67kDa,与电泳结果吻合。

|
| 图 3 纯化后的Bst DNA聚合酶大片段 Figure 3 Purification of the Bst DNA polymerase large fragment MW:Broad spectrum protein marker; 1:Bst DNA polymerase large fragment |
以人类基因组为模板,分别用NEB的Bst DNA polymerase large fragment和自产的Bst DNA聚合酶大片段对其进行扩增,设置三组平行实验,并设置以ddH2O代替酶的空白对照,3h后,用荧光光度计检测各管DNA浓度,结果如表 1所示。
| 聚合酶与其对照 | 平行1 | 平行2 | 平行3 |
| 阴性对照 | 15 | 17 | 13 |
| NEB Bst DNA polymerase large fragment | 35 | 33 | 37 |
| 自产Bst DNA聚合酶大片段 | 55 | 52 | 50 |
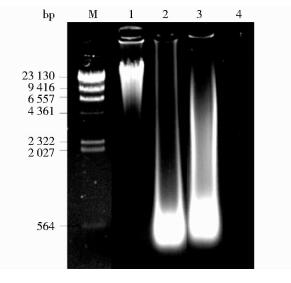
对扩增后产物进行1%琼脂糖凝胶电泳,结果如图 4所示。
|
| 图 4 人类基因组扩增产物 Figure 4 Amplification products of human genome M:λ-HindⅢdigest DNA Marker; 1:Human Genome; 2: NEB Bst DNA polymerase large fragment; 3: Post-purification Bst DNA polymerase large fragment; 4: Negative control |
以人类基因组为模板,dN9为引物,用Bst DNA聚合酶大片段对其在25℃、35℃、45℃、55℃、65℃分别进行扩增,3h后用荧光光度计检测各管DNA浓度,结果如表 2所示。
| 温度 | 25℃ | 35℃ | 45℃ | 55℃ | 65℃ |
| 平行1 | 17 | 17 | 44 | 13 | 21 |
| 平行2 | 12 | 34 | 50 | 21 | 31 |
| 平行3 | 16 | 16 | 48 | 18 | 18 |
向每个反应体系中各加入1/10体积的5mmol/L NaCl和等体积的异丙醇,-20℃放置10min后,14 000r/min离心6min,将扩增产物沉淀下来,并重新溶解于50μl 1×TE溶液中。用Thermo Nanodrop 2000检测各扩增产物的浓度,结果如表 3所示。
| 样品 | 浓度 | A260 | A280 | 260/280 | 260/230 |
| 25℃ | 559.1 | 11.182 | 6.317 | 1.77 | 1.81 |
| 35℃ | 713.7 | 14.275 | 8.224 | 1.74 | 1.7 |
| 45℃ | 789.8 | 15.797 | 9.736 | 1.62 | 1.46 |
| 55℃ | 718.2 | 14.364 | 8.193 | 1.75 | 1.7 |
| 65℃ | 628.6 | 12.572 | 7.092 | 1.77 | 1.82 |
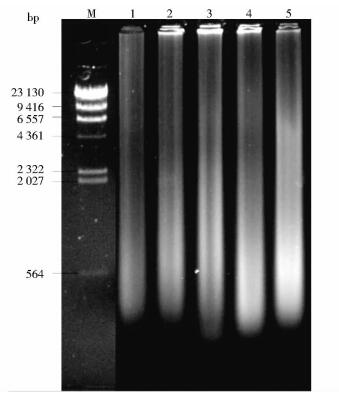
将上述扩增产物进行1%琼脂糖凝胶电泳,结果如图 5所示。
|
| 图 5 不同温度下人类基因组扩增产物 Figure 5 Amplification products of human genome at different temperatures M:λ-HindⅢdigest DNA Marker; 1:25℃; 2:35℃; 3:45℃; 4:55℃; 5:65℃ |
用Thermo Nanodrop 2000对所提取的胃癌样本1和2的DNA进行浓度和纯度测定,结果如表 4所示。
| 样品 | 浓度 | A260 | A280 | 260/280 | 260/230 |
| 胃1 | 775.5 | 15.511 | 9.339 | 1.66 | 1.18 |
| 胃2 | 479.3 | 9.586 | 5.948 | 1.61 | 1.03 |
使用Bst DNA聚合酶大片段对所提取的胃癌石蜡包埋组织DNA基因组进行扩增,3h后用荧光光度计检测各管DNA浓度,结果如表 5所示。
| 平行1 | 平行2 | 平行3 | |
| 胃癌样本1 | 65 | 63 | 60 |
| 胃癌样本2 | 60 | 58 | 56 |
将扩增产物进行1%琼脂糖凝胶电泳,结果如图 6所示。

|
| 图 6 胃癌样本石蜡包埋组织DNA基因组扩增产物 Figure 6 Amplification products of the FFPE gastric cancer DNA M:λ-HindⅢdigest DNA Marker; 1: Gastric cancer sample 1 DNA; 2: Gastric cancer sample 2 DNA; 3: Amplification products of gastric cancer sample 1 DNA; 4: Amplification products of gastric cancer sample 2 DNA |
用微阵列芯片扫读仪对杂交好的胃癌aCGH芯片进行扫读,该芯片中共有7 488个探针点。以样本1为例,样本1 DNA用Cy5标记,对照为正常人类基因组用Cy3标记,杂交后扫读结果如图 7所示。

|
| 图 7 样品1 Cy5和对照Cy3杂交结果 Figure 7 Hybridization of sample 1 Cy5 and Human Genome DNA Cy3 |
样本1 DNA用Cy3标记,对照为正常人类基因组用Cy5标记,杂交后扫读结果如图 8所示。

|
| 图 8 样品1 Cy3和对照Cy5杂交结果 Figure 8 Hybridization of sample 1 Cy3 and Human Genome DNA Cy5 |
选取两张扫读图片中第1行第3个探针区块放大,并对比显示如图 9所示。

|
| 图 9 两张芯片杂交后第1行第3个探针区块 Figure 9 Block 3 on row 1 of the two hybridized chips |
实验结果显示,无论是正常人类基因组还是从胃癌石蜡切片组织提取的DNA经Bst DNA聚合酶大片段扩增后,均可见显著扩增。温度梯度实验结果显示,使用dN9随机引物进行扩增时,该酶的最适温度为45℃。多次扩增结果显示,Bst DNA聚合酶大片段能够实现稳定扩增,平均扩增倍数能达到200倍,扩增产物均匀性差别不大。但是在500~2 000bp有显著扩增条带。
与胃癌aCGH芯片杂交结果显示,扫描得到图像中芯片上的探针点大部分呈现黄色,代表着Cy3与Cy5激发出来的荧光强度基本相同,即样本与对照之间的拷贝数基本相同。这说明扩增产物能与整个芯片上的7 488个探针点进行杂交,并得到较为理想的结果。同时,图 9中箭头指示的三个不同探针点呈现绿色和红色相反的结果,刚好符合荧光交换标记法的结果,其他探针区块的部分探针点也有相同的结果,绿色或者红色点代表着不同程度的拷贝数变异。更加印证了用Bst DNA聚合酶大片段扩增后产物能够应用于aCGH研究中。
Aviel-Ronen等[1]研究表明,从石蜡包埋组织中提取的DNA经Bst DNA聚合酶大片段扩增后,平均扩增倍数能达到803~1 035倍。这比之前报道的Large等[9]从新鲜或者冷冻组织样品中提取的DNA扩增倍数为250倍要高很多。这可能是因为不同的DNA浓度测量方法导致的:前者使用的是NanoDrop分光光度法,后者使用的是PicoGreen DNA浓度测定法。另一种可能的解释是Bst DNA聚合酶大片段会对短片段的DNA进行优先扩增[17-18]。并且使用实时定量PCR(quantitative realtime PCR,qPCR)和aCGH两种方法对未扩增的样本DNA和扩增后产物进行拷贝数差异进行分析,结果显示扩增前后拷贝数差异基本相同,即扩增后不会失真。
本研究中所使用的Bst DNA聚合酶大片段具有高最适温度,能够适应更加苛刻和广泛的扩增条件;同时该酶消除了DNA的二级结构,因而能够准确并且均匀地扩增全基因组序列。使用高度续进性的Bst DNA聚合酶大片段得到长的DNA片段,确保了扩增得到的DNA覆盖了整个基因组,连贯并且无偏地扩增所有的遗传位点,从而能够应用于aCGH研究中,更加真实和准确地反应基因组中DNA拷贝数变异情况。
大量研究结果表明,Bst DNA聚合酶大片段适用于对从石蜡组织中提取DNA的WGA。扩增后产物DNA可用于后续的qPCR和aCGH方法来检测基因拷贝数变异。该扩增方法效率高,技术成熟。使用Bst DNA聚合酶大片段进行全基因组扩增将成为常规分子实验的一部分,随着WGA技术的不断完善,它将会有更广泛的临床和科研应用前景。
| [1] | Aviel-Ronen S, Zhu C, Coe B P, et al. Large fragment Bst DNA polymerase for whole genome amplification of DNA from formalin-fixed paraffin-embedded tissues. BMC Genomics , 2006, 7 (1) : 312. DOI:10.1186/1471-2164-7-312 |
| [2] | Grant S F, Steinlicht S, Nentwich U, et al. SNP genotyping on a genome-wide amplified DOP-PCR template. Nucleic Acids Res , 2002, 30 (22) : e125. DOI:10.1093/nar/gnf125 |
| [3] | Zhang L, Cui X, Schmitt K, et al. Whole genome amplification from a single cell:implications for genetic analysis. Proc Natl Acad Sci USA , 1992, 89 (13) : 5847–5851. DOI:10.1073/pnas.89.13.5847 |
| [4] | Grothues D, Cantor C R, Smith C L. PCR amplification of megabase DNA with tagged random primers (T-PCR). Nucleic Acids Res , 1993, 21 (5) : 1321–1322. DOI:10.1093/nar/21.5.1321 |
| [5] | Dean F B, Hosono S, Fang L, et al. Comprehensive human genome amplification using multiple displacement amplification. Proc Natl Acad Sci USA , 2002, 99 (8) : 5261–5266. DOI:10.1073/pnas.082089499 |
| [6] | Stoecklein N H, Erbersdobler A, Schmidt-Kittler O, et al. SCOMP is superior to degenerated oligonucleotide primed-polymerase chain reaction for global amplification of minute amounts of DNA from microdissected archival tissue samples. Am J Pathol , 2002, 161 (1) : 43–51. DOI:10.1016/S0002-9440(10)64155-7 |
| [7] | Spits C, Le Caignec C, De Rycke M, et al. Optimization and evaluation of single-cell whole-genome multiple displacement amplification. Human Mutation , 2006, 27 (5) : 496–503. DOI:10.1002/(ISSN)1098-1004 |
| [8] | Dean F B, Nelson J R, Giesler T L, et al. Rapid amplification of plasmid and phage DNA using Phi 29 DNA polymerase and multiply-primed rolling circle amplification. Genome Res , 2001, 11 (6) : 1095–1099. DOI:10.1101/gr.180501 |
| [9] | Lage J M, Leamon J H, Pejovic T, et al. Whole genome analysis of genetic alterations in small DNA samples using hyperbranched strand displacement amplification and array-CGH. Genome Res , 2003, 13 (2) : 294–307. DOI:10.1101/gr.377203 |
| [10] | Rook M S, Delach S M, Deyneko G, et al. Whole genome amplification of DNA from laser capture-microdissected tissue for high-throughput single nucleotide polymorphism and short tandem repeat genotyping. Am J Pathol , 2004, 164 (1) : 23–33. DOI:10.1016/S0002-9440(10)63092-1 |
| [11] | 何艳, 蒋涛. 基于链置换反应的DNA等温扩增技术应用进展. 医学综述 , 2010, 1 : 24–27. He Y, Jiang T. Application progress of DNA isothermal amplification based on strand displacement reaction. Medical Recapitulate , 2010, 1 : 24–27. |
| [12] | Lasken R S, Egholm M. Whole genome amplification:abundant supplies of DNA from precious samples or clinical specimens. Trends in Biotechnology , 2003, 21 (12) : 531–535. DOI:10.1016/j.tibtech.2003.09.010 |
| [13] | Mcclary J, Ye S Y, Hong G F, et al. Sequencing with the large fragment of DNA polymerase I from Bacillus stearothermophilus. DNA Seq , 1991, 1 (3) : 173–180. DOI:10.3109/10425179109020768 |
| [14] | Mead D A, Mcclary J A, Luckey J A, et al. Bst DNA polymerase permits rapid sequence analysis from nanogram amounts of template. Biotechniques , 1991, 11 (1) : 76–78. |
| [15] | Phang S M, Teo C Y, Lo E, et al. Cloning and complete sequence of the DNA polymerase-encoding gene (BstpolI) and characterisation of the Klenow-like fragment from Bacillus stearothermophilus. Gene , 1995, 163 (1) : 65–68. DOI:10.1016/0378-1119(95)00387-L |
| [16] | 袁亚婷, 江岳鑫, 尹小文, 等. 四种石蜡包埋组织DNA提取方法的比较. 中国组织工程研究与临床康复 , 2010, 24 : 4430–4434. Yuan Y T, Jiang Y X, Yin X W, et al. Comparison of four methods for DNA extraction from paraffin-embedded tissues. Journal of Clinical Rehabilitative Tissue Engineering Research , 2010, 24 : 4430–4434. |
| [17] | Srinivasan M, Sedmak D, Jewell S. Effect of fixatives and tissue processing on the content and integrity of nucleic acids. Am J Pathol , 2002, 161 (6) : 1961–1971. DOI:10.1016/S0002-9440(10)64472-0 |
| [18] | Siwoski A, Ishkanian A, Garnis C, et al. An efficient method for the assessment of DNA quality of archival microdissected specimens. Mod Pathol , 2002, 15 (8) : 889–892. DOI:10.1097/01.MP.0000024288.63070.4F |
 2016, Vol. 36
2016, Vol. 36




