文章信息
- 阿力玛, 高原, 苏小虎, 周欢敏.
- A Li-ma, GAO Yuan, SU Xiao-hu, ZHOU Huan-min.
- CRISPR/Cas9编辑绒山羊FGF5基因细胞株的建立
- Establishment of CRISPR/Cas9-edited FGF5 Cell Strains in Cashmere Goat
- 中国生物工程杂志, 2016, 36(7): 41-47
- CHINA BIOTECHNOLOGY, 2016, 36(7): 41-47
- http://dx.doi.org/DOI:10.13523/j.cb.20160707
-
文章历史
- 收稿日期: 2016-01-07
- 修回日期: 2016-02-28
我国绒山羊具有世界上品质最好的羊绒纤维,其绒毛产量居世界首位,制作的服饰产品深受人们喜爱[1]。随着现代生活水平的不断提高,人们对羊绒的品质如含绒量、绒质等提出了更高的要求。因此,如何提高绒山羊的产绒量和绒毛品质就显得尤为重要。传统的绒山羊育种已经赶不上市场对绒毛需求变化的步伐,必须将现代生物工程技术应用到家畜的育种上,使羊绒产业能高效生产,资源达到有效的利用。
1994年,Hebert等利用基因打靶技术敲除小鼠ES细胞中的FGF5基因,证实了FGF5基因是通过调节毛发生长周期从而导致被毛增长[2]。FGF5基因是FGF基因家族中的一员,有研究表明,有多种FGFs都在毛囊的发育中起作用[3-5]。2006年,高爱琴等克隆出了全长为925bp的山羊FGF5基因cDNA序列[6],用RT-PCR的方法检测了FGF5基因mRNA在绒山羊皮肤毛囊周期性生长不同时期的表达及分布。由于FGF5基因突变可导致毛发生长周期中生长期的延长使毛发长度增加,我们运用基因编辑技术得到编辑了FGF5基因的内蒙古白绒山羊必将带来十分可观的经济效益。为此,在内蒙古白绒山羊中编辑FGF5基因得到高产绒量的动物将具有重要的研究价值。
规律性重复短回文序列簇(CRISPRs)是一类独特的DNA重复序列,广泛存在于原核生物基因中,包含大多数的细菌和几乎所有的古细菌[7]。CRISPR/Cas系统是继TALEN之后的基因组定点编辑新技术。2005年,Cas系统被发现在原核生物中表现出某种获得性免疫功能,能使宿主获得抵抗噬菌体、质粒等外来DNA入侵的免疫能力[8-9]。2012年,Doudna带领的研究小组把crRNA、tracrRNA二元复合体经过改造加工成了单链的RNA嵌合体,并且利用自己设计的crRNA序列使用原核生物中的CRISPR/Cas9系统能够精确地切割体外的DNA靶序列,而改造完成后的RNA嵌合体也同样能指导Cas9蛋白对特定位点的双链DNA进行剪切[10]。这是对CRISPR/Cas系统的首次改造及应用,随后有大量的报道证明CRISPR/Cas系统在大肠杆菌内能够精确切割外源的双链DNA及外源的质粒[11-12]。2013年,Cong和Mali首次将CRISPR/Cas系统在哺乳动物中成功应用,又一次拓展了动物基因编辑的技术[13-14]。之后有研究表明,利用CRISPR/Cas系统突变一个等位基因的克隆效率是利用TALEN技术的2~5倍,且利用CRISPR/Cas系统更容易产生纯合体突变。此外,应用此系统进行基因敲入产生克隆的比例比TALEN技术高出近10倍[15]。目前,科学家们应用该技术已经对多个物种及模式生物进行了基因组定点敲除或修饰,这些生物包括人类、小鼠、大鼠、斑马鱼、猪、山羊、绵羊、食蟹猴及植物等[16-20]。此外,科学家们还发现由于CRISPR/Cas9系统非常简单可用于对基因组的多个位点同时进行基因编辑,这一特点极大的降低了多基因突变的难度,对基因功能研究及动物育种具有巨大的推动作用[1]。
目前,提高我国绒山羊生产性能的育种研究正在逐步开展,从而满足家畜优势产品的高效生产、特殊性状新品种快速培育的需求[1]。动物基因编辑技术在动物生产及人类疾病治疗等领域都有很高的价值,本研究正是针对家畜育种改良的任务而开展,将动物基因编辑技术CRISPR/Cas9应用于绒山羊的绒毛改良,改善生产性状,迅速改进生产功能,培育具有高产绒量性状的新品系,为生产FGF5基因编辑的绒山羊奠定基础,推动畜牧业的可持续发展。
1 材料与方法 1.1 材料山羊皮肤成纤维细胞、pN1-EGFP载体由内蒙古自治区生物制造重点实验室保存。
大肠杆菌感受态细胞DH5α菌株、质粒抽提试剂盒、基因组DNA提取试剂盒均购自天根生化科技(北京)有限公司;体外转录Cas/gRNA质粒构建试剂盒购自北京唯尚立德生物科技有限公司;高保真GXL酶、各种限制性内切核酸酶、pMD-19T载体及DNA Marker均购自大连TaKaRa公司;T7E1酶购自NEB公司;DMEM/F12购自Hyclone公司;胎牛血清购自Sigma公司;引物合成、基因测序均由上海生物工程公司完成。
1.2 方法 1.2.1 FGF5基因克隆及序列分析根据山羊FGF5基因第一外显子序列设计引物,PCR扩增后进行电泳检测并测序,-80℃保存备用。引物序列如表 1所示。
| Primer name | Primer sequence (5′-3′) |
| PF | CAACCCTGCAAGATGCACTTAGG |
| PR | CAGGTTCTGGAGGAGAGCAAGC |
反应体系:模板(山羊基因组):1μl(58.1ng/μl);上游引物:1μl;下游引物:1μl;GXL polymerase:1μl:10×GXL Buffer 5μl,10mmol/L dNTPs 4μl;补ddH2O至50μl。反应条件:94℃(5min),94℃(30s),58℃(30s),72℃(30s),30个循环,72℃(10min)。
1.2.2 Cas9/gRNA真核表达质粒载体的构建在FGF5基因的第一外显子序列设计2个NGG的gRNA靶点,序列如表 2所示。
| gRNA name | The target sequence (5′-3′) |
| 1 | AGAAGCGCCTCGCACCCAAAGGG |
| 2 | TTTCCATCTGCAGATCTACCCGG |
对选择的gRNA靶点序列设计合成引物,引物序列如下表 3所示。
| Primer name | Primer sequence (5′-3′) |
| 1 PF | ACTCACTATAGGAGAAGCGCCTCGCACCCAAA |
| 1 PR | CTCTAAAACTTTGGGTGCGAGGCGCTTCT |
| 2 PF | ACTCACTATAGG TTTCCATCTGCAGATCTACC |
| 2 PR | CTCTAAAACGGTAGATCTGCAGATGGAAA |
(1)合成相应的oligo二聚体反应体系:上游引物:1μl;下游引物:1μl;Solution I:5μl,补ddH2O至10μl。反应条件:95℃(3min),94~25℃(0.05℃/s),16℃(5min),-20℃保存待用。
(2)将合成的oligo二聚体插入Cas9/gRNA质粒载体反应体系:Cas9/gRNA Vector:1μl;Oligo二聚体:2μl;补ddH2O至10μl,25℃保温5min后转化,挑菌扩增后提质粒DNA进行测序分析,-80℃保存备用。
1.2.3 Cas9/gRNA真核表达质粒载体活性检测P4代细胞生长至完全汇合时,使用NEPA21电转染仪分别转染两个Cas9/gRNA质粒,转染电压150V,脉冲时间5ms。转染2天后,提取细胞基因组DNA,使用前述引物进行PCR,反应体系和反应条件同1.2.1,2%琼脂糖凝胶电泳鉴定,若条带单一进行后续操作。
取Cas9/gRNA质粒转染组与未转染组PCR产物各10μl混合后标记为阳性组,另取未转染组PCR产物20μl标记为对照组。按照以下程序退火:95℃5min,95℃以-2℃/s的速率降至85℃,再以-0.1℃/s的速率降至25℃,16℃保存。
退火完成后每组样品加入1μl T7E1酶与2μl 10×NEB Buffer2,37℃酶切1h,2%琼脂糖凝胶电泳检测。
1.2.4 FGF5基因编辑细胞株的建立及鉴定P4代细胞生长至完全汇合时,使用NEPA21电转染仪转染酶切活性较好的Cas9/gRNA质粒转染细胞,方法同1.2.3。阳性对照为转染pEGFP-N1质粒。
48h后消化收集转染细胞,调整细胞浓度,体视镜下用自制玻璃细针将单个细胞接种于96孔板培养,培养液为15%FBS+85%DMEM/F12,培养条件为37℃、5% CO2、饱和湿度。待细胞长至80%后传至24孔板中,直到细胞传至60mm培养皿。细胞生长至完全汇合时一半冻存、一半按基因组DNA提取试剂盒说明提取基因组DNA并PCR鉴定,反应体系和反应条件同1.2.1,PCR产物送由上海生物工程公司测序。
测序后,将发生突变的单细胞株PCR产物进行纯化,与pMD-19T克隆载体连接,菌落PCR后将15个单菌落送由上海生物工程公司测序,以此评估稳定细胞株的突变纯度。
2 结果 2.1 FGF5基因序列扩增结果根据FGF5序列引物进行PCR扩增,扩增产物经电泳鉴定在431bp处有条带,与目的片段大小相符(图 1)。
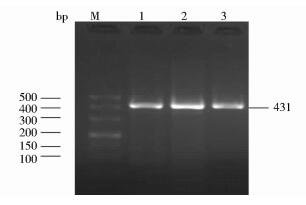
|
| 图 1 FGF5基因PCR扩增产物凝胶电泳结果 Figure 1 The results of PCR amplification products of FGF5 gene M:DL500 marker; 1, 2:The amplification products of FGF5 gene; 3:Negative control |
质粒DNA测序分析后均正确,命名序列正确的质粒为pCas9-FGF5-1、pCas9-FGF5-2。两个载体只有gRNA序列不同,基础质粒图谱如图 2所示。
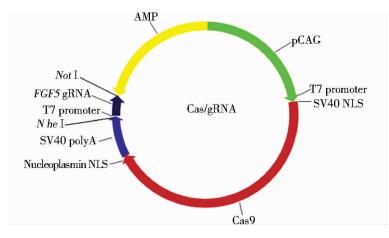
|
| 图 2 Cas/gRNA质粒图谱 Figure 2 The frame of Cas/gRNA plasmid |
电泳检测结果显示,两个pCas9-FGF5载体活性检测均得到相应大小的目的条带(图 3)。从图 3中可以看出,1号有431bp、300bp、131bp 3个条带,2号有431bp、363bp、68bp 3个条带,3号对照组只有431bp一个条带。2个样品均有3条目的条带,即T7E1酶发挥作用将杂合链切断后产生了3个大小不一的目的条带。因此,2个Cas/gRNA质粒载体均具有活性,可用于后续细胞转染实验。根据条带的清晰度,本研究最终确定使用pCas9-FGF5-2质粒用于后续的细胞转染实验。
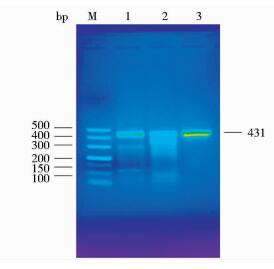
|
| 图 3 T7E1酶切凝胶电泳结果 Figure 3 Enzyme linearization of T7E1 M:DL500 marker; 1:Transfection of pCas9-FGF5-1 vectors; 2: Transfection of pCas9-FGF5-2 vectors; 3:Negative control |
细胞生长至完全汇合时[图 4(a)]用pCas9-FGF5-2真核表达质粒载体转染细胞,转染48h后细胞生长至70%~80%[图 4(b)]开始接种单细胞。阳性对照为pEGFP-N1质粒转染48h观察转染效率([图 4(c)明场、图 4(d)暗场]。由图 4可以看出,细胞转染前后形态基本一致,均呈长梭状,对照组绿色荧光阳性细胞占比较大,转染效率约为60%,该转染条件合适。

|
| 图 4 pCas9-FGF5-2质粒转染细胞结果 Figure 4 The results of transfection of pCas9-FGF5-2 |
单细胞接种2~3天可观察到细胞呈长梭状[图 5(a)],4~5天细胞生长至40%~50%[图 5(b)],6~7天细胞生长至汇合后进行传代培养[图 5(c)]。

|
| 图 5 FGF5基因编辑细胞株的构建 Figure 5 The construction of the cell lines that knockout FGF5 gene |
本实验共培养135个单细胞株,测序结果显示在靶标区域有20个发生突变,总突变率为14.81%。FGF5-/-型突变率为2.96%,FGF5+/-型突变率为11.85%(表 4)。
| CRISPR/cas9 | 单克隆细胞株(个) | 突变细胞株(个) | 突变率(%) | ||
| DNA | 135 | 20 | FGF5-/-型突变率 | FGF5+/-型突变率 | 总突变率(%) |
| 2.96% (4/135) | 11.85% (16/135) | 14.81% | |||
TA克隆测序得知,3个单克隆细胞株的两条等位基因在靶点附近均增添一个T碱基[图 6(a)、(b)],导致该位点以后阅读框移码的改变[图 6(c)],产生错误的蛋白质,导致FGF5失活。

|
| 图 6 单细胞株鉴定结果 Figure 6 The results of monoclones identification (a) Result of peak photo of mutation(the increased T base was marked in the red box) (b) Positive cell line sequence alignment(the following is normal genome sequence) (c) Positive cell lines protein sequence alignment(the following is normal genome sequence) |
TA克隆测序得1个单克隆细胞株在靶点附近发生序列破坏,第一个单克隆细胞株一条等位基因上缺失了23个碱基,导致该位点以后阅读框移码的改变[图 7(b)];另一条等位基因上缺失了12个碱基,缺失4个氨基酸[图 7(c)]。
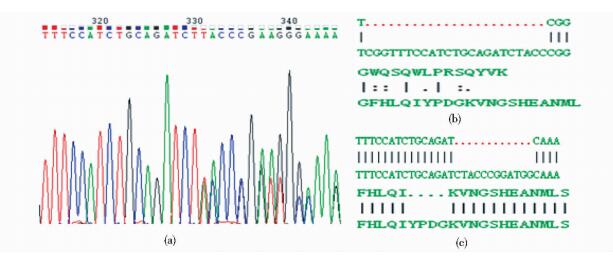
|
| 图 7 单细胞株鉴定结果 Figure 7 The results of monoclones identification (a) Result of peak photo of mutation (b), (c) Positive cell line sequence alignment and protein sequence alignment (the following is normal genome sequence) |
大量研究表明,FGF5基因在绒山羊的毛发生长周期中具有重要的调控作用[21],它的突变可以导致毛发生长周期中生长期的延长,从而使毛发的长度增加。本研究构建了FGF5基因编辑的pCas9-FGF5真核表达质粒载体,将山羊的FGF5基因编辑从而改善绒毛品质,对以后生产出高产绒量的山羊、提高绒毛纺织性能增加绒毛的经济价值具有重要意义。
传统的基因重组技术由于同源重组率低、易脱靶等原因,很难获得双敲除的细胞克隆[22]。CRISPR/Cas9相对于稍早的RNAi、ZFN和TALEN系统,具有操作简单、效率高、成本低、可同时编辑多个数量基因等优点,是生产基因编辑动物的理想工具。还可将其应用于人类基因治疗、新药开发等生物医学研究领域,针对目前人类所高发的重大疾病(如癌症、传染病、遗传病及自体免疫性疾病等)开辟新的治疗途径,并且为临床上的个性化治疗提供方便[23]。本研究采用的pCas9-FGF5真核表达质粒载体成本低且效率高,获得了20个不含筛选标记基因FGF5基因编辑细胞株(包括FGF5-/-和FGF5+/-),FGF5-/-型细胞株为生产FGF5基因编辑的绒山羊奠定了基础。
传统转基因动物研究常利用荧光标记基因和药物筛选制备基因编辑的细胞,考虑到家畜育种的后期应用及生物安全等方面的问题,在设计pCas9-FGF5真核表达质粒载体时没有加入筛选标记基因,导致获得FGF5基因编辑的细胞时不能采用药物筛选法,只能通过有限稀释法和单细胞接种法进行细胞筛选。单细胞接种法比有限稀释法更易获得单克隆细胞[1]。由于有限稀释法只是从理论和概率上说明每个孔内的细胞为一个,而在实际应用中,这种方法获得单克隆的概率很低。而单细胞接种法则是确定将1个细胞传入96孔板。为解决单个细胞不易培养的问题,本研究在单细胞培养时所用的培养液为细胞条件培养液。细胞条件培养液中含有收集自同系细胞生长至80%~90%汇合的细胞培养液,这样的培养液中含有细胞生长所需的各种因子及信号,可以促进细胞生长。
| [1] | 王丙萍. 靶除FGF5基因体细胞克隆绒山羊的研究. 内蒙古: 内蒙古农业大学, 生命科学学院, 2014 . Wang B P. The Study on the Cloned Goat Knockout FGF5 Gene Transferred Somatic Cell. Inner Mongolia: Agricultural University, College of Life Science, 2014 . |
| [2] | Hebert J M, Rosenquist T, Gotz J, et al. FGF5 as a regulator of the hair growth cycle:evidence from targeted and spontaneous mutations. Cell , 1994, 78 (6) : 1017–1025. DOI:10.1016/0092-8674(94)90276-3 |
| [3] | Nguyen H Q, Danilenko D M, Bucay N, et al. Expression of keratinocyte growth factor in embryonic liver of transgenic mice causes changes in epithelial growth and differentiation resulting in polycystic kidneys and other organ malformations. Oncogene , 1996, 12 (10) : 2109–2119. |
| [4] | Rosenquist T A, Martin G R. Fibroblast growth factor signalling in the hair growth cycle:expression of the fibroblast growth factor receptor and ligand genes in the murine hair follicle. Dev Dyn , 1996, 205 : 379–386. DOI:10.1002/(ISSN)1097-0177 |
| [5] | Sundberg J P, Rourk M K, Boggess D, et al. Angora mouse mutation:altered hair cycle, follicular dystrophy, phenotypic maintenance of skin grafts, and changes in keratin expression. Vet Pathol , 1997, 34 (5) : 171–179. |
| [6] | 高爱琴, 李宁, 李金泉, 赵兴波. 山羊FGF5基因单核苷酸多态性群体遗传学分析. 华北农学报 , 2006, 21 (3) : 71–76. Gao A Q, Li N, Li J Q, et al. Analysis on single nucleotide polymorphisms of FGF5 gene in different goat breeds. Acta Agriculturae Boreali-Sinica , 2006, 21 (3) : 71–76. |
| [7] | Lillestøl R, Redder P, Garrett R A, et al. A putative viral defence mechanism in archaeal cells. Archaea , 2006, 2 (1) : 59–72. DOI:10.1155/2006/542818 |
| [8] | Bolotin A, Quinquis B, Sorokin A, et al. Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin. Microbiology , 2005, 151 (8) : 2551–2561. DOI:10.1099/mic.0.28048-0 |
| [9] | Barrangou R, Fremaux C, Deveau H, et al. CRISPR provides acquired resistance against viruses in prokaryotes. Science , 2007, 315 (5819) : 1709–1712. DOI:10.1126/science.1138140 |
| [10] | Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science , 2012, 337 (6096) : 816–821. DOI:10.1126/science.1225829 |
| [11] | Sapranauskas R, Gasiunas G, Fremaux C, et al. The Streptococcus thermophilus CRISPR/Cas system provides immunity in Escherichia coli. Nucleic Acids Res , 2011, 39 (21) : 9275–9272. DOI:10.1093/nar/gkr606 |
| [12] | Magadán A H, Dupuis M È, Villion M, et al. Cleavage of phage DNA by theStreptococcus thermophilus CRISPR3-Cas system. PLoS One , 2012, 7 (7) : e40913. DOI:10.1371/journal.pone.0040913 |
| [13] | Cong L, Ran F A, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems. Science , 2013, 339 (6121) : 819–823. DOI:10.1126/science.1231143 |
| [14] | Mali P, Yang L, Esvelt KM, et al. RNA-guided human genome engineering via Cas9. Science , 2013, 339 (6121) : 823–826. DOI:10.1126/science.1232033 |
| [15] | Ding Q, Regan Stephanie N, Xia Y, et al. Enhanced efficiency of human pluripotent stem cell genome editing through replacing TALENs with CRISPRs. Cell Stem Cell , 2013, 12 (4) : 393–394. DOI:10.1016/j.stem.2013.03.006 |
| [16] | Ni W, Qiao J, Hu S, et al. Efficient gene knockout in goats using CRISPR/Cas9 system. PLoS One , 2014, 9 (9) : e106718. DOI:10.1371/journal.pone.0106718 |
| [17] | Feng Z Y, Zhang B T. Efficient genome editing in plants using a CRISPR/Cas system. Cell Res , 2013, 23 (10) : 1229–1232. DOI:10.1038/cr.2013.114 |
| [18] | Hwang W Y, Fu Y, Reyon D, et al. Efficient genome editing in zebrafish using a CRISPR-Cas system. Nat Biotechnol , 2013, 31 (3) : 227–229. DOI:10.1038/nbt.2501 |
| [19] | Jinek M, East A, Cheng A, et al. RNA-programmed genome editing in human cells. Elife , 2013, 2 : e00471. |
| [20] | Wang H, Yang H, Shivalila C S, et al. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell , 2013, 153 (4) : 910–918. DOI:10.1016/j.cell.2013.04.025 |
| [21] | He X L, Yuan C, Chen Y L. Isolation, characterization, and expression analysis of FGF5 isoforms in Cashmere goat. Small Ruminant Research , 2014, 116 (2~3) : 111–117. |
| [22] | Ma K, Wang J, Shen B, et al. Efficient targeting of FATS at a common fragile site in mice through TALEN-mediated double-hit genome modification. Biotechnol Lett , 2014, 36 (3) : 471–479. DOI:10.1007/s10529-013-1387-z |
| [23] | 李辉, 施振旦. CRISPR/Cas9新型基因打靶系统的研究进展. 江苏农业学报 , 2013, 29 (4) : 907–911. Li H, Shi Z D. Research progress of gene targeting technology of CRISPR/Cas9 system. Jiangsu J of Agr Sci , 2013, 29 (4) : 907–911. |
 2016, Vol. 36
2016, Vol. 36




