文章信息
- 孙文怡, 张素芳, 林心萍, 栾雨时
- SUN Wen-yi, ZHANG Su-fang, LIN Xin-ping, LUAN Yu-shi
- 一种遗传转化方法在海洋红酵母(Rhodotorula mucilaginosa)中的应用
- Application of a Transformation in Rhodotorula mucilaginosa Genetic Engineering
- 中国生物工程杂志, 2016, 36(6): 81-86
- China Biotechnology, 2016, 36(6): 81-86
- http://dx.doi.org/10.13523/j.cb.20160611
-
文章历史
- 收稿日期: 2015-12-24
- 修回日期: 2016-01-28
2. 中国科学院大连化学物理研究所 大连 116023;
3. 大连工业大学食品学院 大连 116034;
4. 吉林师范大学生命科学学院 四平 136000
2. Dalian Institute of Chemical Physics, CAS, Dalian 116023, China;
3. School of Food Science and Technology, Dalian Polytechnic University, Dalian 116034, China;
4. School of Life Sciences, Jilin Normal University, Siping 136000, China
海洋红酵母(Rhodotorula mucilaginosa)属于细胞生物(cellular organisms);真核生物域(Eukaryota);真菌界(Fungi);半知菌亚门(Deuteromycotina),芽孢纲(Blastomycetes),隐球酵母目(Cryptococcales),红酵母属(Rhodotorula)真菌。R. mucilaginosa栖息范围广泛,从发酵牛奶、果实到大马哈鱼内脏、海岸、以及冰川等极端环境[1]都可生存,且具有较强的抗逆性,可用于生物表面活性剂生产、生物防治和环境治理,并应用于医药食品等领域。由于R. mucilaginosa菌体自身特点,尤其在脂肪酸和类胡萝卜素的生产上得到广泛的应用[2, 3]。微生物油脂种质资源丰富,其生产不受季节限制,安全性高,因此通过生物技术手段改造该菌来提高微生物油脂[4]及类胡萝卜素[3]的产量势在必行。
海洋红酵母富含类胡萝卜素,是类胡萝卜素生产的工业菌株,随着类胡萝卜素合成的相关基因已经明确[5],如何利用基因工程手段进行改造,是解决海洋红酵母类胡萝卜素产量问题的关键。由于缺少高效的转化方法,遗传操作的难度大大增加。大肠杆菌,酿酒酵母等模式微生物能够通过合成生物学手段被改造进行类胡萝卜素生产[6],但由于其菌体自身的局限性,无法达到理想的产量。R. mucilaginosa本身具有类胡萝卜素合成的代谢途径,通过从野生菌中分离诱变,优化发酵条件等方面[7]已经使类胡萝卜素产量提高。若想更深入地了解R. mucilaginosa,实现理性设计和改造,就需要对R. mucilaginosa进行基因操作。
R. mucilaginosa没有内源质粒,前期工作使用常规的电转化[8, 9],化学转化法[10, 11]及其他转化法[12, 13]均未能实现对R. mucilaginosa的改造,也未查询到国内外对于其转化方法的研究文献,随着本年度公布的R. mucilaginosa基因组草图[14],海洋红酵母的基因操作研究将迎来崭新的阶段,但目前还没有完整的基因注释,为构建R. mucilaginosa的遗传操作系统增加了难度。目前,R.mucilaginosa既没有高效的遗传转化方法进行基因修饰,又无法通过基因组挖掘内源功能元件,使得R.mucilaginosa的进一步研究遇到瓶颈。农杆菌介导转化是一种来源于植物的转化方法,近年来逐步应用到酵母和真菌中[15, 16],由于其转化效率高,过程简单,稳定遗传等优点,已经逐步代替了其他的转化方法。2014年,Lin(本实验室林心萍博士)等[17]对圆红冬孢酵母通过农杆菌介导转化的方法成功建立其遗传操作体系,本文拟利用其分离自Rhodotorula toruloides NCYC1585并在红冬孢酵母属酵母中验证了功能的启动子等元件,尝试建立R. mucilaginosa的遗传操作方法。
1 材料与方法 1.1 菌株,质粒和培养基海洋红酵母(R. mucilaginosa CGMCC 2.1515)购于中国普通微生物菌种保藏管理中心,农杆菌菌株[18](Agrobacterium tumefaciens AGL1:AGL0 recA∷bla pTiBo542ΔT Mop+CbR)为本实验室保存;质粒PZPK-pGPD-HYG-Tnos,由本实验室林心萍博士构建,PZPK是在pTHR1[19]的基础上通过restriction free (RF)克隆[20]替换aadA基因为kan,并含有潮霉素抗性基因[17] (图 1)。由于R. mucilaginosa基因注释信息的限制,所用的潮霉素抗性基因是根据同科的圆红冬孢酵母密码子使用偏好性进行优化,并委托上海生工生物工程技术服务有限公司进行全基因合成(KF826486),启动子gpd (KF826485)来源于R. toruloides NCYC 1585的3-磷酸-甘油醛脱氢酶启动子[21],终止子来源于pBI121的nos终止子;YEPD液体培养基:葡萄糖20 g/L,蛋白胨10 g/L,酵母浸粉10 g/L,pH 6.0;相应的固体培养基添加15 g/L琼脂粉,YEPDH为带有50 μmol/L潮霉素的YEPD培养基。LB液体培养基:酵母浸粉5 g/L,胰蛋白胨10 g/L,氯化钠10 g/L,pH 7.0;相应的固体培养基添加15 g/L琼脂粉。IM诱导培养基[22]:MM 盐溶液(磷酸氢二钾10 mmol/L,磷酸二氢钾10mmol/L,氯化钠2.5 mmol/L,硫酸镁2 mmol/L,氯化钙0.7 mmol/L,七水硫酸铁9 μmol/L,硫酸铵4mmol/L,葡萄糖10 mmol/L,调节pH至7.0),甘油0.5% (w/v),乙酰丁香酮200 μmol/L,4-吗啉乙磺酸 (pH 5.3) 40 mmol/L,琼脂2.0%。
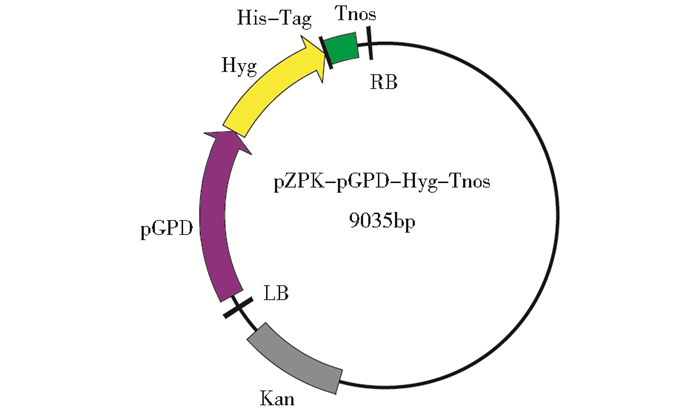
|
| 图 1 PZPK-pGPD-HYG-Tnos质粒图 Fig. 1 Binary vector PZPK-pGPD-HYG-Tnos pGPD: glyceraldehyde- 3-phosphate dehydrogenase promoter; hyg: hygromycin gene; LB: left border; RB: right border; Tnos: nos terminator from the pBI121 vector |
详见参考文献17。
1.3 农杆菌介导转化至酵母细胞取一环活化后的R. mucilaginosa,接于5 ml YEPD中,30℃,200 r/min过夜培养。用无菌水洗涤一遍后,调节至OD600nm=0.4~0.6,备用。农杆菌工程菌株活化后,接于5 ml含有卡那霉素(50 μg/ml) 和利福平(50μg/ml) 的LB液体中,30℃,200 r/min培养过夜。用无菌水洗涤一遍,调节至OD600nm=0.4~0.6,备用。
取上述酵母及农杆菌稀释液各100 μl,混匀,涂于IM诱导平板的膜上,分别采用滤纸膜和纤维素薄膜作为介质,24~25℃,培养2天。待膜上出现红色菌苔时将膜转移至筛选平板YEPDH上,于30℃进行培养,直至转化子出现。
1.4 转化子的鉴定表型鉴定
从筛选平板上挑取转化子划线于新的筛选平板YEPDH上,进一步确定表型,以同时划线海洋红酵母野生型作为对照。随后挑取上述验证平板上生长的菌株,接入含有50 μg/ml潮霉素的YEPD中培养,再次确认表型。
基因型鉴定
酵母基因组模板的制备方法参考文献[23]。提取的转化子的基因组采用GPD-F&HYG-R引物进行扩增,同时以海洋红酵母的基因组作为负对照,以其中一个以构建成功双元载体为正对照。PCR引物为:5′-Primer: GCATGATCCGACCGAATCTGCAG,3′-Primer:CTAGTGATGGTGATGGTGGTG。扩增条件:95℃ 10 min;95℃ 20 s,62℃ (每个循环下降1℃) 20 s,72℃ 1.5 min,10个循环;95℃ 20 s,60℃ 20 s,72℃ 1.5 min,25个循环;72℃ 10 min,4℃结束反应。
1.5 转化子表达水平验证转录水平的表达分析
首先进行RNA提取,采用MP Biomedicals公司的FastRNATM试剂盒,样品离心(5 000×g,10min,4℃),1 ml RNA Pro溶液重悬浮,转移到含有0.1 mm硅珠裂解介质B管中。裂解介质管在FastPrep-24TM仪器上处理5次(3×45 s,6.5 米/秒,循环之间2min冰上冷却),离心,上清液转移至新的离心管中,室温放置5 min,再加入300 μg氯仿,涡旋振荡10 s室温放置5 min后离心,上清再次转移到新的离心管中,加入500μl冰冷的无水乙醇,颠倒混匀,-20℃放置,离心后用DEPC水配制的75 %乙醇重悬,离心去除乙醇,将RNA重悬于DEPC水中。提取后的RNA进行反转录,用TaKaRa的PrimeScriptTM II High Fidelity RT-PCR Kit试剂盒进行反转录,设计引物进行PCR转录水平分析。
Western blot鉴定蛋白水平的表达参考文献[17]。
基因型验证正确的转化子培养24h,直接采用物理机械破碎方法对R. mucilaginosa细胞壁进行破碎处理,分别对全菌体和上清进行SDS-PAGE电泳分离,采用Western blot技术,特异结合潮霉素蛋白C末端的His-tag抗体进行免疫印迹检测。
1.6 转化子稳定性验证将转化子划线于YEPD平板30℃培养,至菌落长起后,再取划线外缘的菌落划线于新鲜YEPD平板上。如此传接5代后,将菌重新划线于含有50 μg/ml潮霉素的YEPD筛选平板上,30℃培养,观察菌体生长情况。
2 结 果 2.1 农杆菌介导转化至R. mucilaginosa我们首先合并诱导培养和共培养的步骤,将在LB/YEPD中已活化好的农杆菌和酵母菌混合,将两种活化的菌株混合,涂布于承载介质上,置于诱导平板上共培养。为了避免相同基因型的菌株重复产生,为后续的T-DNA随机插入筛选增加难度,采用两种承载介质进行比较,滤纸和纤维素薄膜。将承载介质连同生长的菌体一同转移到筛选平板上。生长两天后,结果如图 2所示。两种农杆菌工程菌株与海洋红酵母结合共转化后,均能够在含有潮霉素的平板上成功获得了转化子,而没有和农杆菌工程菌株互相作用的海洋红酵母(WT)不能在筛选平板上生长出转化子。图 2中的Sample 1和Sample 2分别为菌体在具有潮霉素抗性的筛选平板上的生长情况,从图 2可以看出滤纸(Sample 1)由于渗透力比较强,共培养时没有完全晕开,导致菌落生长过于集中,而纤维素薄膜(Sample 2)表观上要优于Sample 1。随机挑取7个单菌落划线于YEPD潮霉素抗性平板进行表型验证(Phenotype),负对照为野生型的海洋红酵母,并对其中的1,2,3,4进行基因组(gDNA)提取,并对gDNA进行PCR验证,图 3结果显示,1,2有正确大小的阳性条带。
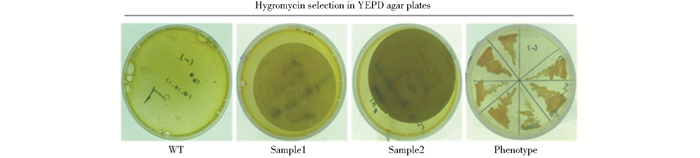
|
| 图 2 R. mucilaginosa农杆菌介导转化表型验证结果 Fig. 2 ATMT results of R. mucilaginosa with the HYG transformation and phenotype confirmation results WT: R. mucilaginosa wild type strain; Sample1: Co-cultured on IM plates with filter paper; Sample2: Co-cultured on IM plates with cellulose acetate film |
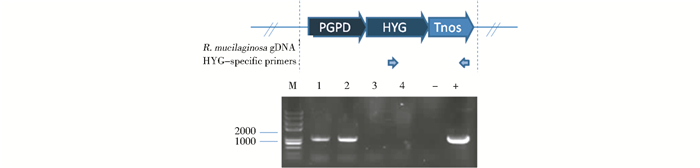
|
| 图 3 R. mucilaginosa的菌落PCR结果 Fig. 3 Primer sets to detect insertion and genotyping results of the hyg casette region-targeted R. mucilaginosa (Lane 1~4) -: gDNA of R. mucilaginosa wild type strain; +: Plamid PZPK-GPD-Hyg-Tnos; M: DNA Marker |
转录水平表达
从图 4(a)中可以看出,通过PCR验证,转录水平都有表达。

|
| 图 4 cDNA PCR及Western blot 检测转化子中的潮霉素蛋白表达 Fig. 4 cDNA PCR(a) and Western blot(b) analysis of the expression of HYG in transformed R. mucilaginosa (a) Lane 1, 2 represent right transformants individual; Lane (-): Total cDNA sample from R. mucilaginosa; Lane (+): A vector with hyg gene; Lane M: DNA marker (b) Lane 1, 2 represent right transformants individual (include whole cell lysate and supernatant) ; Lane WT: Total protein sample from R. mucilaginosa; Lane M: Protein marker |
Western blot用于分析抗性基因蛋白质是否表达,从图 4(b)的Western blot结果中可以看出经过特异性免疫印迹反应,转化子中都含有HYG特异性条带。
2.3 转化子的稳定性分析经鉴定后的潮霉素转化子,在无抗性筛选压力的培养基上连续培养4代(Ⅰ,Ⅱ,Ⅲ,Ⅳ)后,将转化子1,2分别在菌落边缘取菌块重新划线至含有潮霉素的筛选平板上,以野生型菌株为负对照(-)。试验表明,所有的转化子经过4代连续无筛选压力转接后,均能重新在筛选平板(Ⅴ)上生长(图 5)。利用ATMT方法所获得的转化子具有高遗传稳定性。

|
| 图 5 潮霉素的稳定性验证 Fig. 5 Certification of the transfermants phenotypic stability The transformants were tested by culturing on YEPD plates without antibiotics for four generations(Ⅰ, Ⅱ, Ⅲ, Ⅳ). Subsequently, clones were transferred to YEPD plates containing appropriate selection antibiotics(Ⅴ). Lane (-):R. mucilaginosa; Lane 1, 2 represent right transformants individual |
R. mucilaginosa是典型的微生物油脂和类胡萝卜素生产菌株,通过设计代谢途径对其分子机制的探讨,可操作性强的遗传工具是必要的。不同的转化方法都有其优势和局限性,农杆菌介导转化效率高,在酵母中已经成为一种常规的遗传操作方法,但也存在着个体差异,如:转化时间,菌株生长状态等。提高农杆菌的转化效率,将极大提高此转化方法的应用范围。
3.2 表达元件的选择对来源于圆红冬孢酵母单倍体np11[24]的启动子,终止子及根据np11密码子优化的博来霉素抗性基因ble表达盒质粒也分别进行转化,都没有得到正确的转化子,同时也充分证明了使用内源的基因及表达元件要优于外源的,这与文献[25]中启动子的比较得出一致的结论。随着海洋红酵母基因组测序的完成[14],进行基因注释后通过密码子优化或调取内源启动子和终止子,再通过实验优化进一步提高ATMT的转化效率,使得农杆菌介导转化法在海洋红酵母中能够作为一种有力的转化手段。
4 结 论本文采用一种新的遗传转化方法——农杆菌介导转化对R. mucilaginosa菌株进行应用,成功将外源基因插入了染色体基因组中,并建立其遗传转化体系;通过对转化子表型、基因型、蛋白水平的验证,证明其稳定表达具有遗传稳定性。这对研究海洋红酵母基因功能,代谢途径改造,以及构建海洋红酵母完善的遗传操作平台提供了研究基础。
| [1] | Raggi P, Lopez P, Diaz A, et al. Debaryomyces hansenii and Rhodotorula mucilaginosa comprised the yeast core gut microbiota of wild and reared carnivorous salmonids, croaker and yellowtail. Environ Microbiol, 2014, 16(9): 2791-2803. |
| [2] | Li M, Liu G L, Chi Z, et al. Single cell oil production from hydrolysate of cassava starch by marine-derived yeast Rhodotorula mucilaginosa TJY15a. Biomass Bioenerg, 2010, 34(1): 101-107. |
| [3] | Aksu Z, Eren A T. Carotenoids production by the yeast Rhodotorula mucilaginosa: Use of agricultural wastes as a carbon source. Process Biochem, 2005, 40(9): 2985-2991. |
| [4] | 赵宗保. 加快微生物油脂研究为生物柴油产业提供廉价原料. 中国生物工程杂志, 2005, 25(2): 8-11. Zhao Z B. Toward cheaper microbial oil for biodiesel oil. China Biotechnology, 2005, 25(2): 8-11. |
| [5] | 陶俊, 张上隆, 徐昌杰,等. 类胡萝卜素合成的相关基因及其基因工程. 生物工程学报, 2002, 18(3): 276-281. Tao J, Zhang S L, Xu C J, et al. Gene and gene engineering of carotenoid biosynthesis. Chinese Journal of Biotechnology, 2002, 18(3): 276-281. |
| [6] | Steen E J, Kang Y S, Bokinsky G, et al. Microbial production of fatty-acid-derived fuels and chemicals from plant biomass. Nature, 2010, 463 (7280): 559-562. |
| [7] | 刘雅婷,刘宏娟,王艳萍. 常压室温等离子体诱变粘红酵母筛选高产油脂菌株及发酵条件优化. 中国油脂, 2015, 40(1): 83-87. Liu Y T, Liu H J, Wang Y P. Screening of high lipid production strain from Rhodotorula glutinis mutagenized by atmospheric and room temperature plasma and optimization of fermentation condition. China Oils and Fats, 2015, 40(1): 83-87. |
| [8] | Karube I, Tamiya E, Matsuoka H. Transformation of Saccharomy cescerevisiae spheroplasts by high electric pulse. FEBS Lett, 1985, 182(1): 90-94. |
| [9] | Hashimoto H, Morikawa H, Yamada Y, et al. A novel method for transformation of intact yeast-cells by electroinjection of plasmid DNA. Appl Microbiol Bio, 1985, 21(5): 336-339. |
| [10] | Meyer V. Genetic engineering of filamentous fungi-progress, obstacles and future trends. Biotechnol Adv, 2008, 26(2): 177-185. |
| [11] | Gietz R D, Woods R A. Genetic transformation of yeast. Biotechniques, 2001, 30(4): 816-818. |
| [12] | Kuck U, Hoff B. New tools for the genetic manipulation of filamentous fungi. Appl Microbiol Biotechnol, 2010, 86(1): 51-62. |
| [13] | Schiestl R H, Petes T D. Integration of DNA fragments by illegitimate recombination in Saccharomyces cerevisiae. Proc Natl Acad Sci U S A, 1991, 88(17): 7585-7589. |
| [14] | Deligios M, Fraumene C, Abbondio M, et al. Draft genome sequence of Rhodotorula mucilaginosa, an emergent opportunistic pathogen. Genome Announc, 2015, 3(2): e00201-15. |
| [15] | Meyer V. Genetic engineering of filamentous fungi-progress, obstacles and future trends. Biotechnol Adv, 2008, 26(2): 177-185. |
| [16] | Michielse C B, Hooykaas P J, van den Hondel C, et al. Agrobacterium-mediated transformation as a tool for functional genomics in fungi. Current Genetics, 2005, 48(1): 1-17. |
| [17] | Lin X P, Wang Y N, Zhang S F, et al. Functional integration of multiple genes into the genome of the oleaginous yeast Rhodosporidium toruloides. FEMS Yeast Res, 2014, 14(4): 547-555. |
| [18] | Lazo G R, Stein P A, Ludwig R A. A DNA transformation-competent arabidopsis genomic library in Agrobacterium. Nat Biotechnol (NY), 1991, 9(10): 963-967. |
| [19] | Ji L H, Jiang Z D, Liu Y B, et al. A simplified and efficient method for transformation and gene tagging of Ustilago maydis using frozen cells. Fungal Genet Biol, 2010, 47(4): 279-287. |
| [20] | van den Ent, J Löwe. Rf cloning: A restriction-free method for inserting target genes into plasmids. J Biochem Bioph Meth, 2006, 67(1): 67-74. |
| [21] | Liu Y B, Koh C M J, Sun L H, et al. Characterization of glyceraldehyde-3-phosphate dehydrogenase gene rtgpd1 and development of genetic transformation method by dominant selection in oleaginous yeast Rhodosporidium toruloides. Appl Microbiol Biotechnol, 2013, 97(2): 719-729. |
| [22] | Bundock P, den Dulk-RasA, Beijersbergen A, et al. Trans-kingdom T-DNA transfer from Agrobacterium tumefaciens to Saccharomyces cerevisiae. EMBO J, 1995, 14(13): 3206-3214. |
| [23] | Lin X P, Yang F, Zhou Y J, et al. Highly-efficient colony PCR method for red yeasts and its application to identify mutations within two leucine auxotroph mutants. Yeast, 2012, 29(11): 467-474. |
| [24] | Zhu Z W, Zhang S F, Liu H W, et al. A multi-omic map of the lipid-producing yeast Rhodosporidium toruloides. Nat Commun, 2012, 3:1112. |
| [25] | Wang Y N, Lin X P, Zhang S F, et al. Cloning and evaluation of different constitutive promoters in the oleaginous yeast Rhodosporidium toruloides.Yeast, 2015, DOI: 10.1002/yea.3145. |
 2016, Vol. 36
2016, Vol. 36




