文章信息
- 陈青, 朱鸿飞, 郭晓宇.
- CHEN Qing, ZHU Hong-fei, GUO Xiao-yu.
- DNA模式识别受体研究进展
- Progress on DNA Innate Immune Recognition Receptors
- 中国生物工程杂志, 2016, 36(5): 112-117
- China Biotechnology, 2016, 36(5): 112-117
- http://dx.doi.org/DOI:10.13523/j.cb.20160516
-
文章历史
- 收稿日期: 2015-12-09
- 修回日期: 2016-02-03
2. 中国农业科学院北京畜牧兽医研究所 北京 100193
2. Institute of Animal Sciences, Chinese Academy of Agricultural Sciences, Beijing 100081, China
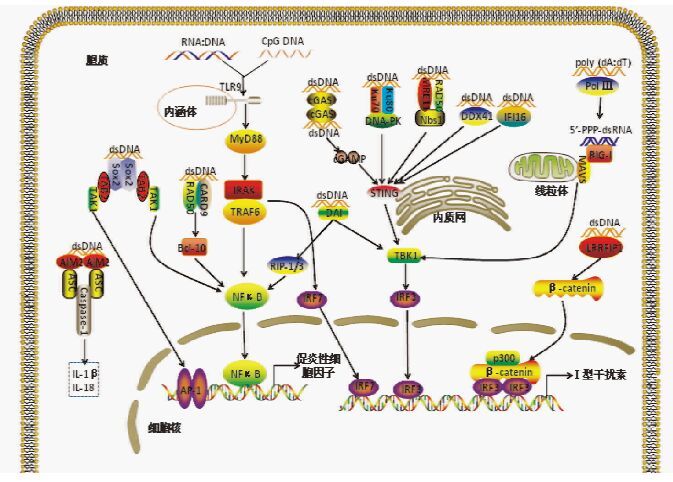
作为一种病原相关分子模式(pathogen-associated molecular pattern,PAMP),外源DNA能够被细胞中的某些模式识别受体(pattern recognition receptor,PRR)识别,激活固有免疫应答信号通路,诱导并上调Ⅰ型干扰素和促炎性细胞因子基因的转录和表达,从而启动天然免疫防御机制,同时促进产生获得性免疫应答[1-2]。 继2000年Hemmi等[3]研究发现DNA胞膜受体——Toll样受体9(toll-like receptor 9,TLR9)之后,迄今为止,共有10余种DNA胞质受体先后被报道,其中包括DNA 依赖的干扰素调节因子激活物(DNA-dependent activator of IFN-regulatory factor,DAI)、干扰素刺激基因(stimulator of interferon gene,STING)、RNA 聚合酶Ⅲ (RNA polymerase III,Pol III)、富亮氨酸重复序列相互作用蛋白1 (Leucine-rich repeat flightless-interacting protein 1,LRRFIP1)、AIM2样蛋白(AIM2-like receptors,ALR)家族、DExD/H解旋酶家族、DNA依赖性蛋白激酶(DNA dependent protein kinase,DNA-PK)、减数分裂重组蛋白11同系物A(meiotic recombination 11 homolog A,MRE11)、环鸟苷酸-腺苷酸合成酶(cyclic GMP-AMP synthase,cGAS)、DNA修复相关蛋白Rad50及转录因子Sox2(sry-related HMG box 2)[4-5]。以上DNA模式识别受体及其介导的信号通路如图 1所示。
|
| 图 1 DNA模式识别受体及其信号通路 Figure 1 DNA innate immune recognition receptors and their signaling pathways |
根据DNA受体在细胞内所处位置的不同,可将其分成两类,第一类命名为胞膜DNA受体,包括TLR9;第二类命名为胞质DNA受体,这一类受体成员较多,包括除TLR9外的其它DNA受体。本文先对两类受体的研究进展分别进行介绍,重点阐述受体介导的固有免疫应答所涉及的信号通路;最后还讨论了DNA的免疫识别与自身免疫病之间的关系。
1 胞膜DNA受体TLR9介导的免疫识别研究进展TLR9是最早被发现的细胞DNA受体,也是迄今为止唯一识别DNA的胞膜受体。TLR9主要表达于浆细胞样树突细胞(plasmacytoid dendritic cell,pDC)和B细胞中,在静息状态下位于细胞内质网上[6]。经CpG DNA诱导,TLR9由内质网转位到内涵体,跨膜蛋白UNC93B1 和高尔基体在转位过程中起了重要的作用。随着内涵体的酸化,TLR9在内涵体中被蛋白水解酶剪切,剪切后的TLR9具备了信号转导功能[7]。
TLR9能够识别微生物及哺乳动物基因组中的CpG DNA序列,以及存在于细胞中的RNA:DNA 杂合体,激活依赖于MyD88分子的信号通路,刺激细胞上调表达Ⅰ型干扰素和促炎性细胞因子,进而调节适应性免疫[8]。近期研究表明,TLR9在内涵体中与CpG DNA形成2∶2的分子复合物,CpG DNA通过磷酸二酯键骨架和碱基同时与两分子的TLR9相互作用;不含CpG基序的抑制性DNA主要以骨架同TLR9相互作用,形成单体。因此,免疫刺激序列(immunostimulatory sequence,ISS)中往往需要加入CpG基序以增强其与TLR9的亲和性[9]。
2 胞质DNA受体介导的免疫识别研究进展通过使用TLR9拮抗剂或TLR9-/-小鼠的研究表明,细胞中还存在着其它DNA受体。自2007年首个DNA胞质受体DAI被发现后,先后有10余种胞质受体被报道,但是关于上述胞质受体通过何种机制诱导产生Ⅰ型干扰素,尚未达成共识。本文对已发现的DNA胞质受体分别进行阐述。
2.1 DAI2007年,日本学者研究发现胞质蛋白DLM-1(又名ZBP1)能够识别胞质DNA,激活IRF3并上调Ⅰ型干扰素的表达,因此将这种蛋白质命名为DNA 依赖的干扰素调节因子激活物 (DAI) [10-11]。进一步的研究表明,DAI对于长度小于100bp的dsDNA仅有很小的反应性,因此DAI对胞质DNA的识别取决于dsDNA的长度,而非序列特异性[12]。
DAI受体在其N端含有Z-α和Z-β结构域,能够结合Z型DNA;受体的D3结构域与Z-β结构域相邻,能够结合B型DNA。DAI通过C端与TBK1和IRF3相互作用形成复合体,介导Ⅰ型干扰素的产生[10-11]。同时,DAI可通过C端与RIP1和RIP3形成复合物,激活NF-κB介导的信号通路,上调多种细胞因子表达,并诱导宿主细胞的程序性坏死[13]。然而,用DNA刺激DAI基因缺失的MEF细胞系或小鼠,Ⅰ型干扰素的分泌并未受到影响,这提示细胞中还有其它DNA识别受体存在[14-15]。
2.2 AIM2和ALR家族除了诱导Ⅰ型干扰素和细胞因子的产生,胞质DNA还能够激活NLRP3非依赖性的炎性复合体,表明胞质中存在其它DNA受体。2009年,多家研究机构几乎同时报道发现黑素瘤缺乏因子2(absent in melanoma 2,AIM2)。AIM2属PYHIN蛋白家族成员,分子结构中包含1个HIN200结构域和1个Pyrin结构域;HIN200结构域能够识别dsDNA,促使AIM2寡聚化和纤维化,Pyrin结构域则介导招募ASC,进而激活caspase 1。活化的caspase 1在IL-1β和IL-18的成熟中具有重要作用[16-20]。
人IFI16及其在小鼠中的同源蛋白p204同属于PYHIN蛋白家族成员,分子结构中包含2个HIN结构域(HIN-A和HIN-B)和1个Pyrin结构域;IFI16能够识别多种病毒的dsDNA,通过STING-TBK1-IRF3 信号通路上调IFN-β的表达。近期研究表明,IFI16在特定条件下能够激活炎性复合体,并介导IFI16依赖性的细胞焦亡[21-22]。
基于上述特点,AIM2、IFI16和p204共同组成了ALR家族,通过HIN结构域结合dsDNA。ALR家族识别DNA不需要后者的序列特异性,仅通过静电作用和DNA骨架相互作用。然而,由于IFI16(p204)主要分布在细胞核中,少量存在于细胞质,其如何区分细胞核DNA和病原DNA,仍有待于进一步研究[23]。有体外研究表明,IFI16需结合150bp左右的裸dsDNA才能实现多聚化并产生活性,科研人员推测IFI16通过“感知”裸dsDNA的长度来区分自身DNA和非自身DNA,因为细胞核DNA通常和组蛋白结合,避免了同IFI16的直接接触[24]。
2.3 RNA聚合酶ⅢRNA聚合酶Ⅲ是由多基因编码的寡聚蛋白,在细胞质和细胞核中均有分布,其生物学功能包括tRNA、5S RNA和7SLRNA等小RNA的转录、合成与加工。2009年,两组科研人员分别报道了RNA聚合酶Ⅲ是一种新型的胞质DNA“感受器”[25-26]。研究发现,RNA聚合酶Ⅲ能够结合胞质中的poly (dA:dT),无需依赖启动子而转录生成5′-3P dsRNA。这种RNA作为RIG-I 的配体,通过激活RIG-I-MAVS信号通路上调Ⅰ型干扰素的表达。
然而,RNA聚合酶Ⅲ对poly (dA:dT)的识别仅限于在人的细胞中,在小鼠细胞中并不存在这种DNA识别机制。因此,该信号通路的广谱性、调控机制及其对病原体DNA的免疫识别作用仍需进一步验证[25]。
2.4 LRRFIP1LRRFIP1属于钙结合微丝蛋白家族成员,最早发现于果蝇体内,在果蝇胚胎发育和肌肉形成期间起重要作用[27]。LRRFIP1主要存在于细胞质中,能够结合dsRNA及富含GC的dsDNA。进一步研究表明,LRRFIPl亦存在于多种动物的组织细胞中,在动物胚胎发育、表皮生长、平滑肌细胞的发育,以及肿瘤和心血管病的发病中发挥重要作用[28-30]。
2010年,我国学者曹雪涛等研究发现LRRFIP1是一种胞质核酸的新型模式识别受体。该研究表明,LRRFIP1识别dsDNA或dsRNA后,促进β-catenin的磷酸化;活化的β-catenin与IRF3结合,招募更多p300乙酰转移酶到IFN-β转录增强体,上调IFN-β的表达水平[31]。这些研究结果证明LRRFIPl在对单增李斯特菌及水疱性口炎病毒的感染产生Ⅰ型干扰素应答过程中起重要作用。
2.5 DExD/H解旋酶家族目前为至,研究发现该家族中共包括59种蛋白质,其成员RIG-I、MDA5、LGP2及DDX1/9/21/33/36介导胞质RNA的识别;DDX29和DDX60既能够识别胞质RNA,也能够识别胞质DNA,并通过RIG-I-MAVS信号通路上调Ⅰ型干扰素表达; DHX36和DHX9在TLR9介导的CpG DNA识别中起重要作用[32-33]。DHX36主要介导浆细胞样树突细胞中IRF7的核转位及IFN-α的产生,而DHX9能够识别B型CpG DNA,刺激细胞上调TNF-α和IL-6,同时激活NF-κB信号通路。
DDX41属于DExD/H解旋酶家族成员之一,体外实验研究表明,DDX41通过其DEADc结构域能够识别胞质中的dsDNA和Ⅰ型单纯疱疹病毒,进而结合STING,激活TBK1- IRF3信号通路和NF-κB信号通路,上调IFN-β的表达[34]。另有文献报道,DDX41能够结合来源于细菌的c-di-GMP或 c-di-AMP,通过STING-TBK1-IRF3信号通路上调Ⅰ型干扰素的表达。亲和力分析实验表明,c-di-GMP或 c-di-AMP结合所需的是DDX41上的中央DExD/H box结构域而不是解旋酶结构域,而DDX41如何进一步同STING相互作用发挥下游功能,仍需通过蛋白质结构分析和基因敲除技术继续探索[35]。
2.6 DNA-PK复合物DNA-PK复合物是一种DNA损伤反应蛋白,属于磷脂酰肌醇-3激酶相关激酶家族,由Ku70、Ku80及DNA-PK催化亚基组成。DNA-PK复合体在细胞中具有多种生物学功能,其中包括免疫细胞V(D)J重组、稳定端粒结构和受损DNA修复等。在静息HEK293T细胞中,DNA-PK复合物存在于细胞质,利用生物素化的DNA进行亲和力下拉试验,证明DNA-PK复合体是一个潜在的DNA传感器。进一步研究显示,缺乏DNA-PK复合物的小鼠成纤维细胞或基因缺失小鼠在接触外源DNA或DNA病毒感染时,I型干扰素和促炎性细胞因子的表达均有减少[36]。
最初研究表明,Ku70的主要功能涉及DNA双链断处的修复,2011年研究人员发现Ku70能够识别胞质中长度500bp以上的dsDNA,进而激活IRF1和IRF7,上调 III型干扰素IFN-λ1的表达[37]。2012年另一组科研人员发现DNA-PK复合物能够识别小鼠胚胎成纤维细胞质中的dsDNA,激活STING-TBK1- IRF3信号通路,上调Ⅰ型干扰素的表达。同时,该研究证明DNA-PK在依赖于DNA的NF-κB信号通路中并不发挥作用,表明细胞中还存在着其它DNA识别途径[36]。
2.7 Mre11和Rad50Mre11是存在于细胞中的另一种DNA损伤反应蛋白,MRE11的N端含有4个磷酸酯酶结构域,邻侧含有2个DNA结合结构域。体外实验表明,MRE11具有单链和双链内切核酸酶的活性及3′→5′外切核酸酶活性[38]。Mre11、RAD50和NBS1形成MRN复合物,在DNA复制、DNA损伤感应、端粒维持、保证染色体的稳定性等过程中具有重要作用[39]。
近期研究表明,Mre11具有识别胞质DNA的能力。MRE11结合胞质中的dsDNA,招募RAD50从细胞核移位到细胞质,形成dsDNA-MRE11-RAD50复合体,进而激活STING-TBK1- IRF3信号通路[40]。最新研究表明,RAD50可同CARD9形成复合体,识别细胞质中的dsDNA和病毒DNA,通过招募Bcl-10激活NF-κB信号通路,上调pro-IL-1β的表达[41]。
2.8 cGAS-STING-TBK1识别通路2013年cGAS的发现,为DNA胞质受体家族增添了新成员,cGAS也被认为在介导胞质DNA识别中起关键作用。对人cGAS分子的晶体结构和生化分析表明,未活化的cGAS以自抑制形式(autoinhibited form)的二聚体存在;当外源或内源DNA进入细胞质后,每个cGAS都能识别并结合一个DNA分子,从而形成2∶2的cGAS-DNA复合物;与DNA结合后,cGAS分子构象发生变化,在其核苷酸转移酶结构域(nucleotidyltransferase domain,NTase domain)形成催化位点,结合并催化ATP、GTP生成cGAMP(cyclic GMP-AMP)[42-43]。
cGAMP与内质网膜上的STING分子二聚体结合,使后者的构象发生改变而活化[44-45]。活化的STING从内质网迁移至高尔基体,然后停留在位于核周的内涵体上。STING通过其C端结构域结合并磷酸化TBK1;随后,STING-TBK1复合体进入内涵体,结合并激活IRF3;活化的IRF3进入细胞核,启动先天免疫相关基因的转录[46]。有研究表明,STING还可以激活NF-κB介导的信号通路,但其中的机制尚未阐明[47]。
2.9 Sox2Sox2是Sox转录因子家族成员之一,后者在动物界中广泛存在,迄今为止,在哺乳动物中已有20个以上SOX蛋白及其编码基因被相继发现。早期研究发现,Sox2在胚胎发生、神经分化和晶状体发育等过程中都起着关键作用[48]。
2015年,中国学者发现在人中性粒细胞中Sox2蛋白能够通过分子中的HMG结构域识别单核细胞增生李斯特菌基因组中特定序列的DNA序列,导致Sox2蛋白二聚化,进而与TAB 2、TAK1结合,二聚化并激活TAK1,TAK1激活下游信号分子MKK4/7和 IKKa/b,再分别通过JNK和NF-κB信号通路,上调促炎性细胞因子的表达[49-50]。
3 DNA的免疫识别与自身免疫病的关系鉴于多种胞质DNA受体对外源及自身DNA具有无差异识别作用,细胞采用多种措施防止因自身DNA识别而造成的自身免疫病。某些DNA受体仅识别外源DNA中的特定序列,如TLR9和Sox2;细胞器的膜结构实现了对受体和自身DNA进行有效的隔离,细胞核中的组蛋白也对基因组DNA进行了有效的保护,避免DNA受体的免疫识别;细胞利用DNase II和DNase III对细胞质中的DNA进行有效降解,使其浓度低于受体感知的阈值[51]。
在人体中,DNase III的突变失活会导致细胞质中DNA片段的大量积累,进而激活STING介导的DNA识别通路,引发Ⅰ型干扰素相关自身免疫病;同样,在小鼠中的研究表明,缺失DNase II基因的小鼠会在胚胎发育阶段由于Ⅰ型干扰素大量产生而造成死亡;经Ⅰ型干扰素受体缺失拯救的小鼠,发育早期也会因其它炎性因子的过量表达而发生多发性关节炎。通过STING分子缺失,上诉症状能够得到缓解乃至清除,说明STING介导的DNA识别在上述疾病致病机制中起重要作用[52]。
4 展 望近10年来,在固有免疫系统对病原DNA的识别领域取得了巨大研究进展。胞质DNA受体介导的识别信号通路也被进一步阐明,cGAS-STING-TBK1信号通路的核心作用得到了广泛认证。然而,其中仍有很多机制有待于进一步研究。未来的研究工作应重点阐明其它DNA受体如何与STING分子相互作用,以及诸多信号通路中的调节机制,尤其是负调节机制。上诉机制的阐明也必将对病毒与宿主的相互作用机制、免疫逃逸、新型疫苗和佐剂研发及自身免疫病的治疗方面提供有力的理论指导作用。
| [1] | Akira S, Uematsu S, Takeuchi O. Pathogen recognition and innate immunity. Cell,2006, 124 (4) : 783 –801. |
| [2] | Takeuchi O, Akira S. Pattern recognition receptors and inflammation. Cell,2010, 140 (6) : 805 –820. |
| [3] | Hemmi H, Takeuchi O, Kawai T, et al. A Toll-like receptor recognizes bacterial DNA. Nature,2000, 408 (6813) : 740 –745. |
| [4] | Dempsey A, Bowie A G. Innate immune recognition of DNA: A recent history. Virology,2015, 479 : 479 –480. |
| [5] | Paludan S R. Activation and regulation of DNA-driven immune responses. Microbiol Mol Biol Rev,2015, 79 (2) : 225 –241. |
| [6] | Yasuda K, Richez C, Uccellini M B, et al. Requirement for DNA CpG content in TLR9-dependent dendritic cell activation induced by DNA-containing immune complexes. J Immunol,2009, 183 (5) : 3109 –3117. |
| [7] | Kawasaki T, Kawai T. Toll-like receptor signaling pathways. Front Immunol,2014, 5 : 461 –468. |
| [8] | Jensen S B, Paludan S R. Sensing the hybrid-a novel PAMP for TLR9. EMBO J,2014, 33 (6) : 529 –530. |
| [9] | Ohto U, Shibata T, Tanji H, et al. Structural basis of CpG and inhibitory DNA recognition by toll-like receptor 9. Nature,2015, 520 (7549) : 702 –705. |
| [10] | Takaoka A, Wang Z, Choi M K, et al. DAI (DLM-1/ZBP1) is a cytosolic DNA sensor and an activator of innate immune response. Nature,2007, 448 (7152) : 501 –505. |
| [11] | Kaiser W J, Upton J W, Mocarski E S. Receptor-interacting protein homotypic interaction motif-dependent control of NF-kappa B activation via the DNA-dependent activator of IFN regulatory factors. J Immunol,2008, 181 (9) : 6427 –6434. |
| [12] | Wang Z, Choi M K, Ban T, et al. Regulation of innate immune responses by DAI (DLM-1/ZBP1) and other DNA-sensing molecules. Proc Natl Acad Sci USA,2008, 105 (14) : 5477 –5482. |
| [13] | Upton J W, Kaiser W J, Mocarski E S. DAI/ZBP1/DLM-1 complexes with RIP3 to mediate virus-induced programmed necrosis that is targeted by murine cytomegalovirus vIRA. Cell Host Microbe,2012, 11 (3) : 290 –297. |
| [14] | Takaoka A, Taniguchi T. Cytosolic DNA recognition for triggering innate immune responses. Adv Drug Deliv Rev,2008, 60 (7) : 847 –857. |
| [15] | Rebsamen M, Heinz L X, Meylan E, et al. DAI/ZBP1 recruits RIP1 and RIP3 through RIP homotypic interaction motifs to activate NF-kappaB. EMBO Rep,2009, 10 (8) : 916 –922. |
| [16] | Burckstummer T, Baumann C, Bluml S, et al. An orthogonal proteomic-genomic screen identifies AIM2 as a cytoplasmic DNA sensor for the inflammasome. Nat Immunol,2009, 10 (3) : 266 –272. |
| [17] | Fernandes-Alnemri T, Yu J W, Datta P, et al. AIM2 activates the inflammasome and cell death in response to cytoplasmic DNA. Nature,2009, 458 (7237) : 509 –513. |
| [18] | Hornung V, Ablasser A, Charrel-Dennis M, et al. AIM2 recognizes cytosolic dsDNA and forms a caspase-1-activating inflammasome with ASC. Nature,2009, 458 (7237) : 514 –518. |
| [19] | Rathinam V A, Jiang Z, Waggoner S N, et al. The AIM2 inflammasome is essential for host defense against cytosolic bacteria and DNA viruses. Nat Immunol,2010, 11 (5) : 395 –402. |
| [20] | Morrone S R, Matyszewski M, Yu X, et al. Assembly-driven activation of the AIM2 foreign-dsDNA sensor provides a polymerization template for downstream ASC. Nat Commun,2015, 6 : 7827 –7839. |
| [21] | Schroder K, Tschopp J. The inflammasomes. Cell,2010, 140 (6) : 821 –832. |
| [22] | Unterholzner L, Keating S E, Baran M, et al. IFI16 is an innate immune sensor for intracellular DNA. Nat Immunol,2010, 11 (11) : 997 –1004. |
| [23] | Jakobsen M R, Paludan S R. IFI16: At the interphase between innate DNA sensing and genome regulation. Cytokine Growth Factor Rev,2014, 25 (6) : 649 –655. |
| [24] | Diner B A, Lum K K, Cristea I M. The emerging role of nuclear viral DNA sensors. J Biol Chem,2015, 290 (44) : 26412 –26421. |
| [25] | Cao X. New DNA-sensing pathway feeds RIG-I with RNA. Nat Immunol,2009, 10 (10) : 1049 –1051. |
| [26] | Chiu Y H, Macmillan J B, Chen Z J. RNA polymerase III detects cytosolic DNA and induces type I interferons through the RIG-I pathway. Cell,2009, 138 (3) : 576 –591. |
| [27] | Fong K S, de Couet H G. Novel proteins interacting with the leucine-rich repeat domain of human flightless-I identified by the yeast two-hybrid system. Genomics,1999, 58 (2) : 146 –157. |
| [28] | Ariake K, Ohtsuka H, Motoi F, et al. GCF2/LRRFIP1 promotes colorectal cancer metastasis and liver invasion through integrin-dependent RhoA activation. Cancer Lett,2012, 325 (1) : 99 –107. |
| [29] | Rikiyama T, Curtis J, Oikawa M, et al. GCF2: expression and molecular analysis of repression. Biochim Biophys Acta,2003, 1629 (1-3) : 15 –25. |
| [30] | Douchi D, Ohtsuka H, Ariake K, et al. Silencing of LRRFIP1 reverses the epithelial-mesenchymal transition via inhibition of the Wnt/beta-catenin signaling pathway. Cancer Lett,2015, 365 (1) : 132 –140. |
| [31] | Yang P, An H, Liu X, et al. The cytosolic nucleic acid sensor LRRFIP1 mediates the production of type I interferon via a beta-catenin-dependent pathway. Nat Immunol,2010, 11 (6) : 487 –494. |
| [32] | Miyashita M, Oshiumi H, Matsumoto M, et al. DDX60, a DEXD/H box helicase, is a novel antiviral factor promoting RIG-I-like receptor-mediated signaling. Mol Cell Biol,2011, 31 (18) : 3802 –3819. |
| [33] | Kim T, Pazhoor S, Bao M, et al. Aspartate-glutamate-alanine-histidine box motif (DEAH)/RNA helicase A helicases sense microbial DNA in human plasmacytoid dendritic cells. Proc Natl Acad Sci USA,2010, 107 (34) : 15181 –15186. |
| [34] | Zhang Z, Yuan B, Bao M, et al. The helicase DDX41 senses intracellular DNA mediated by the adaptor STING in dendritic cells. Nat Immunol,2011, 12 (10) : 959 –965. |
| [35] | Parvatiyar K, Zhang Z, Teles R M, et al. The helicase DDX41 recognizes the bacterial secondary messengers cyclic di-GMP and cyclic di-AMP to activate a type I interferon immune response. Nat Immunol,2012, 13 (12) : 1155 –1161. |
| [36] | Ferguson B J, Mansur D S, Peters N E, et al. DNA-PK is a DNA sensor for IRF-3-dependent innate immunity. Elife,2012, 1 : e00047 –e00063. |
| [37] | Zhang X, Brann T W, Zhou M, et al. Cutting edge: Ku70 is a novel cytosolic DNA sensor that induces type III rather than type I IFN. J Immunol,2011, 186 (8) : 4541 –4545. |
| [38] | Hopfner K P, Karcher A, Craig L, et al. Structural biochemistry and interaction architecture of the DNA double-strand break repair Mre11 nuclease and Rad50-ATPase. Cell,2001, 105 (4) : 473 –485. |
| [39] | Deng Y, Guo X, Ferguson D O, et al. Multiple roles for MRE11 at uncapped telomeres. Nature,2009, 460 (7257) : 914 –918. |
| [40] | Kondo T, Kobayashi J, Saitoh T, et al. DNA damage sensor MRE11 recognizes cytosolic double-stranded DNA and induces type I interferon by regulating STING trafficking. Proc Natl Acad Sci U S A,2013, 110 (8) : 2969 –2974. |
| [41] | Roth S, Rottach A, Lotz-Havla A S, et al. Rad50-CARD9 interactions link cytosolic DNA sensing to IL-1beta production. Nat Immunol,2014, 15 (6) : 538 –545. |
| [42] | Civril F, Deimling T, de Oliveira Mann C C, et al. Structural mechanism of cytosolic DNA sensing by cGAS. Nature,2013, 498 (7454) : 332 –337. |
| [43] | Zhang X, Wu J, Du F, et al. The cytosolic DNA sensor cGAS forms an oligomeric complex with DNA and undergoes switch-like conformational changes in the activation loop. Cell Rep,2014, 6 (3) : 421 –430. |
| [44] | Chin K H, Tu Z L, Su Y C, et al. Novel c-di-GMP recognition modes of the mouse innate immune adaptor protein STING. Acta Crystallogr D Biol Crystallogr,2013, 69 (Pt 3) : 352 –366. |
| [45] | Shang G, Zhu D, Li N, et al. Crystal structures of STING protein reveal basis for recognition of cyclic di-GMP. Nat Struct Mol Biol,2012, 19 (7) : 725 –727. |
| [46] | Ishikawa H, Ma Z, Barber G N. STING regulates intracellular DNA-mediated, type I interferon-dependent innate immunity. Nature,2009, 461 (7265) : 788 –792. |
| [47] | Ishikawa H, Barber G N. STING is an endoplasmic reticulum adaptor that facilitates innate immune signalling. Nature,2008, 455 (7213) : 674 –678. |
| [48] | 陈艳玫, 姚鑫. 转录因子Sox2的研究进展. 生命科学,2004, 16 (3) : 129 –134. |
| [49] | Xia P, Wang S, Ye B, et al. Sox2 functions as a sequence-specific DNA sensor in neutrophils to initiate innate immunity against microbial infection. Nat Immunol,2015, 16 (4) : 366 –375. |
| [50] | Yu Z, Chen T, Cao X. Neutrophil sensing of cytoplasmic, pathogenic DNA in a cGAS-STING-independent manner. Cell Mol Immunol.[2015-04-27].http://www.nature.com/cmi/journal/vaop/ncurrent/full/cmi201534a.htm. |
| [51] | Wu J, Chen Z J. Innate immune sensing and signaling of cytosolic nucleic acids. Annu Rev Immunol,2014, 32 : 461 –488. |
| [52] | Ran Y, Shu H B, Wang Y Y. MITA/STING: a central and multifaceted mediator in innate immune response. Cytokine Growth Factor Rev,2014, 25 (6) : 631 –639. |
 2016, Vol. 36
2016, Vol. 36




