文章信息
- 朱云鹏, 王鹏, 夏博然, 唐延婷, 王权
- ZHU Yun-peng, WANG Peng, XIA Bo-ran, TANG Yan-ting, WANG Quan
- SARS冠状病毒主蛋白酶抑制剂的筛选及抑制动力学研究
- Screening and Inhibition Kinetics of SARS Coronavirus Main Protease Inhibitors
- 中国生物工程杂志, 2016, 36(4): 35-42
- China Biotechnology, 2016, 36(4): 35-42
- http://dx.doi.org/10.13523/j.cb.20160406
-
文章历史
- 收稿日期: 2015-10-21
- 修回日期: 2015-11-24
2. 天津国际生物医药联合研究院 天津 300457
2. Tianjin International Joint Academy of Biomedicine, Tianjin 300457, China
2002年底到2003年,一种严重的急性呼吸综合征肆虐全球,导致全球33个国家和地区的至少8 000人发病,高达近千人死亡[1]。WHO将这种疾病命名为严重急性呼吸综合征(Severe Acute Respiratory Syndromes,SARS),它是由SARS冠状病毒(SARS-CoV)引起的,SARS冠状病毒是一种全新的冠状病毒,为单股正链RNA病毒,是目前报道的RNA病毒中基因组最大的病毒[2]。从2012年夏天开始,另一新型冠状病毒出现并引起严重的中东呼吸综合征(MERS),表明冠状病毒已经对人类健康构成了持续的重大威胁[3]。SARS冠状病毒虽然随着“非典”的过去淡出人们的视野,然而由于自然界野生动物群体中还广泛存在SARS冠状病毒,SARS再次爆发的危险依然存在[4]。因此,阐明并利用它们的复制机制,甄选潜在的药物靶点,筛选、设计与储备抗SARS的有效药物依然是一个严峻的课题[5]。SARS冠状病毒主蛋白酶(SARS-CoV Mpro)作为SARS病毒的主要蛋白水解酶,能够将SARS-CoV复制酶多聚蛋白切割成病毒复制所必需的功能蛋白,在病毒生命周期中发挥重要作用,因此该酶是抗SARS病毒药物的一个理想靶点[6, 7]。
SARS冠状病毒主蛋白酶是一种半胱氨酸蛋白酶,蛋白共有306个氨基酸残基,分子量大小为33kDa左右。其活性形式为二聚体结构,催化活性中心是由145位的半胱氨酸(Cys145)和41位的组氨酸(His41)形成催化二价体[8]。由于冠状病毒主蛋白酶与小核糖体病毒的3C蛋白酶存在一定相似性,因此也被称为3C样蛋白酶(3C-like proteinase,3CL pro)[9]。
到目前为止,依然没有针对SARS的特效药,为了应对SARS再次爆发的可能,有效的抗SARS疫苗或药物是迫切需要的[10]。因此关于SARS-CoV Mpro 抑制剂的研究从未停止过,主要从三个方面进行:基于SARS-CoV Mpro结构的化合物虚拟筛选,化合物(包括小分子片段)及其衍生物的筛选以及天然化合物库的筛选。德国Lubeck大学生物化学研究所[11]根据SARS冠状病毒主蛋白酶的三级结构,得到其底物类似物Cbz-VNSTLQ-CMK(Cbz:苄氧羰基,CMK:氯甲基酮),这种抑制剂可以结合到SARS-CoV Mpro的特定区域,阻止酶发挥作用。Pillaiyar等[12]设计并合成了针对SARS冠状病毒主蛋白酶的低分子肽抑制剂,其中26m和26n表现出较好的抑制活性,其Ki值分别为0.39μmol/L和0.33μmol/L,能够促进强效肽抑制剂的进一步发展。Wu等[13]从1 000多种化合物中筛选出的一种HIV蛋白酶抑制剂显示出了一定的Mpro抑制活性(Ki=0.6μmol/L),模型研究显示这个化合物可以特异地与蛋白酶的活性位点结合。Bacha等[14]报道了一类双活性芳基硼酸化合物可以与Mpro的活性位点附近的丝氨酸残基簇(Ser139,Ser144,Ser147)结合,从而可逆地抑制Mpro的活性。Jon等[15]对NIH分子库进行了高通量筛选,发现了对SARS冠状病毒主蛋白酶具有较好抑制作用的化合物ML188,它是第一类真正的中等分子量的非共价SARS-CoV Mpro抑制剂,并具有IC50小于2 μmol/L的良好的抑制效果,对于通过非共价机制对SARS-CoV Mpro抑制剂的设计和完善,ML188是一个很好的起点。Young等[16]从东北雷公藤中分离出4种化合物,发现iguestrin的抑制效果最好,其IC50值为2.6μmol/L。因此,本文以SARS冠状病毒主蛋白酶为靶点,对我单位收集、设计、储藏的蛋白酶抑制剂聚焦库进行体外药物筛选,以期发现SARS冠状病毒主蛋白酶抑制剂,并着重从酶动力学的角度探讨抑制剂与SARS-CoV Mpro的作用模式。
1 材料与方法 1.1 材 料 1.1.1 试剂携带SARS-Mpro(NP_828863.1)核酸编码片段的pGEX-6p-1-SARS-Mpro重组质粒由清华大学饶子和课题组提供。氯化钠、甘油、三羟甲基氨基甲烷(Tris)、异丙基-β-D-硫代吡喃半乳糖苷(IPTG)均购自上海生工生物工程有限公司,Ni2+离子金属螯合层析柱购自Novagen公司,二甲基亚砜(DMSO)购自天津市化学试剂六厂,其余试剂均为国产分析纯。
1.1.2 仪器JN-300 低温超高压连续流细胞破碎仪购自广州聚能生物技术有限公司,AKTA FPLC 蛋白自动纯化系统购自GE Healthcare公司,Fluoroskan Ascent FL荧光分析仪购自Thermo Labsysterms公司,其余均为国产常规设备。
1.2 方 法 1.2.1 SARS冠状病毒主蛋白酶的异源重组表达与提纯将pGEX-6p-1-SARS-Mpro重组质粒转化至大肠杆菌BL21(DE3)感受态细胞中。挑取单克隆接种于5ml含氨苄(100μg/ml)的LB培养基中,37 ℃过夜培养,转接至800ml含氨苄(100μg/ml)LB培养基的摇瓶中,37 ℃培养至OD600=0.6~1.0。加入终浓度为0.5mmol/L 的IPTG,16 ℃诱导表达16~20h。4℃,5 500r/min离心培养液收集菌体,用裂解缓冲液(20mmol/L Tris-HCl pH 8.0,500mmol/L NaCl,10% 甘油)重悬后由低温超高压细胞破碎仪裂解。12 000r/min高速离心裂解液,将上清液进行镍离子金属螯合层析,以Washing Buffer(20mmol/L Tris-HCl pH8.0,500mmol/L NaCl,10% 甘油,15mmol/L 咪唑)除去杂蛋白,以Elution Buffer(20 mmol/L Tris-HCl pH8.0,500 mmol/L NaCl,10% 甘油,200 mmol/L 咪唑)洗脱目的蛋白。将洗脱的目的蛋白溶液进行Resource Q阴离子交换层析分离。通过SDS-PAGE检测蛋白纯度后将其分装并于-80℃保存。
1.2.2 高通量药物筛选以SARS冠状病毒主蛋白酶为靶点的药物筛选原理:根据SARS冠状病毒主蛋白酶可以识别的天然底物,合成多肽序列AVLQSGFRKK,在多肽的两端分别加上MCA荧光基团和Dnp荧光淬灭基团。SARS冠状病毒主蛋白酶在P1位谷氨酰胺残基(Q)和P1′位丝氨酸残基(S)之间进行序列切割后,荧光基团与淬灭基团分离,荧光基团在320nm激发光下可以产生405nm的发射光;如酶活性被候选药物抑制,序列无法被切割,淬灭基团对荧光基团有抑制作用,不会产生405nm的发射光(图 1)。通过荧光分析仪检测体系中荧光强度的变化,可以反映化合物对SARS冠状病毒主蛋白酶的抑制活性。SARS冠状病毒主蛋白酶体外筛选模型各组分如表 1所示。
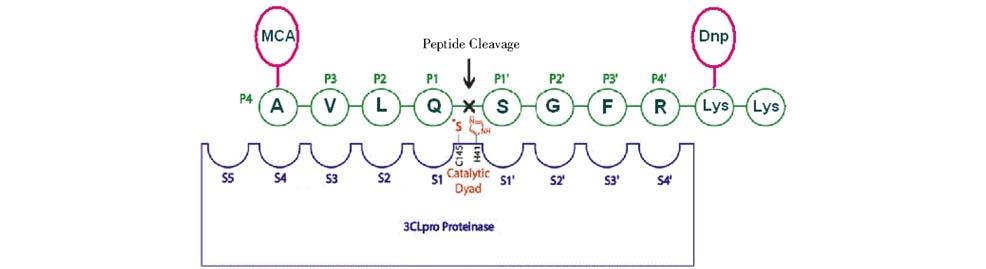
|
| 图 1 以SARS冠状病毒主蛋白酶为靶点的药物筛选原理 Fig. 1 Drug screening principle of SARS coronavirus main proteinase target |
| 体积(μl) | 浓度(μmol/L) | 缓冲液 | |
| SARS Mpro | 97 | 0.5 | 50 mmol/L Tris-HCl pH 7.3 |
| 底物 | 2 | 20 | 1 mmol/L EDTA |
| 化合物 | 1 | 500 | |
| 总体积 | 100 |
具体方法:在U型检测板中,依次加入97μl蛋白溶液,1μl化合物,震荡孵育30s,然后加入底物,振荡10s,进行荧光检测。同时设定不含化合物(以95% DMSO取代)的孔为阴性对照。反应在30℃下进行,平行测定3次。阴性对照组酶反应初速率为V0,实验组为Vi,Vi/V0表示加入化合物后酶的剩余活性,化合物对蛋白酶的抑制率(抑制活性)Ir=1-Vi/V0。为了排除假阳性的干扰,对于抑制率>80%的药物进行荧光淬灭(fluorescence quenching)的检测。首先在体系中加入蛋白和底物让体系的荧光值达到最大值Q1,然后向体系中加入化合物后测定荧光值为Q2,通过公式(Q1-Q2)/Q1计算得到淬灭率Qr。选择抑制率>80%,淬灭率<20%的化合物,为阳性结果。将阳性化合物进行梯度稀释,测定不同浓度下化合物对蛋白酶的抑制率,用GraphPad Prism 5软件进行分析得出化合物的半数抑制浓度(IC50)。
1.2.3 抑制剂的动力学研究按照1.2.2 的酶活检测体系,分析在不同浓度抑制剂存在的条件下,酶活性与酶浓度、酶量的关系,区分化合物的可逆与不可逆抑制作用。
通过Lineweaver-Burk双倒数作图,分析在不同浓度抑制剂存在的条件下,动力学参数Km和Vmax的变化,区分可逆抑制剂对SARS冠状病毒主蛋白酶的抑制类型;通过Dixon作图法,计算竞争型抑制剂的抑制常数Ki的值。
按照1.2.2的酶活检测体系,检测不同底物浓度条件下抑制剂的IC50值;比较抑制常数Ki值与IC50值的关系,对抑制剂的抑制类型进行验证。
2 结果与讨论 2.1 SARS冠状病毒主蛋白酶的原核表达与纯化经Resource Q型阴离子交换层析分离纯化所得蛋白溶液,进行12% SDS-PAGE凝胶电泳纯度检验。从图 2可以看出目的蛋白的纯度可达95%以上。

|
| 图 2 SDS-PAGE 分析结果(a)及Western blot 检测结果(b) Fig. 2 Result of SDS-PAGE analysis (a) and test result of Western blot (b) M: Protein maker; SARS: Purified target protein |
图 3为抑制剂筛选模型,其中横坐标为反应时间,纵坐标表示荧光强度;曲线斜率即为酶促反应速率,其中直线部分(一般认为产物生成量小于总量10%部分)斜率认为是酶促反应初速度;从图 3中我们可发现加入有抑制活性的样品后,酶促反应初速率低于阴性对照,通过计算即可获得抑制率。测定不同底物浓度下酶促反应的初速率,用GraphPad Prism 5软件进行分析得出米氏常数Km值为34.62μmol/L,最大反应速率Vmax为298.2μmol/L·s(图 4)。

|
| 图 3 抑制剂筛选模型 Fig. 3 The model of screening inhibitors |

|
| 图 4 SARS冠状病毒主蛋白酶的动力学参数Km值和Vmax值 Fig. 4 SARS-CoV Mpro kinetic parameter Km and Vmax |
按照1.2.2 的酶活检测体系,对蛋白酶抑制剂库96种化合物进行筛选,其中抑制率大于80%且淬灭率小于20%的化合物共5种,其IC50值分别为0.69±0.05μmol/L、1.19±0.41μmol/L、0.14±0.01μmol/L、1.36±0.07μmol/L、0.36±0.03μmol/L。高通量筛选结果如表 2所示,不同化合物对SARS冠状病毒主蛋白酶的抑制活性的量效关系如图 5所示。
| 化合物编号 | 抑制率 | 淬灭率 | IC50(μmol/L) | HillSlope | Span |
| P-1-08 | 90.36% | 0.00% | 0.69±0.05 | 2.312 | 107.3 |
| P-1-19 | 91.57% | 0.63% | 1.19±0.41 | 0.997 | 111.9 |
| P-2-24 | 100.80% | 2.04% | 0.14±0.01 | 1.402 | 108.6 |
| P-2-28 | 99.34% | 2.04% | 1.36±0.07 | 1.483 | 103.3 |
| P-2-54 | 93.81% | 2.48% | 0.36±0.03 | 1.935 | 108.4 |

|
| 图 5 不同化合物的量效关系图 Fig. 5 Dose-effect relationship of different compounds (a) P-1-08 (b) P-1-19 (c) P-2-24 (d) P-2-28 (e) P-2-54 |
由图 6(a)、6(b)、6(c)、6(e)可知在有抑制剂存在的测活体系中,SARS主蛋白酶的酶活性(反应初速率)与加入酶量的关系为一组平行的直线,随着抑制剂浓度的增加,酶反应速率下降,曲线的斜率不受影响,横坐标截距增大,说明P-1-08、P-1-19、P-2-24、P-2-54这4种化合物在酶促反应初期对SARS冠状病毒主蛋白酶的抑制属于不可逆抑制。这4种抑制剂能够与酶分子某些必需基团以牢固的共价键结合,使酶永久失活减少有效酶量。图 6(d)中均为通过原点的直线,随着抑制剂浓度的增加,直线的斜率减小,酶促反应速率下降,酶活逐步降低,化合物P-2-28对SARS冠状病毒主蛋白酶的抑制属于可逆抑制。抑制剂与酶以非共价键的方式结合而引起酶活降低,但并不导致酶分子结构永久改变。

|
| 图 6 在不同浓度抑制剂下酶活力和酶量的关系 Fig. 6 Relationship of enzyme activity and the amount of enzyme at different inhibitors concentrations (a) P-1-08 (b) P-1-19 (c) P-2-24 (d) P-2-28 (e) P-2-54 |
根据抑制剂、底物和酶三者的相互关系,可逆抑制又可分为竞争性抑制、非竞争性抑制、反竞争性抑制和混合型抑制4种类型,可通过Lineweaver-Burk双倒数作图法进行判断[17]。由图 7的Lineweaver-Burk曲线可以看出,在添加不同浓度的P-2-28时,得到一组相交于Y轴的斜率不同,截距相同的直线,随着P-2-28浓度的增大,Vmax不变,Km值增大,说明化合物P-2-28的抑制类型为竞争性。P-2-28在反应体系中与底物竞争和酶的同一活性中心结合,从而干扰酶与底物的结合,使酶的催化活性降低。

|
| 图 7 P-2-28对SARS冠状病毒主蛋白酶抑制作用的Lineweaver-Burk曲线 Fig. 7 Lineweaver-Burk curve of P-2-28 inhibition for SARS-CoV Mpro |
竞争性抑制的抑制常数Ki可以通过Dixon作图法求得,在不同的底物浓度下以抑制剂的浓度为横坐标,酶活反应速率的倒数为纵坐标作图。两条直线交点的横坐标即为-Ki[18],结果如图 8所示。根据图中两条直线的交点,由横坐标得出抑制剂P-2-28对SARS冠状病毒主蛋白酶的竞争性抑制常数Ki为0.81μmol/L。

|
| 图 8 P-2-28对SARS冠状病毒主蛋白酶抑制作用的Dixon图 Fig. 8 Dixon curve of P-2-28 inhibition for SARS-CoV Mpro |
由Cheng-Prusoff 公式可知,竞争性抑制剂的IC50与Ki的关系,如公式(1)所示[19]。

结合图 4和图 9计算不同底物浓度的IC50值与Ki的关系符合公式(1),IC50随底物浓度升高而增加,验证了化合物P-2-28对SARS冠状病毒主蛋白酶的抑制类型为竞争型。

|
| 图 9 不同底物浓度下P-2-28的IC50值 Fig. 9 IC50 value of P-2-28 under different substrate concentrations |
(1)通过对SARS冠状病毒主蛋白酶(SARS-CoV Mpro)的异源表达与纯化,获得了纯度较高的SARS冠状病毒主蛋白酶,然后以其为靶点,利用基于荧光共振能量转移(FRET)技术的体外药物筛选模型,对蛋白酶抑制剂库96种待测化合物进行了筛选,获得抑制率大于80%、淬灭率小于20%的化合物5种,分别为P-1-08、P-1-19、P-2-24、P-2-28、P-2-54。其半数有效抑制浓度(IC50)分别为:0.69±0.05μmol/L、1.19±0.41μmol/L、0.14±0.01μmol/L、1.36±0.07μmol/L、0.36±0.03μmol/L。
(2)从动力学的角度探讨筛选出来的5种化合物对SARS-CoV Mpro的抑制作用。其中P-1-08、P-1-19、P-2-24、P-2-54这4种化合物对SARS冠状病毒主蛋白酶的抑制作用为不可逆抑制。P-2-28对SARS冠状病毒主蛋白酶的抑制作用为可逆抑制,根据Lineweaver-Burk图和Dixon图的研究,发现它对SARS冠状病毒主蛋白酶呈竞争性抑制,抑制常数Ki为0.81μmol/L。通过对底物浓度,IC50值及Ki值关系的研究,验证了P-2-28的抑制作用为竞争性抑制。该抑制剂的发现为SARS冠状病毒主蛋白酶抑制剂的研究打下基础,为抗SARS病毒药物开发提供了先导化合物。
| [1] | WHO.Cumulative number of reported cases(SARS). http://www.who.int/csr/sars/country/table2004-04-21/en/index.html. |
| [2] | WHO.Cumulative number of reported cases of severe acute respiratory syndrome (SARS). (2003-05-31) . |
| [3] | Rolf Hilgenfeld, Malik Peiris. From SARS to MERS: 10 years of research on highly pathogenic human coronaviruses. Antiviral Research, 2013,100(1):286-295. |
| [4] | Chen Y, Cai H, Pan J, et al. Functional screen reveals SARS coronavirus nonstructural protein nsp14 as a novel cap N7 methyltransferase. PNAS, 2009,106 (9): 3484-3489. |
| [5] | Liang G,Chen Q,Xu J,et al.Laboratory diagnosis of four recent sporadic cases of community-acquired SARS, Guangdong Province. China Emerg Infect Dis, 2004,10(10):1774-1781. |
| [6] | 唐小龙,江振友,林晨,等. SARS相关冠状病毒的基因组和主要蛋白酶的研究进展.第二军医大学学报,2004,25(3):272-276. Tang X L,Jiang Z Y,Lin C,et al. Progress about genome and main protease of the SARS-associated coronavirus. Second Military Medical University, 2004, 25(3):272-276. |
| [7] | Rota P A,Oberste M S, Monroe S S, et al. Characterization of a novel coronavirus association with severe acute respiratory syndrome.Science,2003,300(5626):1394-1399. |
| [8] | Yang H,Bartlam M, Rao Z. Drug design targeting the main proteinase,the Achilles'heel of coronavirus. Curr Pharm Design, 2006, 12(35):4573-4590. |
| [9] | Allaire M,Chernaia M M,Malcolm B A. Picornaviral 3C cysteine proteinases have a fold similar to chymotrypsin-serine proteinases. Nature, 1994, 369(6475):72-78. |
| [10] | Young-Sam Keum, Yong-Joo Jeong. Development of chemical inhibitors of the SARS coronavirus: Viral helicase as a potential target. Biochemical Pharmacology, 2012,84(10):1351-1358. |
| [11] | Anand K, Ziebuhr J, Wadhwani P, et al.Coronavirus main proteinase(3CL pro)structure:basis for design of anti-SARS drug. Science, 2003, 300(5626):1763-1767. |
| [12] | Pillaiyar Thanigaimalai, Sho Konno, Takehito Yamamoto, et al. Design, synthesis, and biological evaluation of novel dipeptide-type SARS-CoV 3CL protease inhibitors: Structure-activity relationship study. European Journal of Medicinal Chemistry, 2013,65:436-447. |
| [13] | Wu C Y, Jan J T, Ma S H, et al. Small molecules targeting severe acute respiratory syndrome human coronavirus. Proc Natl Acad Sci USA, 2004,101(27):10012-10017. |
| [14] | Bacha U, Barrila J ,Velazquez-campoy A, et al. Identification of novel inhibitors of the SARS coronavirusmain proteinase. Biochemistry, 2004,43(17):4906-4912. |
| [15] | Jon Jacobs, Valerie Grum-Tokars, Ya Zhou, et al. Discovery, synthesis, and structure-based optimization of a series of N-(tert-Butyl)-2-(N-arylamido)-2-(pyridin-3-yl) acetamides (ML188) as potent noncovalent small molecule inhibitors of the SARS-CoV 3CL protease. Med Chem, 2013, 56 (2):534-546. |
| [16] | Young Bae Ryu, Su-Jin Park, Young Min Kim, et al. SARS-CoV 3CLpro inhibitory effects of quinone-methide triterpenes from Tripterygium regelii. Bioorganic & Medicinal Chemistry Letter, 2010,20:1873-1876. |
| [17] | 黄熙泰. 现代生物化学. 第3版. 北京:化学工业出版社,2006. 117-125. Huang X T. Modern Biochemistry. 3rd ed. Beijing: Chemical Industry Press, 2006. 117-125. |
| [18] | 沈同,王镜岩. 生物化学. 第3版. 北京:高等教育出版社,2002.368-372. Shen T, Wang J Y. Biochemistry. 3rd ed. Beijing: Higher Education Press, 2002. 368-372. |
| [19] | Yung-chi Cheng, William H Prusoff. Relationship between the inhibition constant(Ki) and the concentration of inhibitor which causes 50 percent inhibition(I50) of an enzymatic reaction. Biochemical Pharmacology, 1973, 22:3099-3108. |
 2016, Vol. 36
2016, Vol. 36




