文章信息
- 蒲强, 罗嘉, 沈林園, 李强, 张谊, 张顺华, 朱砺
- PU Qiang, LUO Jia, SHEN Lin-yuan, LI Qiang, ZHANG Yi, ZHANG Shun-hua, ZHU Li
- CRISPR/Cas9基因组编辑技术的研究进展及其应用
- The Advance and Application of CRISPR/Cas9 Mediated Genome Editing Technique
- 中国生物工程杂志, 2015, 35(11): 77-84
- China Biotechnology, 2015, 35(11): 77-84
- http://dx.doi.org/10.13523/j.cb.20151111
-
文章历史
- 收稿日期: 2015-06-08
- 修回日期: 2015-07-10
2. 四川省畜牧总站 成都 610041;
3. 西昌学院动物科学学院 西昌 615013
2. Sichuan Province General Station of Animal Husbandry, Chengdu 610041, China;
3. Department of Animal Science, Xichang College, Xichang 615013, China
DNA重组技术的发展,标志着生物学新时代的开始,分子生物学家从此可以定向对目的DNA分子进行操纵和修改。在分子生物学的发展历史中,微生物的某些生命活动不断带给人们启示,发展出了对DNA操作具有重要意义的工具,如分子克隆用的限制性内切核酸酶和PCR用的耐热聚合酶。近年来,另一种从细菌的获得性免疫系统发展而来的基因操作工具CRISPR/Cas9受到广泛关注,其应用和影响范围仍在不断扩大。随着测序技术的不断进步,越来越多物种的全基因组序列被获得。面对这些海量的基因组数据,基因定点编辑技术将是研究基因功能的一种高效手段,研究人员可以直接编辑或调节DNA序列的功能,迅速获得基因功能和应用信息,使这些生物基因信息得到充分利用。研究发现CRISPR/Cas9作用精确,可以用于自身目标基因的敲除、激活或抑制,也可以插入外源目标基因,这些基因包括人、老鼠、斑马鱼、细菌、果蝇、酵母、线虫和农作物细胞内的基因,这也意味着基因编辑是一种可以广泛使用的生物技术。
1 CRISPR/Cas9系统简介 1.1 CRISPR/Cas9系统的发现CRISPR系统是细菌的一种获得性免疫系统。细菌被病毒侵染之后,可以捕获其DNA片段整合进入基因组并形成记忆,当再次遭到相同病毒入侵时,对其形成免疫。早在1987年,Nakata等就在大肠杆菌中发现有29nt串联间隔重复序列,但一直不清楚其功能[1]。随着DNA测序技术的飞速发展,微生物基因组测序数据显著增多,Mojica等发现这种重复序列广泛存在于细菌和古细菌中,据估计超过40%的细菌基因组和90%的古细菌基因组中具有CRISPR结构[2]。早期的这些发现开始吸引研究者们对这样的微生物重复元素进行研究。2002年将其正式命名为CRISPR(clustered regulatory interspaced short palindromic repeat)[3]。直到2005年,3个研究组同时发现CRISPR间隔序列和侵染细菌的病毒或噬菌体间高度同源[4, 5, 6]。从而推测,这一系统可能类似于siRNA,是细菌抵抗噬菌体的一种生理机制。它们与植物的siRNA及高等动物的获得性免疫一样,都是获取入侵病毒的某个片段形成记忆,当再次遭到入侵时可以对抗入侵的一种方式。2010年CRISPR的基本功能和机制变得清晰,开始利用CRISPR系统研究各种生物技术[7, 8, 9]。然而,还没有探索基因组编辑的应用。2012 年,由RNA介导的基因组编辑技术 CRISPR/Cas9(CRISPR/CRISPR-associate 9)开始发展起来[10, 11]。2013年,两项研究同时实现从嗜热链球菌和酿脓链球菌中设计II型CRISPR系统,并完成在哺乳动物细胞中的基因组编辑[12, 13, 14]。
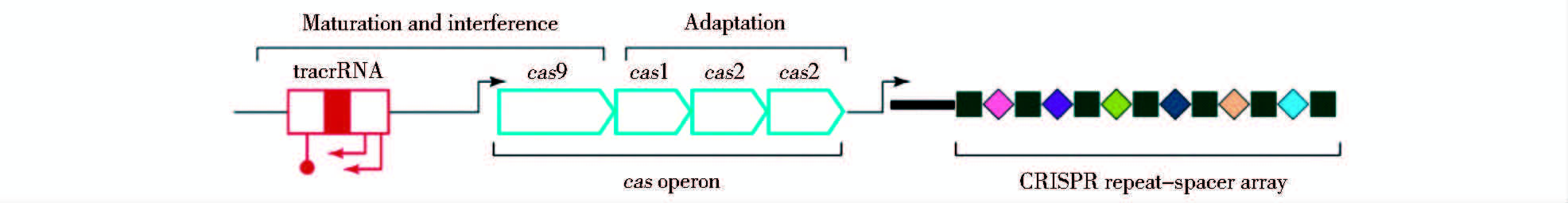
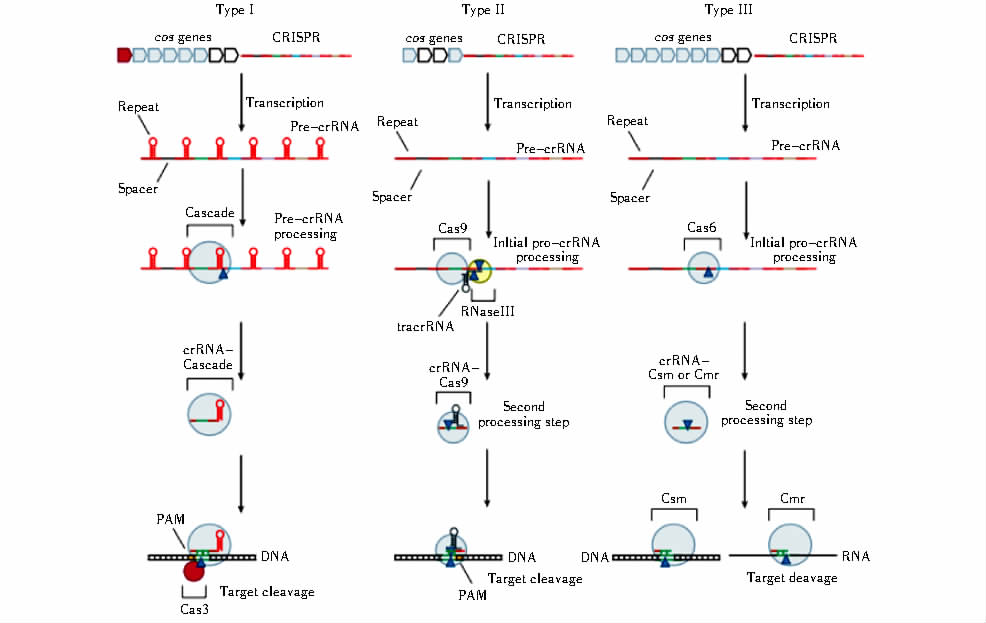
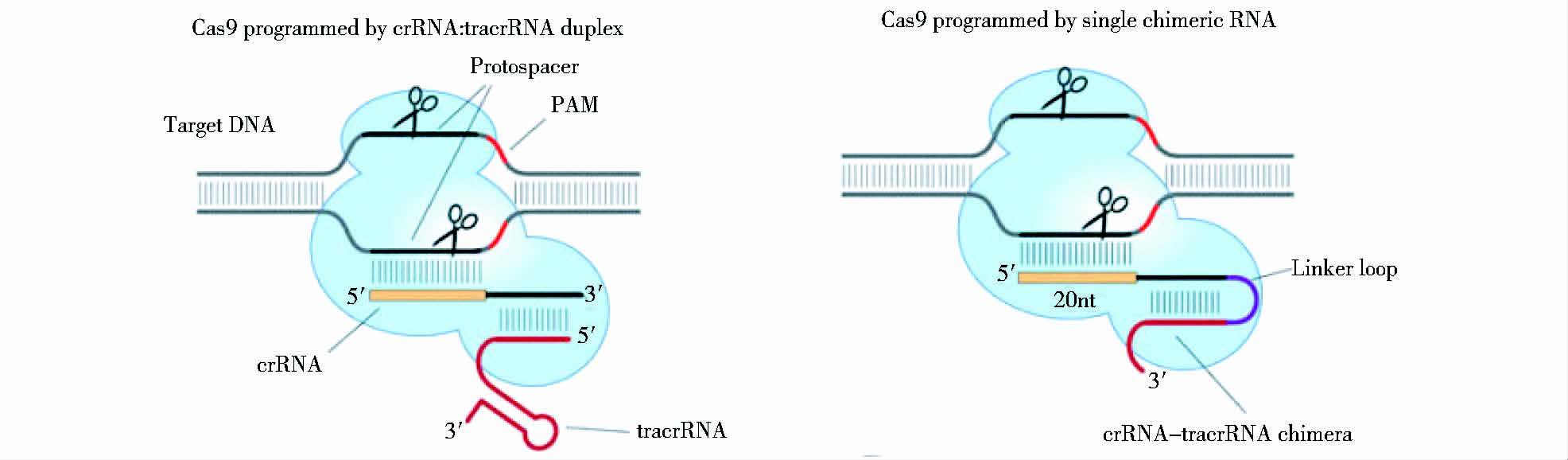
1.2 CRISPR/Cas9 的基因座结构及编辑原理CRISPR 由一系列短的高度保守的正向重复序列(repeat) 与长度相似的间隔序列(spacer)间隔排列组成[15](图 1)。Cas基因是一类基因家族,编码的蛋白质具有与核酸结合的功能以及与核酸酶、聚合酶、解旋酶等结合的活性。根据Cas基因的不同,将CRISPR/Cas9系统分为I~III 3种类型(图 2)。在Ⅰ型与Ⅲ型 CRISPR/Cas系统中,pre-crRNA 初级转录本被 CRISPR 相关RNA 酶切割成重复序列,释放小的成熟 crRNA。成熟 crRNA 再与相关蛋白质结合形成 RNA-蛋白质复合体指导内切核酸酶对外源 DNA 序列的识别与降解。II型CRISPR/Cas9系统是目前最常用于人工基因组编辑的CRISPR/Cas系统。根据Cas蛋白的类型不同分为3个亚类:II-A型含有Cas1、Cas2、Cas9和Csn2蛋白质;II-B型含有Cas1、Cas2、Cas4和Csx12样Cas9 4种蛋白质;II-C型则有Cas1、Cas2及Cas9 3种蛋白质。其中,II型CRISPR/Cas9系统最早在改造后用于小鼠和人类基因组编辑,是目前研究最为充分的系统。它只需要单独的Cas9蛋白即可在gRNA(guide RNA)的引导下完成对DNA的定点切割。Cas9蛋白行使功能需CRISPR转录而来的crRNA与反式激活的、与CRISPR 重复区互补的tracrRNA(trans-activating crRNA,tracrRNA) 形成的复合物参与[18]。为了操作简便,研究者将crRNA和tracrRNA 融合进同一条单链中,设计出单链引导RNA(single guide RNA,sgRNA)。在引导 RNA 的指导下,Cas9 定位于特定 DNA 序列上,进行 DNA 双链切割,实现基因组的定向编辑。对靶序列有效的切割还需靶序列相邻的原型间隔序列毗邻基序,即PAM(protospacer-associated motif)的参与[19, 20]。PAM提供两个冗余的检查点,确保Cas9不会错误地破坏它自身的基因组DNA。CRISPR/Cas9基因组编辑技术的基本原理为将 tracrRNA: crRNA 设计为引导RNA(sgRNA),sgRNA包含位于 5′端的靶DNA的互补序列,以及位于3′端的 tracrRNA: crRNA 的类似序列,crRNA或sgRNA包含一个20nt指导序列可以直接匹配的靶位点,利用靶DNA 的互补序列来定位需编辑的位点,利用tracrRNA: crRNA 的类似序列与Cas9 结合,实现目标基因定向基因组改造[12, 13, 20, 21],如图 3所示。
1.3 CRISPR/Cas9的特点及应用
CRISPR本身是一种防御系统,用以保护细菌和古细菌细胞不受病毒的侵害。在这些生物基因组中的CRISPR位点能表达与入侵病毒基因组序列相匹配的小分子RNA 。当微生物感染了这些病毒,CRISPR RNA就能通过互补序列结合病毒基因组,并表达CRISPR相关酶,也就是Cas,这些酶都属于核酸酶,能切割病毒DNA,阻止病毒完成其功能。
将CRISPR/ Cas系统用于其他非细菌细胞需要满足两个条件:一个是Cas酶,用于切断靶标DNA,如目的基因中的DNA片段;另一个是称为引导RNA(gRNA)的RNA分子,这种分子能通过互补结合靶标。 其中Cas酶至关重要,为了能更好的利用这一技术工具,不少实验室解析了这种酶的精确分子机制,探索其如何靶向和作用于DNA。
近期Sternberg等[22]就确定了Cas9在病毒感染过程中是如何在RNA序列的引导下识别和降解外源DNA,以及在动物和植物细胞中诱导位点特异性遗传改变的。CRISPR/Cas9系统相关的最新研究也证实其能构建更高效的基因定点修饰技术。例如,在猴子胚胎干细胞中利用CRISPR/Cas9系统通过同源重组实现对基因的打靶[23];在哺乳动物细胞系中利用CRISPR/Cas9系统来进行一代基因突变[24];利用CRISPR/Cas9系统实现对范可尼贫血疾病患者机体的基因编辑[25];在人类的造血干细胞和效应细胞中实现有效的基因剔除[26];对动物和植物基因组进行编辑[27, 28, 29];全基因组测序技术揭示了CRISPR/Cas9系统可以在人类诱导性多功能干细胞(iPSC)中实现高效的基因编辑[30]。因此,它在生命科学基础研究、生物技术和医药领域有广泛的应用前景。在生物学基础研究领域,CRISPR/Cas9系统主要被用来定点敲除一些基因,有助于快速解析基因功能及基因间的相互作用;在生物技术领域,它有助于定向育种,有助于抗逆、高产或有特殊经济性状的作物或菌种的培育,对一些关键性状基因的编辑能够大大加快良种的育种速度;在医药领域,结合iPSC技术,可以将通过基因编辑修复的iPSC细胞重新发育为正常组织和器官来供患者使用,从而快速建立疾病模型,并进行基因治疗和新药开发。
2 CRISPR/Cas9基因组编辑的结果检验及脱靶效应分析编码Cas9和sgRNA的片段通过质粒或其他途径转染进细胞后,Cas9在特异的位点对目标基因组进行切割,引起DNA双链断裂 (double-strand break,DSB),通过非同源末端连接(nonhomologous end joining,NHEJ)或同源重组(homologous recombination,HR)产生DNA的插入或删除(indel),或者依赖于含有特异突变的同源模板,进行目的位点的定向改造。然而基因组编辑的结果需要确认,CRISPR/Cas9的特异性是人们对该系统关心的一个问题。它被关注后不久就有报道提出了CRISPR/Cas9的脱靶现象[31, 32, 33]。通过改造其系统元件或采用不同策略能增加基因组编辑的特异性[34]。例如,Cas9-D10A切口酶配合一对gRNA使用,可以减少意外的脱靶基因修饰,它分别在相对的两条DNA链上产生切口,从而形成功能性的双链断裂。这种设计能最大限度降低脱靶效应,同时保持高效而又特异的基因修饰。对于目标位点或预测的脱靶位点,除了用核酸酶进行电泳分析检测外,还可以用DNA测序方法进行确认。当需要检测的样品较少或靶位点较少时,可以采用Sanger测序[32]。当样品或靶位点较多时,适合采用高通量测序。例如,利用全基因组测序鉴别CRISPR/Cas9 在人基因组中的脱靶点[35],检测CRISPR/Cas9在人干细胞克隆个体中的靶基因突变[36]。
尽管一些报道指出对人类iPSC进行CRISPR/Cas9编辑具有很高的特异性,人体多能干细胞中的脱靶突变发生率很低[37],但基因组编辑的安全性仍然是人们担心的一个问题,尤其在基因治疗领域。CRISPR/Cas9会在相关位点引入不容忽视的脱靶突变[31, 38]。有研究者利用Ion Torrent公司的PGM测序仪,在小鼠胚胎和体外培养的细胞中,对已知的脱靶位点进行深度测序,结果并未在这些位点发现Cas9切割酶引入的脱靶突变[39]。全基因组测序(whole genome sequencing )可以对整个基因组进行单碱基分辨率的检测,当CRISPR/Cas9用于基因治疗等对安全性要求较高的领域时,有必要采用这一方法检测基因组编辑的效果。当前已有不少对CRISPR/Cas9编辑后的基因组进行全测序的实例。例如,中国科学院的研究团队揭示了CRISPR/Cas系统在水稻中产生突变的机制,研究小组测试了2个水稻亚种11个靶基因中CRISPR/Cas9诱导产生突变的效率、特点、遗传性及特异性,通过全基因组重测序及检测与靶序列高度同源的序列,仅在只有一个碱基不同的潜在脱靶位点检测到突变,表明CRISPR/Cas9系统在水稻中有很高的特异性[40]。Veres等[36]利用全基因组测序鉴定了CRISPR/Cas9介导的人类干细胞克隆,发现即使基因编辑具有很高的特异性,定向克隆仍然可能在基因组的其他地方存在差异,因此严谨的克隆研究需要全基因组测序来描述突变。CRISPR/Cas9技术是将一种蛋白质传送到细胞中,使其在精确的特异位点切割基因组。删除缺陷基因后可以让细胞的DNA重新融合到一起,或是将新的基因插入到删除基因的位置中。Yu等[41]成功地鉴别出了两种小分子(β3-肾上腺素受体激动剂L755507和一种细胞类蛋白质转运抑制剂Brefeldin A),它们可以显著地促进新的遗传物质插入到细胞DNA中,在研究过程中还发现了两种胸腺嘧啶类似物(叠氮胸苷和三氟尿苷)可抑制插入而促进DNA删除的化合物,表明这两个过程在细胞中发挥了竞争作用,能够在维持技术精确性的同时提高CRISPR/Cas9的基因编辑效率,利用这种方法可以简单有效地对基因组进行精准编辑。
对于基因组比较大的生物体来说,对目标区域进行富集,然后对富集片段进行高通量测序,是一种检测脱靶的策略。对DSB区域的富集与检测,曾报道过BLESS (breaks labeling,enrichment on streptavidin,and next generation sequencing)法[42]。最近,J. Keith Joung的小组报道了一种检测方法,称为Guide-seq (genome-wide,unbiased Identification of DSB enabled by sequencing)[43]。这种方法的过程是,使活体细胞吸收双链寡核苷酸(double-stranded oligodeoxynucleotide,dsODN)标签并整合进基因组断裂位点,抽提基因组DNA,进行随机打断和末端修复加高通量测序接头等操作,然后依据dsODN的专有序列进行PCR扩增,富集出包含dsODN的片段,再对收获的片段进行高通量测序,分析Cas9的切割位点。它适合临床前对所采用的CRISPR/Cas9在某类细胞中的应用风险进行评估。
在进行CRISPR/Cas9脱靶效应分析时,最近张昱实验室的研究人员建立了染色体免疫沉淀和二代高通量测序(ChIP-seq)的方法,研究了Cas9蛋白和特异靶向RNA在人类细胞基因组中的结合图谱[35]。研究发现,对于不同靶向RNA,Cas9蛋白在人类基因组中具有特异的结合脱靶效应。另外,发现Cas9蛋白的结合脱靶效应具有细胞特异性,揭示染色质构象或其他表观遗传因子可能参与了调控。这项研究为进一步研究CRISPR/Cas9基因组编辑的分子机制及优化其在体内和体外的应用提供了重要的工具和方法。
3 CRISPR/Cas9技术与高通量测序结合用于研究基因功能Cas9包含两个活性区域,即RuvC 和 HNH 结构域,各负责切割一条DNA单链。当只有一个结构域引入突变时,Cas9可被改造成为切口酶(nickase),即nCas9。nCas9与两条不同的sgRNA联用可用于较大片段的置换,也可增强编辑的特异性;当两个结构域同时引入突变时,Cas9内切酶活性丧失,成为dead Cas9(dCas9),dCas9仍能与靶区域结合,用来开启或关闭某些基因的表达。它与目的基因启动子区域或编码区段作用时,可能由于空间位阻效应阻挡了转录因子或 RNA 聚合酶与 DNA 的结合,从而抑制转录。将dCas9与转录调控因子的抑制区融合,可抑制靶基因,这就是CRISPR干扰(CRISPR interference,CRISPRi)[44];dCas9也可与反式激活结构域VP64的激活区结合,可激活靶基因[45, 46]。dCas9的这种特异性调控功能,与 RNA-seq相结合,可以用于许多基因功能相关的研究[45, 47, 48]。Gilbert 等[48]通过RNA-seq分析表明CRISPRi调节的转录抑制是非常特异的,证实CRISPRi作为通用工具在真核细胞精确调控基因表达的潜力。
另外,CRISPR/Cas9技术与高通量DNA测序的结合也可进行功能基因组学研究。一些研究者已经通过CRISPR/Cas9技术建立可能与某类功能相关的突变体库,通过功能性筛选和富集、PCR扩增及深度测序分析,确认与这类功能相关的基因。例如,魏文胜等开发了一种基于 CRISPR/Cas9系统的慢病毒聚焦型人源细胞文库、功能性基因筛选平台及基于高通量深度测序技术解析数据的完整技术路线[49]。开发出了一个新型的CRISPR/Cas9 sgRNA文库,提出了一种基于sgRNA文库功能筛查和高通量测序分析的基因鉴别新方法,通过哺乳细胞CRISPR/Cas9敲除文库实现高通量功能性基因筛选。
新一代测序技术既可以在全基因组或全转录组范围内筛选出基因或其转录产物的变化,也可以在某个局部范围(如外显子组内)考察基因的变异。这项技术能筛选出与某些功能相关的、变异的目标基因,而这些目标基因的具体功能可以用CRISPR/Cas9技术进行研究。最近新发展起来的一项技术 SunTag 类似于一套分子挂钩,能够将多个拷贝的生物活性分子挂到可用来靶向一些基因或其他分子的蛋白质支架上,整合SunTag 后的分子生物活性能够显著放大[50, 51]。Weissman 等证实,整合SunTag后的 CRISPR分子可用于控制基因组内大量基因的抑制和激活,利用这些规则,鉴别出了阻止癌细胞生长及调控组织发育的一些基因,并对细菌毒素损伤细胞机制有了一些新的认识[51]。这些研究表明,可以利用CRISPR 技术来了解基因在生物细胞的作用机制、致病的作用机制。例如,CRISPR/Cas9技术与高通量测序技术结合,可以在药物开发中快速发现药物的靶标并进行有效、直观的验证,即先用药物处理细胞,选出药物抗性克隆,对这些克隆进行高通量测序,找出候选的基因突变,这些基因的产物可能是药物的靶标。对于如何确认候选基因的突变导致药物抗性,可以采用CRISPR/Cas9技术来突变这些基因,观察细胞是否出现抗性。在类似的研究中,使用高通量测序能在大范围快速发现候选的基因突变,可以通过对基因组测序发现侯选突变[52, 53],为了兼顾研究基因表达情况,可采用转录组测序的方法进行突变检测[47],也有一些研究为了能够直接发现与蛋白质功能变异相关的遗传突变而采用了外显子组测序技术[54]。
4 CRISPR/Cas系统新类型的发现CRISPR/Cas 是一类基于基因序列的功能系统,它自身还存在很多未知的信息亟待探索,可以采用高通量测序技术对其加以研究。如前所述,自然存在的CRISPR/Cas 系统有多种类型及亚型,CRISPR/Cas9只是其中的一种。因此,深入研究和了解不同类型和种类的CRISPR/Cas系统对于充分认识和利用这类系统非常重要。例如,可以利用高通量测序技术研究CRISPR位点间隔区[55],也可以利用RNA-seq 探索CRISPR/Cas系统中RNA 在微生物中的加工和调节机制[56]。
目前,对CRISPR/Cas系统只有Ⅱ型研究应用比较深入,其他类型的研究相对缺乏,然而,为了更加有效的利用这一作用深远的技术,对其进行不断的探索显得尤为必要。
5 结 语CRISPR/Cas9基因组编辑技术,是进入21世纪后影响深远的遗传学研究技术,它大大推动了生命科学相关领域的研究,促进了生物学的发展。在生命科学基础研究、生物技术和医药领域有广泛的应用前景。
| [1] | Ishino Y, Shinagawa H, Makino K, et al. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. Journal of Bacteriology, 1987, 169(12): 5429-5433. |
| [2] | Wei C, Liu J, Yu Z, et al. TALEN or Cas9-rapid, efficient and specific choices for genome modifications. Journal of Genetics and Genomics, 2013, 40(6): 281-289. |
| [3] | Jansen R, Embden J, Gaastra W, et al. Identification of genes that are associated with DNA repeats in prokaryotes. Molecular Microbiology, 2002, 43(6): 1565-1575. |
| [4] | Pourcel C, Salvignol G, Vergnaud G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. Microbiology, 2005, 151(3): 653-663. |
| [5] | Bolotin A, Quinquis B, Sorokin A, et al. Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin. Microbiology, 2005, 151(8): 2551-2561. |
| [6] | Mojica F J, García-Martínez J, Soria E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. Journal of Molecular Evolution, 2005, 60(2): 174-182. |
| [7] | Quiberoni A, Moineau S, Rousseau G M, et al. Streptococcus thermophilus bacteriophages. International Dairy Journal, 2010, 20(10): 657-664. |
| [8] | Horvath P, Romero D A, Coûté-Monvoisin A C, et al. Diversity, activity, and evolution of CRISPR loci in Streptococcus thermophilus. Journal of Bacteriology, 2008, 190(4): 1401-1412. |
| [9] | Horvath P, Coûté-Monvoisin A C, Romero D A, et al. Comparative analysis of CRISPR loci in lactic acid bacteria genomes. International Journal of Food Microbiology, 2009, 131(1): 62-70. |
| [10] | Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816-821. |
| [11] | Gasiunas G, Barrangou R, Horvath P, et al. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proceedings of the National Academy of Sciences, 2012, 109(39): E2579-E2586. |
| [12] | Cong L, Ran F A, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339(6121): 819-823. |
| [13] | Mali P, Yang L, Esvelt K M, et al. RNA-guided human genome engineering via Cas9. Science, 2013, 339(6121): 823-826. |
| [14] | Sander J D, Joung J K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology, 2014, 32(4): 347-355. |
| [15] | Karginov F V, Hannon G J. The CRISPR system: small RNA-guided defense in bacteria and archaea. Molecular Cell, 2010, 37(1): 7-19. |
| [16] | Doudna J A, Charpentier E. The new frontier of genome engineering with CRISPR-Cas9. Science, 2014, 346(6213): 1258096. |
| [17] | Westra E R, Swarts D C, Staals R H, et al. The CRISPRs, they are a-changin': how prokaryotes generate adaptive immunity. Annual Review of Genetics, 2012, 46: 311-339. |
| [18] | Jinek M, Jiang F, Taylor D W, et al. Structures of Cas9 endonucleases reveal RNA-mediated conformational activation. Science, 2014, 343(6176): 1247997. |
| [19] | Deveau H, Barrangou R, Garneau J E, et al. Phage response to CRISPR-encoded resistance in Streptococcus thermophilus. Journal of Bacteriology, 2008, 190(4): 1390-1400. |
| [20] | Nishimasu H, Ran F A, Hsu P D, et al. Crystal structure of Cas9 in complex with guide RNA and target DNA. Cell, 2014, 156(5): 935-949. |
| [21] | Cho S W, Kim S, Kim J M, et al. Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease. Nature Biotechnology, 2013, 31(3): 230-232. |
| [22] | Sternberg S H, Redding S, Jinek M, et al. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature, 2014, 507(7490): 62-67. |
| [23] | Zhu S, Rong Z, Lu X, et al. Gene targeting via homologous recombination in monkey embryonic stem cells using CRISPR/Cas9 system. Stem Cells and Development, 2015, 4(10):1147-1149. |
| [24] | Bauer D E, Canver M C, Orkin S H. Generation of genomic deletions in mammalian cell Lines via CRISPR/Cas9. Journal of Visualized Experiments, 2015, (95): e52118-e52118. |
| [25] | Osborn M J, Gabriel R, Webber B R, et al. Fanconi anemia gene editing by the CRISPR/Cas9 system. Human Gene Therapy, 2014, 26(2):114-126. |
| [26] | Mandal P K, Ferreira L M, Collins R, et al. Efficient ablation of genes in human hematopoietic stem and effector cells using CRISPR/Cas9. Cell Stem Cell, 2014, 15(5): 643-652. |
| [27] | Belhaj K. Editing plant genomes with CRISPR/Cas9. Open Access Overview.[2014-11-29]. http://www.sciencedirect.com/science/article/pii/S0958166914001943. |
| [28] | Fan Z, Li W, Lee SR et al. Efficient gene targeting in golden Syrian hamsters by the CRISPR/Cas9 system. PLoS One, 2014, 9(10): e109755. |
| [29] | Kimura Y. Efficient generation of knock-in transgenic zebrafish carrying reporter/driver genes by CRISPR/Cas9-mediated genome engineering. Open Access Overview.[2014-10-8]. http://www.nature.com/articles/srep06545?WT.ec_id=SREP-631-20141014. |
| [30] | Smith C, Gore A, Yan W, et al. Whole-genome sequencing analysis reveals high specificity of CRISPR/Cas9 and TALEN-based genome editing in human iPSCs. Cell Stem Cell, 2014, 15(1): 12-13. |
| [31] | Fu Y, Foden J A, Khayter C, et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nature Biotechnology, 2013, 31(9): 822-826. |
| [32] | Ran F A, Hsu P D, Lin C Y, et al. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity. Cell, 2013, 154(6): 1380-1389. |
| [33] | Pattanayak V, Lin S, Guilinger J P, et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity. Nature Biotechnology, 2013, 31(9): 839-843. |
| [34] | Shen B, Zhang W, Zhang J, et al. Efficient genome modification by CRISPR-Cas9 nickase with minimal off-target effects. Nature Methods, 2014, 11(4): 399-402. |
| [35] | Duan J, Lu G, Xie Z, et al. Genome-wide identification of CRISPR/Cas9 off-targets in human genome. Cell Research, 2014, 24(8): 1009-1012. |
| [36] | Veres A, Gosis B S, Ding Q, et al. Low incidence of off-target mutations in individual CRISPR-Cas9 and TALEN targeted human stem cell clones detected by whole-genome sequencing. Cell Stem Cell, 2014, 15(1): 27-30. |
| [37] | Suzuki K, Yu C, Qu J, et al. Targeted gene correction minimally impacts whole-genome mutational load in human-disease-specific induced pluripotent stem cell clones. Cell Stem Cell, 2014, 15(1): 31-36. |
| [38] | Cradick T J, Fine E J, Antico C J, et al. CRISPR/Cas9 systems targeting β-globin and CCR5 genes have substantial off-target activity. Nucleic Acids Research, 2013,43(18):1093. |
| [39] | Bell C C, Magor G W, Gillinder K R, et al. A high-throughput screening strategy for detecting CRISPR-Cas9 induced mutations using next-generation sequencing. BMC Genomics, 2014, 15(1): 1002. |
| [40] | Zhang H, Zhang J, Wei P, et al. The CRISPR/Cas9 system produces specific and homozygous targeted gene editing in rice in one generation. Plant Biotechnology Journal, 2014, 12(6): 797-807. |
| [41] | Yu C, Liu Y, Ma T, et al. Small molecules enhance CRISPR genome editing in pluripotent stem cells. Cell Stem Cell, 2015, 16(2): 142-147. |
| [42] | Crosetto N, Mitra A, Silva M J, et al. Nucleotide-resolution DNA double-strand break mapping by next-generation sequencing. Nature Methods, 2013, 10(4): 361-365. |
| [43] | Tsai S Q, Zheng Z, Nguyen N T, et al. GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. Nature Biotechnology, 2015,33(2):187-197. |
| [44] | Larson M H, Gilbert L A, Wang X, et al. CRISPR interference (CRISPRi) for sequence-specific control of gene expression. Nature Protocols, 2013, 8(11): 2180-2196. |
| [45] | Perez-Pinera P, Kocak D D, Vockley C M, et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nature Methods, 2013, 10(10): 973-976. |
| [46] | Maeder M L, Linder S J, Cascio V M, et al. CRISPR RNA-guided activation of endogenous human genes. Nature Methods, 2013, 10(10): 977-979. |
| [47] | Qi L S, Larson M H, Gilbert L A, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell, 2013, 152(5): 1173-1183. |
| [48] | Gilbert L A, Larson M H, Morsut L, et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell, 2013, 154(2): 442-451. |
| [49] | Zhou Y, Zhu S, Cai C et al. High-throughput screening of a CRISPR/Cas9 library for functional genomics in human cells. Nature, 2014, 509(7501): 487-491. |
| [50] | Tanenbaum M E, Gilbert L A, Qi L S, et al. A protein-tagging system for signal amplification in gene expression and fluorescence imaging. Cell, 2014, 159(3): 635-646. |
| [51] | Gilbert L A, Horlbeck M A, Adamson B, et al. Genome-scale CRISPR-mediated control of gene repression and activation. Cell, 2014, 159(3): 647-661. |
| [52] | Puente XS, Pinyol M, Quesada V, et al. Whole-genome sequencing identifies recurrent mutations in chronic lymphocytic leukaemia. Nature, 2011, 475(7354): 101-105. |
| [53] | Berger M F, Hodis E, Heffernan T P, et al. Melanoma genome sequencing reveals frequent PREX2 mutations. Nature, 2012, 485(7399): 502-506. |
| [54] | Quesada V, Conde L, Villamor N, et al. Exome sequencing identifies recurrent mutations of the splicing factor SF3B1 gene in chronic lymphocytic leukemia. Nature Genetics, 2012, 44(1): 47-52. |
| [55] | Savitskaya E, Semenova E, Dedkov V, et al. High-throughput analysis of type IE CRISPR/Cas spacer acquisition in E. coli. RNA Biology, 2013, 10(5): 716-725. |
| [56] | Richter H, Zoephel J, Schermuly J, et al. Characterization of CRISPR RNA processing in Clostridium thermocellum and Methanococcus maripaludis. Nucleic Acids Research, 2012, 40(19): 9887-9896. |
 2015, Vol. 35
2015, Vol. 35