文章信息
- 白利平, 姜蓉, 郭连宏, 张洋, 李元
- BAI Li-ping, JIANG Rong, GUO Lian-hong, ZHANG Yang, LI Yuan
- ste3与ste4基因双敲除对依博素生物合成的影响
- The Effects of ste3 and ste4 Genes Double Disruption in Ebosin Biosynthesis
- 中国生物工程杂志, 2015, 35(11): 23-28
- China Biotechnology, 2015, 35(11): 23-28
- http://dx.doi.org/10.13523/j.cb.20151104
-
文章历史
- 收稿日期: 2015-06-25
- 修回日期: 2015-07-23
依博素是由链霉菌139产生的一种新型胞外多糖,该多糖由半乳糖、阿拉伯糖、甘露糖、岩藻糖、木糖、鼠李糖、半乳糖醛酸和葡萄糖共8种单糖组成,其重复单元的结构已经确定[1]。药效学研究显示该多糖具有显著的抗类风湿性关节炎活性且毒性较低,目前正在申报临床研究,有可能发展成为新型药物[2]。我们已确定依博素生物合成基因簇ste,该基因簇大小为32kb,包括27个开放阅读框架(ste1~ste27) [3, 4, 5]。生物信息学分析表明链霉菌139中的ste1、ste2、ste3、ste4基因与天蓝色链霉菌和灰色链霉菌的dasR、dasA、dasB、dasC有较高的同源性,其中ste1对应于dasR,而ste2、ste3、ste4分别对应于dasA、dasB、dasC,转录方向相反[6]。
对天蓝色链霉菌和灰色链霉菌的研究发现 DasR 属于葡萄糖酸操纵子的负调节蛋白(a negative regulator of the gluconate operon,GntR家族),与其下游的 DasA、DasB、DasC 转运蛋白超家族共同调控上述两种链霉菌的孢子形成及营养利用[7, 8];其中 DasA[9]可能负责结合胞外营养物质,并通过两个膜蛋白(DasB、DasC的编码产物)转运入胞内。
我们最近研究确定Ste1属于DasR类蛋白,首次发现dasR类基因能够调控胞外多糖的生成及菌株形态分化,确定ste1为一个新的bld基因。调控分子机制研究显示,Ste1能够结合依博素生物合成基因簇中的启动子发挥其负调控功能,同时调控大量途径特异基因转录,共同调节依博素的生物合成[10]。为了进一步研究Ste3 和Ste4在依博素的生物合成中的功能,通过基因同源重组双交换及回复实验,获得ste3&ste4双基因缺失突变株和回复株。通过对各菌株的依博素产量分析,确定了ste3&ste4参与依博素的生物合成。
1 材料与方法 1.1 菌株和质粒本实验所用菌株和质粒见表 1。其中,链霉菌139(Streptomyces sp. 139)为胞外多糖依博素产生菌;E. coli DH5α为基因克隆宿主菌;E. coli ET12567 用于质粒去甲基化修饰后进行链霉菌原生质体转化;pKC1139为E. coli-Streptomyces穿梭载体;pUC19-kan提供同源双交换所用的卡那霉素抗性基因片段;pGEM-3zf-ermE*提供回复实验所需强启动子ermE*。
| Strain,Plasmid | Description | Source |
| Strain | ||
| Streptomyces sp.139 | Ebosin producing strain | Our lab |
| Strain D34 | The ste3&ste4 deficient strain of Streptomyces sp. 139 | This study |
| Strain C34 | The ste3&ste4 complemented strain of Streptomyces sp. 139 | This study |
| E. coli DH5α | supE44 Δ lacU169 (φ80 lacZ ΔM15) hsdR17 recA1 endA1 gyrA96 thi-1 relAI | Our lab |
| E. coli ET12567 | Methylation-deficient E.coli dam-dcm-hsdM. | Our lab |
| Plasmid | ||
| pKC1139 | Shuttle plasmid (E. coli-Streptomyces) pSG5,pBR322; aac(3)IV lacZa oriTRK2; Amr | Our lab |
| pKC34D | pKC1139 derived plasmid carrying F1,F2 and Kmr fragments. Kmr Amr | This study |
| pKC34C | pKC1139 derived plasmid carrying promoter ermE* and F3. Amr | This study |
| pUC19-kan | pUC19 derived plasmid carrying kanamycin resistance gene (XbaI). Kmr Ampr | Our lab |
| pGEM-3zf-ermE* | pGEM-3zf carrying promoter ermE* from Streptomyces erythraeus Ampr | Our lab |
| Note:Kmr: Kanamycin resistance; Amr: Apramycin resistance; Ampr: Ampicillin resistance | ||
Streptomyces sp. 139 DNA提取使用TSB 培养基(加0.5%甘氨酸)于28℃培养;其种子和发酵培养基均为F19 培养基(葡萄糖1%、淀粉2%、黄豆粉2%、胰蛋白胨0.2%、牛肉膏0.2%、酵母膏0.4%、K2PO4 0.05%、CaCO3 0.3%、微量盐溶液0.1ml)。E. coli 于LB 培养基 (或加抗生素) 37℃培养。限制性内切核酸酶、pfu DNA聚合酶、T4 DNA连接酶购自Promega公司;卡那霉素、安普霉素、氨苄青霉素购自Sigma公司;DNA分子质量标准均购自Fermentas公司;DNA胶回收试剂盒购自Qiagen公司;Southern杂交试剂盒购自Amersham公司。
1.3 构建ste3&ste4双基因敲除株以Streptomyces sp.139 总DNA为模板,以P1/P2为引物,PCR扩增ste3基因的上游片段F1(932bp);利用引物P3/P4 PCR扩增获得ste4基因的下游片段F2(595bp)(表 2)。PCR反应条件为:95℃变性60s,58℃复性30s,72℃延伸2min,共30个循环,最后72℃延伸10min,10℃保持。F1、F2片段与pGEM-T载体连接后进行序列测定,将与pGEM-T载体连接的F1、F2片段分别用EcoRI-XbaI和XbaI-HindIII双酶切、XbaI单酶切pUC19-kan,回收的F1、F2 和kan片段与EcoRI-HindIII酶切的质粒pKC1139进行连接,获得用于ste3&ste4基因阻断的重组质粒pKC34D。将质粒pKC34D转化甲基化缺失的E. coli ET12567并提取质粒,在聚乙二醇(PEG 6000)介导下,将pKC34D转化至Strpetomyces sp.139的原生质体[11],筛选步骤按报道方法进行[10],挑选对卡那霉素有抗性对安普霉素敏感的菌株(KmrAms),获得ste3&ste4双基因敲除株。
| Primer application | Primers | Nucleotide sequences | Restriction enzyme site | Location | Size (bp) |
| PCR F1(Upstream fragment of ste3) | P1 | 5′-CTGGAATTCCCGCGTCGTCCTCTACAA-3′ | EcoRI | 3018~3035 | 932 |
| P2 | 5′-GCATCTAGATTCGGTCTGCACGGTCAT-3′ | XbaI | 3932~3949 | ||
| PCR F2(Downstream fragment of ste4) | P3 | 5′-GCATCTAGATTCTCCCTGGGCATCG-3′ | XbaI | 5161~5176 | 595 |
| P4 | 5′-AGCAAGCTTGGTGGCGTTATCCCTTC-3′ | HindIII | 5739~5755 | ||
| PCR F3(ste3&4 and promoter region) | P5 | 5′-CGCGGATCCTGGCGCAGCAGCAGTAAC-3′ | BamHI | 3827~3844 | 1932 |
| P6 | 5′-CCCAAGCTTGGTGGCGTTATCCCTTC-3′ | HindIII | 5739~5755 |
用BamHI和HindIII 双酶切Streptomyces sp. 139 和ste3&ste4双基因敲除株总DNA,以荧光标记的932bp 的F1 片段为探针,按试剂盒(Amersham)提供操作步骤进行Southern blot。探针标记的方法如下:利用大肠杆菌DNA聚合酶中的Klenow大片段,以变性的DNA(探针)为模板,利用荧光(fluorescein)标记的11-dUTP代替模板DNA中的dTTP,得到荧光标记的探针。
1.5 ste3& ste4双基因突变株基因互补株的获得以Streptomyces sp.139基因组DNA为模板,采用引物P5/P6 PCR扩增获得ste3&ste4基因及其上游启动区片段F3共1 932bp(表 2),PCR的扩增条件同前。回收PCR产物与pGEM-T载体连接后进行序列测定,BamHI-HindIII双酶切质粒获得F3片段,以EcoRI-BamHI双酶切pGEM-3zf-ermE*重组质粒得到0.45kb的ermE*启动子片段。将链霉菌强启动子ermE*和F3片段与EcoRI-HindIII酶切的质粒pKC1139进行连接,得到互补质粒pKC34C。将质粒pKC34C转化至ste3&ste4双基因缺失突变株,提取质粒并进行酶切验证后得到基因互补菌株。
1.6 胞外多糖依博素的产量分析分别将Streptomyces sp.139、ste3&ste4双基因缺失突变株及回复株发酵液离心,取等体积上清液分别经X5大孔树脂柱层析,流出液再行007x7阳离子交换树脂柱层析,流出液以60%乙醇沉淀,反复两次。离心去上清液,沉淀物经挥发去除乙醇,以适量水溶解,经DEAE-Dextran A-25柱层析,蒸馏水洗脱,洗脱液经超滤(Mw 80000超滤膜)后冷冻干燥,称重后分别计算产量,重复3次。
2 结 果 2.1 Ste3和Ste4的同源性分析ste3的大小为986bp,终止密码子与ste4的起始密码子有重叠,编码一个328aa的蛋白质,Ste3蛋白与很多膜糖转运蛋白(sugar transporter integral membrane protein)具有很高的同源性。与来自Streptomyces coelicolor A3(2) 的SCO5233(CAB94618) 氨基酸序列具有89%的一致性,与来自Streptomyces griseus的DasB (BAB79298) 氨基酸序列具有35%的一致性(图 1)。ste4的大小为834bp,编码一个277aa的蛋白质,Ste4蛋白与来自Streptomyces coelicolor A3(2) 的SCO5234(CAB94619) 氨基酸序列具有87%的一致性,与来自Streptomyces griseus的DasC (BAB79299) 氨基酸序列具有41%的一致性(图 2)。Ste3蛋白和Ste4蛋白C端均具有TM_PBP2保守结构域[12],TM_PBP2 为胞质结合蛋白的跨膜亚单位[Transmembrane subunit (TM) found in periplasmic binding protein (PBP)],能够特异转运胞外营养物质,如麦芽糖或麦芽糊精等进入胞内,同时伴随ATP的水解。TM_PBP2最初发现在革兰氏阴性菌的壁膜间隙,随后在革兰氏阳性菌及真核细胞中也发现了TM_PBP2的存在,其在营养物质转运、代谢及能量利用等方面发挥重要作用[13]。Streptomyces coelicolor A3(2) 的SCO5233和SCO5234(也称为DasB、DasC)是以同一个转录单位转录,在二乙酰壳二糖( N,N′-diacetylchitobiose)入胞过程中发挥转运功能[14, 15];Streptomyces griseus中的DasB、DasC也发挥着类似功能[9]。

|
| 图 1 Ste3蛋白同源性分析 Fig. 1 Homology analysis of Ste3SCO5233: Putative sugar transporter integral membrane protein from Streptomyces coelicolor A3(2) (CAB94618); DasB: Putative sugar transporter inner membrane protein from Streptomyces griseus (BAB79298) |

|
| 图 2 Ste4蛋白同源性分析 Fig. 2 Homology analysis of Ste4SCO5234: Putative sugar transporter integral membrane protein from Streptomyces coelicolor A3(2) (CAB94619); DasC: Putative sugar transporter inner membrane protein from Streptomyces griseus (BAB79299) |
采用前述方法通过pKC34D对Streptomyces sp.139进行基因同源重组双交换[图 3(a)],共筛选获得32个KmrAms阳性克隆菌株。随机选择1个阳性克隆和链霉菌139提取基因组DNA,采用前述方法进行Southern杂交,结果显示,Streptomyces sp.139基因组DNA处出现一条5.8kb的阳性杂交条带,而ste3&ste4双基因缺失突变株基因组DNA处出现一条3.9kb的阳性杂交信号条带 [图 3(b)],与预期相符。表明通过基因同源重组双交换Streptomyces sp.139中的ste3和ste4基因内部的大部分片段已被卡那霉素抗性基因片段(kan)取代,命名为Strain D34。
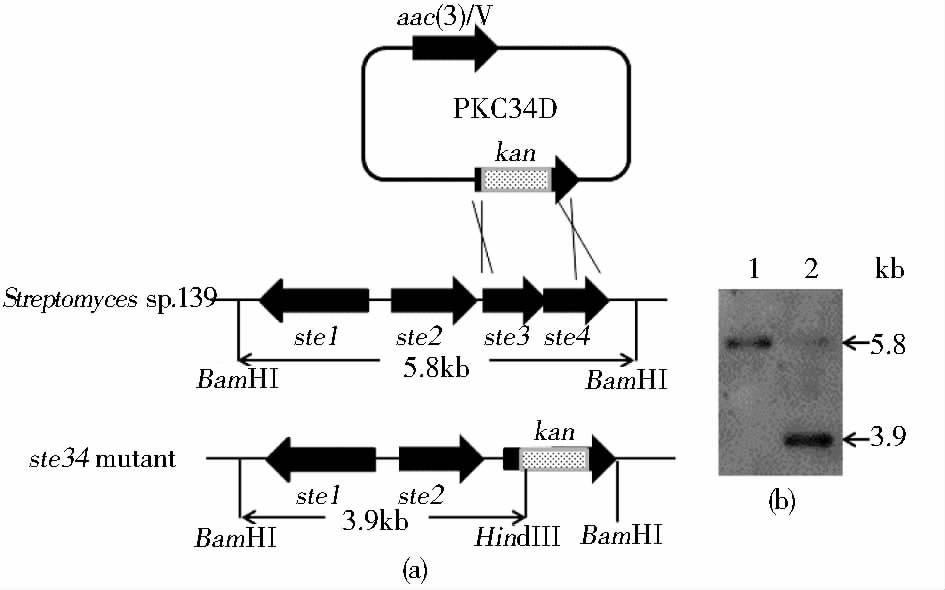
|
| 图 3 基因同源重组双交换示意图及缺失株的Southern 杂交验证 Fig. 3 The diagram of disruption of ste3&ste4 by double crossover and Southern blot of ste3&ste4 mutants(a) The diagram of gene disruption via double crossover (b) Southern blot of Strain D34 1: Chromosome DNA of Streptomyces sp. 139; 2: Chromosome DNA of Strain D34 |
将链霉菌强启动子ermE*和ste3&ste4基因及其上游启动区片段克隆于pKC1139,获得互补质粒pKC34C。互补质粒pKC34C转化至ste3&ste4双基因缺失突变株Strain D34,以安普霉素(Amr)为抗性标记筛选得到40多个转化子,随机挑选2个转化子提取质粒并酶切验证,将包含重组质粒的克隆命名为Strain C34。
2.3 依博素的产量分析分别从Streptomyces sp.139、StrainD34及StrainC34发酵液上清中提取胞外多糖依博素,重复3次实验进行产量定量分析。结果显示,Streptomyces sp.139的依博素平均产量为319mg/L,而ste3&ste4双基因缺失突变株的依博素平均产量为153mg/L,产量降低1倍以上。ste3和ste4基因互补后,依博素平均产量上升到299mg/L(图 4)。
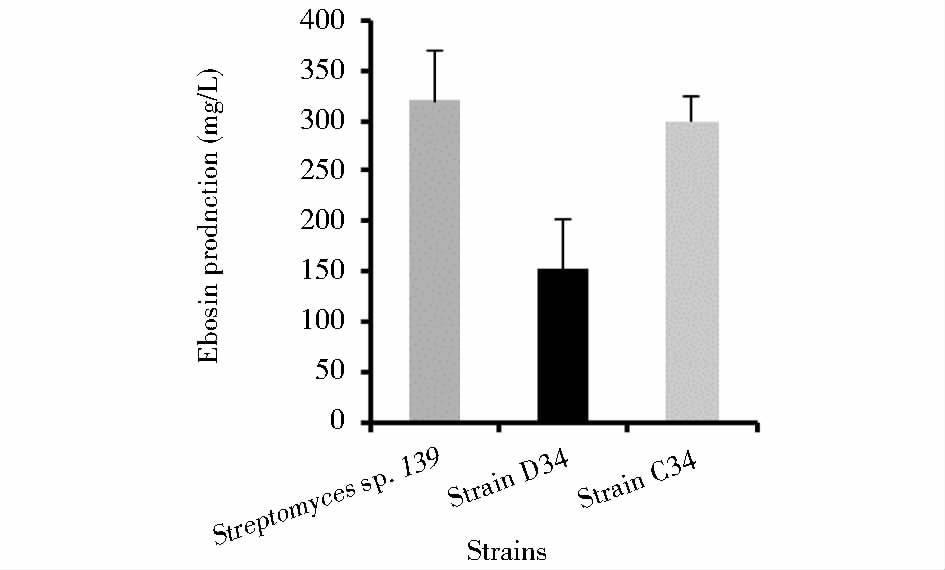
|
| 图 4 不同菌株中依博素产量分析 Fig. 4 The production of Ebosin from Streptomyces sp. 139,StrainD34, and Strain C34 |
链霉菌不同寡糖摄取系统的编码基因已被鉴定。例如,网链霉菌摄取纤维二糖和纤维三糖的cebEFG、天蓝色链霉菌摄取麦芽糖的 malEFG,以及摄取海藻糖和蜜二糖的aglEFG;橄榄绿链霉菌摄取GlcNAc和(GlcNAc)2的ngcEFG;Streptomyces thermoviolaceus摄取木二糖的bxlEFG。所有这些基因与dasA、dasB、dasC 一样,都以ABC转运蛋白超家族的形式行使其功能,其中E基因的编码产物为糖结合蛋白,FG基因的编码产物为两个膜蛋白[6]。但到目前为止,尚无DasB、DasC参与寡糖转运并与次级代谢产物生物合成相关的实验证据。
本研究通过生物信息学分析,在依博素生物合成基因簇中发现了与天蓝色链霉菌和灰色链霉菌的DasB、DasC 高度同源的膜蛋白Ste3、Ste4。由于依博素是由8种单糖组成的杂多糖,因此研究胞外营养物质,尤其是糖的转运与其生物合成的相关性显得尤为重要。利用基因同源重组双交换,我们获得了ste3&ste4双基因突变株,发酵结果显示,阻断突变株胞外多糖依博素产量降低1倍以上;通过导入携带强启动子和ste3&ste4基因的重组质粒可重建ste3&ste4双基因突变株的生物合成途径,依博素产量显著提高,基本达到野生株水平。
ste3&ste4双基因突变株依博素产量只是降低而没有完全消失,推测可能有以下两方面原因。一是基因组中有类似功能的基因发挥部分代偿功能,目前我们正在进行Streptomyces sp.139的基因草图分析,预计能够发现某些的功能类似基因;二是与其他化合物生物合成相比,多糖生物合成相对复杂,在乳酸菌研究中发现,即使敲除胞外多糖生物合成中最重要的引导糖基转移酶(welE),都不能彻底废除胞外多糖的生物合成[16]。综上所述,本研究首次证明链霉菌中参与营养物质转运的膜蛋白与胞外多糖依博素的生物合成密切相关,为研究Streptomyces sp.139的初级代谢和次级代谢之间的相关性奠定了基础。
| [1] | Jing C, Jianbo W, Yuan L, et al. A new IL-1 receptor inhibitor 139A: fermentation, isolation, physico-chemical properties and structure. J Antibiot (Tokyo), 2003, 56(2):87-90. |
| [2] | Zhang Y, Wang L F, Bai J Y, et al. Anti-inflammatory effect of Ebosin on rat collagen-induced arthritis through suppessing production of interleukin-1β, interleukin-6 and tumor necrosis factor. Eur J Inflamm, 2013, 11(3):677-688. |
| [3] | Wang L Y, Li S T, Li Y. Identification and characterization of a new exopolysaccharide biosynthesis gene cluster from Streptomyces. FEMS Microbiol Lett, 2003, 220(1):21-27. |
| [4] | Bai L, Chang M, Shan J, et al. Identification and characterization of a novel spermidine/spermine acetyltransferase encoded by gene ste26 from Streptomyces sp. 139. Biochimie, 2011, 93(9):1401-1407. |
| [5] | Zhang Y, Zhou J, Chang M, et al. Characterization and functional evidence for Ste27 of Streptomyces sp. 139 as a novel spermine/spermidine acetyltransferase. Biochem J, 2012, 443(3):727-734. |
| [6] | 白利平,李元. 链霉菌调控蛋白DasRABC的研究进展. 微生物学通报, 2010, 37(9):1369-1373. Bai L P, Li Y. Study of DasRABC in Streptomyces. Microbiology China, 2010, 37(9):1369-1373. |
| [7] | Rigali S, Titgemeyer F, Barends S, et al. Feast or famine: the global regulator DasR links nutrient stress to antibiotic production by Streptomyces. EMBO Rep, 2008, 9(7):670-675. |
| [8] | Rigali S, Nothaft H, Noens E E, et al. The sugar phosphotransferase system of Streptomyces coelicolor is regulated by the GntR-family regulator DasR and links N-acetylglucosamine metabolism to the control of development. Mol Microbiol, 2006, 61(5):1237-1251. |
| [9] | Seo J W, Ohnishi Y, Hirata A, et al. ATP-binding cassette transport system involved in regulation of morphological differentiation in response to glucose in Streptomyces griseus. J Bacteriol, 2002, 184(1):91-103. |
| [10] | Bai L, Qi X, Zhang Y, et al. A new GntR family regulator Ste1 in Streptomyces sp. 139. Appl Microbiol Biotechnol, 2013, 97(19):8673-8682. |
| [11] | Kieser T, Bibb M J, Butter M J, et al. Practical Streptomyces Genetics. Norwich:The John Innes Foundation, 2000. |
| [12] | Marchler-Bauer A, Derbyshire M K, Gonzales N R, et al. CDD: NCBI's conserved domain database. Nucleic Acids Res, 2015, 43(Database issue):D222-226. |
| [13] | Herrou J, Crosson S. Myo-inositol and D-ribose ligand discrimination in an ABC periplasmic binding protein. J Bacteriol, 2013, 195(10):2379-2388. |
| [14] | Colson S, van Wezel G P, Craig M, et al. The chitobiose-binding protein, DasA, acts as a link between chitin utilization and morphogenesis in Streptomyces coelicolor. Microbiology, 2008, 154(Pt 2):373-382. |
| [15] | Saito A, Shinya T, Miyamoto K, et al. The dasABC gene cluster, adjacent to dasR, encodes a novel ABC transporter for the uptake of N,N'-diacetylchitobiose in Streptomyces coelicolor A3(2). Appl Environ Microbiol, 2007, 73(9):3000-3008. |
| [16] | Lebeer S, Verhoeven T L, Francius G, et al. Identification of a gene cluster for the biosynthesis of a long, galactose-rich exopolysaccharide in Lactobacillus rhamnosus GG and functional analysis of the priming glycosyltransferase. Appl Environ Microbiol, 2009, 75(11):3554-3563. |
 2015, Vol. 35
2015, Vol. 35




