文章信息
- 裴智勇, 侯仙慧, 桂小柯, 陈禹保
- PEI Zhi-yong, HOU Xian-hui, GUI Xiao-ke, CHEN Yu-bao
- Primer Spanner:一个高效的定点突变PCR引物设计在线工具
- Primer Spanner: A Web-based Platform to Design PCR Primers for High Efficient Site-directed Mutagenesis
- 中国生物工程杂志, 2015, 35(10): 53-58
- China Biotechnology, 2015, 35(10): 53-58
- http://dx.doi.org/10.13523/j.cb.20151008
-
文章历史
- 收稿日期: 2015-06-05
- 修回日期: 2015-07-02
2. 中国科学院北京基因组研究所 北京 100101;
3. 北京大学北京核磁共振中心 北京 100871;
4. 北京大学化学与分子工程学院 北京 100094
1) 用户可通过http://PS1.biocloud.org.cn 免费访问PS并实现在线引物设计。
2. Beijing Institute of Genomics, Chinese Academy of Sciences, Beijing 100101, China;
3. Beijing Nuclear Magnetic Resonance Center, Peking University, Beijing 100871, China;
4. College of Chemistry and Molecular Engineering, Peking University, Beijing 100871, China
作为一项成熟的、强有力的生物技术手段,PCR在现代分子生物学中具有广泛的应用,如定点突变,不使用连接酶的载体构建、线性DNA片段拼接等[1]。定点突变(SDM)技术在DNA序列修饰、体外定向进化、蛋白结构与功能研究等方面有着广泛的应用。近三十年来,不断有新的工作改进这一实验技术[2, 3, 4, 5, 6, 7, 8]。目前,最为常用的方法之一是使用QuikChange®(Agilent Technologie)定点突变试剂盒,将该实验分为三个简单的步骤完成,即基于PCR的突变链合成,模板质粒的DpnI酶切降解和突变质粒的转化。其中突变引物设计需要用户根据目标序列要求自行完成,该公司也提供了在线的引物设计工具 (http://www.genomics.agilent.com/primerDesignProgram.jsp)帮助注册的用户进行突变引物设计。然而,该工具设计的每对引物是完全反向互补的,突变位点位于引物序列的中间区域,造成引物序列较长,因而在PCR退火过程中引物间更倾向于形成二聚体,而不是与模板结合。这一情况在需要进行多个突变时更为严重。该试剂盒的说明书指出,对3个位点同时突变时,成功率只有55%。目前,也有其它设计突变引物的在线工具如PrimerX (http://www.bioinformatics.org/primerx/index.htm),但采取的是相同的设计策略,同样存在形成引物二聚体的问题。此外,该方法设计的引物容易与模板错配,特别是在一对引物中引入多个突变的情况下,极易造成PCR不成功。
为了提高定点突变的效率,克服引物二聚体的形成非常重要。本工作采用5′端部分重叠(partial overlapping)的策略来设计突变引物,两条引物的3′端分别与模板完全匹配。已有一些报道,如 Zheng等[9]和Liu等[10]的工作描述相似的设计思路。然而,他们并没有提供易用化的工具。本工作提供了一个基于网页的在线工具Primers Spanning(http://PS1.biocloud.org.cn/),可以根据用户的目标序列和拟突变位点自动化地设计定点突变引物。同时,引物的相关参数,如Tm值、GC百分比、序列长度、二级结构等也纳入设计考虑中。可实现的突变类型包括一个或多个碱基的替换、插入和删除。经过我们的实验验证,PS设计的引物具有很高的突变成功率。
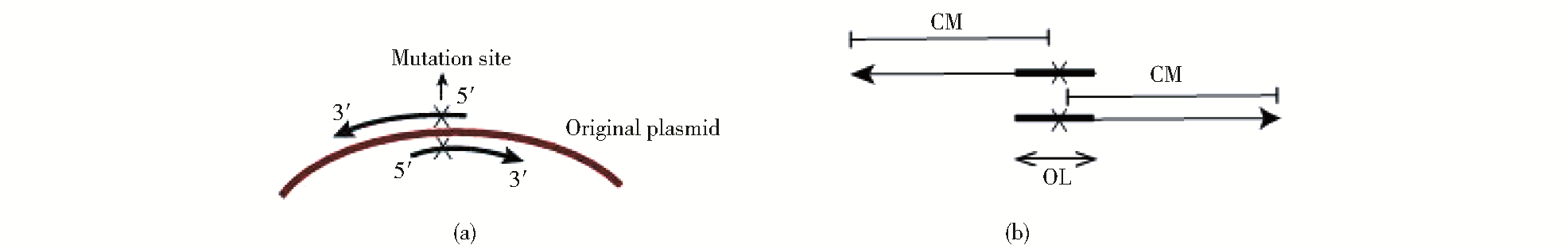
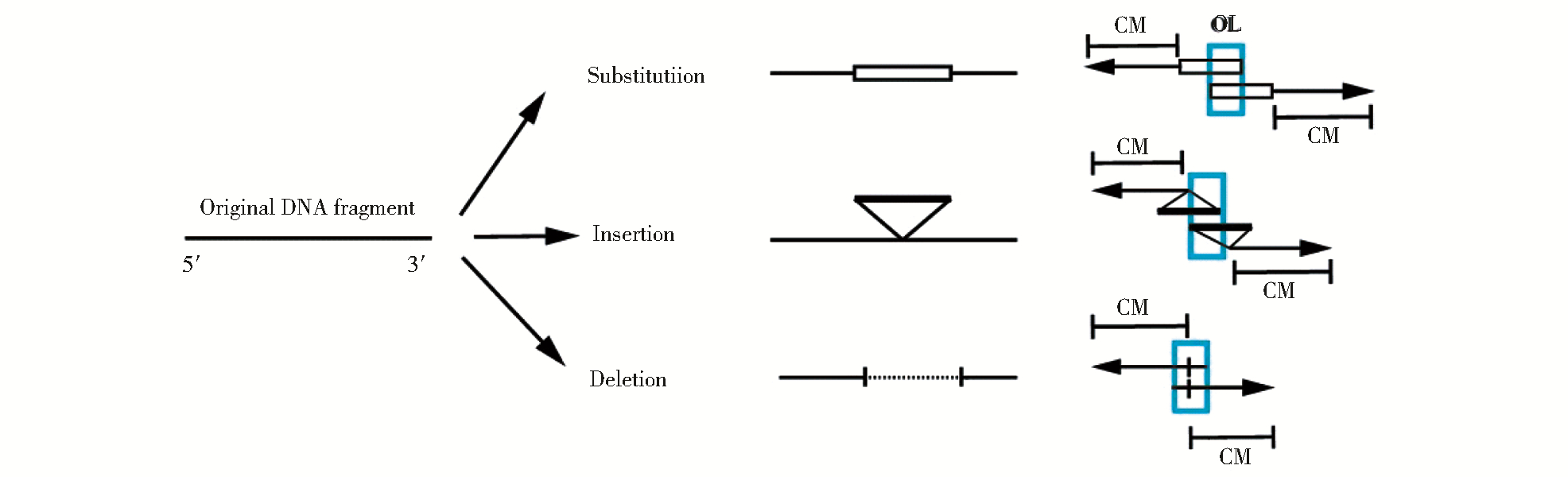
1 材料与方法 1.1 引物设计拟设计的引物,在5′端部分互补,因此,两条引物分为与模板完全互补的CM区(completely match region)和彼此反向互补的OL区(over lapping region),如图 1所示,图 1a为突变引物及相对位置示意,图 1b为引物各区段划分及命名,其中CM表示 3′端完配对区;OL表示 5′端重叠区。对于不同突变类型,OL区序列确定方法有所区别(图 2)。Tm值计算在引物设计中具有重要地位。PCR缓冲液中的离子强度对DNA退火温度计算影响很大[11, 12],其中Mg2+浓度影响最大[13].本工作采用Ahsen等[11]改进过的经验公式,基于nearest-neighbor (N-N)模型[14]并用 monovalent cation,Mg2+,dNTPs 和DMSO的浓度进行了校正:

|
| 图 1 引物设计示意 Fig. 1 Schematic diagram for mutation primer designing (a) The relative position of a pair of mutation primers (b) The crosses represent mutation sites CM: 3′-complete matching part; OL: 5′-overlapping part |
|
| 图 2 不同突变类型示意图(方框中为OL区) Fig. 2 Primer designing diagram for different types of mutations OL: Overlapping part, indicated by box |
其中,

该公式中,[Monovalent cations]是PCR缓冲液中Na+,K+ 和Tris+ 等一价阳离子的浓度。
关于参数控制,CM区的Tm值在58~68℃(长度约为18~24bp),保证PCR过程中引物与模板良好匹配;OL区的Tm在45~53℃(长度约为9~14bp),一方面避免引物二聚体形成,另一方面在PCR结束后两条产物的OL区可以退火形成缺刻的质粒,在模板被DpnI切割后,可以直接转化大肠杆菌。
1.2 实验验证材料及方法本工作中用到的引物合成和质粒测序由AuGGT DNA-SYN Biotechnology (Beijing)完成;质粒模板是从大肠杆菌TOP10菌株中扩增提取;KOD Plus高保真扩增酶购于TOYOBO公司(Japan);DpnI内切酶购于NEB公司(USA)。文中定点突变实验分两步进行,第一步PCR扩增目的质粒,20μl 反应体系中含有10ng模板质粒,0.5U KOD DNA 扩增酶,0.2mmol/L的dNTPs,2μl 10×KOD 反应缓冲液,2mmol/L MgSO4,以及0.25μmol/L由PS设计的突变引物。PCR 反应程序如下: 94℃预变性 2min,之后16到18个扩增循环,94℃ 25s变性,55℃ 25s退火,68℃延伸时间为“N”min,N由模板长度和KOD扩增速度(1kB/min)确定,最后,加68℃后延伸10min。第二步用Dpn1消化质粒模板,反应体系中含有9μl PCR产物和1μl 10×反应缓冲液和 0.5μl DpnI (NEB)。37℃消化1h以上,此产物可以直接转化感受态大肠杆菌细胞。
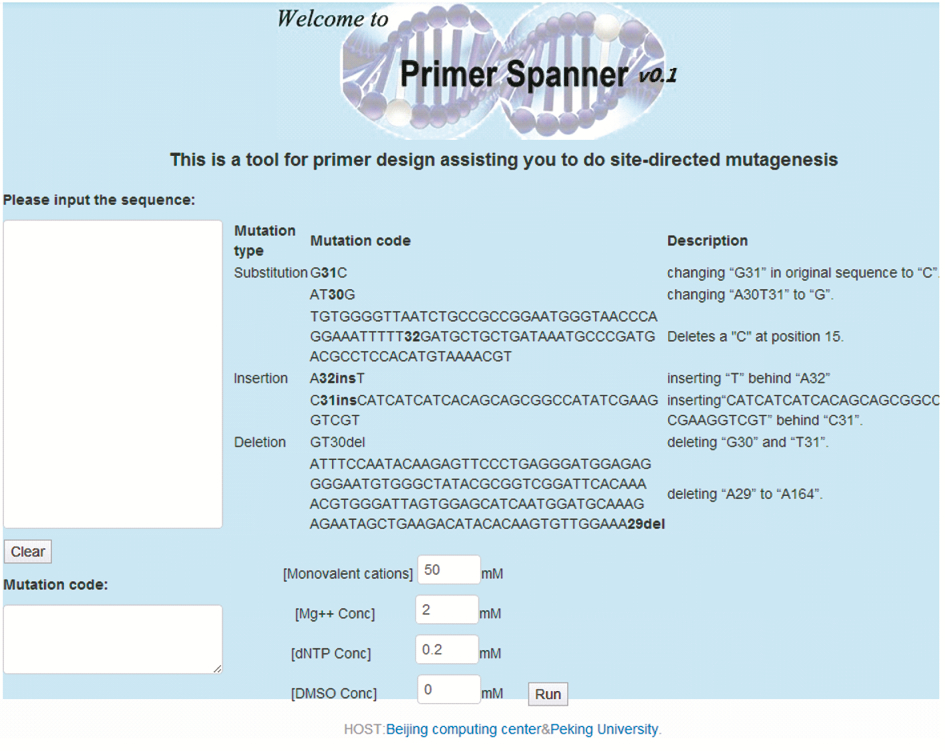
2 结 果 2.1 PS用户界面介绍在PS的主界面(图 3),用户需要输入原始的DNA序列,并在突变代码(Mutation code)中填写待突变的核苷酸字母和序号。突变代码是拟进行突变的位置和类型的描述,须按照指定规则填写。主页面给出了例子对不同类型的突变代码给出了直观的解释。如“A30”,表示原始序列中的第30位碱基为A。
|
| 图 3 Primer Spanner主页用户界面 Fig. 3 Main interface of Primer Spanner |
表 1中有对各种突变代码的详细描述,包括单个碱基突变和连续多碱基突变。在连续多碱基突变的代码中,数字为连续待突变碱基的第一个碱基的序号。对于替换和插入突变,可突变的序列长度,受限于引物的长度,一般不超过50nt。对于删除突变,长度没有特别限制。
 |
根据用户输入的原始序列和拟突变位点,可能会初步生成几十甚至几百对可能的引物。因而一个重要的步骤是对初步设计的待选引物进行评估排序,从而给出最好的引物对供用户选择。在本工具中,PS采用了一系列的标准对初选引物打分排序,最终将排名前5对引物在网页中呈现。引物评价标准包括GC百分含量,两条引物CM区Tm值间的差值,引物序列3′端稳定性和特异性,二级结构形成可能性,引物长度等 ,都进行了加权打分。其中二级结构形成可能性,包括序列形成发夹结构的可能性和局部引物二聚体的可能性,这些都会影响扩增效率和突变成功率,因此在打分时,关于二级结构形成可能性,给于较大的权重。
引物得分的计算公式如下:
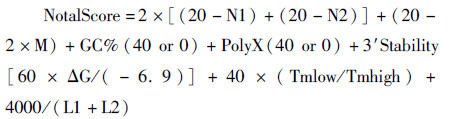
其中,N1和N2分别为两条引物各自形成发夹结构的最多核苷酸数,M代表引物之间形成二聚体的最多核苷酸数,PolyX代表完全相同的核苷酸数,3′Stability 代表 3′端稳定性(计算3′端5个核苷酸的ΔG),Tmlow 和Tmhigh 分别为两条引物CM区的Tm值,L1 和 L2 分别为两条引物核苷酸数。
关于引物二聚体形成的不同类型,在评估中也做了考虑,包括同二聚体和异二聚体,以及不同方向形成二聚体的可能性。 设计中考虑的另一个重要影响因子是3′端的稳定性。在本工作中,我们计算了3′端末5个碱基的热动力参数[15]。最后将以上各项打分加和后对各个引物对排序,给出得分最高的前5对引物,在页面中返回。
2.3 突变引物设计为了展示PS设计的突变引物的特征,在表 2中,我们给出了三种类型的突变引物示例,分别为替换、插入和删除。这些引物都已通过实验验证,具有非常高的扩增效率。
 |
我们应用PS进行了大量突变引物设计和测试实验,达到很高的成功率。下面为两个定点突变实例,待突变的序列为枯草芽孢杆菌(Bacillus subtilis)的ArsC基因,该基因已连入pET-28a (+) 载体,针对Lys97和Arg98设计突变引物,分别将Lys97突变为Glu,Arg98突变为Met,突变引物序列如下(加粗斜体字母标识突变位点):
K97E_F:5′-ACATGTAGAACGTGAGCATTGGGGTTT-3′
K97E_R:5′-CACGTTCTACATGTGGAGGCGTCATC-3′
R98M_F: 5′-GTAAAAATGGAGCATTGGGGTTTTGATG-3′
R98M_R: 5′-TGCTCCATTTTTACATGTGGAGGCGTC-3′
两对引物均可扩增出相应大小的目的载体片段,如图 4a所示,PCR产物经过DpnI酶切消化后不需要进行胶回收,直接转化大肠杆菌感受态细胞,分别挑取一个单克隆进行测序,结果显示二者均成功突变成目标序列,如图 4b和图 4c所示。
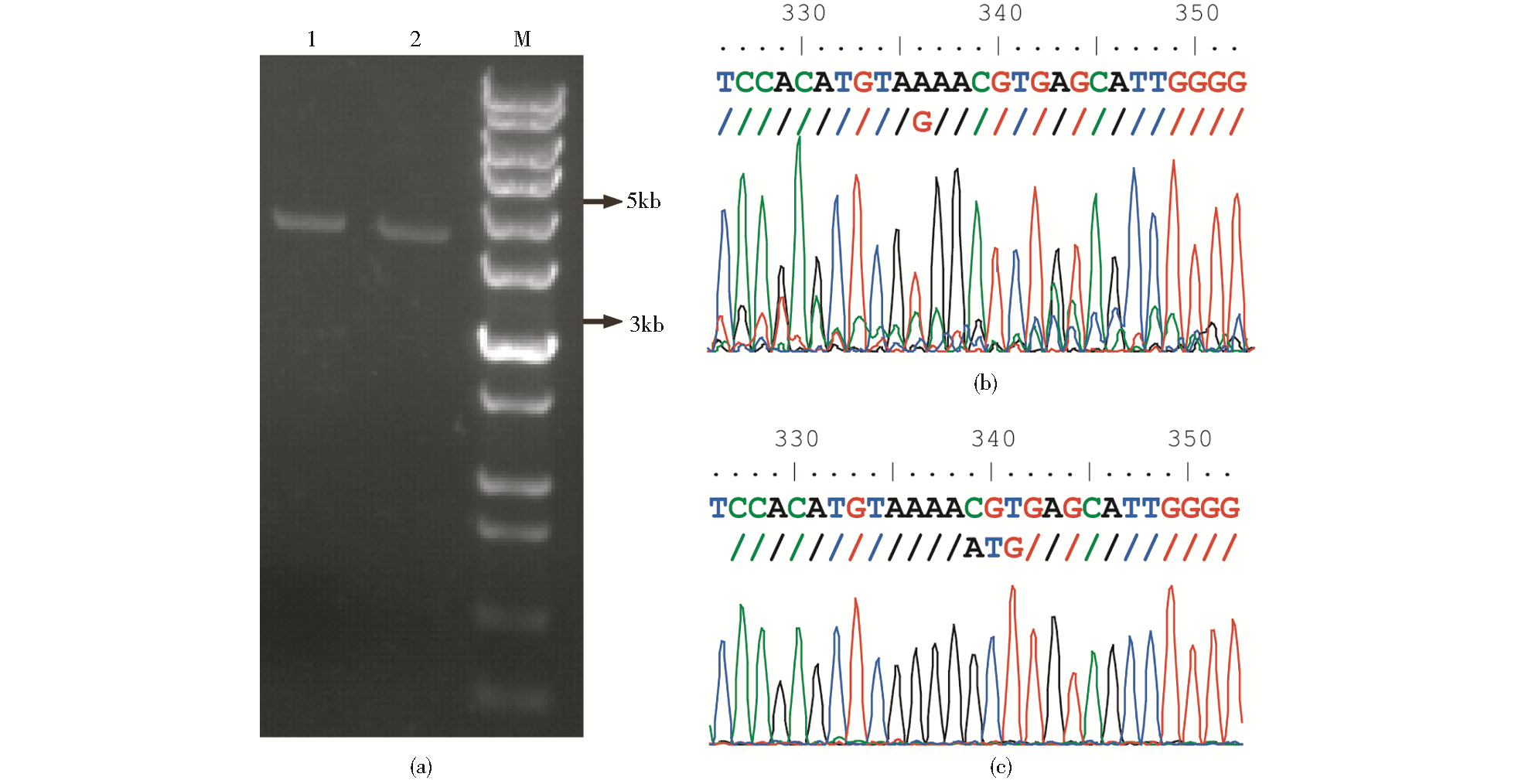
|
| 图 4 应用PS设计引物进行定点突变实验验证结果 Fig. 4 The results of site-directed mutagenesis experiments examination using primers designed by PS (a)Primers PCR (b)Lys97 mutation DNA sequencing (c)Arg98 mutation DNA sequencing |
经过测序验证,如图 4所示,图 4a为两对ArsC突变引物的PCR结果图,泳道1是用Lys97突变引物扩增得到的目的DNA电泳图,泳道2是Arg98突变引物扩增得到的目的DNA电泳图,对应的条带大小如泳道M标示;图 4b和图 4c分别为目的DNA经过DpnI酶切消化后转化大肠杆菌感受态细胞,挑取其中一个单克隆的测序结果,所测序列与野生型相同的部分用“/”表示,不同的位置显示相应碱基,测序结果与目标序列一致。
3 结 论引物在基于PCR的实验技术中起着十分重要的作用。本工作中,我们采用了一种基于Tm计算的策略设计非对称突变引物。在此基础上,我们开发了PS 在线工具,能够自动化地完成突变引物的设计。该工具设计的引物可以高效地完成PCR扩增,并且可以完成替换、插入、删除等不同类型的定点突变。经过实验验证,该工具设计的引物成功率非常高,并且不需要加入特殊酶的处理,简单高效。在未来的工作中,我们将进一步开发并扩展PS的功能及应用,如线性DNA拼接、载体构建等。
| [1] | Wetmur J G. DNA probes: applications of the principles of nucleic acid hybridization. Critical Reviews in Biochemistry and Molecular Biology, 1991, 26: 227-259. |
| [2] | Hemsley A, Arnheim N, Toney M D, et al. A simple method for site-directed mutagenesis using the polymerase chain reaction. Nucleic Acids Research, 1989, 17: 6545-6551. |
| [3] | Datta A K. Efficient amplification using ‘megaprimer’ by asymmetric polymerase chain reaction. Nucleic Acids Research, 1995, 23:4530-4531. |
| [4] | Ke S H, Madison E L. Rapid and efficient site-directed mutagenesis by single-tube ‘megaprimer’ PCR method. Nucleic Acids Research, 1997, 25: 3371-3372. |
| [5] | Brons-Poulsen J, Petersen N E, Horder M,et al. An improved PCR-based method for site directed mutagenesis using megaprimers. Molecular and Cellular Probes, 1998, 12: 345-348. |
| [6] | Angelaccio S, Bonaccorsi M C. Site-directed mutagenesis by the megaprimer PCR method: variations on a theme for simultaneous introduction of multiple mutations. Analytical Biochemistry, 2002, 306: 346-349. |
| [7] | Wei D, Li M, Zhang X, et al. An improvement of the site-directed mutagenesis method by combination of megaprimer, one-side PCR and DpnI treatment. Analytical Biochemistry, 2004, 331: 401-403. |
| [8] | Wu W, Jia Z, Liu P, et al. A novel PCR strategy for high-efficiency, automated site-directed mutagenesis. Nucleic Acids Research, 2005, 33: e110. |
| [9] | Zheng L, Baumann U, Reymond J L. An efficient one-step site-directed and site-saturation mutagenesis protocol. Nucleic Acids Research, 2004, 32: e115-e115. |
| [10] | Liu H, Naismith J H. An efficient one-step site-directed deletion, insertion, single and multiple-site plasmid mutagenesis protocol. BMC Biotechnology, 2008, 8: 91. |
| [11] | von Ahsen N, Wittwer C T, Schutz E. Oligonucleotide melting temperatures under PCR conditions: nearest-neighbor corrections for Mg2+, deoxynucleotide triphosphate, and dimethyl sulfoxide concentrations with comparison to alternative empirical formulas. Clinical Chemistry, 2001, 47: 1956-1961. |
| [12] | Owczarzy R, You Y, Moreira B G, et al. Effects of sodium ions on DNA duplex oligomers: improved predictions of melting temperatures. Biochemistry, 2004, 43: 3537-3554. |
| [13] | Owczarzy R, Moreira B G, You Y, et al. Predicting stability of DNA duplexes in solutions containing magnesium and monovalent cations. Biochemistry, 2008, 47: 5336-5353. |
| [14] | Santa Lucia J. A unified view of polymer, dumbbell, and oligonucleotide DNA nearest-neighbor thermodynamics. Proceedings of the National Academy of Sciences of USA, 1998, 95: 1460-1465. |
| [15] | Santa Lucia J J, Hicks D. The thermodynamics of DNA structural motifs. Annual Review of Biophysics and Biomolecular Structure, 2004, 33: 415-440. |
 2015, Vol. 35
2015, Vol. 35




