文章信息
- 温赛, 刘怀然, 续丹丹
- WEN Sai, LIU Huai-ran, XU Dan-dan
- 溶菌酶及其分子改造研究进展
- Advances in Research on Lysozyme and Strategies for New Antimicrobial Activity
- 中国生物工程杂志, 2015, 35(8): 116-125
- China Biotechnology, 2015, 35(8): 116-125
- http://dx.doi.org/10.13523/j.cb.20150817
-
文章历史
- 收稿日期:2015-04-27
- 修回日期:2015-05-06
溶菌酶(lysozyme,EC 3.2.1 .17 )又称胞壁质酶(muramidase)或N-乙酰胞壁质聚糖水解酶(N-acetylmuramide glycanohydrase),是一种能特异性水解原核细菌细胞壁中的主要成分肽聚糖(peptidoglycan,也称胞壁质)的糖苷水解酶。这种酶可以分解微生物的细胞壁,使细菌失去细胞壁的保护并在胞内高渗透压的作用下破裂死亡,实现杀菌的目的。该酶最早于1921年由著名的英国细菌学家Alexander Fleming在人的鼻液中发现,后被证实广泛存在于鸟类和禽类的蛋清、哺乳动物的各种器官组织和体液、植物、及软体动物、昆虫体内[1]。
溶菌酶作为高等有机体的组织及体液中最强大的抗菌剂之一,是生物机体对抗外源病原菌侵袭的重要防御因子。如人溶菌酶是一种小分子碱性球蛋白,它由上皮细胞和单核-巨噬细胞分泌,可识别和破坏病原菌细胞结构,并通过信号级联反应吸引白细胞集中到感染部位,最终消灭侵染人体的病原菌[2, 3]。此外,溶菌酶还可与带负电荷的病毒蛋白直接结合,和DNA、RNA、脱辅基蛋白形成复盐,使病毒失活[4, 5]。在植物中,如番木瓜、凤梨和无花果的鲜汁中均含有丰富的溶菌酶,推测其与植物的抗病毒作用有密切关系[6]。另一方面,溶菌酶也广泛存在于微生物中,如各种细菌和噬菌体[7]。噬菌体溶菌酶与噬菌体侵染过程中细菌细胞壁的分解有关[8]。细菌溶菌酶的主要功能是参与细胞生长、分裂、形态改变等与细胞壁相关的代谢过程[9]。因此,细菌不会对溶菌酶这种细菌中广泛含有的自溶素(autolysin),产生真正意义上的抵抗性。
近年来,由于抗生素滥用导致“超级细菌”的出现和畜牧产品抗生素残留等食品安全问题日益突出,人们迫切需要开发新型杀菌剂。而溶菌酶作为一种天然杀菌蛋白,能有效杀灭各种有害微生物,如食源性细菌中的嗜热脂肪芽孢杆菌(Bacillus stearothermophilus)、肉毒梭状芽孢杆菌(Clostridium botulinum)、热解糖梭菌(C. thermosaccharolyticum)、酪丁酸梭菌(C. tyrobutyricum)和单核细胞增多性李斯特氏菌(Listeria monocytogenes)等[10, 11, 12]。更重要的是,溶菌酶不会导致细菌耐药性、对人体无毒副作用,因此与其相关的科研和应用已成为学术热点。
1 溶菌酶的分类及胞壁质酶性质溶菌酶来源广泛、种类繁多,根据水解肽聚糖骨架具体部位的不同主要分为三类:N-乙酰胞壁质酶(N-acetylmuramidase)、N-乙酰胞壁酰-L-丙氨酸酰胺酶(N-acetylmuraxnyl-L-alanine amidase)和内肽酶(endopeptidase)。
(1)N-乙酰胞壁质酶主要作用于细菌细胞壁所含肽聚糖的N-乙酰胞壁酸(N-acetylmuramic acid,NAM) 的1位碳原子和N-乙酰葡萄糖胺(N-acetylglucosamine,NAG) 的4位碳原子之间的β-1,4糖苷键,使细胞壁的不溶性聚多糖分解成可溶性糖肽,导致细胞壁结构被破坏,细菌破裂死亡。
(2)N-乙酰胞壁酰-L-丙氨酸酰胺酶,又称MA酰胺酶,可分解N-乙酰胞壁酸其乳酸基与五肽侧链上1位氨基酸残基之间的酰胺键。该酶受阳离子肽的影响,对于微生物细胞的自溶具有重要作用[13]。目前多来源于噬菌体,如T7噬菌体[14]、蜡状芽孢杆菌噬菌体12826和TP21[15]等。
(3)内肽酶则作用于多肽侧链中的肽键,也可在葡聚糖酶的协同作用下促进真菌细胞壁的溶解。目前已发现许多微生物来源的内肽酶,如仿葡萄球菌(Staphylococcus simulans) 产生的溶葡球菌素(Lysostaphin)、球孢链霉菌(Streptomyces globisporus)产生的变溶菌素中含有的内肽酶[16]、米氏链球菌(Streptococcus milleri)所产的内肽酶“millericin B”等[17]。
三者在肽聚糖骨架上作用位点的区别如图 1所示。其中,N-乙酰胞壁质酶在自然界中分布广泛,是主要的溶菌酶类,也被称为狭义的溶菌酶,目前对该类酶的研究最为深入。

|
| 图 1 三种广义溶菌酶的作用位点 Fig. 1 The action site of three kinds of lysozymes |
根据底物的乙酰化程度不同,N-乙酰胞壁质酶又可分为β-1,4-N-乙酰胞壁质酶(β-1,4-N-acetylmuramidases)和β-1,4-N,6-O-二乙酰基胞壁质酶(β-1,4-N,6-O-diacetylmuramidases)两类。
1.1.1 β-1,4-N-乙酰胞壁质酶目前,科学界研究最多的β-1,4-N-乙酰胞壁质酶是鸡卵清溶菌酶(hen egg-white lysozyme,HEWL)。该酶在鲜鸡蛋中的含量最高,约占 3.5%,易于分离纯化,已经实现商业化生产。鸡卵清溶菌酶由129个氨基酸残基组成,分子量为14.4 kDa,是一种碱性小分子蛋白。1966年,Phillips[18]首次根据X射线晶体学方法研究并提出了鸡卵清溶菌酶的底物结合方式和催化机制。鸡卵清溶菌酶分子呈椭圆形结构,肽链上的亲水基团向外处于分子表面,而疏水基团聚集在内部,在椭圆形体上有一条明显的狭长裂缝,为溶菌酶的活性中心。研究表明,处于溶菌酶活性中心的氨基酸残基Glu35和Asp52对“底物-酶中间体”的形成起着重要作用,且活性中心的狭长裂缝(catalytic groove)内含有6个单糖的结合位点(A-F),因此底物不同的话其结合方式也会存在差异。当溶菌酶发挥催化作用时,肽聚糖糖链正好嵌入椭球形上的裂缝,溶菌酶“抓住”糖链并沿其移动,直至所有的糖苷键被打断[19]。因此,溶菌酶特殊的空间结构和催化特点决定了溶菌酶具有分解细菌肽聚糖骨架的能力。
在微生物来源中,β-1,4-乙酰胞壁质酶最典型的代表是来源于海氏肠球菌(Enterococcus hirae,也称为Streptococcu faecium ATCC9790)的两种N-乙酰胞壁质酶。第一种海氏肠球菌胞壁质酶的水解作用一直以恒定速率进行,直到90%的糖苷键都发生断裂水解速率才减慢。这种酶的功能就像“外-胞壁质酶”(exo-muramidase),起始于糖链的非还原末端,即N-乙酰葡萄糖胺端,之后酶逐步向前地水解N-乙酰胞壁酸和N-乙酰葡萄糖胺所组成的亚单位之间的β-1,4-糖苷键。因此,这种酶也称为“外-二糖酶”(exo-disaccharidase)[20]。研究证明,该酶的一个分子会紧密结合一个多糖链,并且直到几乎所有的键都被水解才会释放,而酶的释放环节是潜在的限速步骤[21]。
第二种海氏肠球菌胞壁质酶,与溶解海氏肠球菌细胞壁相比,能更迅速地溶解藤黄微球菌(Micrococcus luteus)的细胞壁。该酶的分子量为74 kDa,存在着典型的SXXK基序以及青霉素-相互作用蛋白(penicillin-interacting protein)的其它特征氨基酸序列,并能以低亲和力结合青霉素[22, 23, 24, 25]。研究发现该酶由一段含49个氨基酸的前导肽、胞壁质酶结构域、青霉素结合域以及含有6个重复单元(每个单元包含45个氨基酸且由富含丝氨酸和苏氨酸的序列间隔开)的底物结合域组成[26]。这种结构被称为蛋白质模块化设计,即每个组成蛋白质的不同模块都负责一个特定的功能[27, 28],因此,E. hirae的第二种胞壁质酶是蛋白质模块化设计的一个典型例子,这种模块化结构中的特异性底物结合域有助于溶菌酶与肽聚糖这种不溶性的高分子底物形成功能酶-底物复合物,可以使酶更好地发挥催化作用,溶解底物。
1.1.2 β-1,4-N,6-O-二乙酰基胞壁质酶肽聚糖的差异对溶菌酶的活性会产生较大影响。研究发现,一些细菌(如金黄色葡萄球菌)对鸡卵清溶菌酶的杀菌作用不敏感是因为其细胞壁中N-乙酰胞壁酸第6位碳原子上的羟基氧原子也发生了乙酰化,影响了溶菌酶的活性[29]。而来自于真菌拟内串生孢霉(Chalaropsis)的溶菌酶被最早发现含有β-1,4-N,6-O二乙酰基胞壁质酶活性,可以水解O-,N-乙酰化的NAM与NAG之间的β-1,4糖苷键,即能够裂解细胞壁中NAM发生了O-乙酰化的细菌(图 2)[30]。对Chalaropsis溶菌酶的研究显示,其成熟肽分子量为22.4 kDa,由211个氨基酸残基组成,N末端氨基酸为Thr,C末端氨基酸为Gly,在Cys108和Cys147之间具有单一的二硫键,为弱碱性球蛋白(等电点7.5),能够彻底分解金黄色葡萄球菌、变形链球菌等多种革兰氏阳性菌,但对革兰氏阴性菌没有作用[31]。
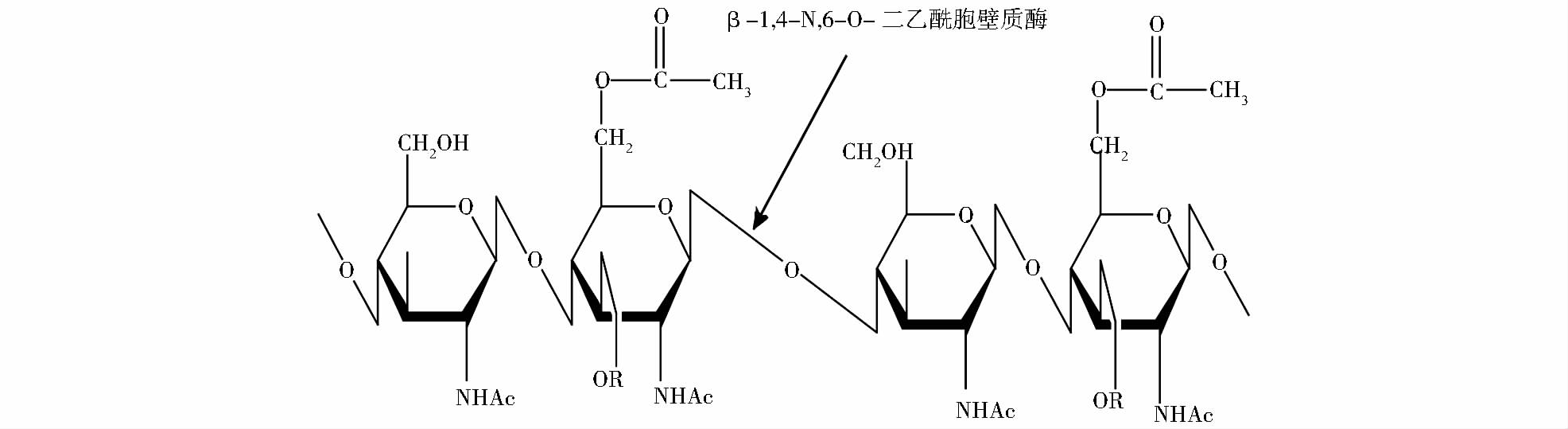
|
| 图 2 β-1,4-N,6-O-二乙酰胞壁质酶的作用底物示意图(NAM第 6位羟基的氧原子被乙酰化) Fig. 2 Substrate of β-1,4-N,6-O-diacetylmuramidase (6-O-acetylated bacterial peptidoglycan) |
继Chalaropsis之后,链霉菌属(Streptomyces)、嗜酸乳杆菌(Lactobacillus acidophilus)、丙酮丁醇梭菌(Clostridium acetobutylicum)以及一些细菌的噬菌体中也发现了β-1,4-N,6-O二乙酰基胞壁质酶的存在(表 1)。其中,对球孢链霉菌(Streptomyces globisporus)N,6-O二乙酰基胞壁质酶M1的研究较多:该酶由acm基因编码,翻译产生包括多达77个氨基酸的前导肽和217个氨基酸的成熟肽。M1溶菌酶分子量约为23kDa,分子中含有一个二硫键,等电点为10.88。对N端序列进行分析显示,M1溶菌酶与Chalaropsis溶菌酶有47.3%的相同度和61%的相似性[32, 33]。Seo等[34]在大肠杆菌中对M1溶菌酶基因进行了重组表达,重组酶被表达至周质空间并具有生物活性,但是表达量非常低,原因在于β-1,4-N,6-O二乙酰基胞壁质酶可能会对宿主菌产生一定的毒性。目前,来自球孢链霉菌的多种不同溶菌酶所组成的混合物“变溶菌素(mutanolysin)”已经实现了商业化生产。这类β-1,4-N,6-O二乙酰基胞壁质酶在快速抑制牙齿表面变形链球菌的生长、预防治疗龋齿,从细胞壁提取免疫活性物质等方面都有良好的效果和应用前景[32, 33, 34, 35, 36]。
| Organism | Specificity | |
| β-1,4-N-acetylmuramidase | β-1,4-N,6-O-diacetyl-muramidase | |
| Arthrobacter crystallopoites | + | - |
| Bacillus thuringensis | + | - |
| Bacillus subtilis | + | - |
| Clotridium acetobutylicum | + | + |
| Enterococcus hirae M-1 | + | - |
| Enterococcus hirae M-2 | + | - |
| Lactobacillus acidophilus | + | + |
| Pseudomonas aeruginosa | + | - |
| Streptomyces coeficolor | + | + |
| Streptomyces erythraeus | + | + |
| Streptomyces globisporus | + | + |
| Streptomyces griseus | + | + |
基于溶菌酶具有的特异性催化肽聚糖发生水解的胞壁质酶活性(muramidase activty),在很长一段时间里人们都认为溶菌酶只能裂解革兰氏阳性菌(Gram-positive bacteria,G+),而对革兰氏阴性菌(Gram-negative bacteria,G-)没有杀菌作用。这是因为革兰氏阳性菌的细胞壁包覆在细胞质膜(cytoplasmic memberane)的外层,主要由交联度高、厚度为20~80nm的肽聚糖层组成;而革兰氏阴性菌的细胞壁除含有交联度低、厚度约为2nm肽聚糖层外,还额外包覆着一层厚约8nm的外膜(outer memberane)且成分复杂(图 3)。由此推断,革兰氏阴性菌外膜的存在会阻碍溶菌酶与肽聚糖层直接接触,使得溶菌酶失效。但是近年来,越来越多的研究证实溶菌酶的胞壁质酶活性并不是溶菌酶杀菌的唯一机制[38, 39, 40],而溶菌酶的作用对象也不仅局限于革兰氏阳性菌。

|
| 图 3 革兰氏阳性菌和革兰氏阴性菌细胞壁组成 Fig. 3 Structure of Gram-positive and Gram-negative cell wall |
1996年,Ibrahim 等[41]在对蛋清溶菌酶加热后发现,变性后的溶菌酶不仅仍然能够作用于革兰氏阳性菌,对革兰氏阴性菌的杀菌作用甚至得到了提高。该研究指出,部分解折叠后的变性溶菌酶形成疏水性二聚体,该二聚体可以插入细菌细胞膜内并造成膜损伤,与抗菌肽机制类似。此后,Pellegrini等[42]研究发现了蛋清溶菌酶第 98~112位的氨基酸序列能抑制多种G+菌和G-菌的生长,该多肽片段不具有胞壁质酶活性,其对G+菌的作用稍逊于溶菌酶,但将Asn106突变为Arg106后,其杀菌活性得到了极大的提高。这部分序列随后被证明是定位于活性中心的狭长裂缝的上部边缘(upper lip)的一段“螺旋-环-螺旋”结构中,该二级结构在体外实验中证实可以利用其C端的螺旋结构穿透革兰氏阴性菌的外膜,并通过形成一个离子通道对细菌的内膜造成破坏。与之类似,During等[40]研究发现,热失活的T4溶菌酶其C端的两亲性螺旋结构除细菌外,对真菌也具有抑制作用。在最近的研究中,Thammasirirak 等[43]模拟肠道消化过程利用消化液蛋白酶对鹅蛋溶菌酶进行了酶解,其水解产物经纯化后得到两条抗菌肽,分别对G+菌、G-菌具有杀菌作用。此后的扫描电镜结果证实,细菌的细胞膜被完全破坏,这进一步验证了在溶菌酶分子中存在抗菌片段。
这些研究表明溶菌酶的抗菌活性并不完全依赖于其胞壁质酶活性,其特殊的局部结构特征和氨基酸序列也对抗菌作用有直接的贡献[40, 41, 42, 43, 44]。具体而言,溶菌酶的非胞壁质酶杀菌活性来自以下两种可能的机制:第一,溶菌酶与阳离子抗菌肽(cationic antimicrobial peptide)类似,都利用磷脂-蛋白相互作用原理(proteinphospholipid interaction mechanism),以物理的方式作用于细菌的细胞膜,使细胞膜形成穿膜离子通道,并破坏细胞膜的完整性,导致细胞质外溢而达到杀菌的目的[38, 44, 45]。第二种机制认为溶菌酶裂解细胞壁时会激活细胞壁中所谓自溶蛋白(autolysin enzyme)从而导致细胞裂解[44]。目前,溶菌酶的这种非酶活性杀菌能力仍有待更深入的研究。
3 溶菌酶的分子改造研究研究溶菌酶作用机制所揭示的信息,为研究人员利用现代蛋白质工程技术对溶菌酶进行分子修饰、改造,构建具有新型杀菌性质的溶菌酶提供了可行性依据。
3.1 改造溶菌酶的胞壁质酶性质通过对溶菌酶的氨基酸残基进行人为突变和定向筛选,可以有效改变溶菌酶的水解酶活、热稳定性、最适pH、底物特异性等性质,开发出更加适应现代生产需求的新型溶菌酶。例如,通过定点突变技术,Hayashi[46]证明了对于鸡卵清溶菌酶,当其活性中心的氨基酸Trp62被Tyr、Phe、His等替代后,突变酶的细菌裂解活性提高了2倍以上[46]。除了Trp62外,活性中心Asp101和Asn37残基也与“酶-底物”的结合方式密切相关。在经过定点突变后,Trp62His/Asp101Gly/Asn37Gly突变体与原始酶相比裂解酶活提高了3倍[47]。另一方面,溶菌酶所带的净(正)电荷分布也直接影响溶菌酶酶活。如鸡卵清溶菌酶突变体Val73Arg/Gln125Arg由于增加了带正电的残基,使得酶与细菌表面的负电荷之间的相互作用增强,因此突变体在离子强度更高、pH值更低的环境中具有更高的酶活。还有研究者通过替换了噬菌体溶菌酶CpI-7的15个氨基酸残基,使得CpI-7的细胞壁结合域所带电荷种类发生转变(中性pH条件下,电荷量由-14.93变为+3.0),从而影响了该酶对带负电荷的肺炎双球菌(Diplococcus pneumoniae)细胞壁的结合能力。除了对肺炎双球菌耐药菌株的致死活性显著提高外,突变酶对大肠杆菌和恶臭假单胞菌(Pseudomonas putida)也表现出了致死作用[48]。
而针对T4噬菌体溶菌酶,Mathews等也进行了大量的深入研究。例如,在前人的基础上,他们建立了一种筛选热稳定性T4噬菌体溶菌酶突变体的方法。该方法利用M13噬菌体表达系统和禽流感骨髓瘤逆转录病毒构建T4溶菌酶的突变文库,并通过平板抑菌圈法筛选得到Tm(melting temperature)改变值在1℃以下的突变酶,筛选方法灵敏度高。此后,Mathews等借助蛋白质工程技术先后研究了700余个T4噬菌体溶菌酶的热稳定性突变体(包括负突变)及其晶体学参数[8],促使该溶菌酶成为了研究蛋白质稳定性的一种模式分子。
除此之外,许多溶菌酶被发现具有典型的模块式的组成结构(modular design),包含了功能上各自独立的催化结构域、底物结合域和识别域(一般含有若干个重复的基序)[7, 48, 49]。这种模块式的结构特点与纤维素酶、几丁质酶和木聚糖酶是类似的:对于不溶性的高分子底物(如肽聚糖)的降解,特异性的底物结合域更有利于“酶-底物复合中间体”的形成[7]。有学者通过功能域洗牌(domain shuffling)以及模块设计(module engineering)的方法有效提高了噬菌体溶菌酶的水解活性和底物结合力[50]。
对溶菌酶的改良还包括化学修饰方法,如通过干热(dry-heating)处理使溶菌酶发生磷酸化(phosphorylation),显著提高了卵清溶菌酶的热稳定性、乳化能力和凝胶化学特性[51]。糖基化法(glycosylation)也是对溶菌酶进行修饰的主要方法之一,在溶菌酶分子上连接糖链有效提高了酶的稳定性和对E. coli的杀菌能力[52, 53, 54]。在此基础上,Enomoto等[55]通过将两种方法结合起来,先使用麦芽五糖对鸡卵清溶菌酶进行糖化,再通过干热法和磷酸盐使溶菌酶磷酸化,实现了溶菌酶稳定性、磷酸钙增溶性的提升和免疫原性的降低。
以上这些研究方法不仅为人们提供了更多具有优异酶学性质的溶菌酶,也为更深入地揭示溶菌酶分子结构与功能关系提供了理论基础。
3.2 改造溶菌酶的非胞壁质酶性质目前溶菌酶的抑菌谱具有很强的局限性,而围绕溶菌酶的非胞壁质酶活性进行改造是开发新型广谱溶菌酶的有效途径之一。可以设想,溶菌酶需要穿透外膜、分解肽聚糖层,再插入磷脂分子组成的质膜才能实现杀灭革兰氏阴性菌的目的,因此,必须在不影响胞壁质酶活性的前提下,强化溶菌酶对细胞膜的作用。以此为出发点,对溶菌酶的主要改造途径可以归结为以下两类。
第一类改造方法是将溶菌酶变为膜穿透模式(membrane-penetrating form)。即在溶菌酶蛋白质结构中加入一个外露的疏水结构域(surface-exposed hydrophobic domain),该结构可以与外膜发生融合,将溶菌酶定位至肽聚糖层,随后插入质膜中发挥杀菌作用(图 4a)。例如有研究报道了通过DNA重组技术在溶菌酶的羧基端附加一段适当大小的疏水肽,增加了溶菌酶对革兰氏阴性菌的杀菌效果[56]。Ibrahim [57]也通过在溶菌酶的羧基端插入一个短小的疏水性五肽(Phe-Phe-Val-Ala-Pro)使溶菌酶对大肠杆菌的杀菌效果得到了提高。这些方法模拟了膜融合蛋白(fusogenic protein)这种可以穿过生物膜的天然蛋白,使溶菌酶对G-的作用得到了加强[58]。与之类似,除了在C端添加疏水性多肽外,还可以利用化学法在赖氨酸残基上连接不同长度的饱和脂肪酸,以模拟双向蛋白(amphitropic proteins)与组成膜的脂质(可逆)结合(lipid-binding)的能力[59]。例如,有研究证明当鸡卵清溶菌酶的赖氨酸残基上分别连接了硬脂酸(C18)或者棕榈酸(C16)等饱和脂肪酸后,这些氨基酸将发挥疏水性载体的作用将溶菌酶送至质膜层。该研究发现,当酰化程度不超过摩尔比2∶1(脂肪酸残基∶溶菌酶)时,对溶菌酶的胞壁质酶活性影响程度较小,而对革兰氏阴性菌Escherichia coli K-12的杀菌能力有了显著提高[60]。此外,在溶菌酶分子上连接中、短链的饱和脂肪酸也成功拓展了溶菌酶对革兰氏阴性菌的抑菌谱[61]。但亲脂化修饰的问题在于酶活会出现一定损失。针对该问题,Liu等[62]提出在对鸡卵清溶菌酶亲脂化处理前先进行糖基化修饰,可以有效提高酶活的回收效率。
第二类改造方法则是将疏水性抗菌素与溶菌酶相结合,构建新的融合抗菌剂。研究已知,食用绿叶中含有许多种可靶向作用于质膜、具有一定杀菌能力的酚醛分子,但这类天然抗菌物质的问题在于具有疏水性,因此会固定于革兰氏阴性菌的疏水性外膜中无法接近质膜,或者被革兰氏阳性菌亲水性的细胞壁所排斥。而另一方面,此前的研究证明溶菌酶可以与革兰氏阴性菌的外膜中的脂多糖(lipopolysaccharide,LPS)发生相互作用,使得LPS与磷脂分子之间的空间结构发生形变[63],由此帮助溶菌酶穿透外膜。加之溶菌酶的胞壁质酶活性可以分解破坏肽聚糖层,因此将溶菌酶与酚醛抗菌素相结合可以帮助疏水抗菌素同时克服外膜及肽聚糖的阻碍,最终达到并富集于质膜以发挥其杀菌作用(图 4b)。以紫苏醛为例,该物质来自中国传统草药紫苏,研究证明它对革兰氏阴性菌的作用强于革兰氏阳性菌,但由于具有疏水性质,杀菌能力仅为中等水平。通过化学法将紫苏醛与卵清溶菌酶中赖氨酸残基的ε-氨基相偶联,形成的复合物对E. coli K-12和金黄色葡萄球菌均有杀菌作用,且高于两者分别作用于这两种细菌时的杀菌效力,说明紫苏醛和溶菌酶在偶联后产生了一种新的协同作用机制[64]。类似的例子还有肉桂醛,这种来自肉桂的广谱杀菌物质[65]也可以通过醛基与溶菌酶的残基发生共价交联,增强溶菌酶对E. coli和金黄色葡萄球菌的杀菌作用[66]。
4 展 望溶菌酶作为一种广泛存在的天然抗菌蛋白,不仅安全无毒、稳定性好,具有抗菌消炎、抗病毒、增加免疫力等药理作用[68, 69, 70],还可与其它抗菌剂发挥协同杀菌作用,最重要的是不会使致病菌株产生耐药性。但溶菌酶的抗菌谱较窄是其存在的一个不足。因此,开发广谱、高效的新型溶菌酶制剂,对于控制抗生素滥用、建立绿色防腐技术、维护食品药品安全等方面具有重要的应用价值。
目前,对溶菌酶进行蛋白质改造的技术手段仍集中于化学法和分子生物学方法,但技术策略不尽相同。其中,以提高溶菌酶对革兰氏阴性菌的杀菌能力为例,一种有效的策略是在已知抑菌原理的基础上对现有抗菌因子进行组合,这使得该类酶在食品、药品等方面拥有极大的应用安全优势。这些研究同时也证明了,蛋白质可以通过移植一个合适的功能域至现有的分子结构中以实现特定的生物功能。因此,延续这个策略并进一步探索溶菌酶与其它生物活性分子的结合,很有可能为我们提供更多更有价值的新产品。
此外,值得注意的是溶菌酶几乎都是大分子,具有潜在的免疫原性,对其在医药和食品中的应用可能会造成一定的限制。因此,有学者致力于研究溶菌酶中抗菌肽的分离与制备[20, 71, 72, 73, 74]。虽然实验结果显示这些短肽的杀菌活性与溶菌酶相比较低,但小分子肽的生产成本更低,因此可以平衡酶活较低的问题。基于这一点,笔者认为通过生物信息学数据合理设计获得抗性短肽的策略也会成为未来溶菌酶的研究方向之一。
| [1] | Jolles P, Jolles J. What’s new in lysozyme research? Always a model system, today as yesterday. Mol Cell Biochem, 1984, 63 (2): 165-189. |
| [2] | Davis K M, Weiser J N. Modifications to the peptidoglycan backbone help bacteria to establish infection. Infection and Immunity, 2010, 79 (2): 562-570. |
| [3] | Rubio C A. The natural antimicrobial enzyme lysozyme is up-regulated in gastrointestinal inflammatory conditions. Pathogens, 2014, 3 (1): 73-92. |
| [4] | Hasselberger F X. Uses of Enzymes and Immobilized Enzymes. Chicago:Nelson-Hall, 1978. |
| [5] | 王佃亮. 重组人溶菌酶研究进展. 中国生物工程杂志, 2003, 23 (9): 59-62. Wang D L.Research progress in recombinant human lysozyme.China Biotechnology,2013,23(9):59-62. |
| [6] | During K. Can lysozymes mediate antibacterial resistance in plants? Plant Mol Biol, 1993, 23 (1): 209-214. |
| [7] | Jollès P. Lysozymes: Model Enzymes in Biochemistry and Biology. Birkhäuser Verlag, 1996. |
| [8] | Baase W A, Liu L, Tronrud D E, et al. Lessons from the lysozyme of phage T4. Protein Science, 2010, 19 (4): 631-641. |
| [9] | Shockman G D, Holtje J-V. Buereriul Cell Wall. Elsevier, 1994. |
| [10] | Hughey V L, Johnson E A. Antimicrobial activity of lysozyme against bacteria involved in food spoilage and food-borne disease. Applied and Environmental Microbiology, 1987, 53 (9): 2165-2170. |
| [11] | Dias R, Vilas-Boas E, Campos F M, et al. Activity of lysozyme on Lactobacillus hilgardii strains isolated from Port wine. Food Microbiology, 2015, 49: 6-11. |
| [12] | Guzzo F, Cappello M S, Azzolini M, et al. The inhibitory effects of wine phenolics on lysozyme activity against lactic acid bacteria. International Journal of Food Microbiology, 2011, 148 (3): 184-190. |
| [13] | Harding R L, Henshaw J, Tilling J, et al. Thioester analogues of peptidoglycan fragment MurNAc-L-Ala-[gamma]-D-Glu as substrates for peptidoglycan hydrolase MurNAc-L-Ala amidase. Journal of the Chemical Society, Perkin Transactions 1, 2002, (14): 1714-1722. |
| [14] | Cheng X, Zhang X, Pflugrath J W, et al. The structure of bacteriophage T7 lysozyme, a zinc amidase and an inhibitor of T7 RNA polymerase. Proceedings of the National Academy of Sciences of the United States of America, 1994, 91 (9): 4034-4038. |
| [15] | Loessner M J, Maier S K, Daubek-Puza H, et al. Three Bacillus cereus bacteriophage endolysins are unrelated but reveal high homology to cell wall hydrolases from different bacilli. Journal of Bacteriology, 1997, 179 (9): 2845-2851. |
| [16] | Brnneke V, Fiedler F. Production of bacteriolytic enzymes by Streptomyces globisporus regulated by exogenous bacterial cell walls. Applied and Environmental Microbiology, 1994, 60 (3): 785-791. |
| [17] | Beukes M, Bierbaum G, Sahl H G, et al. Purification and partial characterization of a murein hydrolase, Millericin B, produced by Streptococcus milleri NMSCC 061. Applied and Environmental Microbiology, 2000, 66 (1): 23-28. |
| [18] | Phillips D C. The three dimensional structure of an enzyme molecule. Scientific American, 1966, 215 (5): 78-90. |
| [19] | Zlesnierowski G, Kijowski J, Lysozyme, in: Huopalahti R, López-Fandiño R. Bioactive Egg Compounds. Heidelberg:Springer, 2007.33-40. |
| [20] | Barrett J F, Schramm V L, Shockman G D. Hydrolysis of soluble, linear, un-cross-linked peptidoglycans by endogenous bacterial N-acetylmuramoylhydrolases. Journal of Bacteriology, 1984, 159 (2): 520-526. |
| [21] | Barrett J F, Dolinger D L, Schramm V L, et al. The mechanism of soluble peptidoglycan hydrolysis by an autolytic muramidase. A processive exodisaccharidase. Journal of Biological Chemistry, 1984, 259 (19): 11818-11827. |
| [22] | Kariyama R, Shockman G D. Extracellular and cellular distribution of muramidase-2 and muramidase-1 of Enterococcus hirae ATCC 9790. Journal of Bacteriology, 1992, 174 (10): 3236-3241. |
| [23] | Ghuysen J M. Serine beta-lactamases and penicillin-binding proteins. Annual Review of Microbiology, 1991, 45 (1): 37-67. |
| [24] | Joris B, Englebert S, Chu C P, et al. Modular design of the Enterococcus hirae muramidase-2 and Streptococcus faecalis autolysin. FEMS Microbiology Letters, 1992, 91 (3): 257-264. |
| [25] | Shockman G D. The autolytic (‘suicidase’) system of Enterococcus hirae: from lysine depletion autolysis to biochemical and molecular studies of the two muramidases of Enterococcus hirae ATCC 9790. FEMS Microbiology Letters, 1992,100 (1-3): 261-267. |
| [26] | Chu C P, Kariyama R, Daneo-Moore L, et al. Cloning and sequence analysis of the muramidase-2 gene from Enterococcus hirae. Journal of Bacteriology, 1992, 174 (5): 1619-1625. |
| [27] | García P, García J, García E, et al. Modular organization of the lytic enzymes of Streptococcus pneumoniae and its bacteriophages. Gene, 1990, 86 (1): 81-88. |
| [28] | Diaz E, López R, Garcia J L. Chimeric pneumococcal cell wall lytic enzymes reveal important physiological and evolutionary traits. Journal of Biological Chemistry, 1991, 266 (9): 5464-5471. |
| [29] | Brumfitt W, Wardlaw A C, Park J T. Development of lysozyme-resistance in Micrococcus lysodiekticus and its association with an increased o-acetyl content of the cell wall. Nature, 1958, 181 (4626): 1783-1784. |
| [30] | Felch J W, Inagami T, Hash J H. The N, O-diacetylmuramidase of Chalaropsis species. V. The complete amino acid sequence. Journal of Biological Chemistry, 1975, 250 (10): 3713-3720. |
| [31] | Shih J W, Hash J H. The N,O-diacetylmuramidase of Chalaropsis Species: III. amino acid composition and partial structural formula. Journal of Biological Chemistry, 1971, 246 (4): 994-1006. |
| [32] | Lichenstein H S, Hastings A E, Langley K E, et al. Cloning and nucleotide sequence of the N-acetylmuramidase M1-encoding gene from Streptomyces globisporus. Gene, 1990, 88 (1): 81-86. |
| [33] | Seo H J, Kitaoka M, Ohmiya K, et al. Substrate specificity of the N,6-O-diacetylmuramidase from Streptomyces globisporus. Journal of Bioscience and Bioengineering, 2003, 95 (3): 313-316. |
| [34] | Seo H J, Shimonishi T, Ohmiya K, et al. Characterization of N-acetylmuramidase M-1 of Streptomyces globisporus produced by Escherichia coli BL21(DE3)pLysS. Journal of Bioscience and Bioengineering, 2001, 92 (5): 472-474. |
| [35] | 曹涛, 刘同军, 王艳君. 微生物溶菌酶的研究及应用. 中国调味品, 2011, 36 (3): 23-26,32. Cao T, Liu T J, Wang Y J. Research and application of microbial lysozyme. China Condiment, 2011, 36 (3): 23-26,32. |
| [36] | 刘同军, 徐文琳, 张玉臻. 变溶菌素Mutanolysin研究历史和发展前景. 微生物学报, Liu T J, Xu W L, Zhang Y Z. History and prospects of the research on mutanolysin. Acta Microbiologica Sinica, 2000, 40 (2): 224-227. |
| [37] | Glynn L E. Lysozymes: Model Enzymes in Biochemistry and Biology. Basel:John Wiley & Sons, 1997. |
| [38] | Pellegrini A, Thomas U, von Fellenberg R, et al. Bactericidal activities of lysozyme and aprotinin against gram-negative and gram-positive bacteria related to their basic character. J Appl Bacteriol, 1992, 72 (3): 180-187. |
| [39] | Ibrahim H R, Matsuzaki T, Aoki T. Genetic evidence that antibacterial activity of lysozyme is independent of its catalytic function. FEBS Lett, 2001, 506 (1): 27-32. |
| [40] | During K, Porsch P, Mahn A, et al. The non-enzymatic microbicidal activity of lysozymes. FEBS Lett, 1999, 449 (2-3): 93-100. |
| [41] | Ibrahim H R, Higashiguchi S, Juneja L. A structural phase of heat-denatured lysozyme with novel antimicrobial action. J Agric Food Chem, 1996, (44): 1416-1423. |
| [42] | Pellegrini A, Thomas U, Bramaz N, et al. Identification and isolation of a bactericidal domain in chicken egg white lysozyme. J Appl Microbiol, 1997, 82 (3): 372-378. |
| [43] | Thammasirirak S, Pukcothanung Y, Preecharrama S. Antimicrobial peptides drived from goose egg white lysozyme. Comp Biochem Phy, 2010, 151: 84-91. |
| [44] | Ibrahim H R, Higashiguchi S, Juneja L R, et al. A structural phase of heat-denatured lysozyme with novel antimicrobial action. Journal of Agricultural and Food Chemistry, 1996, 44: 1416-1423. |
| [45] | Ibrahim H R, Higashiguchi S, Koketsu M, et al. Partially unfolded lysozyme at neutral pH agglutinates and kills gram-negative and Gram-positive bacteria through membrane damage mechanism. Journal of Agricultural and Food Chemistry, 1996, 44: 3799-3806. |
| [46] | Hayashi K. The position of the active tryptophan residue in lysozyme. J Biochem, 1965, 58: 227-235. |
| [47] | Kumagai I. Redesign of the substrate-binding site of hen egg white lysozyme based on the molecular evolution of C-type Iysozymes. J BioI Chem, 1992, 267: 4608-4612. |
| [48] | Diez-Martinez R, de Paz H, Bustamante N, et al. Improving the lethal effect of Cpl-7, a Pneumococcal phage lysozyme with broad bactericidal activity, by inverting the net charge of its cell wall-binding module. Antimicrobial Agents and Chemotherapy, 2013, 57 (11): 5355-5365. |
| [49] | Hermoso J, Monterroso B, Albert A, et al. Structural basis for selective recognition of Pneumococcal cell wall by modular endolysin from phage Cp-1. Structure, 2003, 11 (10): 1239-1249. |
| [50] | Schmelcher M, Tchang V S, Loessner M J. Domain shuffling and module engineering of Listeria phage endolysins for enhanced lytic activity and binding affinity. Microbial Biotechnology, 2011, 4 (5): 651-662. |
| [51] | Li C P, Salvador A S, Ibrahim H R, et al. Phosphorylation of egg white proteins by dry-heating in the presence of phosphate. Journal of Agricultural and Food Chemistry, 2003, 51: 6808-6815. |
| [52] | Nakamura N K, Furukawa N, Matsuoka M, et al. Enzyme activity of lysozyme-dextran complex prepared by high-pressure treatment. Food Science and Technology International, 1997, 3: 235-238. |
| [53] | Seo S, Karboune S, Yaylayan V, et al. Glycation of lysozyme with galactose, galactooligosaccharides and potato galactan through the Maillard reaction and optimization of the production of prebiotic glycoproteins. Process Biochemistry, 2012, 47 (2): 297-304. |
| [54] | Seo S, Karboune S, L'Hocine L, et al. Characterization of glycated lysozyme with galactose, galactooligosaccharides and galactan: Effect of glycation on structural and functional properties of conjugates. LWT - Food Science and Technology, 2013, 53 (1): 44-53. |
| [55] | Enomoto H, Nagae S, Hayashi Y, et al. Improvement of functional properties of egg white protein through glycation and phosphorylation by dry-heating. Asian-Australasian Journal of Animal Sciences, 2009, 22 (4): 591-597. |
| [56] | Hideyuki A. Bactericidal action of lysozymes attached with various sizes of hydrophobic peptides to the C-terminal using genetic modification. FEBS Letters, 1997, 415: 114-118. |
| [57] | Ibrahim H R. Enhanced bactericidal action of lysozyme to Escherichia coli by inserting a ydrophobic pentapeptide into its C terminus. The Journal of Biological Chemistry, 1993, 269: 5059-5063. |
| [58] | Tucker A D, Parker M W, Tsernoglou D, et al. Crystallization of a proform of aerolysin, a hole-forming toxin from Aeromonas hydrophila. Journal of Molecular Biology, 1990, 212 (4): 561-562. |
| [59] | Burn P, Dalle Carbonare B H, Lipid-protein interactions in biological membranes, in: Bittar E E, Neville B, ed. Principles of Medical Biology, Elsevier, 1997.39-66. |
| [60] | Ibrahim H R, Kobayashi K, Kato A. Length of hydrocarbon chain and antimicrobial action to Gram-negative bacteria of fatty acylated lysozyme. Journal of Agricultural and Food Chemistry, 1993, 41 (7): 1164-1168 |
| [61] | Liu S T, Sugimoto T, Azakami H, et al. Lipophilization of lysozyme by short and middle chain fatty acids. Journal of Agricultural and Food Chemistry, 2000, 48: 265-269. |
| [62] | Liu S, Azakami H, Kato A. Improvement in the yield of lipophilized lysozyme by the combination with Maillard-type glycosylation. Food / Nahrung, 2000, 44 (6): 407-410. |
| [63] | Ibrahim H R, Yamada M, Matsushita K, et al. Enhanced bactericidal action of lysozyme to Escherichia coli by inserting a hydrophobic pentapeptide into its C terminus. Journal of Biological Chemistry, 1994, 269 (7): 5059-5063. |
| [64] | Ibrahim H R, Hatta H, Fujiki M, et al. Enhanced antimicrobial action of lysozyme against gram-negative and gram-positive bacteria due to modification with perillaldehyde. Journal of Agricultural and Food Chemistry, 1994, 42: 1813-1817. |
| [65] | de Oliveira M, Brugnera D, do Nascimento J, et al. Cinnamon essential oil and cinnamaldehyde in the control of bacterial biofilms formed on stainless steel surfaces. Eur Food Res Technol, 2012, 234 (5): 821-832. |
| [66] | Valenta C, Bernkop-Schnürch A, Schwartz M. Modification of lysozyme with cinnamaldehyde: A strategy for constructing novel preservatives for dermatics. International Journal of Pharmaceutics, 1997, 148 (2): 131-137. |
| [67] | Ibrahim H R, Aoki T, Pellegrini A. Strategies for new antimicrobial proteins and peptides: lysozyme and aprotinin as model molecules. Current Pharmaceutical Design, 2002, 8: 671-693. |
| [68] | Nakatsuji T, Gallo R L. Antimicrobial Peptides: Old Molecules with New Ideas. Journal of Investigative Dermatology, 2011, 132 (3): 887-895. |
| [69] | May K D, Wells J E, Maxwell C V, et al. Granulated lysozyme as an alternative to antibiotics improves growth performance and small intestinal morphology of 10-day-old pigs. Journal of Animal Science, 2012, 90 (4): 1118-1125. |
| [70] | Oliver W T, Wells J E. Lysozyme as an alternative to antibiotics improves growth performance and small intestinal morphology in nursery pigs. Journal of Animal Science, 2013, 91: 3129-3136. |
| [71] | Abdou A M, Higashiguchi S, Aboueleinin A M, et al. Antimicrobial peptides derived from hen egg lysozyme with inhibitory effect against Bacillus species. Food Control, 2007, 18 (2): 173-178. |
| [72] | You S-J, Udenigwe C C, Aluko R E, et al. Multifunctional peptides from egg white lysozyme. Food Research International, 2010, 43 (3): 848-855. |
| [73] | Memarpoor-Yazdi M, Asoodeh A, Chamani J. A novel antioxidant and antimicrobial peptide from hen egg white lysozyme hydrolysates. Journal of Functional Foods, 2012, 4 (1): 278-286. |
| [74] | Ibrahim H R, Inazaki D, Abdou A, et al. Processing of lysozyme at distinct loops by pepsin: A novel action for generating multiple antimicrobial peptide motifs in the newborn stomach. Biochimica et Biophysica Acta (BBA) - General Subjects, 2005, 1726 (1): 102-114. |
 2015, Vol. 35
2015, Vol. 35





