文章信息
- 张瑞祥, 杨忠
- ZHANG Rui-xiang, YANG Zhong
- 海底沉积物中几丁质酶的筛选、分离及活性分析
- Screening, Identification and Enzymatic Properties Studies of Chitinase from Submarine Sediments
- 中国生物工程杂志, 2015, 35(8): 76-82
- China Biotechnology, 2015, 35(8): 76-82
- http://dx.doi.org/10.13523/j.cb.20150811
-
文章历史
- 收稿日期:2015-06-05
- 修回日期:2015-06-23
几丁质是自然界中总量仅次于纤维素的天然多糖,降解后得到的衍生物在生物、能源、医疗、农业等各个方面具有广泛的应用价值[1]。工业化制备几丁质衍生物必须首先获得壳聚糖,这通常需要对贝壳类原材料进行一定的预处理(采用强酸或强碱除去蛋白杂质和色素等[2]),在得到壳聚糖之后普遍采用三种处理方法以获得衍生物,分别为:化学法、物理法和酶解法。其中化学法过程简单,速度快,但是缺点在于其产物中单糖较多,浓酸消耗大,易发生副反应。物理法虽然能耗低,但是设备要求高,并且产物不均一[3]。利用几丁质酶(chitinase)特异性降解壳聚糖的酶解法比前两种方法过程简单,条件温和,产物的分子量易控制,是一种更为合理、高效的手段。几丁质酶广义上是指所有与几丁质降解有关的酶,而狭义上是指能特异性催化水解β-1,4-糖苷键的酶。工业中普遍使用的几丁质酶是从沙雷氏菌属(Serratia)中分离得到的,但是其分泌的几丁质酶活性较低或不稳定[4]。因此寻找能分泌高稳定、高活性的几丁质酶产生菌是几丁质酶研究领域的热点,同时也是几丁质生产工业化亟需解决的问题。
海洋是巨大的微生物资源库,海洋微生物分泌的几丁质酶因为其独特的自然环境,大多具有比较宽的pH值适应范围或者温度的耐受力[4]。Rashad等[5]以埃及沿海地区的海水、沉积物及虾壳为样本筛选得到一株灰色链霉菌(Streptomyces);Subhash等[6]从深海沉积物中筛选得到一株盐水微菌(Salinimicrobium strain JC207),以上两株几丁质酶产生菌均具有工业化应用的价值。中国同样具有丰富的海洋资源,目前在汕头海域[7],南海海域[8]及黄海沿岸[9]等都有几丁质酶产生菌筛选工作的相关报道。本研究以北部湾沉积物为样本,筛选得到一株几丁质酶产生菌蜡样芽孢杆菌B04株,并优化了其产几丁质酶的发酵条件,成功分离纯化菌株分泌的几丁质酶,并初步研究了该酶的酶学性质,为大规模筛选新型几丁质酶奠定了基础。
1 材料与方法 1.1 材料与试剂中国北部湾沉积物由上海交通大学生命科学技术学院王风平教授课题组惠赠。沉积物分别来自海底以下6~8cm(A组)和12~15cm(B组)。胶体几丁质根据Kabana等[10]的方法制备并略有修改,使用磷酸盐缓冲液(20 mmol/L,pH 6.0)调节至浓度为1%(w/v)。DNS试剂根据Miller[11]的方法制备并略有修改。其他试剂为国产分析纯。
1.2 培养基分离纯化培养基:3 g/L 蛋白胨,0.5 g/L MgSO4·7H2O,0.02 g/L FeSO4·7H2O,0.01 g/L ZnSO4·7H2O,0.3 g/L KH2PO4,0.7 g/L K2HPO4,1 g/L 胶体几丁质,1.5% 琼脂,人工海水[12]定容,pH 7.0,高压蒸汽灭菌备用。发酵培养基:2 g/L 蛋白胨,1 g/L 葡萄糖,2 g/L 酵母粉,0.5 g/L MgSO4·7H2O,0.01 g/L FeSO4·7H2O,0.3 g/L KH2PO4,0.7 g/L K2HPO4,1 g/L 胶体几丁质,去离子水定容,pH 7.0,高压蒸汽灭菌备用。
1.3 几丁质酶活性的测定以胶体几丁质为底物采用DNS法测几丁质酶的活性。将100 μl 酶液加入100 μl 底物中,置于37℃反应2h,反应结束后煮沸10 min终止反应。取上清,加入DNS试剂,煮沸10 min,冷却至室温后测量反应体系在540 nm处的吸光值,以失活的酶液作为空白对照。以N-乙酰氨基葡萄糖(GlcNAc)为底物制作标准曲线,根据标准曲线得到反应产生还原糖的含量。根据还原糖的量计算其相应的酶活力。
一个酶活力单位(U)的定义为:在37℃,pH 6.0的20 mmol/L磷酸盐缓冲液中每分钟产生1μmol 还原糖所需的酶量。本实验中采用Bradford法测定蛋白质的浓度。
1.4 微生物分离培养及几丁质酶粗酶液的收集取适量海底沉积物,用无菌水进行梯度稀释,选取合适浓度涂布于分离纯化培养基。37℃培养3~5天后挑取产生透明圈的菌落进行多次划线分离,得到单克隆。将分离得到的菌株进行发酵培养,测量发酵液中蛋白质浓度和几丁质酶活性,选取比活力最高的菌株进行后续实验。提取该菌株基因组,使用27F和1492R通用引物扩增菌株的16S rDNA序列,构建系统发育树。
将该菌株接种于发酵培养基中,单因子实验优化菌株的最适发酵产酶条件,按照最适条件发酵培养,离心收集发酵液上清,作为几丁质酶纯化的粗酶液。
1.5 几丁质酶的纯化纯化实验均在4℃条件下进行。
使用缓冲液 A (20 mmol/L Tris-HCl,300 mmol/L NaCl,5%甘油,pH 8.0)预平衡含有几丁质结合珠的重力柱。将粗酶液与磁珠混合,孵育1h。弃掉未结合蛋白,之后用0.3 mol/L NaOH洗脱结合蛋白,即为粗酶液中含有几丁质结合域的蛋白。
采用硫酸铵沉淀的方法对粗酶液中的几丁质酶进行初步纯化,确定硫酸铵沉淀的最佳浓度范围。将沉淀得到的蛋白复溶到缓冲液B(20 mmol/L Tris-HCl,5%甘油,pH 8.0)中,过夜透析,以备后续纯化。将透析后的上清依次过HiTrap Butyl FF柱,HiTrap Q FF柱及Superdex 75 10/300柱。在每一步层析过程中收集具有几丁质酶活性的组分进行下一步的纯化。
1.6 几丁质酶的酶学性质分析对纯化得到的酶进行酶学性质分析。使用不同的缓冲液体系研究该酶的最适pH,分别是50 mmol/L: glycine-HCl 缓冲液(pH 3.0),sodium acetate缓冲液(pH: 4.0~5.0),phosphate缓冲液(pH: 6.0~8.0)和glycine-NaOH缓冲液(pH: 9.0~10.0)。
研究该酶在4~70℃范围内最适反应温度[13],20~120 min范围内的最佳反应时间,0.5%~3% (w/v)范围内的最佳底物浓度,以及不同金属离子、不同底物对几丁质酶活性的影响。研究的金属离子包括:Mn2+ (MnSO4),Ni2+ (NiSO4),Fe3+ (FeCl3),Zn2+ (ZnSO4),Co2+ (CoSO4),Cu2+ (CuSO4),Ca2+ (CaCl2),Ba2+ (BaCl2),Sr2+ (SrCl2),Mg2+ (MgCl2),Ag+ (AgNO3),浓度均为5mmol/L。
2 结 果 2.1 几丁质降解菌的筛选及鉴定涂板发现10-5为最佳稀释浓度,挑取产生透明圈的阳性菌落进行多次划线分离,共筛选得到21株产几丁质酶的菌株。将菌株分别进行发酵培养,测量发酵液中蛋白质的含量以及还原糖的含量,根据标准曲线(图 1a,R2=0.9958)计算其相应酶活力,其中菌株B04分泌的几丁质酶比活力最高(图 1b)。将该菌株的16S rDNA序列在NCBI数据库中进行BLAST比对发现,B04与Bacillus anthracis(Accession number: KF973290.1)和Bacillus cereus(Accession number: KF672365.1)等的相似度为98%以上,采用邻位连接法(Neighbor-joining)构建系统发育树(图 1c),鉴定B04菌株为蜡样芽孢杆菌(Bacillus cereus),命名为Bacillus cereus strain B04。
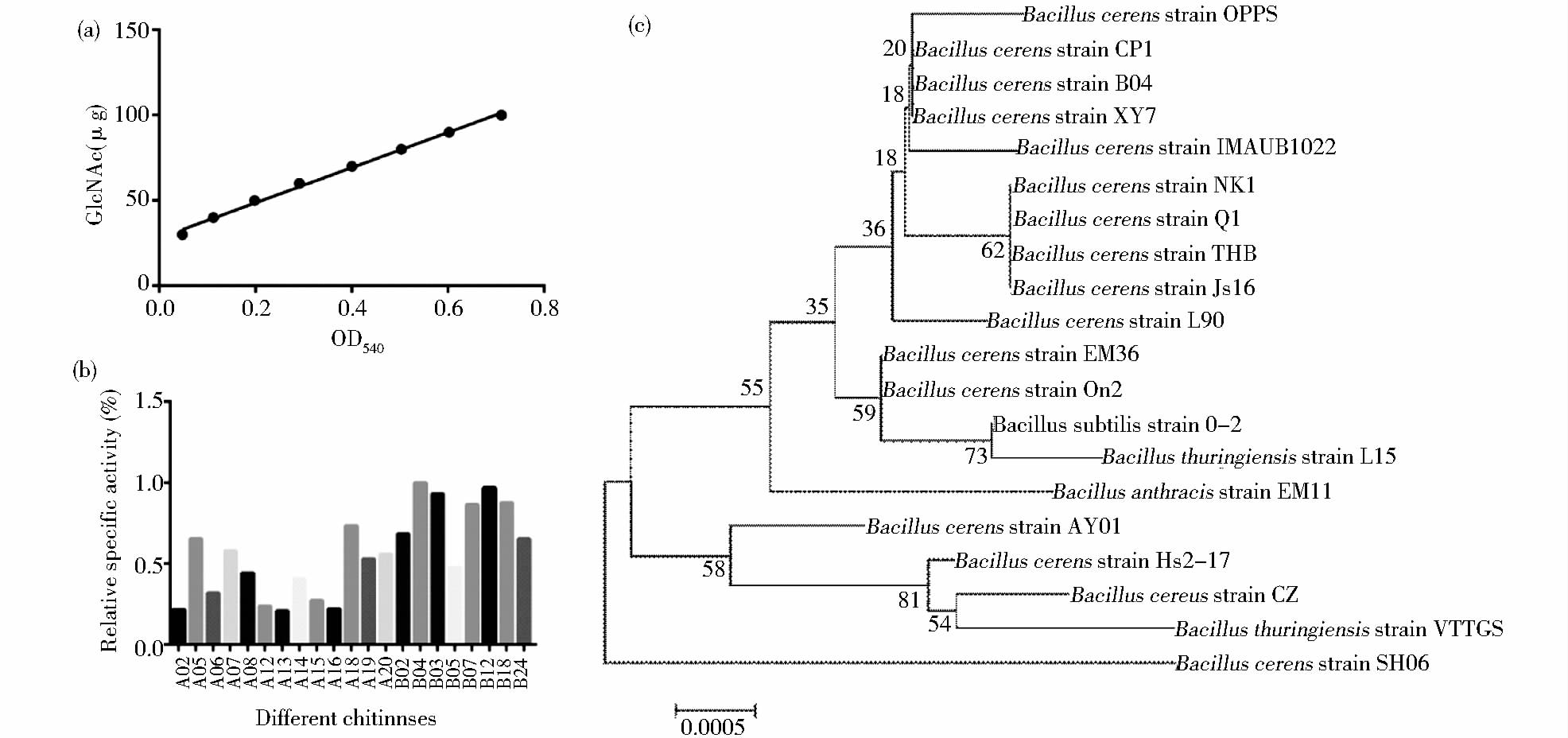
|
| 图 1 N-乙酰氨基葡萄糖标准曲线(a),B. cereus strainB04菌株的筛选(b)及系统发育树(c) Fig. 1 Standard curve of N-acetylglucosamine(a),the screening(b) and phylogenetic tree of B. cereus strain B04(c) |
单因子实验优化菌株的发酵产酶条件,以5%的接种量接种到250ml摇瓶中,220 r/min,26℃恒温培养,培养一定时间后,测菌株产酶的活性。
如图 2所示,初始pH对B. cereus strain B04菌株发酵产酶的影响比较明显,在pH5.0~9.0范围内随着pH的升高,几丁质酶的比活力也开始上升,在碱性条件下,产酶的比活力更高,当初始pH为9.0时活性最高。使用250ml的摇瓶时,装液量在40~70ml的范围内,对几丁质酶比活力的影响并不明显,但当高于70ml时,比活力出现了明显的下降。当胶体几丁质浓度低于2g/L时,产酶的比活力较低。浓度为2~4g/L时,发酵产酶的比活力最高,但当浓度高于4g/L时,酶的比活力出现了明显的下降。在氮源的利用方面,B. cereus strain B04比蛋白胨对无机氮源的利用率更高,而在无机盐中硝酸盐的利用率要比铵盐的略高。
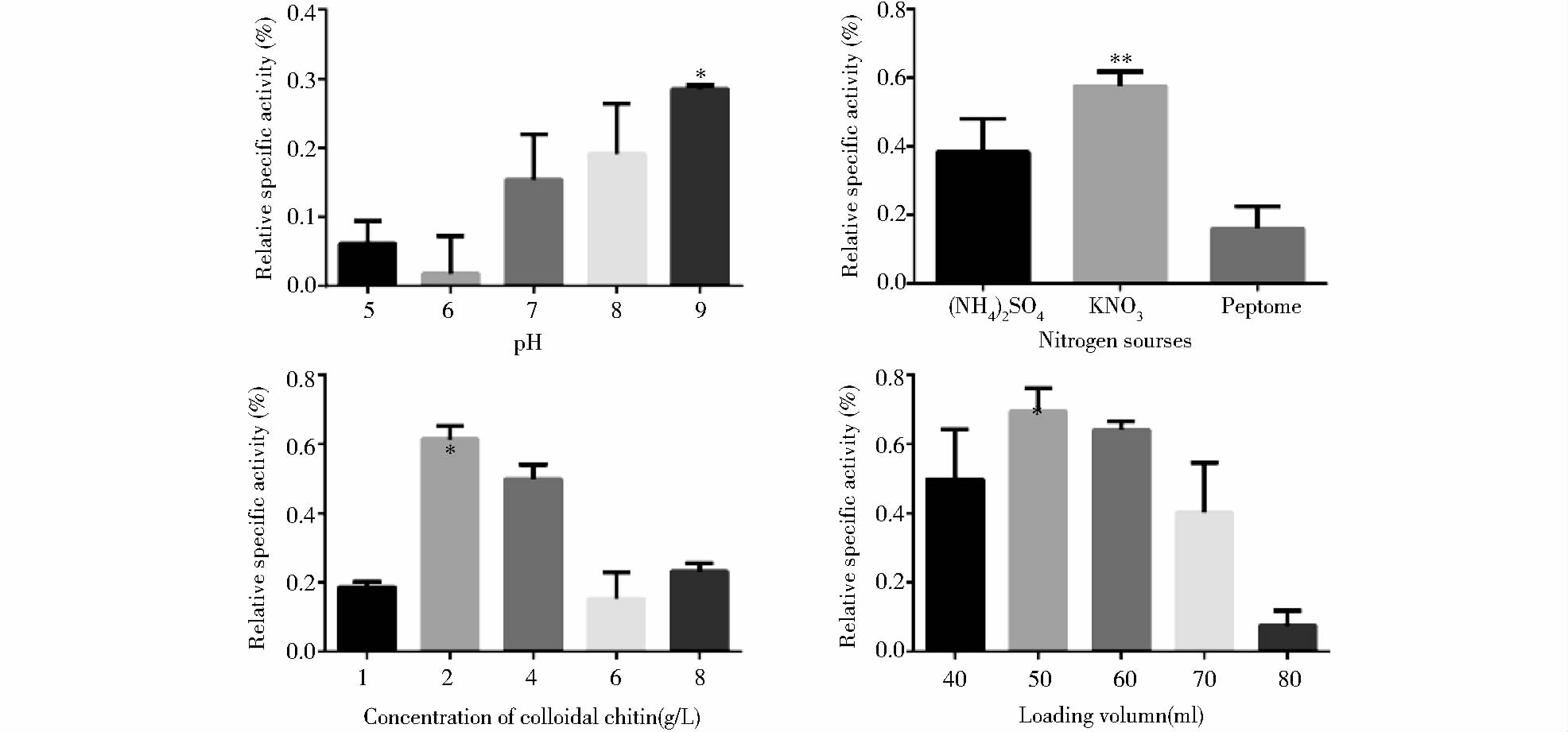
|
| 图 2 单因子对B. cereus strainB04发酵产酶的影响 Fig. 2 The effect of single factor on the produced chitinase activity of B. cereus strainB04 (* : P<0.05,** : P<0.01) |
B. cereus strain B04菌株的发酵液经过与几丁质珠特异结合后,洗脱出6个明显条带(图 3),即B. cereus strain B04的发酵液上清中共有6种不同的含有几丁质结合域的蛋白,蛋白分子量大小在35.0~66.2 kDa之间。
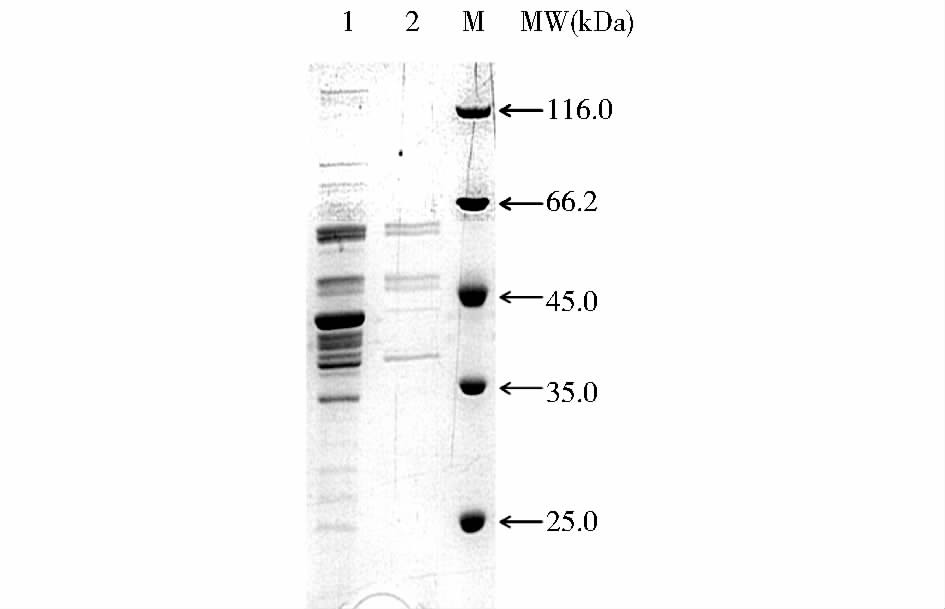
|
| 图 3 几丁质珠亲和层析电泳图 泳道1:粗酶液;泳道2:亲和层析洗脱液;泳道M:蛋白Marker Fig. 3 The electrophoretogram of chitin beads affinity chromatography |
B. cereus strain B04菌株的发酵液上清经过硫酸铵沉淀(最佳沉淀浓度为20%~85%)、疏水层析、离子交换层析和分子筛层析之后(表 1),纯化得到的几丁质酶,大小为36 kDa,纯度达到电泳纯,比活力为2.12 U/mg,活力回收率为15.27%,纯化倍数达到21.20。将纯化得到的酶进行N端氨基酸测序发现,该几丁质酶为Chi36。
| 纯化步骤 | 总酶活(U) | 总蛋白(mg) | 比活力(U/mg) | 活力回收率(%) | 纯化倍数 |
| 粗酶液 | 46.94 | 478.78 | 0.10 | 100.00 | 1.00 |
| 硫酸铵沉淀 | 26.51 | 79.44 | 0.33 | 56.48 | 3.30 |
| HiTrap Butyl FF | 10.87 | 14.76 | 0.74 | 23.16 | 7.40 |
| HiTrap Q FF | 9.90 | 8.63 | 1.15 | 21.10 | 11.50 |
| Superdex 75 | 7.17 | 3.39 | 2.12 | 15.27 | 21.20 |
本研究考察了多种因素对几丁质酶活性的影响(图 4)。几丁质酶的最适pH为4.0,在pH值为:6.0~10.0的范围内,能保留大约50%的活力。最佳反应温度为60℃,测量酶活力的最佳反应时间为80min。当胶体几丁质的浓度为1.5%(w/v)时,几丁质酶的酶活力最高,而浓度过高或者过低都会抑制它的活力。Co2+对几丁质酶的酶活力有明显的促进作用,而Ba+、Sr2+、Mg2+和Ag+对几丁质酶的活力则有抑制作用。在底物特异性方面,分离纯化的酶能够高效利用胶体几丁质,而对天然几丁质及壳聚糖则没有明显的降解作用。
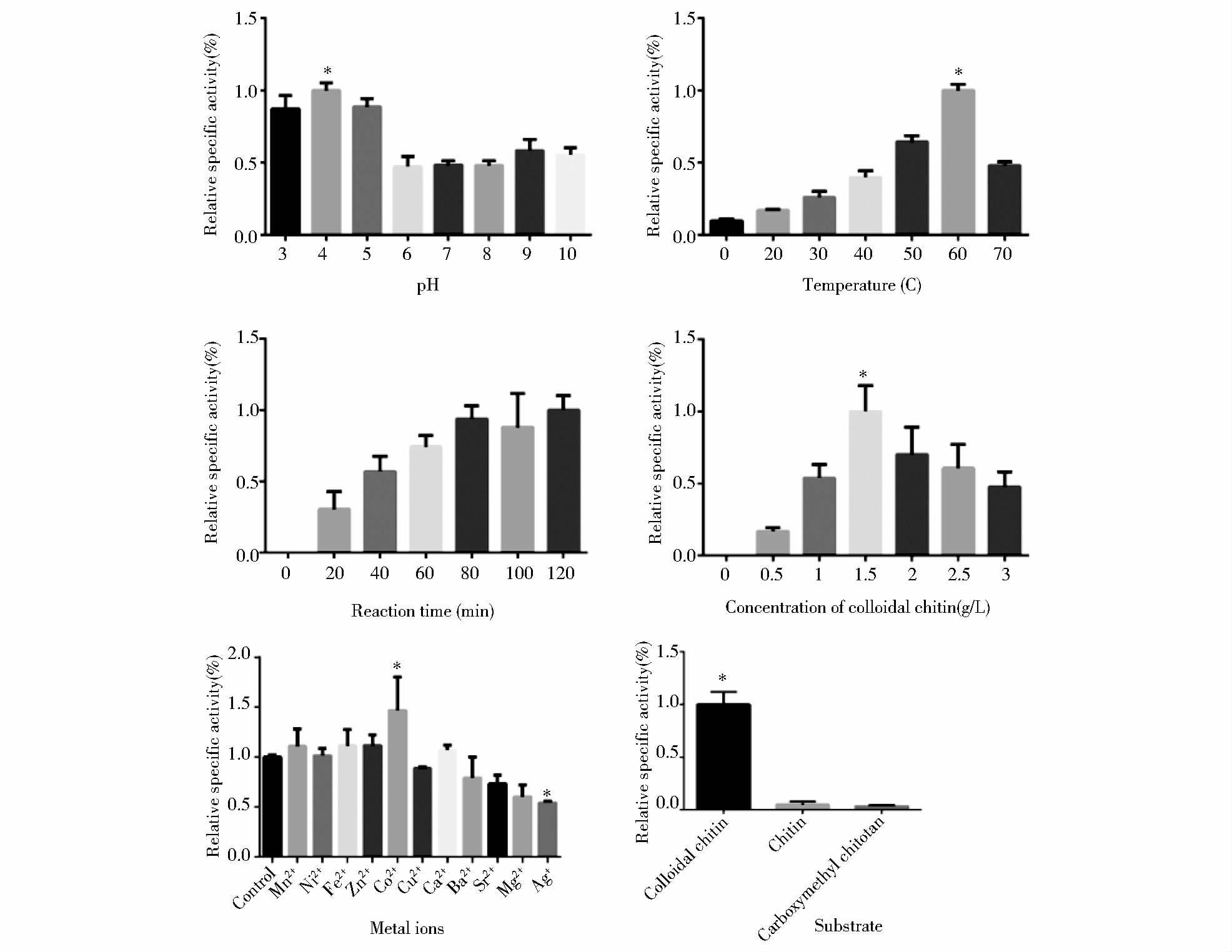
|
| 图 4 几丁质酶的酶学性质分析 Fig. 4 Effect of various factors on chitinase activity and stability * : P<0.05 |
本研究首次进行了中国南海北部湾海底沉积物样本中几丁质酶降解菌的筛选工作。共筛选了两组样本,分别是A组和B组。这两组样本位于同一位点,但是来自于不同的海底深度。研究发现,B组筛选得到菌株产生的几丁质酶活力普遍比A组高(图 1b)。产生这种现象的可能原因是,当虾蟹等壳类动物的残骸沉到海底之后,首先被表层沉积物中的微生物降解成容易吸收利用的几丁质,之后再被更深层的微生物进行充分的降解和利用。
对分离得到的高酶活菌株进行了16S rDNA序列分析,通过构建系统发育树,初步判断该菌隶属于蜡样芽孢杆菌属。其他筛选得到的菌株经16S rDNA序列分析后发现多数为蜡样芽孢杆菌属或者假交替单胞菌属。孙菽蔚等[14]研究发现,在胶州湾海泥中,假交替单胞菌是重要的优势菌群。这说明,在海底沉积物中,蜡样芽孢杆菌及假交替单胞菌可能是可培养几丁质酶产生菌中的优势菌株。
对Bacillus cereus strain B04菌株分泌的几丁质酶进行初步研究后发现,纯化得到的酶为Chi36,将Chi36B04的酶学性质与已报道的从B. cereus分离出来的几丁质酶进行比较。Arora等[15]得到的Chi36,其最适pH为6.5,最适反应温度为65℃;Wang等[16]得到的Chi36,其最适pH为5.8,最适反应温度为35℃,纯化倍数为3.39倍。本研究选择的纯化路线较为合理,纯化倍数达到21.2,高于一般Chi36的纯化方法,而且纯化蛋白的比活力远远高于纯化倍数较高的蛋白[17, 18, 19, 20]。目前,对于几丁质酶的研究过程中,酶活测定方法、底物甚至酶活单位的定义等都各不相同,没有统一的酶活力标准[21],不利于几丁质酶酶活力大小的比较。工业上大多采用强酸溶解的方法对几丁质进行预处理,微生物分泌的几丁质酶其最适pH范围广,但大多为酸性[4],从B. cereus中分离得到的几丁质酶最适pH普遍在5.0~6.5之间,而本研究分离得到的Chi36B04最适pH为4.0,这使得该酶在工业化应用上具有更显著的优势。
本研究建立了从海底沉积物出发,通过透明圈及DNS法筛选出可培养的几丁质酶产生菌,并通过对菌株分泌的几丁质酶进行分离纯化的技术体系,为几丁质酶产生菌及几丁质酶的筛选提供了基础。
| [1] | Oh S, Kim D H, Patnaik B B, et al. Molecular and immunohistochemical characterization of the chitinase gene from Pieris rapae granulovirus. Archives of Virology, 2013, 158(8): 1701-1718. |
| [2] | 李海浪. 壳聚糖衍生物的制备及其在药物载体中的应用研究. 上海: 中国科学院上海应用物理研究所, 2014. Li H L. Study on the preparation of chitosan derivatives and their applications in drug delivery. Shanghai: Institute of Applied Physics, Chinese Academy of Sciences, 2014. |
| [3] | 陈海燕, 张彬. 壳寡糖的制备及其生理功能. 中国饲料, 2007, 4: 19-21. Chen H Y, Zhang B. The preparative methods and physiological function of oligochiaosan. China Feed, 2007, 4: 19-21. |
| [4] | Bhattacharya D, Nagpure A, Gupta R K. Bacterial chitinases: properties and potential. Critical Reviews in Biotechnology, 2007, 27(1): 21-28. |
| [5] | Rashad F M, Fathy H M, El-Zayat A S, et al. Isolation and characterization of multifunctional Streptomyces species with antimicrobial, nematicidal and phytohormone activities from marine environments in Egypt. Microbiological Research, 2015,175:34-47. |
| [6] | Subhash Y, Sasikala C, Ramana C V. Salinimicrobium sediminis sp. nov., isolated from a deep-sea sediment. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(Pt 3): 984-988. |
| [7] | 王海东, 刘韫滔, 林伯坤, 等. 汕头海域几丁质酶产生菌的筛选及其几丁质酶编码基因的研究. 应用与环境生物学报, 2009, 15(4): 511-514. Wang H D, Liu Y T, Lin B K, et al. Isolation of chitinolytic bacteria from the Shantou Bay, China and study on their chitinase coding genes. Chinese Journal of Applied and Environmental Biology, 2009, 15(4): 511-514. |
| [8] | 蔡亚萍, 苏建强, 谢忠, 等. 南海海域几丁质降解菌的筛选及其特性研究. 厦门大学学报(自然科学版), 2008, 47(2): 259-263. Cai Y P, Su J Q, Xie Z, et al. Isolation and characterization of chitin-degrading bacteria from South China Sea. Journal of Xiamen University(Natural Science), 2008, 47(2): 259-263. |
| [9] | 陶树兴, 王婷婷, 徐珊, 等. 黄海沿岸及内陆盐湖几丁质酶产生菌的筛选及特性. 陕西师范大学学报(自然科学版), 2010, 38(4): 73-78. Tao S X, Wang T T, Xu S, et al. Isolation and characterization of chitinase-producing microorganisms from the Yellow Sea coast and inland salt lake. Journal of Shaanxi Normal University (Natural Science Edition), 2010, 38(4): 73-78. |
| [10] | Kabana R R, Godoy G, Morgan J G, et al. The determination of soil chitinase activity: conditions for assay and ecological studies. Plant and Soil, 1983, 75(1): 95-106. |
| [11] | Miller G L. Use of dinitrosalicylic acid reagent for determination of reducing sugar. Analytical Chemistry, 1959, 31: 426-428. |
| [12] | Kester D R, Duedall I W, Connors D N, et al. Preparation of artificial seawater. Limnology and Oceanography, 1967, 12(1): 176-179. |
| [13] | 张军霞,丛大鹏,李雅华, 等. 棘孢木霉几丁质酶tachi2基因的原核表达及酶学性质研究. 中国生物工程杂志, 2013, 33(6): 45-51. Zhang J X, Cong D P, Li Y H, et al. Prokaryotic expression of tachi2 gene from Trichoderma asperellum and characterization of recombinant enzyme. China Biotechnology, 2013, 33(6): 45-51. |
| [14] | 孙菽蔚, 王子峰, 岳海东, 等. 一种海洋几丁质酶产生菌的筛选及其产酶条件的初步研究. 海洋科学, 2007, 31(5):10-16. Sun S W, Wang Z F, Yue H D, et al. Screening of chitinase-producing marine bacterial strains and preliminary studied on chitinase-producing conditions. Marine Sciences, 2007, 31(5):10-16. |
| [15] | Arora N, Ahmad T, Rajagopal R, et al. A constitutively expressed 36 kDa exochitinase from Bacillus thuringiensis HD-1. Biochemical and Biophysical Research Communications, 2003, 307(3): 620-625. |
| [16] | Wang S Y, Wu S J, Thottappilly G, et al. Molecular cloning and structural analysis of the gene encoding Bacillus cereus exochitinase Chi36. Journal of Bioscience and Bioengineering, 2001, 92(1): 59-66. |
| [17] | Hammami I, Siala R, Jridi M, et al. Partial purification and characterization of chiIO8, a novel antifungal chitinase produced by Bacillus cereus IO8. Journal of Applied Microbiology, 2013, 115(2): 358-366. |
| [18] | Dai D H, Huang G R, Li W. Purification and characterization of a novel extracellular chitinase from thermophilic Bacillus sp. Hu1. African Journal of Biotechnology, 2011, 10(13): 2476-2485. |
| [19] | Wang S L, Chao C H, Liang T W, et al. Purification and characterization of protease and chitinase from Bacillus cereus TKU006 and conversion of marine wastes by these enzymes. Marine Biotechnology, 2009, 11(3): 334-344. |
| [20] | Chen W M, Chen G H, Chen C S, et al. Cloning, expression and purification of Bacillus cereus endochitinase in the Escherichia coli AD494(DE3)pLysS expression system. Bioscience Biotechnology and Biochemistry, 2009, 73(5): 1172-1174. |
| [21] | Ferrari A R, Gaber Y, Fraaije M W. A fast, sensitive and easy colorimetric assay for chitinase and cellulase activity detection. Biotechnology for Biofuels, 2014, 7(1): 37-44. |
 2015, Vol. 35
2015, Vol. 35




