文章信息
- 李鹏鹏, 于浩, 许平, 唐鸿志
- LI Peng-peng, YU Hao, XU Ping, TANG Hong-zhi
- 6-羟基-3-琥珀酰吡啶单加氧酶HspB的结构研究
- The Structural Studies of 6-Hydroxy-3-succinoyl-pyridine Monooxygenase
- 中国生物工程杂志, 2015, 35(8): 68-75
- China Biotechnology, 2015, 35(8): 68-75
- http://dx.doi.org/10.13523/j.cb.20150810
-
文章历史
- 收稿日期:2015-03-13
- 修回日期:2015-05-06
2. 青岛农业大学生命科学学院 青岛 266109
2. Collage of Life Science, Qingdao Agriculture University, Qingdao 266109, China
杂环化合物是目前数目最为庞大的一类有机化合物,其中吡啶及其衍生物广泛存在于环境、化学合成工业,以及工农业废弃物中[1]。虽然吡啶衍生物是一类有毒的环境污染物,但是在具有生物药学活性的自然产物以及具有特殊功能的材料中颇为常见[2, 3],因此吡啶衍生物的生物代谢研究意义重大。工业界现已开发出大量的吡啶衍生物合成方法,但是这些方法都伴随有大量副产物的产生[4]。生物催化法是具有应用价值的工业合成技术,它能够专一、高效地完成许多经典化学合成手段实现不了的反应[5]。迄今为止,有关吡啶代谢的酶的研究有限,限制了吡啶衍生物生物合成的研究。已报道的吡啶单加氧酶通常很难转化相应的苯环化合物,而很少有苯环化合物代谢的酶能够特异性地转化吡啶。吡啶单加氧酶可以根据吡啶羟化的位置分为邻位氧化、对位氧化、间位氧化三类。目前,仅有3个吡啶对位单加氧酶被报道,其中有两个6-羟基尼古丁酸3-单加氧酶[6, 7]和一个2,6-二羟基吡啶3-单加氧酶[8],这三个酶均为黄素蛋白单加氧酶。2,6-二羟基吡啶3-单加氧酶的晶体结构阐述了FAD依赖的芳香环羟化酶家族之间的结构关系[8],但是这类酶的生化性质,以及蛋白结构与辅因子之间的关系并未得到研究。
6-羟基-3-琥珀酰吡啶单加氧酶(HspB)是在恶臭假单胞菌S16尼古丁降解基因簇中发现的一个黄素单加氧酶(Swiss-Prot数据库编号为F8G0M4.1),长度为393个氨基酸,分子质量约为41.5kDa。HspB含有一个FAD辅因子,并在辅酶NADH存在下,能将6-羟基-3-琥珀酰吡啶(HSP)转化为2,5-二羟基吡啶(DHP)(图 1)[9, 10, 11]。虽然该酶在尼古丁降解过程中的作用以及催化机制已经得到解析,但是有关其催化功能与结构的关系还未得到研究。

|
| 图 1 HspB催化6-羟基-3-琥珀酰吡啶生成2,5-二羟基吡啶 Fig. 1 HspB breaks 6-hydroxy-3-succinoyl-pyridine into 2,5-dihydroxy-pyridine |
在本研究中,设计了HspB蛋白的关键位点的定点突变以及N-端和C-端截尾突变,并对HspB及其突变体的重组蛋白进行构建、表达、纯化以及活性检测。目的在于:通过对突变体的研究,探索关键氨基酸位点对HspB结构稳定性与催化活性的关系,为该酶在吡啶衍生物的生物合成应用提供结构信息。
1 材料与方法 1.1 材 料 1.1.1 质粒和菌种恶臭假单胞菌(Pseudomonas putida)S16,菌株保藏号:DSM 28022,CCTCC No. 205038。质粒pET28a、E. coli DH5α和E. coli BL21(DE3)为本实验室保存。
1.1.2 试 剂基因组提取试剂盒(美国Promega公司);琼脂糖凝胶回收试剂盒(Axygen,Union City,CA,USA);Ex-Taq PCR Polymerase[宝生物工程(大连)有限公司]。限制性内切酶(Nco I/Xho I)购自NEB 公司。引物由苏州金唯智生物科技有限公司合成,测序由上海美吉生物有限公司完成。
1.1.3 仪 器洁净工作台(VD-850,苏州净化设备有限公司);恒温摇床(HYG-C,HZQ-F100,太仓市华美生化仪器厂);PCR仪(22331 Hamburg,德国Eppendorf公司);离心机(5417R,德国Eppendorf公司);立式压力蒸汽灭菌(YXQ-LS-SII,上海博讯实业有限公司);蛋白电泳仪、核酸电泳仪和凝胶成像仪均是天能生物有限公司产品,紫外可见分光光度计UV 2550 购自日本Shimadzu 公司。
1.2 方 法 1.2.1 HspB突变体重组质粒的构建根据(表 1)设计合成引物。以Pseudomonas putida S16 的基因组为模板,进行PCR 扩增。反应体系:Ex-Taq PCR Polymerase (1.0 U/μl) 1.0 μl,10×PCR plus缓冲液 5.0 μl,dNTPs混合液 3 μl,模板2 μl,前向、后向引物(10 nmol/L)各1.0 μl,用ddH2O 补加至50 μl。PCR条件: 94℃ 5min;94℃ 30 s,58℃ 30 s,68℃ 1 min,共30个循环;68℃ 10 min;4℃10 min。
| Mutations | Primer(5′-3′) |
| hspB-H-F | ATA CCATGGTGAGCATGAAACAGCGCGTAAT |
| hspB-H-R | GTG CTCGAGAAAGGTTTCCATAGTCTCTGGAA |
| Δ1-15 (F) | ATA CCATGGGGCTGCTGACTGCCTTGGGCCT |
| Δ334-393 (R) | GTG CTCGAGGTCGGCTTCGAACACGTCGAGA |
| Δ364-393 (R) | GTG CTCGAGATTTTTTCCTTCACCAGGCTTC |
| Δ379-393 (R) | GTG CTCGAGCCTATCTATATCCTTTGAAATG |
| Δ389-393 (R) | GTGCTCGAGAGTCTCTGGAAAACGTAAAGCA |
| Underlined is the recognition site | |
将PCR 产物和pET28a 质粒用内切酶Nco I 和Xho I 进行双酶切。纯化DNA 片段和pET28a 载体,用T4连接酶连接。转化至DH5α中,筛选,测序。
1.2.2 HspB突变体的表达和纯化将重组质粒转化入感受态细胞BL21中,筛选单克隆培养,作为种子。 种子培养,诱导表达,细胞破碎,蛋白纯化等步骤可参考文献[12]。
1.2.3 HspB突变体酶活测定方法HspB催化HSP转化为2.5-二羟基吡啶需要消耗NADH,NADH在340 nm处有特征收峰值,当NADH转化为NAD+后340 nm处特征吸收峰会消失。配置HspB酶反应体系,测量反应1min后340 nm波长处紫外吸收值的降低,从而计算HspB酶的活性。
1.2.4 HspB突变体动力学常数测定利用UV2550测定HSP的动力学常数(Km、kcat值),测定方式为:配置HspB酶反应体系,固定NADH终浓度为250 μmol/L,调整HSP终浓度分别为25 μmol/L,50 μmol/L,125 μmol/L,250 μmol/L,500 μmol/L和1 000 μmol/L,利用UV2550测定340nm处紫外吸收的变化,反应温度为25℃。每组实验做三组平行,根据实验结果进行线性回归,计算Km、Kcat值。
测定NADH的动力学常数(Km、kcat 值)方法一样,固定HSP的终浓度为250 μmol/L,调整NADH终浓度分别为3 μmol/L,5 μmol/L,12.5 μmol/L,25 μmol/L,50 μmol/L,100 μmol/L,150 μmol/L和250 μmol/L。反应温度为25℃。每组实验做三组平行,根据实验结果进行线性回归,计算Km、kcat值。
2 结果与分析 2.1 HspB蛋白建模和序列比对从Swiss-Prot和PDB数据库比对出序列相似或者功能相近的黄素单加氧酶,利用ClustalW软件进行序列比对,并参考已结晶蛋白的晶体结构注释部分二级结构:α螺旋、β折叠以及FAD结合位点Rossmann fold模体(图 2)。根据Pseudomonas putida KT2440和Pseudomonas fluorescens TN5 中的6-羟基尼古丁酸-3-单加氧酶的序列分析,蛋白质N-端区域的核酸结合位点即是辅因子FAD结合位点[Rossmann fold GXGXXG(X)16-17(E/D) 模体]。

|
| 图 2 HspB与同源蛋白之间的多序列比对和二级结构注释 图中红色底色表示完全相同的氨基酸,蓝框红字的氨基酸表示保守氨基酸,α螺旋和β折叠以及Rossmann fold可以参考下面的图标,菱形表示的是定点突变的氨基酸。其中PHBH、MtmOIV 和 PagE来自PDB数据库,对硝基苯酚羟化酶H0C429、H0C413、M3D543、C1I201(PnpA)和单加氧酶G7GLR6、H5THA5来自Swiss-Prot数据库 Fig. 2 Sequence alignment of HspB with homologous proteins by ClustalW Secondary structure of HspB and selected amino acids for site-directed mutagenesis were marked above the sequences. PHBH,MtmOIV and PagE come from PDB database. Para-nitrophenol 4-monooxygenase H0C429,H0C413,M3D543,C1I201 (PnpA) and putative monooxygenase G7GLR6、H5THA5 come from Swiss-Prot database |
HspB与同类蛋白的相似性较低,在Swiss-Prot数据库中与HspB相似性是最高的、已报道功能的蛋白是不同家族的对硝基苯酚羟化酶PnpA(Swiss-Prot数据库编号C1I201),相似性只有43.7%,该酶催化对硝基苯酚的对位羟化,与HspB催化的吡啶环对位羟化反应相似;HspB与已获得晶体结构的相似蛋白,如对羟基苯甲酸羟化酶(Swiss-Prot数据库编号P20586)的相似性只有25%。鉴于HspB与已知结构蛋白的低相似性,本研究采用同源建模和从头建模相结合的方式来对HspB的蛋白结构进行建模,同时利用Discovery Studio3.5软件对HspB和HSP进行分子对接模拟,寻找底物HSP与HspB酶的结合位点,并利用PyMOL软件将对接模型的空间位置作图(图 3)。

|
| 图 3 HspB蛋白建模及与底物HSP分子对接图示 红色区域为设计突变位点,蓝色区域为FAD结合位点,中心小分子为HSP,HSP所在的区域即为HspB的活性中心 Fig. 3 Graph of HspB protein modeling and docking with HSP The red region is site-directed mutagenesis,and the blue region is the Rossmann fold which FAD binds to. Where the molecule HSP stays is the activity center of HspB |
蛋白的结构与功能是相互关联的,HspB的结构信息将有助于研究其催化机理的研究[13, 14],但是HspB的结晶目前并没有得到。因此本研究决定构建多个HspB蛋白的突变体,通过突变体与原始蛋白的生化性质的差别,结合生物信息学的结构建模等手段对HspB的结构信息进行初步的探索。本研究通过三种途径来设计突变体:(1) 根据HspB蛋白的比对结果,对保守氨基酸和保守二级结构进行定点突变。(2)根据HspB蛋白和底物HSP的分子对接模型,将HspB蛋白催化活性中心附近的氨基酸进行定点突变。(3)对羟基苯甲酸羟化酶PHBH是结构与功能研究最为透彻的一种黄素单加氧酶,Baeyer-Villiger单加氧酶MtmOIV是另一个已报道并结晶的同时拥有两个Rossmann fold的group A黄素单加氧酶,PagE是已报道晶体结构的芳香环羟化酶。本研究根据PHBH、MtmOIV 和 PagE的晶体结构和突变性质研究[15, 16, 17],同时参考Swiss-Prot数据库中有文献报道的对硝基苯酚羟化酶H0C429、H0C413、M3D543、C1I201(PnpA)的蛋白质序列,以及相似性较高的单加氧酶G7GLR6和H5THA5的序列比对情况,最终结合HspB蛋白本身的氨基酸序列,设计突变位点。
最终,我们将Rossmann fold,保守位点和活性位点处的氨基酸进行突变,共设计了20个HspB定点突变体(表 1,表 2)。另外为了确定N-端和C-端区域对于酶的活性的影响,还设计了截除N-端的15个氨基酸和C-端的5,15,30和60个氨基酸的突变体。之后对所有的突变体蛋白进行了诱导表达、纯化和酶学性质检测。
| Mutations | Primer(5′-3′) |
| G13A (F) | CGTAGGAGG GGCGCCGGTAGGG |
| G13A (R) | CCCTACCGGC GCCCCTCCTACG |
| E34A (F) | GGTGGTGCT GGCAGCTGAGAGC |
| E34A (R) | GCTCTCAGCT GCCAGCACCACC |
| E36A (F) | GCTGGAAGCT GCGAGCCAACCC |
| E36A (R) | GGGTTGGCTC GCAGCTTCCAGC |
| R44A (F) | CGATAGTCCG GCCGCGCTCGTT |
| R44A (R) | AACGAGCGCG GCCGGACTATCG |
| A45G (F) | TAGTCCGCGC GGGCTCGTTTAT |
| A45G (R) | ATAAACGAGC CCGCGCGGACTA |
| V137Q (F) | GATTGCGAA CAGGGCCCGCGGT |
| V137Q (R) | CCGCGGGCC CTGTTCGCAATCA |
| D164A (F) | ATCGGAGCT GCTGGCGCTAATA |
| D164A (R) | TATTAGCGC CAGCAGCTCCGAT |
| N167A (F) | TGATGGCGC TGCTAGCTTCGTG |
| N167A (R) | CACGAAGCT AGCAGCGCCATCA |
| N167R (F) | GATGGCGCT CGTAGCTTCGTGC |
| N167R (R) | CACGAAGCT ACGAGCGCCATCA |
| R171A (F) | TAGCTTCGT GGCTCGAGAGGTT |
| R171A (R) | AACCTCTCG AGCCACGAAGCTA |
| F179A (F) | GAATCAAAA TGCCTTTGGCATA |
| F179A (R) | TATGCCAAA GGCATTTTGATTC |
| R194A (F) | TACAAATAC CGCGTTCGACTTT |
| R194A (R) | AAAGTCGAA CGCGGTATTTGTA |
| K204A (F) | TGGTTTTGG AGCAACTACTATG |
| K204A (R) | CATAGTAGT TGCTCCAAAACCA |
| R272A (F) | AATGCACCA GGCCGTTACGGAT |
| R272A (R) | ATCCGTAAC GGCCTGGTGCATT |
| R281A (F) | GCGCAATGG TGCTGTGATCCTT |
| R281A (R) | AAGGATCAC AGCACCATTGCGC |
| D287A (F) | CCTTATAGG TGCTGCTGCACAC |
| D287A (R) | GTGTGCAGC AGCACCTATAAGG |
| P294A (F) | CGTAACCAA CGCAACTGGAGGG |
| P294A (R) | CCCTCCAGT TGCGTTGGTTACG |
| G299A (F) | TGGAGGGCT TGCTTTGACGGGG |
| G299A (R) | CCCCGTCAA AGCAAGCCCTCCA |
| D320A (F) | GTCATACAC GCCGGTCGTAGTG |
| D320A (R) | CACTACGACC GGCGTGTATGAC |
| Forward (F) and reverse (R) primers are listed. Underlined is the mutation site | |
25个突变体表达纯化结果见表 3所示,部分突变体蛋白电泳图见图 4。由表 3可知:N-端和C-端缺失的突变体均没有表达出可溶性蛋白。N-端缺失了15个氨基酸的突变体Δ1-15既没有可溶性蛋白也没有包涵体出现[18],这说明了N-端的改变可能直接影响到了蛋白的正常表达,C-端缺失的突变体都是以包涵体的形式表达的,没有可溶性蛋白,这说明C-端的氨基酸参与了蛋白质的正确折叠。
| HspB Mutants | Expression condition |
| Δ1-15 | 不表达 |
| Δ334-393,Δ364-393,Δ379-393,Δ389-393 | 表达蛋白以包涵体形式存在 |
| G13A,D164A,R171A,F179A,R272A,D287A,P294A | 表达蛋白以包涵体形式存在 |
| E34A,E36A,R44A,A45G,V137Q,N167A,N167R,R194A,K204A,R268A,R281A,G299A,D320A | 能够表达和纯化 |
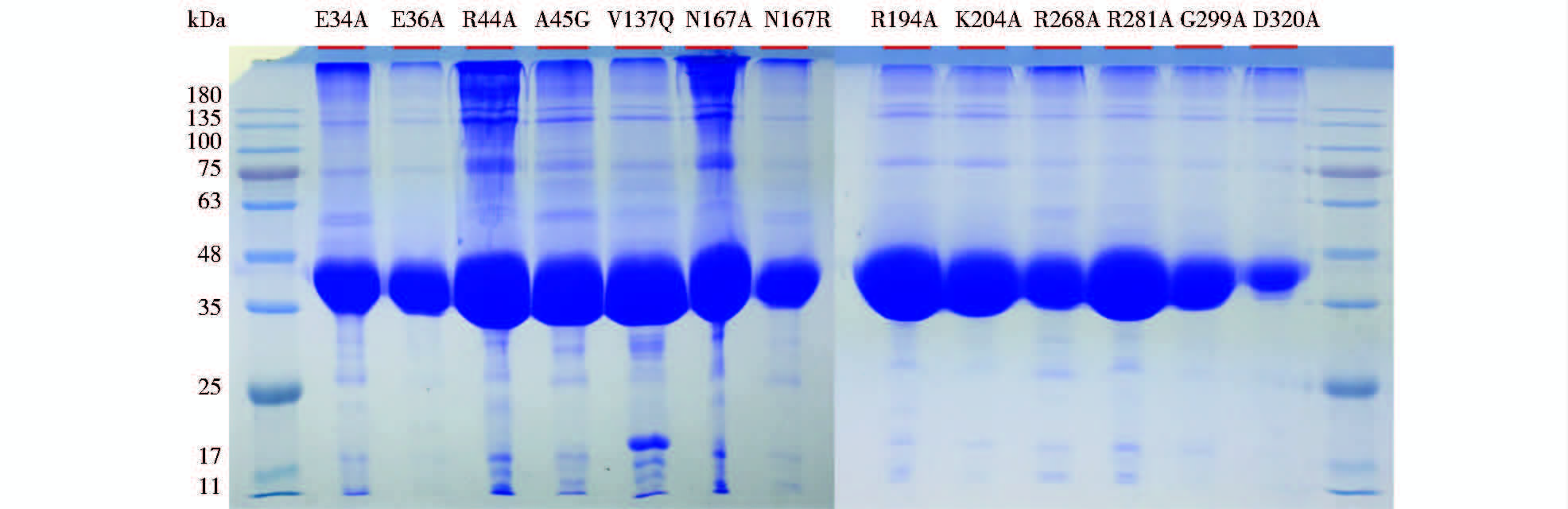
|
| 图 4 可表达纯化的HspB突变体蛋白SDS-PAGE电泳 分子质量在40kDa左右与野生型HspB蛋白分子质量一致,从左到右分别为E34A,E36A,R44A,A45G,V137Q,N167A,N167R,R194A,K204A,R268A,R281A,G299A,D320A突变体 Fig. 4 The SDS-PAGE graph of HspB mutants From left to right: E34A,E36A,R44A,A45G,V137Q,N167A,N167R,R194A,K204A,R268A,R281A,G299A,D320A |
HspB属于NADH依赖的黄素单加氧酶,前期研究表明黄素单加氧酶中辅酶或者辅因子往往在维持蛋白酶构象和催化反应等多方面起到了重要的作用[19]。利用已经报道的脱辅因子的制作方法无法获得可溶性的HspB蛋白,由此推测在HspB中,FAD在维持HspB结构稳定方面也起到了重要的作用。G13A,D164A,R171A,F179A,P294A,R272A和D287A这几个突变体也是全部以包涵体形式表达,其中Gly-13 (G13A)是预测的FAD结合motif的重要位点[20];D-164 (D164A)是另一个在HspB中预测的FAD结合模体(GADGA)中的氨基酸[9];与R-171(R171A)对应的酶PHBH中的氨基酸R-166的突变也导致了PHBH不能结合FAD[16];与F-179(F179A)对应的酶MtmOIV中的氨基酸F-180的突变降低了与FAD的结合能力[15];与P-294(P294A)对应的蛋白PagE中的氨基酸P-282在底物结合位点附近,与FAD还原密切相关[17]。以上的几个氨基酸都与FAD的结合有关,这与我们之前的猜测结果相一致,FAD的结合可能是维持HspB蛋白正常构象的一个必要条件,因此当与FAD的结合相关的氨基酸发生突变时,HspB就不能正常折叠。当然以上的氨基酸也有可能不是与FAD结合直接相关,而是通过影响蛋白构象从而影响蛋白折叠。
2.4 可纯化HspB突变体的酶学性质检测为了更好地了解突变的氨基酸在蛋白质功能和结构方面的作用,研究中对可纯化的13个可溶性蛋白的动力学常数进行了测定。
突变体E34A,E36A,V137Q,N167R,R194A,R281A和D320A的HSP和NADH的Km值均没有明显改变(是原始蛋白的0.7~1.4倍)。并且Km值的变化趋势一致:HSP与NADH的Km值上升或者下降是同步的。例如在突变体E34A,R194A,R281A和D320A中HSP和NADH的Km值均出现了上升,而在突变体E36A,V137Q和N167R中HSP和NADH的Km值都出现了下降。
突变体A45G,N167A,K204A,G299A的Km值变化很大,但是也呈现了相同的变化规律,在A45G和N167A中两种底物的Km值均有较明显上升(上升至原来的1.2~2.5倍);而在K204A和G299A中两种底物的Km值显著上升,是原始蛋白中该值的2.5~27倍(表 4)。根据已经解析的2,6-二羟基吡啶 3-羟化酶(2,6-Dihydroxypyridine 3-hydroxylase)的晶体结构可以看到,辅酶NADH结合的位置并不是很清楚[8],在group A黄素单加氧酶中NADH将蛋白酶还原后就会脱离酶复合体,并不是与底物一起始终结合在酶复合体上。综合上面的结果我们可以认为,NADH与HSP的结合位置可能相近,位于同样的一个活性作用中心,因此结构的改变对于两者结合的影响趋势是一致的。
| HSP | NADH | |||||
| Mutants | Km(μM) | Vmax(n kat mg-1) | Vmax/Km | Km(μM) | Vmax(n kat mg-1) | Vmax/Km |
| Wild type | 172.84 | 189.14 | 1.094 | 35.77 | 121.93 | 3.409 |
| E34A | 175.88 | 50.4 | 0.287 | 46.303 | 25.039 | 0.541 |
| R194A | 239.7 | 118.02 | 0.492 | 45.3 | 49.23 | 1.087 |
| R281A | 207.83 | 131.69 | 0.634 | 37.13 | 45.36 | 1.222 |
| D320A | 191.6 | 121.11 | 0.632 | 41.26 | 57.54 | 1.395 |
| E36A | 123.21 | 148.93 | 1.209 | 24.7167 | 82.5692 | 3.341 |
| V137Q | 165.21 | 172.6 | 1.045 | 17.45 | 57.23 | 3.280 |
| N167R | 167.6 | 181.9 | 1.085 | 29.58 | 94.01 | 3.178 |
| A45G | 231.98 | 45.23 | 0.195 | 69.09 | 25.41 | 0.368 |
| N167A | 207.98 | 166.88 | 0.802 | 86.56 | 72.77 | 0.800 |
| K204A | 4714 | 10.9 | 0.0023 | 98.73 | 1.87 | 0.019 |
| G299A | 434.05 | 22.78 | 0.052 | 92.7 | 5.777 | 0.062 |
| R44A | 2450 | 1.63 | 0.00067 | 12.94 | 0.5259 | 0.041 |
| R268A | 130.15 | 17.76 | 0.136 | 156.7 | 13.3 | 0.085 |
突变体R44A和R268A对两种底物的Km值呈现了不同的变化。在突变体R44A中,HSP的Km值上升到原来的14倍,而NADH的Km值下降至原来的0.36倍;而在R268A中正好相反,NADH的Km值上升至原本的4.4倍,HSP的Km值轻微下降到原来的0.75倍。另外这两个突变体的Vmax值均出现了下降,降至野生型蛋白的10%以下。在酶MtmOIV中,与R-44(R44A)对应的氨基酸R52与FAD的异咯嗪环形成了一个氢键[15],同时R52突变成为丙氨酸后,MtmOIV的酶活也下降至原本的10%以下。对突变体R44A和R268A而言,它们对某一底物的Km值没有明显变化,根据前面的推断说明活性中心没有明显的构象的变化;但是另一底物的Km值却变化明显,这说明了这两个氨基酸可能直接与底物结合,R44与HSP结合催化相关,而R268与NADH的结合及还原过程直接相关。根据经验精氨酸常常出现在活性中心附近,很容易与其他底物作用产生氢键等相互作用力,如果这个推断是正确的,那么在HspB中HSP与NADH具有相同的催化中心,但是直接进行相互作用的氨基酸不同。
3 讨 论研究酶的结构性质,以及酶与辅因子、辅酶和底物之间的相互作用关系最好的方式是解析蛋白晶体结构。但是,HspB高分辨率的晶体结构难以获得[12],因此我们采用了构建突变体的方式来探究HspB的结构性质。本研究中,通过对HspB蛋白的突变体的设计和研究,说明了在HspB中,FAD的正确结合与蛋白的稳定性密切相关。同时根据实验推测,在HspB中,底物HSP和辅酶NADH共享相同的催化中心,但是与不同的氨基酸相互作用。当然此结果只是根据特定氨基酸位点突变后的蛋白的活性改变来推测的,并没有真实的晶体结构数据来证明,这是本研究比较遗憾的地方。
在能够对芳香环进行修饰的芳香环羟化酶中,苯环羟化酶的生化性质已经研究得十分清楚,例如对羟基苯甲酸羟化酶PHBH。但是同源的吡啶羟化酶的生化性质和催化机理的研究十分有限。在接下来的工作中,将根据苯环羟化酶的结构信息,参考HspB蛋白建模的结果,设计、筛选反应活性更高的突变体,以此进一步研究HspB的反应机理。同时结合分子模拟手段,对HspB酶进行改造,致力于将其打造成高效的、催化底物更广泛的吡啶单加氧酶。
| [1] | Reddy D M, Reddy G. Microbial Degradation of Pyridine and Its Derivatives, in: Microorganisms in Environmental Management. Berlin:Springer, 2012. 249-262. |
| [2] | Bull J A, Mousseau J J, Pelletier G, et al. Synthesis of pyridine and dihydropyridine derivatives by regio-and stereoselective addition to N-activated pyridines. Chemical Reviews, 2012, 112(5): 2642-2713. |
| [3] | Wu X F, Neumann H, Beller M. Synthesis of heterocycles via palladium-catalyzed carbonylations. Chemical Reviews, 2012, 113(1): 1-35. |
| [4] | Liu C, Luo J, Xu L, et al. Synthesis of 2-substituted pyridines from pyridine N-oxides. Arkivoc, 2013(1): 154-174. |
| [5] | Petersen M, Kiener A. Biocatalysis. Green Chemistry, 1999, 1(2): 99-106. |
| [6] | Nakano H, Wieser M, Hurh B, et al. Purification, characterization and gene cloning of 6-hydroxynicotinate 3-monooxygenase from Pseudomonas fluorescens TN5. European Journal of Biochemistry, 1999, 260(1): 120-126. |
| [7] | Jiménez J I, Canales á, Jiménez-Barbero J, et al. Deciphering the genetic determinants for aerobic nicotinic acid degradation: the nic cluster from Pseudomonas putida KT2440. Proceedings of the National Academy of Sciences, 2008, 105(32): 11329-11334. |
| [8] | Treiber N, Schulz G E. Structure of 2, 6-dihydroxypyridine 3-hydroxylase from a nicotine-degrading pathway. Journal of Molecular Biology, 2008, 379(1): 94-104. |
| [9] | Tang H, Yao Y, Zhang D, et al. A novel NADH-dependent and FAD-containing hydroxylase is crucial for nicotine degradation by Pseudomonas putida. Journal of Biological Chemistry, 2011, 286(45): 39179-39187. |
| [10] | Wang S N, Liu Z, Tang H Z, et al. Characterization of environmentally friendly nicotine degradation by Pseudomonas putida biotype A strain S16. Microbiology, 2007, 153(5): 1556-1565. |
| [11] | Yu H, Tang H, Wang L, et al. Complete genome sequence of the nicotine-degrading Pseudomonas putida strain S16. Journal of Bacteriology, 2011, 193(19): 5541-5542. |
| [12] | 胡传明, 于浩, 唐鸿志, 等. 6-羟基-3-琥珀酰吡啶单加氧酶的纯化与结晶条件. 微生物学通报, 2014, 41(9): 1779-1784. Hu C M, Yu H, Tang H Z, et al. Purification and crystallization of 6-hydroxy-3-succinoyl-pyridine monooxygenase. Microbiology China, 2014, 41(9): 1779-1784. |
| [13] | Joosten V,van Berkel W J. Flavoenzymes. Current Opinion in Chemical Biology, 2007, 11 (2):195-202. |
| [14] | Crozier-Reabe K, Moran G R. Form follows function: structural and catalytic variation in the class A flavoprotein monooxygenases. International Journal of Molecular Sciences, 2012, 13(12): 15601-15639. |
| [15] | Beam M P, Bosserman M A, Noinaj N, et al. Crystal structure of Baeyer-Villiger monooxygenase MtmOIV, the key enzyme of the mithramycin biosynthetic pathway. Biochemistry, 2009, 48(21): 4476-4487. |
| [16] | Eppink M H, Bunthof C, Schreuder H A, et al. Phe161 and Arg166 variants of p-hydroxybenzoate hydroxylase: Implications for NADPH recognition and structural stability. FEBS Letters, 1999, 443(3): 251-255. |
| [17] | Koskiniemi H, Mets?-Ketel? M, Dobritzsch D, et al. Crystal structures of two aromatic hydroxylases involved in the early tailoring steps of angucycline biosynthesis. Journal of Molecular Biology, 2007, 372(3): 633-648. |
| [18] | Goodman D B, Church G M, Kosuri S. Causes and effects of N-terminal codon bias in bacterial genes. Science, 2013, 342(6157): 475-479. |
| [19] | Romero E, Fedkenheuer M, Chocklett S W, et al. Dual role of NADP(H) in the reaction of a flavin dependent N-hydroxylating monooxygenase. Biochimica et Biophysica Acta (BBA)-Proteins and Proteomics, 2012, 1824(6): 850-857. |
| [20] | Van Berkel W J, Kamerbeek N M, Fraaije M W. Flavoprotein monooxygenases, a diverse class of oxidative biocatalysts. Journal of Biotechnology, 2006, 124(4): 670-689. |
 2015, Vol. 35
2015, Vol. 35




