文章信息
- 贾翠丽, 张华伟, 王斌斌, 朱宏吉, 乔建军
- JIA Cui-li, ZHANG Hua-wei, WANG Bin-bin, ZHU Hong-ji, QIAO Jian-jun
- 革兰氏阳性菌自然感受态生理机制的研究进展
- Advances in Research on Natural Competence of Gram-positive Bacteria and Its Physiological Properties
- 中国生物工程杂志, 2015, 35(6): 90-100
- China Biotechnology, 2015, 35(6): 90-100
- http://dx.doi.org/10.13523/j.cb.20150614
-
文章历史
- 收稿日期:2015-03-18
- 修回日期:2015-04-08
自然感受状态指菌体在自然生长过程中自主吸附、摄取和内化外源DNA的生理状态[1],该状态是外源DNA受体快速获取新的遗传性状,增加生存优势的一种方式[2]。大部分细菌的自然感受状态是短暂存在的[3],只有少数细菌比如奈瑟氏淋球菌(Neisseria gonorrheae)的自然感受状态贯穿整个生命周期[4]。自从1928年Griffith[5]首次在肺炎链球菌(Streptococcus pneumonia)中发现自然感受状态以来,多种具有自然感受状态的细菌被发现,目前已发现80多种细菌具有自然感受状态[6],其中变形链球菌(Streptoccus mutans)的自然感受状态于1981年被Perry等[7]首次发现。外源DNA是一种遗传物质,对细菌的遗传多样性和表型发挥着重要作用;同时在应激环境下还可以充当一种营养物质,对增强受体菌的适应能力起了促进作用[8]。自然感受方面的生理和分子机制研究已经相当广泛,本文从革兰氏阳性菌的DNA摄取机制、自然感受的调控路径和自然感受与其他细胞生理特性的内在关系等方面详细介绍自然感受的研究进展。
1 细菌的DNA摄取机制 1.1 外源DNA的摄取大部分自然感受细菌具有相似的DNA摄取机制[9],其中肺炎链球菌和奈瑟氏淋球菌的DNA摄取机制研究较为成熟。随着肺炎链球菌感受机制的深入研究,Balaban等[10]提出了更为精确的DNA摄取机制,其大致过程如图 1所示:感受过程中诱导的ComGC位于细胞内膜上,经过PilD肽酶加工后在ComGB表面不断聚集成簇,ComGA是一种ATP酶,为该过程提供能量[11]。当ComGC聚集到一定程度在ComG菌毛蛋白的帮助下分泌到培养基中,ComGC簇团离开后留下一个跨膜的细通道[12],此时外源DNA与膜内的DNA结合蛋白ComEA结合,使得外源DNA吸附到细胞膜上,然后在EndA核酸酶的作用下,双链DNA(double- stranded DNA,dsDNA) 的一条链降解为酸溶性产物返回培养基中[13],另外一条互补链在DNA转座酶ComFA作用下通过ComEC膜蛋白进入细胞内[9]。肺炎链球菌摄入的单链DNA(single-stranded DNA,ssDNA)具有3′~5′的极性如图 2所示,而枯草芽孢杆菌摄入的ssDNA没有方向性,3′~5′和5′~3′方向的ssDNA都可以摄入。其原因可能是枯草芽孢杆菌中有至少两种核酸酶,而肺炎链球菌中只有一种核酸酶EndA[14]。
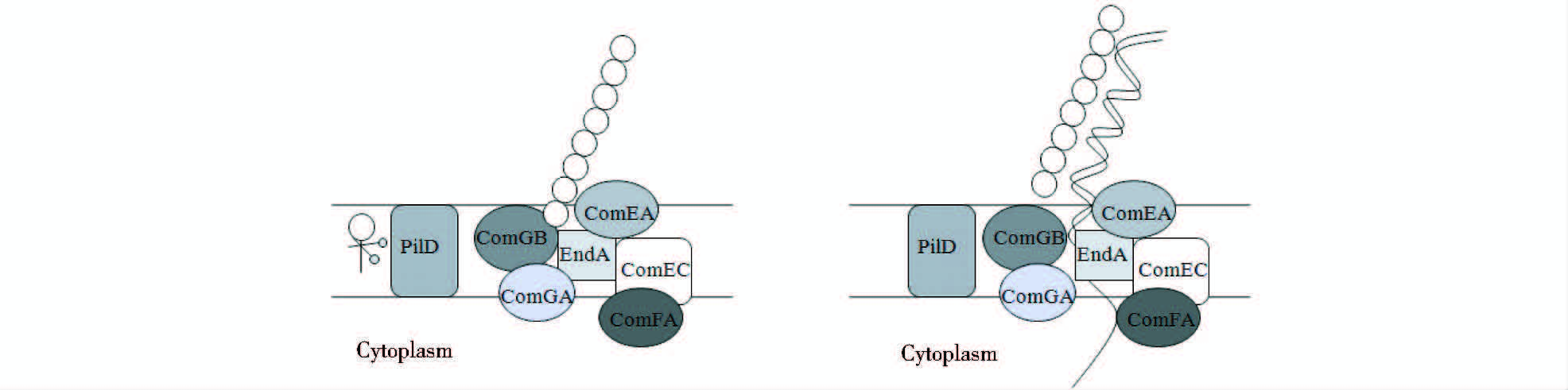
|
| 图 1 肺炎链球菌中外源DNA降解为单链DNA并进入细胞的过程 Fig. 1 The process of the degradation of exogenous DNA into ssDNA and the entrance of ssDNA into cytoplasm in Streptococcus pneumonia |

|
| 图 2 肺炎链球菌中外源DNA的摄取过程 Fig. 2 The DNA-uptake process in Streptococcus pneumoniae |
进入胞内的ssDNA并非都能整合到细菌染色体上,大部分ssDNA被胞内的DNA酶降解为核苷酸,作为原料参与染色体的复制过程(如图 2所示)。Mortier-Barrière等[15]发现DprA可以形成二聚体,与ssDNA结合后使其免受胞内核酸酶的降解。Quevillon-Cheruel等[16]发现细胞内DprA单体和二聚体都可以与ssDNA结合,但是只有后者能形成对自然感受至关重要的核酸蛋白复合物(nucleoprotein complexes,NPC),因为DprA蛋白尾尾相连形成二聚体中具有与RecA蛋白相互作用的结构域,从而促进与RecA的结合进而促进ssDNA的整合过程。为此他们提出ssDNA整合到染色体的大致过程如图 2所示:进入胞内的ssDNA与DprA二聚体结合,然后RecA与该复合物结合形成NPC,RecA结合到NPC上便引起DprA构象的改变,致使二聚体相结合的残基部分暴露,DprA二聚体降解为单体,而RecA蛋白不断聚集(nucleation),当聚集到一定程度该复合物在RecA的作用下进行同源序列的寻找与重组。Attaiech等[17]发现当外源DNA浓度较高时,SsbB可以与ssDNA形成一种DprA和RecA易于接近的储备池(reservoir),以便进行连续的外源DNA整合(如图 2所示),因此SsbB在ssDNA整合过程中也发挥着重要作用。Yadav等[18]发现枯草芽孢杆菌中DprA可以促进RecA与SsbB包裹的ssDNA的结合,对DNA链的退火发挥重要作用,进而促进外源DNA的整合。
细菌自然感受过程需要多种蛋白的参与(表 1),其中信号传导和感应蛋白主要通过自然感受调控发挥作用,大多数革兰氏阳性菌的ComX(枯草芽孢杆菌中的ComK)是自然感受的核心调控因子,对参与DNA摄取的基因具有重要的调控作用,在ComX(或ComK)的调控下,DNA摄取机制中的感受蛋白完成对外源DNA的摄取与重组。肺炎链球菌和枯草芽孢杆菌是革兰氏阳性菌中研究自然感受的模式菌株,各个感受蛋白的功能表征比较完整(表 1)。而乳酸乳球菌尚未鉴定为自然感受菌株,但是乳酸乳球菌含有多种重要的感受蛋白,因此表 1对其他菌株感受蛋白的功能表征具有重要的参考价值,如乳酸乳球菌。目前变形链球菌的自然感受调控路径研究较为广泛,已经发现多种调控路径。下文以变形链球菌为例,结合其他革兰氏阳性菌,从comX的调控系统、ComX对后期感受基因的调控以及自然感受与生物膜形成、耐酸性和细菌素合成的内在关联等方面出发,介绍革兰氏阳性菌自然感受态生理机制的研究进展。
| 感受蛋白功能 | 肺炎链球菌 | 枯草芽孢杆菌 | 变形链球菌 | 乳酸乳球菌 |
| 信号传导与感应 | ||||
| 信号传导肽 | ComC | ComX1) | ComC(44%) | ?2) |
| 转运蛋白 | ComA | ComQ | CslA(63%) | ? |
| ComB | CslB(30%) | ? | ||
| 组氨酸激酶 | ComD | ComP(25%) | ComD (33%) | TcsK(28%) |
| 反应调节因子 | ComE | ComA(22%) | ComE(41%) | TcsR(25%) |
| 核心调控因子 | ComX | ComK(28%) | ComX(46%) | ComX(28%) |
| DNA摄取机制蛋白 | ||||
| 菌毛亚基 | ComGC | ComGC (39%) | ComYC(63%) | ComGC (52%) |
| DNA 结合蛋白 | ComEA | ComEA (42%) | ComEA(52%) | ComEA (52%) |
| 核酸酶 | EndA | NucA | EndA(66%) | YbfB(29%) |
| 膜通道蛋白 | ComEC | ComEC (31%) | ComEC(50%) | ComEC (43%) |
| DNA转座酶 | ComFA | ComFA (40%) | ComFA(59%) | ComFA (53%) |
| 菌毛肽酶 | PilD | ComC (28%) | PulO(43%) | ComC (43%) |
| ATP酶 | ComGA | ComGA (38%) | ComYA(66%) | ComGA (52%) |
| 膜蛋白 | ComGB | ComGB (25%) | ComYB(58%) | ComGB (51%) |
| ssDNA保护蛋白 | DprA | DprA(45%) | DprA(63%) | DprA(54%) |
| 重组酶A | RecA | RecA(68%) | RecA(83%) | RecA(79%) |
| ssDNA结合蛋白 | SsbB | SsbB(52%) | SsbB(76%) | SsbB(51%) |
| 1)表示该菌的相关蛋白与肺炎链球菌中的相似度较低,没有标出;2)在该菌中尚未发现该功能的蛋白 | ||||
Dong等[19]发现变形链球菌中ComX蛋白的C端可以与超级家族蛋白ClpC结合,N端与衔接蛋白MecA结合形成 ComX -MecA-ClpC三联体复合物(图 3),在ClpP存在的情况下ComX会发生依赖于ATP的降解,从而使变形链球菌逃离自然感受状态。进一步的研究发现,当衔接蛋白MecA的C端或N端缺失后,三联体复合物便无法形成,因此ComX是在MecA介导下降解的。但是当变形链球菌生长在化学合成培养基(chemical defined medium,CDM)中时,即ComX诱导肽(ComX-inducing peptide,XIP)诱导自然感受态的形成,不存在MecA介导的ComX降解,这种环境下可能存在其他未知的ComX降解机制,因此变形链球菌中MecA介导的ComX降解方式与comX的调控系统有一定关系。枯草芽孢杆菌是研究自然感受的模式菌株,其感受调控中的ComK具有核心调控作用,类似于变形链球菌中的ComX,当枯草芽孢杆菌未进入自然感受状态时,感受核心调控蛋白ComK以ComK-MecA-ClpC的三联复合体形式存在,活性受到抑制,只有当小分子蛋白质ComS存在时ComK才能被激活释放,诱导后期感受基因的转录[20]。

|
| 图 3 变形链球菌自然感受的调控路径 Fig. 3 Regulatory circuits of natural competence in Streptococcus mutans |
双组分系统是细菌重要的信号传导系统,主要通过蛋白质的磷酸化进行信号的传导。ComDE系统是变形链球菌的一种双组分系统,对自然感受的调控发挥着重要作用。ComD为一种跨膜组氨酸激酶,ComE是与ComD成对存在的反应调节蛋白,存在于细胞质中。ComDE系统对自然感受的调控研究较为深入和成熟,ComDE系统对comX的调控还需要comC、cslAB(类似于肺炎链球菌中comAB)和comX基因的转录表达,其中comC基因编码含有45个氨基酸的感受刺激肽(competence-stimulating peptide,CSP)前体,cslA编码ATP结合转运蛋白,cslB编码CslA的辅助蛋白,CslAB共同构成CSP前体的特异转运蛋白[21]。CslAB在转运CSP前体过程中,通过酶切其结构中的2个甘氨酸前导肽将其转化成熟的CSP分泌到细胞外[22]。当变形链球菌生长到对数期时,CSP积累到一定浓度激活临近细胞跨膜组氨酸激酶ComD的磷酸化,磷酸基团传递给反应调节因子ComE,磷酸化后的ComE可以激活多个基因的转录,其中ComE对cslAB和comC基因的激活构成了一个反馈路径(图 3)。
肺炎链球菌中也有相似的调控路径,只是变形链球菌ComE不能直接激活comX的转录,而肺炎链球菌中ComE可以激活comX的转录。枯草芽孢杆菌中也有类似ComDE的调控路径,其中参与该过程的主要蛋白及其对应功能如表 1所示。其大致过程如下,comX基因编码55个氨基酸的ComX前体,该前体经过ComQ的酶切和异戊烯化形成含有10个氨基酸的感受信息素ComX。当ComX积累到一定浓度,经过双组分系统ComP和ComA的磷酸化激活comS基因的表达,ComS阻止ComK -MecA-ClpC三联体的降解[23],ComK是枯草芽孢杆菌感受态的核心调控因子,ComK可以识别相应基因启动子的ComK-box区域,从而激活DNA摄取和加工相关基因的保护[24]。
磷酸化的ComE是早期感受过程的核心调控因子,可以激活多种早期感受基因的转录,这些基因上游具有ComE的结合位点[25]。通过定量结合实验与DNase I印迹实验发现[26],ComE调控基因的启动子区域具有两对保守的直接重复序列(direct repeat,DR):DRI与DRII(图 4),其中只有DRII是与ComE结合必不可少的序列,该序列大致可以概括为TCBTAAAYSGT。虽然变形链球菌中comX基因的上游存在ComE的假定结合位点,但是Huang等[27]发现变形链球菌中的ComE不能直接激活comX的转录,其原因可能为comX基因上游ComE的假定结合位点位于起始密码子上游的-453至-420位,而comX保守的启动子序列在-79至-27位点之间[26]。虽然变形链球菌中ComE不能直接调控comX,但是CSP介导的ComDE系统是最重要、最广泛的自然感受调控方式。

|
| 图 4 ComE调控基因的上游序列比对结果 Fig. 4 Sequence alignment of upstream regions from ComE-regulated genes in Streptococcus mutans DRI and DRII are highlighted with pale grey and dark grey respectively. Highlighted regions are putative ComE binding sites. N is one of A,T,C and G. The number of unshown nucleotides is listed in subscript of N |
经典的ComDE调控系统不能直接调控comX,而ComX对自然感受调控发挥着核心作用,寻找comX的直接调控系统将对自然感受的调控研究具有里程碑式的意义。Mashburn-Warren等[28]发现变形链球菌中ComRS传导系统对自然感受的调控,该调控系统中的ComR是Rgg家族中的独立调控因子,ComS编码一种17个氨基酸的疏水性小肽 (small hydrophobic peptide,SHP),与含有2个甘氨酸的CSP前体不同,SHP的前导肽含有2个色氨酸。与双组分系统的调控方式截然不同,ComR应答胞内信号肽不需要表面受体,仅仅需要寡肽渗透酶Opp的转运[29],ComRS系统的具体调控路径如图 3所示,当变形链球菌在CDM中生长时,comS基因编码的SHP前体经过未知转运蛋白的加工处理分泌到细胞外,以7个氨基酸(即ComS11-17)的活性形式XIP存在。XIP通过OppD寡肽渗透酶系统进入细胞内与ComR二聚体结合成复合物,该复合体不仅可以直接激活comS的转录,构成正反馈调控途径,还能够直接激活comX的转录,促进自然感受态的形成。
变形链球菌中ComRS调控系统能够直接激活comX的转录,其中ComR蛋白是直接调控comX的主要蛋白。Fontaine等[30]发现comX和comS基因上游的20bp序列是ComR与XIP形成复合物后的识别结合位点,该序列称为ComR-box,是ComR调控comX和comS基因必不可少的(图 5)。与ComDE感受调控系统相比,ComRS系统诱导的自然感受范围更广,可以使整个细胞群都具有感受状态,ComDE途径只能诱导部分细胞亚群具有感受状态[31]。变形链球菌形成自然感受过程中,这两种调控系统并非平行存在的,究竟是XIP还是CSP诱导comX的转录主要取决于培养基的成分[32],在CDM培养基中主要通过XIP途径形成自然感受状态,CSP途径无法诱导自然感受状态的形成,而在正常培养基中则主要是CSP诱导自然感受的形成[33]。

|
| 图 5 comX与comS基因的上游序列 Fig. 5 Upstream sequences of comX and ComS The ComR-box are highlighted in light grey. N is one of A,T,C and G. The number of unshown nucleotides is listed in subscript of N |
Guo等[34]发现CSP和XIP诱导自然感受都与菌体的生长情况密切相关,XIP诱导自然感受时,在菌体生长的不同时期添加合成的XIP(synthetic XIP,sXIP),研究发现对数早期comX的表达量最高,在对数中期和对数晚期comX的表达量分别降低了7倍和23倍,CSP诱导下的comX也有类似的现象,尽管ComDE和ComRS调控系统的结构和调控方式不同,但是这两种情况下自然感受诱导的最佳时机都在对数早期。他们还发现只有在中性环境下XIP诱导的comX表达量最高,pH降低或升高都会降低comX的表达量,不利于XIP诱导自然感受的形成,因此中性环境是XIP诱导自然感受的最佳时机,但是菌体生存环境的pH对CSP诱导的自然感受却影响不大。疏水性的XIP是ComS的活性形式[35],但是细胞内大量的ComS却比大量的XIP更能增强comX和comS的表达,该现象支持了ComS也可以激活ComR的观点(图 3),因此ComS和ComR互相调控共同完成自然感受的调控[36]。
2.4 HdrRM和BrsRM调控网络研究发现缺失hdrM的变形链球菌能够增强自然感受效率,而缺失hdrR几乎对菌株的表型没有影响,同时缺失hdrM和hdrR却降低了自然感受效率,因此HdrRM系统对自然感受具有一定的调控作用,其中HdrR是LytTR家族的转录调控因子,HdrM是一种假定膜蛋白[37]。Okinaga等[38]进一步研究发现hdrR过表达菌株和hdrM缺陷株均提高了自然感受效率,也就是说hdrR过表达菌和hdrM缺陷株具有相似的表型,因此HdrM对HdrR转录因子具有抑制活性。另外这两株突变株都能引起comX和后期感受基因的表达,为了研究HdrRM的作用机制,他们构造了一株过表达hdrR但缺失comX的突变株,该突变株的后期感受基因发生不同程度的降低,因此HdrRM系统可以通过调控comX影响后期感受基因的转录。基因芯片实验发现HdrRM系统没有诱导cslAB,comC和comDE基因的转录,因此HdrRM调节子能够绕过感受态基因comC、comDE,通过调控comX激活后期感受基因,独立于ComDE系统发挥调控功能,然而HdrRM调控comX的详细路径仍然需要进一步探索。
Xie等[39]在变形链球菌中发现一对与HdrRM结构相似的BrsRM调控系统也可以调控自然感受,其中BrsR(SMU.2080)类似于HdrR也编码一个LytTR家族转录因子,BrsM类似于HdrM编码一种假定膜蛋白。体外电泳迁移率(EMSA)实验发现,BrsR可以调控多个细菌素及部分感受基因,该序列是LytTR家族转录因子调控基因共有的一种特征,可能为BrsR的结合调控位点。HdrRM和BrsRM均可以调控自然感受与细菌素合成,但是两者各有侧重,BrsRM侧重于细菌素的调控,而HdrRM主要调控自然感受。
2.5 RcrRPQ操纵子对ComX稳定性的影响Seaton等[40]发现rcrRPQ操纵子也能调控自然感受,该操纵子中rcrR(SMU_835)基因编码一种类似MarR家族的转录抑制因子,rcrPQ(SMU_836和SMU_837)编码ABC外排泵蛋白,在这个操纵子中RcrR是核心调控因子。他们构建了非极性的卡那霉素替代rcrR的变形链球菌突变株(△rcrR-NP),研究发现该突变株无法摄取外源DNA,后期感受基因comYA的表达量大幅下降,但是rcrPQ基因表达量却提高了100倍;与此相对,用极性卡那霉素敲除掉rcrR的变形链球菌突变株(△rcrR-P)不仅大幅提高了自然转化效率,而且增加了comYA基因的表达量,但是rcrPQ基因的表达量与原始菌相比几乎没有变化。Ahn等[41]认为突变株通过RcrPQ的表达量影响自然感受效率,在变形链球菌中过表达rcrPQ基因的进一步实验证实了该观点的合理性。RcrPQ蛋白对自然感受效率的影响机理如图 6所示,△rcrR-P突变株的rcpPQ基因表达量较低,细胞内某未知作用因子无法及时排出细胞而得到积累,大量的作用因子能够抑制MecA介导的ComX降解,未被降解的ComX增强comYA等后期感受基因的转录,从而使突变株的感受转化效率得到大幅提高;而△rcrR-NP突变株的rcrPQ表达量较高,使得作用因子排出细胞外,大部分ComX在MecA-ClpC复合体的作用下降解,无法激活感受基因的转录,最终导致自然感受效率大幅下降,因此RcrPQ通过影响MecA介导的ComX降解调控自然感受,而MecA介导的ComX降解方式存在于CSP诱导自然感受的情况下,XIP诱导自然感受时可能存在其他影响ComX稳定性的作用机制。除此之外变形链球菌中27个氨基酸的837pep1小肽和42个氨基酸的837pep2小肽也可以调控自然感受,837pep1和837pep2主要通过增强ComX的降解,或者干扰ComX对后期感受的调控发挥作用(图 6),△rcrR-P突变株中837pep1和837pep2表达量较少,影响较小;而△rcrR-NP突变株中837pep1和837pep2表达量增加,在一定程度上增强了ComX的降解[42]。

|
| 图 6 rcrPQ、837prep1和837prep2表达量影响自然感受的机理 Fig. 6 Working model for the regulation of competence development by the level of rcrPQ expression,837prep1 and 837prep2 |
自然感受过程中具有σ因子活性的ComX是后期感受的核心调控因子,与RNA聚合酶(RNA polymerase,RNAP)结合成全酶后可以激活后期感受基因的转录,这些感受基因的表达参与DNA的摄取、转运与重组等过程,对自然感受态的形成发挥着重要作用[30, 43]。Campbell等[44]发现ComX调控基因的启动子区域含有一致的保守序列,称为cin-box(competence induced box,cin-box),Cin-box位于起始密码子附近,其上游区域多为胸腺嘧啶核苷酸(图 7)。不同物种的cin-box存在一定差异,比如乳酸乳球菌的cin-box为CGTATA[9],变形链球菌的cin-box为CGAATA(图 7)。Cin-box保守序列为ComX和RNAP的识别结合位点(图 3),是ComX调控基因不可或缺的一部分,同时也是ComX调控基因的一个重要特征。
ComX可以调控多种基因的转录,图 7显示了部分受ComX调控的基因。其中comYA是特异感受基因,变形链球菌共有9个comY基因(comYA-YI),Merritt等[45]发现comYH和comYI基因突变菌株对自然感受效率没有影响,而其余7个基因任何一个基因缺失都会大幅降低自然感受效率,因此这7个ComY蛋白类似于枯草芽孢杆菌中的ComG蛋白,在DNA摄取过程中发挥着重要作用;又有人发现recA、ssbB、dprA和coiA基因的表达可以保护ssDNA免受核酸酶的降解,对ssDNA成功整合到染色体中发挥着重要作用;Okinaga等[46]发现SMU_431、SMU_507、SMU_539、SMU_769、SMU_836、SMU_1055 和SMU_1904等单个基因缺陷的菌株自然转化效率均有不同程度的降低,其中SMU_539突变株完全丧失了自然转化能力,因此这些基因对自然感受具有不同程度的影响。受ComX调控的基因也可以参与细胞内的其他生理活动,比如SMU_925编码一种细菌素免疫蛋白[46],对细菌素合成具有一定的影响;SMU_836编码一种转运蛋白,对菌体耐受性具有一定的影响,因此受ComX调控的基因不仅影响自然感受,对其他生理活动也具有一定的影响作用。

|
| 图 7 变形链球菌中受ComX调控的基因启动子序列比对结果 Fig. 7 Alignment of the promoter regions from ComX-regulated genes in Streptococcus mutans The conserved sequences referred to as cin-box are shaded in dark grey. N means one of A、T、C and G. The number of unseen nucleotides are listed in subscript of N |
变形链球菌中comX的调控系统既有明确的分工,又彼此相互交织,相互影响,比如HdrRM和BsrRM系统都可以调控自然感受和细菌素的合成,但HdrRM侧重于自然感受的调控,而BsrRM系统侧重于细菌素的合成。虽然变形链球菌中的自然感受状态是一个短暂的状态,但是自然感受的调控和形成与生物膜形成、耐酸性和细菌素合成等毒力因子密切相关,对细胞内的生理活动具有重要的影响作用。
4.1 自然感受与生物膜的联系变形链球菌中comDE调控是CSP介导的群感效应,而细菌间的群感效应是生物膜形成的主要调控系统[47],ComDE系统不仅在自然感受过程中发挥着重要的作用,也对生物膜的形成发挥着重要作用。张恺等[48]通过扫描电镜发现,加入CSP信号肽的变形链球菌细胞膜聚集成团,而不加CSP的对照组细胞膜分布较为稀疏,因此CSP信号肽对细胞膜具有重要的影响作用。Zhang等[49]发现在变形链球菌生长过程中额外添加CSP,可以增加生物膜的合成量,CSP添加12h和24h后生物膜中菌体的死亡量和生存量都高于不添加CSP的情况,细胞死亡后裂解释放的基因组DNA是一种胞外粘附物,对细胞间的粘附、细胞膜的稳定发挥着重要作用;而生物膜中的活菌则能持续产生CSP进而诱导自然感受态的形成,该状态下的细胞摄取外源DNA能够增强黏附力,有利于细胞膜的形成,因此无论是死亡的还是活着的细胞都有利于生物膜的形成,同时这也阐释了 CSP介导的ComDE调控系统在变形链球菌生物膜形成中所起的作用。
Senadheera等[50]发现VicRK双组分系统影响gtfB、gtfC、gtfD、gbpB、ftf多个基因的表达,而VicRK和这些基因的上游都具有ComE的假定结合位点(图 4),有可能VicRK与ComE作用后一起调控这些基因的表达。gtf基因编码一种葡萄糖基转移酶,GtfB和GtfC能够利用蔗糖合成不溶于水的葡聚糖,该水不溶的葡聚糖可以促进细胞粘附和聚集,是生物膜形成过程中的重要因子;另外ftf基因编码一种果糖基转移酶,有助于果聚糖的合成,而果聚糖可以促进细菌聚集[51],从而有利于生物膜的形成。因此变形链球菌中ComDE调控途径可以通过早期核心调控因子ComE调节生物膜的形成。
4.2 自然感受与耐酸性的联系Ahn等[52]发现在细菌密度较高时,变形链球菌 BM71细胞的耐酸性明显高于低细菌密度时的细胞,comC、comD和comE单基因缺陷株对外界酸性物质的感应和耐受都存在明显的缺陷,但是在comC缺陷株培养基中添加化学合成的CSP后,该菌株便可恢复一定的耐酸性;许春雁[53]发现耐酸相关基因comD和gbpB点突变影响着变形链球菌的耐酸能力,而gbpB受ComE的调控,因此变形链球菌中的ComDE调控系统对耐酸性有一定的影响。Senadheera等[50]发现变形链球菌vicK基因缺陷菌的乳酸产量减少,但在pH为3.5以下vicK基因缺陷株的存活率却大幅提高,因此VicRK 对变形链球菌的产酸耐酸有一定影响。Ahn等[41]发现rcr操纵子、comX、comYA和relP的启动子区域均具有RcrR的结合位点,可以被RcrR调控。其中RelP和RelRS双组分系统由同一个操纵子编码,RcrR通过调控relP影响(p)ppGpp的代谢,而(p)ppGpp的含量影响rcrRPQ基因的表达,RcrRPQ对自然感受效率与耐酸性均有一定的影响,因此rcrRPQ操纵子通过RelPRS系统将自然感受与耐酸性紧密联系起来。
4.3 自然感受与细菌素合成的关系Kreth等[54]在变形链球菌UA140中发现CSP可以同时诱导自然感受态和细菌素的合成,van der Ploeg等[55]发现变形链球菌在细胞浓度较高时,额外添加CSP情况下能够诱导多个细菌素合成基因的表达,比如细菌素nlmAB、bsmABC以及免疫蛋白基因immB。生物信息学分析发现这些基因上游都具有2段被10bp间隔的11bp的保守序列(图 8),该序列可以被早期感受核心调控因子ComE识别,因此早期感受调控因子ComE将自然感受调控和细菌素合成联系起来。CipB细菌素对自然感受和细菌素合成都具有影响作用,CSP诱导表达的CipB在细胞内外的功能是不同的,NlmTE转运蛋白能将CipB分泌到细胞外[56],胞外成熟的CipB可以抑制竞争者(如乳酸乳球菌)的生长,从而增加自身的生存优势,但是不参与自然感受的调控;而胞内CipB的积累能够促使菌体自溶[57](图 9),菌体裂解释放的DNA被存活的细胞所摄取,促进自然感受态的形成[16]。有研究者发现CipB细菌素不仅具有裂解细胞的作用,对自然感受也有一定的调控作用[40]。当细胞浓度较低时comC编码的CSP浓度较低,ComDE调控下的CipB含量较低,而CipI在双组分系统LiaSR调控下表达量增加,总体上CipI的含量多于CipB,细胞内少量的CipB被大量的CipI所束缚而无法调控自然感受(图 9);随着细胞的生长,胞外的CSP浓度增加,ComDE调控系统大幅激活cipB的转录使细胞内CipB的含量大于CipI免疫蛋白的量,除去被CipI束缚的CipB,仍有剩余的CipB通过调控comE和comR激活感受基因的表达,(图 9)CipB不仅可以通过调控comE参与ComDE调控系统,也可以通过调控comR参与到ComRS调控系统中,因此CipB成功地将ComDE与ComRS两大comX调控系统联系起来,促进了自然感受调控系统间复杂网络的构建[40]。

|
| 图 8 变形链球菌中bsmA,bsmB,bsmC,nlmAB,nlmC和immB基因的上游序列 Fig. 8 The upstream regions of bsmA,bsmB,bsmC,nlmAB and immB of Streptococuus mutans Pale grey and dark grey highlighted the possible ComE binding sites |
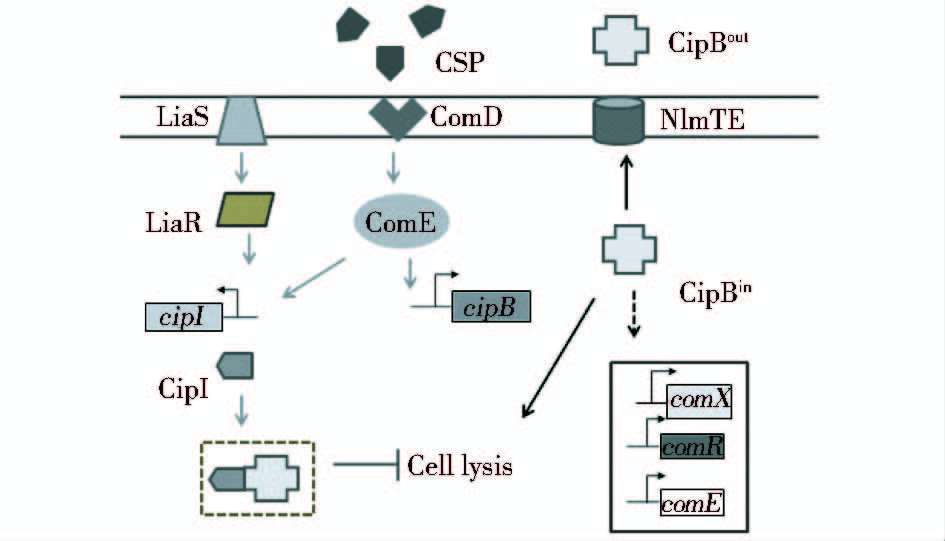
|
| 图 9 变形链球菌中CipB和CipI的调控方式 Fig. 9 Regulatory model of CipB and CipI in Streptococcus mutans |
自然感受的调控系统错综复杂,目前变形链球菌中已经发现ComDE、ComRS、HdrRM、BsrRM和RcrRPQ等多种系统通过comX对自然感受进行调控,但是这些调控系统之间的内在联系却鲜有报道,如图 3所示,HdrRM系统与ComDE、ComRS等系统之间的联系点尚未明确,RcrRPQ系统(如图 6所示)与其他comX调控系统之间的关系研究也较少。随着各个调控系统的不断完善,不难发现不同调控路径中有许多反馈调控,比如ComDE和ComRS调控路径(如图 3所示);即使是同一调控路径也有许多可逆调控[44],例如ComR和ComS之间可以彼此调控,这些信息对comX其他调控路径的探索具有重要的参考价值。目前许多调控细节值得深入研究,如CSP诱导自然感受中ComE如何作用于comX基因,CipB细菌素如何调控ComDE和ComRS系统等等[44],这些调控细节的探索对解读各调控系统间的关系具有一定的启示作用。调控系统内在联系是从总体上深入了解自然感受调控不可或缺的一部分,有可能成为未来自然感受的研究热点。
目前革兰氏阳性菌的自然感受研究逐步完善,但是自然感受的基础研究还需要进一步深入,比如外源DNA为什么需要降解为ssDNA才能进入细胞,哪些性质决定了菌株能否成为自然感受菌株。自然感受菌株能够在自然生长过程中摄取外源DNA改变遗传性状,而自然感受与多种生理特性密切相关,那么人工利用自然感受状态有可能成为改造菌株生理特性的手段,比如过表达或敲除相关感受基因改造菌株耐酸性、提高菌株细菌素合成产量。随着自然感受研究的深入与合成生物学、基因工程的不断发展,自然感受与这些生物技术相结合有望成为加快细菌进化的有效方式。
| [1] | Claverys J P, Prudhomme M, Martin B. Induction of competence regulons as a general response to stress in gram-positive bacteria. Microbiology, 2006, 60(1): 451. |
| [2] | Slager J, Kjos M, Attaiech L, et al. Antibiotic-induced replication stress triggers bacterial competence by increasing gene dosage near the origin. Cell, 2014, 157(2): 395-406. |
| [3] | Claverys J P, Martin B, Polard P. The genetic transformation machinery: composition, localization and mechanism. FEMS Microbiology Reviews, 2009, 33(3): 643-656. |
| [4] | 孙东昌, 张衍梅, 施跃峰. 细菌自然转化的分子机制研究进展. 微生物学报, 2012, 52(1): 6-11. Sun D C, Zhang Y M, Shi Y F. Advances in the molecular mechanism of natural bacterial transformation. Acta Microbiologica Sinica, 2012, 52(1): 6-11. |
| [5] | Griffith F. The significance of pneumococcal types. Journal of Hygiene, 1928, 27(02): 113-159. |
| [6] | Johnston C, Martin B, Fichant G, et al. Bacterial transformation: distribution, shared mechanisms and divergent control. Nature Reviews Microbiology, 2014, 12(3): 181-196. |
| [7] | Perry D, Kuramitsu H K. Genetic transformation of Streptococcus mutans. Infection and Immunity, 1981, 32(3): 1295-1297. |
| [8] | Finkel S E, Kolter R. DNA as a nutrient: novel role for bacterial competence gene homologs. Journal of Bacteriology, 2001, 183(21): 6288-6293 |
| [9] | Claverys J P, Martin B. Bacterial 'competence' genes: signatures of active transformation, or only remnants?. Trends in Microbiology, 2003, 11(4): 161-165. |
| [10] | Balaban M, Bättig P, Muschiol S, et al. Secretion of a pneumococcal type II secretion system pilus correlates with DNA uptake during transformation. Proceedings of the National Academy of Sciences, 2014, 111(7): e758-e765. |
| [11] | Laurenceau R, Péhau-Arnaudet G, Baconnais S, et al. A type IV pilus mediates DNA binding during natural transformation in Streptococcus pneumoniae. PLoS Pathogens, 2013, 9(6): e1003473. |
| [12] | Muschiol S, Balaban M, Normark S, et al. Uptake of extracellular DNA: Competence induced pili in natural transformation of Streptococcus pneumoniae. BioEssays, 2015,37(4):426-435. |
| [13] | Mejean V, Claverys J P. DNA processing during entry in transformation of Streptococcus pneumoniae. Journal of Biological Chemistry, 1993, 268(8): 5594-5599. |
| [14] | Midon M, Schäfer P, Pingoud A, et al. Mutational and biochemical analysis of the DNA-entry nuclease EndA from Streptococcus pneumoniae. Nucleic Acids Research, 2011,39(2):623-634. |
| [15] | Mortier-Barrière I, Velten M, Dupaigne P, et al. A key presynaptic role in transformation for a widespread bacterial protein: DprA conveys incoming ssDNA to RecA. Cell, 2007, 130(5): 824-836. |
| [16] | Quevillon-Cheruel S, Campo N, Mirouze N, et al. Structure-function analysis of pneumococcal DprA protein reveals that dimerization is crucial for loading RecA recombinase onto DNA during transformation. Proceedings of the National Academy of Sciences, 2012, 109(37): e2466-e2475. |
| [17] | Attaiech L, Olivier A, Mortier-Barrière I, et al. Role of the single-stranded DNA-binding protein SsbB in Pneumococcal transformation: Maintenance of a reservoir for genetic plasticity. PLoS Genetics, 2011, 7(6): e1002156. |
| [18] | Yadav T, Carrasco B, Hejna J, et al. Bacillus subtilis DprA recruits RecA onto single-stranded DNA and mediates annealing of complementary strands coated by SsbB and SsbA. Journal of Biological Chemistry, 2013, 288(31): 22437-22450. |
| [19] | Dong G F, Tian X L, Gomez Z A, et al. Regulated proteolysis of the alternative sigma factor SigX in Streptococcus mutans: implication in the escape from competence. BMC Microbiology, 2014, 14(1): 183. |
| [20] | Liu J, Zuber P. A molecular switch controlling competence and motility: competence regulatory factors ComS, MecA, and ComK control cD-dependent gene expression in Bacillus subtilis. Journal of Bacteriology, 1998, 180(16): 4243-4251. |
| [21] | Petersen F C, Scheie A. Genetic transformation in Streptococcus mutans requires a peptide secretion-like apparatus. Oral Microbiology and Immunology, 2000, 15(5): 329-334. |
| [22] | Li Y H, Lau P C, Lee J H, et al. Natural genetic transformation of Streptococcus mutans growing in biofilms. Journal of Bacteriology, 2001, 183(3): 897-908. |
| [23] | Berka R M, Hahn J, Albano M, et al. Microarray analysis of the Bacillus subtilis K-state: genome-wide expression changes dependent on ComK. Molecular Microbiology, 2002, 43(5): 1331-1345. |
| [24] | Turgay K, Hahn J, Burghoorn J, et al. Competence in Bacillus subtilis is controlled by regulated proteolysis of a transcription factor. The EMBO Journal, 1998, 17(22): 6730-6738. |
| [25] | 彭磊, 吴丹, 黄博, 等. 变形链球菌反应调控蛋白ComE结合序列的初步筛选. 中国生物工程杂志. 2010, 30(5): 27-53. Peng L, Wu D, Huang B, et al. Preliminary screening of ComE binding sequences in Streptococuus mutans. China Biotechnology, 2010, 30(5): 27-53. |
| [26] | Hung D C I, Downey J S, Ayala E A, et al. Characterization of DNA binding sites of the ComE response regulator from Streptococcus mutans. Journal of Bacteriology, 2011, 193(14): 3642-3652. |
| [27] | Hung D C, Downey J S, Kreth J, et al. Oligomerization of the response regulator ComE from Streptococcus mutans is affected by phosphorylation. Journal of Bacteriology, 2012, 194(5): 1127-1135. |
| [28] | Mashburn-Warren L, Morrison D A, Federle M J. A novel double-tryptophan peptide pheromone controls competence in Streptococcus spp. via an Rgg regulator. Molecular Microbiology, 2010, 78(3): 589-606. |
| [29] | McIver K S. Stand-alone response regulators controlling global virulence networks in Streptococcus pyogenes. Contrib Microbiol, 2009, 16: 103-119. |
| [30] | Fontaine L, Goffin P, Dubout H, et al. Mechanism of competence activation by the ComRS signalling system in Streptococci. Molecular Microbiology, 2013, 87(6): 1113-1132. |
| [31] | Fontaine L, Wahl A, Fléchard M, et al. Regulation of competence for natural transformation in Streptococci. Infection, Genetics and Evolution, 2014. |
| [32] | Desai K, Mashburn-Warren L, Federle M J, et al. Development of competence for genetic transformation of Streptococcus mutans in a chemically defined medium. Journal of Bacteriology, 2012, 194(15): 3774-3780. |
| [33] | Kaspar J, Ahn S J, Palmer S R, et al. A unique open reading frame within the comX gene of Streptococcus mutans regulates genetic competence and oxidative stress tolerance. Molecular Microbiology, 2015,96(3):463-482. |
| [34] | Guo Q, Ahn S J, Kaspar J, et al. Growth phase and pH influence peptide signaling for competence development in Streptococcus mutans. Journal of Bacteriology, 2014, 196(2): 227-236. |
| [35] | Khan R, Rukke H V, Ricomini Filho A P, et al. Extracellular identification of a processed type II ComR/ComS pheromone of Streptococcus mutans. Journal of Bacteriology, 2012, 194(15): 3781-3788. |
| [36] | Son M, Ahn S J, Guo Q, et al. Microfluidic study of competence regulation in Streptococcus mutans: environmental inputs modulate bimodal and unimodal expression of comX. Molecular Microbiology, 2012, 86(2): 258-272. |
| [37] | Nikolskaya A N, Galperin M Y. A novel type of conserved DNA-binding domain in the transcriptional regulators of the AlgR/AgrA/LytR family. Nucleic Acids Research, 2002, 30(11): 2453-2459. |
| [38] | Okinaga T, Niu G, Xie Z, et al. The hdrRM operon of Streptococcus mutans encodes a novel regulatory system for coordinated competence development and bacteriocin production. Journal of Bacteriology, 2010, 192(7): 1844-1852. |
| [39] | Xie Z, Okinaga T, Niu G, et al. Identification of a novel bacteriocin regulatory system in Streptococcus mutans. Molecular Microbiology, 2010, 78(6): 1431-1447. |
| [40] | Seaton K, Ahn S J, Sagstetter A M, et al. A transcriptional regulator and ABC transporters link stress tolerance, (p) ppGpp, and genetic competence in Streptococcus mutans. Journal of Bacteriology, 2011, 193(4): 862-874. |
| [41] | Ahn S J, Seaton K, Burne R A. Regulation of competence and gene expression in Streptococcus mutans by the RcrR transcriptional regulator. Molecular Oral Microbiology, 2015,30(2):147-159. |
| [42] | Ahn S J, Kaspar J, Kim J N, et al. Discovery of novel peptides regulating competence development in Streptococcus mutans. Journal of Bacteriology, 2014, 196(21): 3735-3745. |
| [43] | Eaton R E, Jacques N A. Deletion of competence-induced genes over-expressed in biofilms caused transformation deficiencies in Streptococcus mutans. Molecular Oral Microbiology, 2010, 25(6): 406-417. |
| [44] | Campbell E A, Choi S Y, Masure H R. A competence regulon in Streptococcus pneumoniae revealed by genomic analysis. Molecular Microbiology, 1998, 27(5): 929-939. |
| [45] | Merritt J, Qi F, Goodman S D, et al. Mutation of luxS affects biofilm formation in Streptococcus mutans. Infection and Immunity, 2003, 71(4): 1972-1979. |
| [46] | Okinaga T, Xie Z, Niu G, et al. Examination of the hdrRM regulon yields insight into the competence system of Streptococcus mutans. Molecular Oral Microbiology, 2010, 25(3): 165-177. |
| [47] | 陈伟. 链球菌生物膜形成中的群感效应. 中国动物传染病学报, 2010, 18(4): 81-86. Cheng W. Quorum sensing in Stereptococcal biofilm formation. Chinese Journal of Animal Infections Disease, 2010, 18(4): 81-86. |
| [48] | 张恺, 霍丽珺, 凌均棨. 口腔变形链球菌感受态刺激因子的蛋白合成及活性检测. 中华口腔医学研究杂志, 2012, 6(6):484-489. Zhang K, Huo L J, Ling J Q. Chemical synthesis and biological activities of competence stimulated peptide of quorum sensing system in Streptococcus mutans UA159. Chin J Stomatol Res, 2012, 6(6):484-489. |
| [49] | Zhang K, Ou M, Wang W, et al. Effects of quorum sensing on cell viability in Streptococcus mutans biofilm formation. Biochemical and Biophysical Research Communications, 2009, 379(4): 933-938. |
| [50] | Senadheera D, Krastel K, Mair R, et al. Inactivation of VicK affects acid production and acid survival of Streptococcus mutans. Journal of Bacteriology, 2009, 191(20): 6415-6424. |
| [51] | Rozen R, Bachrach G, Bronshteyn M, et al. The role of fructans on dental biofilm formation by Streptococcus sobrinus, Streptococcus mutans, Streptococcus gordonii and Actinomyces viscosus. FEMS Microbiology Letters, 2001, 195(2): 205-210. |
| [52] | Ahn S J, Wen Z T, Burne R A. Multilevel control of competence development and stress tolerance in Streptococcus mutans UA159. Infection and Immunity, 2006, 74(3): 1631-1642. |
| [53] | 许春雁. 变形链球菌耐氟菌株中耐酸相关基因comD及gbpB突变的检测, 吉林: 吉林大学, 2013. Xu C Y, Detection of mutation in acid-resistant genes comD and gbpB of the fluoride-resistant of Streptococcus mutants. Jilin: Jilin University, 2013. |
| [54] | Kreth J, Merritt J, Shi W, et al. Co-ordinated bacteriocin production and competence development: a possible mechanism for taking up DNA from neighbouring species. Molecular Microbiology, 2005, 57(2): 392-404. |
| [55] | van der Ploeg J R. Regulation of bacteriocin production in Streptococcus mutans by the quorum-sensing system required for development of genetic competence. Journal of Bacteriology, 2005, 187(12): 3980-3989. |
| [56] | Dufour D, Cordova M, Cvitkovitch D G, et al. Regulation of the competence pathway as a novel role associated with a streptococcal bacteriocin. Journal of Bacteriology, 2011, 193(23): 6552-6559. |
| [57] | Perry J A, Jones M B, Peterson S N, et al. Peptide alarmone signalling triggers an auto-active bacteriocin necessary for genetic competence. Molecular Microbiology, 2009, 72(4): 905-917. |
 2015, Vol. 35
2015, Vol. 35




