文章信息
- 高瑞平, 程隆斌, 李振秋
- GAO Rui-ping, CHENG Long-bin, LI Zhen-qiu
- 一种简便快速的单引物PCR定点突变方法
- A Simple and Rapid Single Primer PCR Method for Site-directed Mutagenesis
- 中国生物工程杂志, 2015, 35(5): 61-65
- China Biotechnology, 2015, 35(5): 61-65
- http://dx.doi.org/10.13523/j.cb.20150509
-
文章历史
- 收稿日期:2015-02-05
- 修回日期:2015-03-31
定点突变是研究蛋白结构与功能的重要手段。定点突变方法中应用最为广泛的是设计两条包含突变位点的完全互补方向相反的引物,以双链质粒为模板进行PCR扩增。扩增产物用Dpn I酶切去除甲基化的质粒模板,再转化DH5α感受态细胞,即可得到突变质粒的克隆[1]。Stratagene公司的QuickChange Site-directed Mutagenesis kit是此种方法的代表。
我们在开展紫穗槐二烯合酶(amorpha-4,11-diene synthase,ADS)的结构与功能之间的关系研究时,需要创建大量点突变,多数位点用QuickChange方法可以正确突变,但是少数几个位点的突变实验中遇到了出人意料的问题,测序结果显示,突变位点虽成功突变,但是在突变处插入了额外的引物序列,且数目不等(1~7个)(图 1所示示额外插入1个引物序列,方框内为引物序列)。经过反复实验,均为相同结果。为解决此问题,我们详细分析了此方法的原理和测序结果,虽然没有完全解析造成此现象的机理,但是推测可能的原因是双引物的存在造成的。另外还发现,在PCR产物中,可能存在着大量非目的片段,且是指数增加(图 2),而目的片段是线性增加的,所以非目的片段的数量要远远大于目的片段。指数扩增的原因是双引物的存在,如果将引物分开分别做单引物PCR,则非目的片段会大量减少,从理论上分析,单引物PCR产物混合后,转化大肠杆菌应该能够获得突变质粒,但是未见此方法的报道。因此,我们对传统的方法进行了改良,设计了单引物PCR方法,一为验证此方法是否可行,另外也希望通过方法的改进来解决实验中遇到的引物插入的问题。
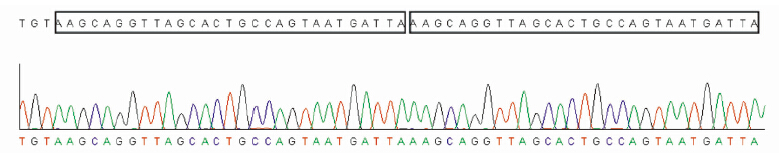
|
| 图 1 ADS G401S突变测序结果示插入的额外引物序列 Fig. 1 Sequence analysis of G401S mutations of ADS denoteing additional inserting prime sequence |
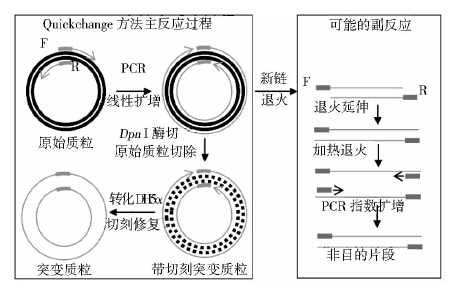
|
| 图 2 Quickchange点突变方法主反应和可能副反应示意图 Fig. 2 Schematic diagram of Quickchange main reaction and side reaction |
含有ADS基因的pET30a载体为本实验室保存。Pyrobest DNA聚合酶,dNTP mixture,限制性内切酶Dpn I均购自TaKaRa公司,引物由上海生工生物工程有限公司合成,DH5α感受态细胞购自鑫博公司,测序由中美泰和生物技术(北京)有限公司完成。
1.2 方 法 1.2.1 引物设计根据ADS基因序列设计点突变引物如表 1。
| 引物名称 | 引物序列 |
| W20R-F | 5′ TTCCTCCAAGCATTAGGGGAGATCAGTTT 3′ |
| W20R-R | 5′ AAACTGATCTCCCCTAATGCTTGGAGGAA 3′ |
| L515F-F | 5′ ATTTGTGCCAGTTTTTTGAAGTTCAATAT 3′ |
| L515F-R | 5′ ATATTGAACTTCAAAAAACTGGCACAAAT 3′ |
| G401S-F | 5′ TAATCATTACTGGCAGTGCTAACCTGCTT 3′ |
| G401S-R | 5′ AAGCAGGTTAGCACTGCCAGTAATGATTA 3′ |
50μl反应体系,其中含pET30a-ADS质粒0.5μl(约50 ng),10×buffer 5μl,dNTP mixture(各2.5mmol/L) 4μl,引物F和引物R各 2μl,Pyrobest DNA聚合酶(5U/μl) 0.5μl,无菌去离子水加至50μl,PCR扩增。PCR反应条件:94℃,10min,1循环;94℃,40s,60℃/61℃/65℃,40s,72℃,15min,15循环;72℃,10min,1循环。
1.2.3 传统QuickChange定点突变方法改进(PCR单引物定点突变方法)对传统的QuickChange定点突变方法进行改进,构建两个PCR体系,其中分别加入引物F和引物R 2μl,其它成分及反应条件同传统方法,PCR后,将两组PCR产物混和,94℃,10 min;37℃,30min进行变性复性。
1.2.4 Dpn I消化原始模板将传统方法的PCR产物和改进方法获得的混和产物进行Dpn I酶切,在其中分别加入1 μl Dpn I,37℃保温6h。将原始模板质粒消化切除。
1.2.5 转化DH5α感受态细胞按大肠杆菌感受态细胞说明书进行操作,取消化原始模板质粒后的PCR混合物3μl转化大肠杆菌DH5α,转化液涂布于卡那霉素LB固体培养基,37℃倒置过夜,次日挑取抗性单克隆于含卡那霉素的LB液体培养基中,37℃,200r/min过夜培养后送测序公司测序。
1.2.6 DNA含量测定方法DNA含量采用德国艾本德BioPhotometer plus测定。
2 结 果 2.1 定点突变PCR产物分析利用传统方法和改进方法对ADS的三个位点进行突变,经过PCR循环,变性复性Dpn I酶切后,各取2μl琼脂糖电泳EB染色紫外检测和BioPhotometer plus测定含量。电泳检测结果如图 3,DNA含量测定结果发现传统方法获得的DNA含量为20~30ng/μl,而改进后的单引物PCR获得的DNA含量为2~6ng/μl,这说明双引物PCR过程中发生了副反应,使得反应产物中存在大量非目的片段,而改进后的单引物PCR没有发生副反应,因此含量较低,这与前面的分析相一致。
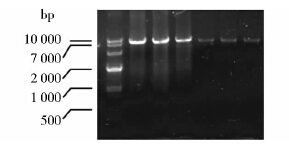
|
| 图 3 突变PCR的琼脂糖电泳检测 Fig. 3 Electrophoresis gel analysis of mutation PCRM: DNA Marker; 1-3: traditional mutation PCR of W20R,L515F,G401S; 4-6: modified mutation PCR of W20R,L515F,G401S |
Dpn I酶切反应后的PCR产物和PCR混合物转化DH5α感受态细胞,经过抗性筛选后,各挑取20个单克隆于含有卡那霉素抗性的LB液体培养基中扩大培养,送测序公司测序。
测序结果统计(表 2)发现,传统的突变方法获得的W20R、G401S抗性克隆均为未突变和插入了额外引物突变体,突变失败,L515F则有15个克隆为正确突变,无插入引物突变。而改进后的方法获得的W20R、G401S、L515F阳性克隆正确突变分别为11、12和10个,无引物插入突变体。正确突变的测序结果见图 4。
| W20R | G401S | L515F | ||||
| 传统 方法 | 改进 方法 | 传统 方法 | 改进 方法 | 传统 方法 | 改进 方法 |
|
| 正确突变 | 0 | 9 | 0 | 8 | 15 | 10 |
| 无突变 | 3 | 11 | 4 | 12 | 5 | 10 |
| 插入引物 | 17 | 0 | 16 | 0 | 0 | 0 |
| 正确突变率率 | 0% | 45% | 0% | 40% | 75% | 50% |
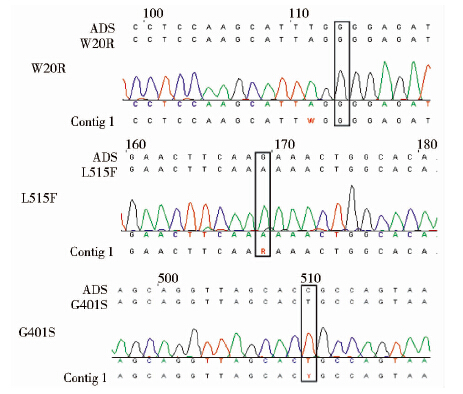
|
| 图 4 成功突变的测序结果 黑框示突变成功位点 Fig. 4 Gene sequencing analysis of succeed mutations Black frames denote the mutational sites |
QuickChange定点突变方法应用广泛,也非常成功。本实验室利用此方法构建了大量的点突变。但是在一些特殊的位点进行突变时遇到了问题,多次突变未获成功,虽然突变碱基成功引入,但是还引入了额外的序列,额外序列和引物序列一致,有的额外引物序列完整无缺,也有的缺少一、两个碱基,没有规律可循。插入的额外引物序列有时还不止一个。我们尚未解析造成此现象的真正原因。但初步推断首先与特定的引物序列有关。如本实验结果所示,L515F传统突变即可获得高效率的突变体,而另两组突变无法获得突变体,均有引物插入。这说明突变引物的插入和特定的引物有关,但进一步的引物序列分析并未找到插入引物序列突变体的引物有何规律。其次,另两组突变可以经过改进后单引物PCR突变获得正确突变体,因此可以推断插入引物的原因之一是两个完全互补引物在一个反应体系里进行。
因为QuickChange定点突变方法非常特殊,两个引物完全互补,模板是环形质粒,造成整个PCR过程复杂。为解决引物插入问题,我们从突变方法入手,创新的把两个引物分开,分别进行延伸反应,延伸产物混合后再酶切转化(流程见图 5)。通过此方法我们成功突变了之前插入额外引物序列的几个位点,并对ADS其他 29个位点进行突变,结果无一例插入额外引物序列。证明我们的推断是正确的。与QuickChange法相比,修改后的方法将两个突变引物分开进行单引物PCR扩增,避免了引物结合引起的其它不可预料的反应,完全避免了突变质粒中插入额外引物序列的现象。另外本方法还减少了非目的片段的产生,目的片段所占比率增加,为筛选阳性结果增加了机率。
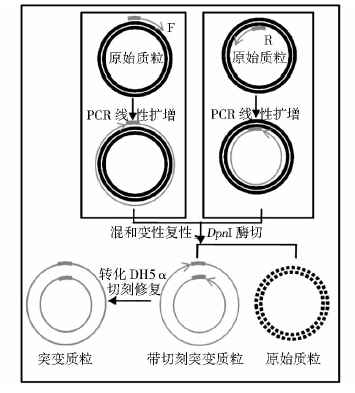
|
| 图 5 改进的定点突变方法示意图 Fig. 5 Schematic diagram of modified Quickchange mutation method |
近年来随着研究的不断深入,定点突变的方法也在不断改进,有适用于多个位点突变的方法[2, 3, 4, 5],以及通过连接突变片段达到突变目的[6, 7, 8]、重叠延伸突变法的改良[9]等。有的在节省试剂和时间方面要优于传统方法[10]。张敏等报道,用单引物二次PCR法对重组质粒HBV前C-C基因成功进行突变,提高了突变成功率。其方法和本文报道的方法不同。本文方法两个单引物PCR反应的模板均是原始质粒,而张敏等方法是将正向引物加入反应体系中进行首轮PCR扩增,再以扩增产物为模板,加入反向引物进行第二轮扩增。他们认为在第二轮扩增中,反向引物可以与正向原模板链、正向变异链结合,但由于反向引物与正向变异链完全互补,故与正向变异链的结合几率较高[11]。我们认为其分析有误。因为定点突变方法设计的引物是一对反向互补序列,其能与模板质粒的某一条链结合并延伸,其虽然也可以与新生片段相结合,但是由于其结合在新生片段的5′端,因此不能进行延伸(如图 6所示)。
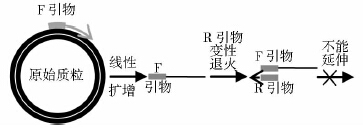
|
| 图 6 张敏等报道的突变方法流程示意图 Fig. 6 Schematic diagram of mutation method reported by ZhangMin et al |
我们推测其报道的突变成功率提高的原因不是将两个引物分开,而是由于在第二轮PCR中,还存在着原始质粒模板,其浓度降低的结果,张敏报道中反应体积为25μl,首轮PCR模板量为200ng,第二轮稀释后为40ng。其实验第一轮利用模板链合成了其中一种突变链,而第二轮则合成了另一种突变链,两链退火后即可和传统方法获得的突变切刻链相同。QuickChange Site-directed Mutagenesis kit手册建议50μl反应体积中模板量为5~50ng,模板量过多会降低突变效率。在Liu[12]和Braman[13]的方法中,也对模板量做了要求,不宜太多。
对QuickChange法的改良也有报道,Liu[12]引进了新的引物设计方案,新合成的链也可作为模板继续扩增,提高了扩增效率。Liu等[14]设计了Tc-PCR解决了DpnI对半甲基化产物消化不完全的问题。本研究建立的单引物定点突变方法从根本上解决特殊的引物序列突变时插入额外引物序列的问题。由于方法初步建立,实验例数较少,在突变效率方面还不是特别高,需要在今后的实验中不断优化改进,以提高突变效率。
| [1] | Weiner M P, Costa G L, Schoettlin W, et al. Site-directed mutagenesis of double-stranded DNA by the polymerase chain reaction. Gene, 1994, 151(1-2): 119-123. |
| [2] | Tian J, Liu Q, Dong S, et al. A new method for multi-site-directed mutagenesis. Analytical Biochemistry, 2010, 406(1): 83-85. |
| [3] | Wei H, Hu J, Wang L, et al. Rapid gene splicing and multi-sited mutagenesis by one-step overlap extension polymerase chain reaction. Analytical Biochemistry, 2012, 429(1): 76-78. |
| [4] | Seyfang A, Huaqian J J. Multiple site-directed mutagenesis of more than 10 sites simultaneously and in a single round. Analytical Biochemistry, 2004, 324(2): 285-291. |
| [5] | Liang X, Peng L, Li K, et al. A method for multi-site-directed mutagenesis based on homologous recombination. Analytical Biochemistry, 2012, 427(1): 99-101. |
| [6] | Yan P, Gao X, Shen W, et al. Parallel assembly for multiple site-directed mutagenesis of plasmids. Analytical Biochemistry, 2012, 430(1): 65-67. |
| [7] | Wan H, Li Y, Fan Y, et al. A site-directed mutagenesis method particularly useful for creating otherwise difficult-to-make mutants and alanine scanning. Analytical Biochemistry, 2012, 420(2): 163-170. |
| [8] | Adachi Y, Fukuhara C. TA strategy for rapid and efficient site-directed mutagenesis. Analytical Biochemistry, 2012, 431(1): 66-68. |
| [9] | 刘欣毅, 张惠展, 袁勤生. 一种新型的基因定点突变方法及其在DdsA突变研究中的应用. 中国生物化学与分子生物学报, 2007, 23(5): 415-418. Liu X Y,Zhang H Z,Yuan Q S. A novel method for site-directed mutagensis and its application in DdsA mutatnt research. Chinese Journal of Biochemistry and Molecular Biology,2007,23(5):415-418. |
| [10] | Hsieh P C, Vaisvila R. Protein engineering: single or multiple site-directed mutagenesis. Methods in Molecular Biology, 2013, 978:173-186. |
| [11] | 张敏, 辛绍杰, 段学章, 等. 单引物二次PCR法对重组质粒中HBV前C-C基因进行点突变的研究. 中华检验医学杂志, 2007, 30(4): 444-446. Zhang M, Xin S J, Duan X Z, et al. Establishment of a new "twice single primer PCR method" and appllication for site-directed mutation in HBV pre-core region.Chin J Lab Med, 2007,30(4):444-446. |
| [12] | Liu H, Naismith J H. An efficient one-step site-directed deletion, insertion, single and multiple-site plasmid mutagenesis protocol. BMC Biotechnology, 2008, 8:91. |
| [13] | Braman J, Papworth C, Greener A. Site-directed mutagenesis using double-stranded plasmid DNA templates. Methods in Molecular Biology, 1996, 57:31-44. |
| [14] | Liu Y, Wu T, Song J, et al. A mutant screening method by critical annealing temperature-PCR for site-directed mutagenesis. BMC Biotechnology, 2013, 13:21. |
 2015, Vol. 35
2015, Vol. 35




