文章信息
- 高越, 檀硕, 任兆瑞, 张敬之
- GAO Yue, TAN Shuo, REN Zhao-rui, ZHANG Jing-zhi
- 原位染色检测慢病毒载体转录通读方法的建立
- The Establishment of the Methodology used for In Situ Detecting Lentiviral Vector Transcriptional Read-through
- 中国生物工程杂志, 2015, 35(5): 55-60
- China Biotechnology, 2015, 35(5): 55-60
- http://dx.doi.org/10.13523/j.cb.20150508
-
文章历史
- 收稿日期:2015-02-09
- 修回日期:2015-03-09
2. 卫生部医学胚胎分子生物学重点实验室暨上海市胚胎与生殖工程重点实验室 上海 200040;
3. 生物治疗协同创新中心 四川大学 华西医院 成都 610041
2. Key Laboratory of Embryo Molecular Biology, Ministry of Health & Shanghai Key Laboratory of Embryo and Reproduction Engineering, Shanghai 200040, China;
3. Collaborative Innovation Center for Biotherapy, West China Hospital, Sichuan University, Chengdu 610041, China
慢病毒(lentivirus,LV)属于反转录病毒科慢病毒属,经功能性删减和拆分而被研制成只有一次性感染能力的慢病毒载体(lentiviral vector,LVV)。目前,最常用的LVV是以HIV-1病毒为基因骨架改造而成的[1]。转录通读(transcriptional read-through,TRT)是LVV在应用过程中存在的潜在不安全因素之一,是指载体因丧失部分或全部转录终止功能,致使其在转录过程中,越过终止信号而继续转录下游基因序列的现象。因此,TRT可能激活病毒载体整合位点下游基因,甚至激活原癌基因的表达。
在研制LVV过程中,为了降低其产生重组性LV的可能性,Zufferey等[2]删除了长末端重复序列(long terminal repeat,LTR)中U3区的启动子核心序列、TATA框等,从而研制出第三代自灭活慢病毒载体(self-inactivating lentiviral vector,SIN-LVV)。但在此删除过程中同时也删除了与启动子重叠的转录终止上游序列(upstream element,USE),使得3′ -LTR(△LTR)转录通读率升高[3]。为了降低SIN-LVV的TRT,提高其生物安全性,研究人员曾在LVV中插入一些终止辅助元件来降低TRT[4, 5]。我们也对降低TRT的方法进行探索,取得了一定的成果[6],并为此建立了通过qRT-PCR在转录水平检测TRT的方法[7]。
然而SIN-LVV若发生TRT现象造成安全隐患会在蛋白质水平表现出来,因此建立一种在蛋白质水平直观、准确检测TRT的方法对于检测方法的完善以及检测结果更加精确有着重要的意义。
为了进一步在蛋白质水平上进行SIN-LVV的TRT结果的验证,我们根据病毒载体整合入基因组的状态构建了模拟原病毒载体,在3′ △LTR 和下游poly A之间插入经过密码子优化的LacZ(optimized LacZ,OLacZ)基因,载体经转染293T细胞后,通过β-半乳糖苷酶原位染色的方法分别对模拟原病毒载体及单个△LTR的TRT进行检测,发生TRT现象的细胞将被染成蓝色。结果表明,针对单个△LTR和原病毒载体TRT的检测均有细胞被染成蓝色。不仅验证了qRT-PCR在转录水平的检测结果,而且说明原位染色检测法是一种直观、有效且准确地在蛋白质水平检测慢病毒载体转录通读的方法。
1 材料与方法 1.1 材 料 1.1.1 质粒、菌株、细胞株质粒pEGFP-N1-△LTR、proFUGW由本所保存;经密码子优化的OLacZ基因由Invitrogen公司合成,形成具有自主知识产权的OLacZ,连接入pGEM-T载体中(T-OLacZ中间载体);TOP10感受态细胞购自天根生化科技有限公司;293T细胞(人胚肾细胞)购自美国ATCC(American Type Culture Collection)细胞库。
1.1.2 主要试剂限制性内切核酸酶Sph Ⅰ、Sca Ⅰ、Sal Ⅰ,T4 DNA Ligase购自TaKaRa公司;PmeⅠ、EcoRⅠ,T4 DNA 聚合酶,碱性磷酸酶购自New England Biolabs公司;β-半乳糖苷酶原位染色试剂盒购自碧云天生物技术研究所;LipofectamineTM 2000转染试剂、Trizol购自Invitrogen公司;DPBS、DMEM培养液、胎牛血清购自Gibco公司。
1.2 方 法 1.2.1 载体构建在T-OLacZ中间载体中,由于OLacZ片段与pGEM-T载体片段大小接近,使用Sph Ⅰ、Sca Ⅰ、SalⅠ对T-OLacZ载体进行三酶切,获得OLacZ片段。使用T4 DNA 聚合酶对OLacZ片段进行补平处理。分别使用BamHⅠ对pEGFP-N1-△LTR载体进行单酶切,PmeⅠ对proFUGW载体[图 1(a)]进行单酶切。载体经补平、去磷酸化处理后与OLacZ片段进行平端连接,得到N1-△LTR-OLacZ[图 1(b)]载体和proFUGW-OLacZ[图 1(c)]载体。所构建的载体经转染细胞后,可通过β-半乳糖苷酶原位染色的方法分别对单个△LTR的转录终止作用,对模拟病毒整合入基因组的状态而构建的原病毒载体的通读状况进行检测。
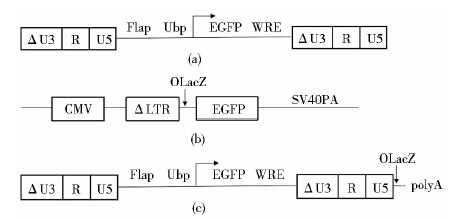
|
| 图 1 载体构建图 Fig. 1 Schematic diagram of the constructed vectors (a) proFUGW (b) N1-△LTR-OLacZ (c) proFUGW-OLacZ |
将293T细胞接种至6孔板,待细胞生长至平板40%~50%密度。使用10 μl LipofectamineTM 2000将4μg质粒N1-△LTR-OLacZ、proFUGW-OLacZ转染293T细胞,转染步骤参照转染说明书进行。在37℃、5% CO2条件下培养6h后更换含胎牛血清的培养液继续培养。
1.2.3 荧光显微镜检测质粒转染细胞48h后,小心吸去上清培养液,加入800μl DPBS覆盖细胞。置于荧光显微镜下镜检,根据细胞GFP表达情况判断质粒的转染效率。
1.2.4 FACS检测转染效率质粒转染细胞48h后,经0.25%胰酶消化细胞2min后用含血清的培养液终止反应。2 000r/min离心2min,弃上清液,加入300μl以PBS为主的鞘液,经流式细胞仪检测,计数2×104个细胞,检测表达GFP的阳性细胞数。
1.2.5 β-半乳糖苷酶原位染色法在蛋白质水平检测转录通读质粒N1-△LTR-OLacZ、proFUGW-OLacZ转染细胞48h后,按照β-半乳糖苷酶原位染色试剂盒说明书对细胞进行染色。质粒转染进入细胞,发生通读现象的细胞会表达β-半乳糖苷酶基因(OLacZ)。以X-Gal为底物,在β-半乳糖苷酶催化下会生成深蓝色产物,从而在光学显微镜下很容易观察到变成蓝色的细胞。染色完成后,在倒置显微镜下观察,并统计被染成蓝色的细胞数目。
1.2.6 qRT-PCR检测转录通读率质粒载体pEGFP-N1-△LTR、proFUGW转染293T细胞48h后收集细胞,Trizol法抽提细胞RNA。取相同量的RNA,使用特异性引物RP-R、LTR-R进行反转录。获得的cDNA分别作为模板,以JP-R、RP-R为引物,LTR-probe为探针进行荧光定量PCR。引物探针序列见表 1,原理及方法同参考文献[7]。通读率=发生通读的mRNA的量/总mRNA的量。
| Primer | Primer sequence (5′-3′) |
| LTR-RJP-FRP-RLTR-probe | GAGAGCTCCCAGGCTCAGATC
TTTAAGACCAATGACTTACAAGGCA
TTGTCTTCGTTGGGAGTGAATTAG
CTGTAGATCTTAGCCACTTT |
通过对T-OlacZ中间载体进行三酶切可得到OLacZ片段[图 2(a)],约3 139bp。将OLacZ片段平端接入pEGFP-N1-△LTR、proFUGW载体3′△LTR下游。使用限制性内切核酸酶EcoRⅠ对获得的载体N1-△LTR-OLacZ、proFUGW-OLacZ进行酶切[图 2(b)、(c)]来鉴定片段接入的正反向。酶切结果表明片段为正向接入,经测序证实载体构建成功。
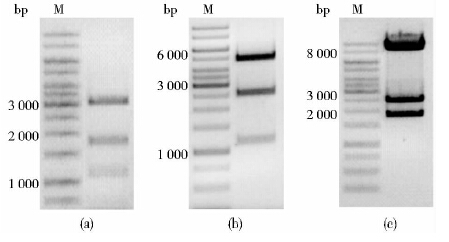
|
| 图 2 载体的酶切鉴定 Fig. 2 Proof of the OLacZ cloning by restriction endonuclease reaction(a) SphⅠ/ScaⅠ/SalⅠdigestion of the T-OLacZ vector resulted in the generation of a 3 139bp,a 1 800bp and a 1 151bp fragments,respectively (b) EcoRⅠdigestion of the N1-△LTR-OLacZ revealed a 4 763bp,a 2 508bp and a 1 109bp fragments,respectively (c) EcoRⅠdigestion of the proFUGW-OLacZ generating a 8 399bp,a 2 508bp and a 1 848bp fragments,respectively |
SIN-LVV感染细胞后,病毒RNA在自身反转录酶 作用下,最终形成两端带有两个相同LTR的双链DNA,经短暂环化后通过病毒整合酶作用整合入细胞基因组中。proFUGW质粒是根据相应病毒整合入染色质后的状态,构建而成的模拟原病毒质粒载体。
质粒N1-△LTR-OLacZ、proFUGW-OLacZ分别转染293T细胞48h后,通过流式细胞技术与荧光显微镜观察并检测其转染效率。N1-△LTR-OLacZ载体由于OLacZ基因插入报告基因GFP上游致使GFP表达较弱。故相同条件下同时转染pEGFP-N1作为对照,来判定N1-△LTR-OLacZ载体的转染效率。通过FACS检测得出pEGFP-N1与proFUGW-OLacZ转染细胞后表达GFP的细胞阳性率分别为79.6%与63.4%(图 3)。
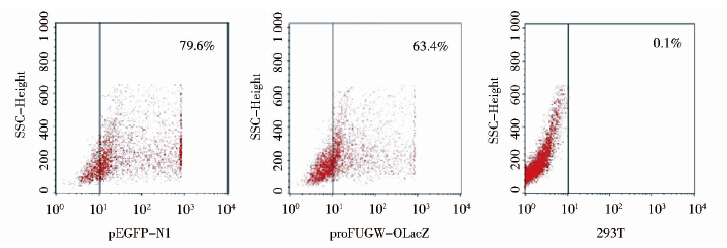
|
| 图 3 流式细胞技术检测载体转染细胞后表达GFP细胞的阳性率 Fig. 3 GFP positive rate of cells infected by vectors |
荧光显微镜下观察载体转染细胞GFP表达情况[图 4(a)、(c)],对细胞进行β-半乳糖苷酶原位染色,显微镜下观察OLacZ基因的表达情况[图 4(b)、(d)],即各载体的转录通读情况。并对蓝色细胞数目进行统计(图 5)。载体N1-△LTR-OLacZ所转染的细胞部分被染成蓝色,蓝色细胞所占比例为16%(n=3),说明单个△LTR在转录过程中不能够起到完全终止的作用。模拟原病毒载体proFUGW-OLacZ所转染的细胞则有更少量的细胞被染成蓝色,蓝色细胞所占比例仅为6%(n=3),明显低于针对单个△LTR的检测结果,这与LVV在临床应用中并没有严重不良事件发生的事实是相符合的。说明相比于单个△LTR,原位染色针对模拟原病毒载体TRT的检测结果更接近于病毒载体的实际情况。
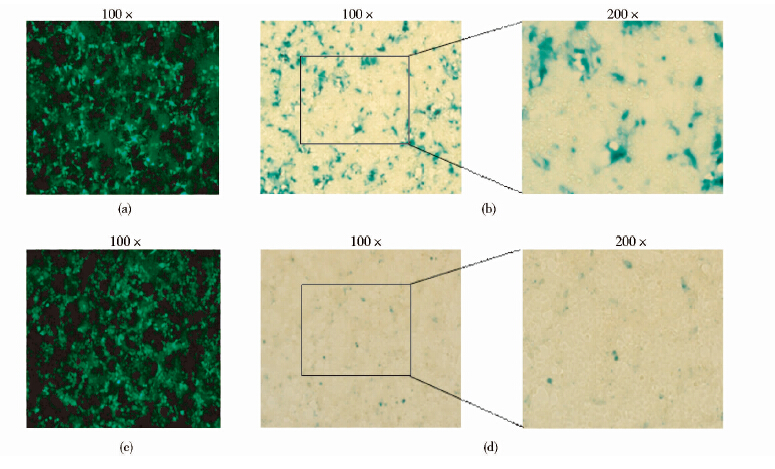
|
| 图 4 N1-△LTR-OLacZ、proFUGW-OLacZ质粒转染细胞荧光镜检和β-半乳糖苷酶原位染色 Fig. 4 Fluorescence microscopy and β-galactosidase staining of the 293T cells transfected with N1-△LTR-OLacZ and proFUGW-OLacZ(a),(b) Fluorescence microscopy and β-galactosidase staining of the 293T cells transfected with N1-△LTR-OLacZ (c),(d) Fluorescence microscopy and β-galactosidase staining of the 293T cells transfected with proFUGW-OLacZ |

|
| 图 5 转染不同载体的293T细胞β-半乳糖苷酶原位染色后蓝色细胞计数 Fig. 5 Cell counting of β-galactosidase stained 293T cells transfected by different vectors |
我们通过qRT-PCR对质粒载体pEGFP-N1-△LTR、proFUGW的TRT进行检测,结果如图 6所示,得出单个△LTR 与SIN-LVV在转录水平的转录通读率分别为46%和62%左右(n=3)。
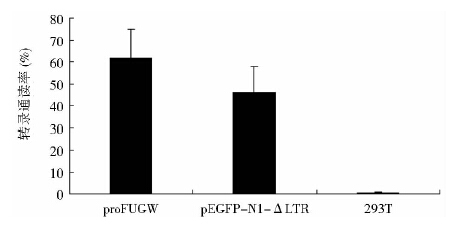
|
| 图 6 qRT-PCR检测质粒载体pEGFP-N1-△LTR、proFUGW转录通读率 Fig. 6 Read-through rate of pEGFP-N1-△LTR and proFUGW detected by qRT-PCR |
在临床基因治疗试验中,起初研究人员常使用γ反转录病毒载体作为基因转移工具。在对奥尔德利希综合征(Wiskott-Aldrich syndrome,WAS)、X连锁重度复合型免疫缺陷病(X-linked severe combined immunodeficiency,X-SCID)等患者进行治疗过程中,载体基因整合入LMO2等原癌基因的附近,由于TRT的影响,激活其下游表达,导致多名患者罹患白血病[8, 9]。
与γ反转录病毒载体相比,LVV更倾向均匀地分布于染色质转录活性区域[10]。到目前为止,全球范围内有近百例获得批准的临床基因治疗试验方案使用了LVV作为基因转移工具[11],并且在异染性脑白质营养不良(meta-chromatic leuko-dystrophy,MLD)[12]、β-地中海贫血[13]、WAS[14]等疾病的基因治疗过程中,都取得了良好的治疗效果。虽然在这个过程中没有严重不良事件的发生,但其潜在的安全隐患依然存在,这也可能是多数基于LVV的基因治疗仍处于临床一期,以及多应用于少数重症疾病的原因之一。它的安全隐患主要来源于以下3个方面:(1)治疗过程中重组活病毒产生的可能性,通过对病毒的改造,这方面的可能性已基本上被消除;(2)随机整合引起插入性突变,由于编码序列只占总基因组的5%左右,因此,有害插入性突变的可能性也很小;(3)TRT和异常剪切引起下游基因的表达[15, 16, 17, 18]。
为了便于定量检测SIN-LVV的通读率,以及验证降低通读率的方案是否有效,我们先前已经建立了在转录水平基于qRT-PCR的检测方法。然而SIN-LVV发生通读现象后,其潜在的安全隐患是在蛋白质水平体现的。Yang等[19]通过引进Cre-loxP系统对SIN-LVV单个△LTR的TRT在蛋白质水平上进行检测,证明了△LTR的TRT明显高于野生病毒LTR (wLTR)。但单个△LTR的TRT是否能够代表完整的SIN-LVV还存在着疑问。我们通过β-半乳糖苷酶原位染色的方法对单个△LTR和原病毒载体的TRT进行检测。结果发现均有细胞被染为蓝色,说明均有TRT现象的发生,证明了原位染色方法的可行性。其中针对单个△LTR的检测结果发现蓝色细胞较多且OLacZ基因表达强烈,可能与表达载体所使用的启动子较强有关。而原病毒载体模拟了病毒载体整合入染色质后的状态,因此针对原病毒载体的检测结果更接近于病毒载体的实际情况。原位染色结果与qRT-PCR所得结果相比较,虽然在转录水平SIN-LVV的TRT高于蛋白质水平,但两者的结论是平行一致的。这可能与通读RNA下游基因缺乏核糖体内部入口造成翻译效率较差,以及通读RNA的稳定性受到RNA链长度及多聚腺苷酸化的影响,翻译成蛋白质前可能被降解等因素有关。在针对某些降低SIN-LVV TRT改造方案的检测中,原位染色法比qRT-PCR检测方法的应用范围更为广泛且限制条件较少,对其是一个很好的补充与改进。
我们所建立的原位染色方法进一步完善了慢病毒载体转录通读的检测方案,使检测结果更加直观、准确。为降低慢病毒载体转录通读方案的鉴定,提高慢病毒载体生物安全性的研究提供了技术支持。慢病毒载体拥有众多的优点,在提高其生物安全性的基础上,会成为未来科学研究和临床基因治疗的重要工具。
| [1] | Buchschacher G L Jr, Wong-Staal F. Development of lentiviral vectors for gene therapy for human diseases. Blood, 2000, 95(8): 2499-2504. |
| [2] | Zufferey R, Dull T, Mandel R J, et al. Self-inactivating lentivirus vector for safe and efficient in vivo gene delivery. J Virol, 1998, 72(12): 9873-9880. |
| [3] | Zaiss A K, Son S, Chang L J. RNA 3' readthrough of oncoretrovirus and lentivirus: implications for vector safety and efficacy. Journal of Virology, 2002, 76(14): 7209-7219. |
| [4] | Higashimoto T, Urbinati F, Perumbeti A, et al. The woodchuck hepatitis virus post-transcriptional regulatory element reduces readthrough transcription from retroviral vectors. Gene Ther, 2007, 14(17): 1298-1304. |
| [5] | Schambach A, Galla M, Maetzig T, et al. Improving transcriptional termination of self-inactivating gamma-retroviral and lentiviral vectors. Mol Ther, 2007, 15(6): 1167-1173. |
| [6] | Fang Y, Gong X, Xu M, et al. A self-deletion lentiviral vector to reduce the risk of replication-competent virus formation. J Gene Med, 2013, 15(2): 102-112. |
| [7] | 何佳平, 方彧聃, 张帆, 等. 慢病毒载体转录通读率检测方法的建立. 生物工程学报, 2013, 29(007): 1006-1015. He J P, Fang Y D, Zhang F, et al. Establishment of the methodology for quantifying lentiviral vector transcriptional read-through rate. Chinese Journal of Biotechnology, 2013, 29(007): 1006-1015. |
| [8] | Galy A, Thrasher A J. Gene therapy for the Wiskott-Aldrich syndrome. Curr Opin Allergy Clin Immunol, 2011, 11(6): 545-550. |
| [9] | Hacein-Bey-Abina S, von Kalle C, Schmidt M, et al. A serious adverse event after successful gene therapy for X-linked severe combined immunodeficiency. N Engl J Med, 2003, 348(3): 255-256. |
| [10] | Wu X, Li Y, Crise B, et al. Transcription start regions in the human genome are favored targets for MLV integration. Science, 2003, 300(5626): 1749-1751. |
| [11] | Wiley J. Vectors Used in Gene Therapy Clinical Trials.[2015-02-06]. http://www.wiley.com//legacy/wileychi/genmed/clinical/. |
| [12] | Biffi A, Montini E, Lorioli L, et al. Lentiviral hematopoietic stem cell gene therapy benefits metachromatic leukodystrophy. Science, 2013: 341(6148):1233158. |
| [13] | Cavazzana-Calvo M, Payen E, Negre O, et al. Transfusion independence and HMGA2 activation after gene therapy of human beta-thalassaemia. Nature, 2010, 467(7313): 318-322. |
| [14] | Aiuti A, Biasco L, Scaramuzza S, et al. Lentiviral hematopoietic stem cell gene therapy in patients with Wiskott-Aldrich syndrome. Science, 2013: 341(6148):1233151. |
| [15] | Schambach A, Baum C. Clinical application of lentiviral vectors - concepts and practice. Curr Gene Ther, 2008, 8(6): 474-482. |
| [16] | Escors D and Breckpot K. Lentiviral vectors in gene therapy: their current status and future potential. Arch Immunol Ther Exp(Warsz), 2010, 58(2): 107-119. |
| [17] | Dropulic B. Lentiviral vectors: their molecular design, safety, and use in laboratory and preclinical research. Hum Gene Ther, 2011, 22(6): 649-657. |
| [18] | Pauwels K, Gijsbers R, Toelen J, et al. State-of-the-art lentiviral vectors for research use: risk assessment and biosafety recommendations. Curr Gene Ther, 2009, 9(6): 459-474. |
| [19] | Yang Q, Lucas A, Son S, et al. Overlapping enhancer/promoter and transcriptional termination signals in the lentiviral long terminal repeat. Retrovirology, 2007, 4: 4. |
 2015, Vol. 35
2015, Vol. 35




