文章信息
- 梁丽珠, 孙佳楠, 李恺, 刘明伟, 丁琛, 秦钧
- LIANG Li-zhu, SUN Jia-nan, LI Kai, LIU Ming-wei, DING Chen, QIN Jun
- 蛋白质组分析油酸对HepG2细胞转录因子DNA结合活性的影响
- Proteome-wide Screening of Transcription Factor DNA Binding Activity in HepG2 Cells after Oleic Acid Treatment
- 中国生物工程杂志, 2015, 35(5): 22-31
- China Biotechnology, 2015, 35(5): 22-31
- http://dx.doi.org/10.13523/j.cb.20150504
-
文章历史
- 收稿日期:2015-01-30
- 修回日期:2015-02-12
2. 军事医学科学院放射与辐射医学研究所 北京 100850;
3. 国家蛋白质组学重点实验室 北京蛋白质组学研究中心 北京 102206;
4. 沈阳药科大学药学院 沈阳 110016;
5. 河北联合大学生命科学学院 唐山 063009
2. Institute of Radiation Medicine, Academy of Military Medical Sciences, Beijing 100850, China;
3. Beijing Proteome Research, State Key Laboratory of Proteomics, Beijing 102206, China;
4. College of Pharmacy of Shenyang Pharmaceutical University, Shenyang 110016, China;
5. School of Life Sciences of Hebei United University, Tangshan 063009, China
当摄入的能量高于机体消耗的能量时,在脂肪组织就会形成大量的甘油三酯,造成肥胖[1];在非脂肪组织,如肝脏、心脏,就会引起脂肪变性[2]。在病理学上对于肝脏脂肪变性的诊断标准是肝脏内脂类的含量占总肝重的5%~10%[3, 4, 5]。肝脏脂肪变性是肝脏病变的早期阶段,进而会发展为非乙醇性脂肪肝、脂肪性肝炎、肝硬化及肝癌[6]。
由于脂类氧化主要在线粒体内完成,不少研究者已经通过同位素示踪、磁共振波谱分析及线粒体定量蛋白质组学等多种手段研究了非乙醇性脂肪肝及其动物模型中线粒体内糖异生、TCA循环的变化[7, 8]。饮食摄入的长链脂肪酸在转录水平上调控肝脏内基因的表达,自20世纪90年代起就有所研究[9, 10, 11],但由于转录因子丰度低且处于动态变化的过程中,很难有全面的观察。
转录因子参与了机体内包括细胞周期、器官发育在内的几乎全部的生理过程的调控。它们行使功能是通过与DNA结构中的启动子结合,影响转录起始复合物的形成,从而激活或抑制基因的表达。在人类基因组中约有6%的基因编码转录因子[12],这些转录因子的含量仅占细胞总蛋白质的0.001%~0.01%。利用传统的方法对转录因子进行质谱鉴定需要数百升的细胞培养物。通过特异性抗体进行的亲和纯化是研究转录因子及其复合物的有效手段,得到的转录因子约占总数的5%。对于目前已知的约1 500种转录因子,根据DNA结合结构域进行分类,可以分为约50个家族,每个家族都结合特异的DNA序列,如核受体家族的转录因子的特异性结合序列为5′-AGAACA-3′或5′-AGGTCA-3′。因此,利用特异的串联DNA序列进行转录因子的富集,有效提高了转录因子在蛋白质水平上的鉴定率[13]。但是这种方法尚未得到广泛应用。
目前,对脂肪酸在转录水平上影响基因表达研究较为清楚的是脂肪酸可以调控包括核受体在内的多种转录因子。其中,属于核受体家族的PPAR家族(PPARα、PPARβ、PPARγ1、PPARγ2)与饱和、不饱和脂肪酸都可以结合。PPAR再与RXRs形成二聚体,共同结合在下游基因的启动子区,行使调节功能。其中在肝中主要是PPARα起作用,控制脂肪酸的延伸、去饱和、氧化及转运[14]。在PPARα敲除鼠中发现缺少PPARα除了影响脂肪酸相关的代谢途径外对糖类代谢、脂肪生成影响不大[15, 16]。另外的研究则表明,脂肪酸可以在转录水平调控肝内糖代谢、脂代谢及蛋白质代谢的关键环节。脂肪酸的摄入抑制ChREBP和MLX二聚体在核内的积累,在转录水平上抑制糖酵解的过程,减少了将糖类化合物向脂肪酸转化[17]。同样,脂肪酸的摄入也可以控制炎症反应和细胞的氧化应激,但是不同的脂肪酸所起的作用却是不同的。ω-3多不饱和脂肪酸可以通过抑制NF-κB信号通路的膜受体TLRs(Toll-like receptors)[18],并且通过活化PPARα来激活IκBα,从而抑制NF-κB在核内的积累,进而抑制炎症反应以及抑制肝脏纤维化的进程,这对机体是有益的。但是,在非酒精性脂肪肝患者中也发现,饱和脂肪酸会激活TLRs[19],并且会引起内质网应激反应,导致未折叠蛋白质的蓄积,进而引发细胞脂毒性凋亡[20]。
脂肪酸摄入导致的HepG2细胞内脂类积累是常用的研究脂肪变性的体外模型[21, 22, 23]。油酸属于ω-9单不饱和脂肪酸,以甘油酯的形式存在于一切动植物油脂中,是日常饮食中摄入最多的脂肪酸(动物脂肪中占40%~50%,花生油中占54%,橄榄油中占55%~83%)。但是目前对于油酸对细胞作用的研究还很局限。作为不饱和脂肪酸的一员,油酸确实能够缓解饱和脂肪酸引起的细胞毒性[24],但是与ω3、ω6等多不饱和脂肪酸相比,油酸确实可以引起细胞的氧化损伤与胰岛素抵抗[22, 25]。油酸在细胞内究竟引起了哪些变化还有待深入研究。另外,已有的关于脂肪酸对转录因子影响的研究,往往基于转录因子在核内的积累量来反映脂肪酸对其的影响,而忽略了其与DNA结合的动态过程。基于对不同家族转录因子DNA结合位点的大量研究所设计的转录因子结合元件可以有效地对转录因子进行富集[13],还原转录因子与DNA结合的动态过程。利用这种高效的转录因子富集的方法与深度覆盖的蛋白质组学技术相结合,本文首次全面地描绘了油酸引发的HepG2细胞脂肪变性前、后转录因子DNA结合活性的差异,并探讨了这些变化调控的生物过程。 1 材料与方法 1.1 主要材料
人肝癌HepG2细胞,购自北京协和细胞资源中心;油酸(oleic acid)、油红O均购自Sigma公司;牛血清白蛋白(BSA),购自北京欣经科生物技术有限公司;DMEM(dulbecco’s minimum essential medium)培养基,购自北京钮因华信科技发展有限公司;胎牛血清(FBS),购自Biowest公司;核蛋白提取试剂盒,购自Thermo公司;pGEX4T2-catTFRE由Sigma公司合成;Biotin标记的引物由华大基因公司合成;Dynal M280链霉亲和素磁珠,购自Invitrogen公司;乙腈和水,购自Avantor Performance Materials公司。 1.2 主要仪器
CO2恒温培养箱、LTQ-Orbitrap Velos质谱仪,均购自Thermo Scientific公司;光学显微镜及照相系统,购自奥林巴斯公司;SDS-PAGE凝胶电泳系统,购自Bio-Red公司。 1.3 方 法 1.3.1 HepG2细胞培养
传代HepG2细胞用含10%胎牛血清和1%青霉素、链霉素的DMEM培养基在37℃、5%CO2培养箱中培养。取对数生长期的细胞,在细胞融合度为80%时,换为无血清的DMEM培养基培养,6h后,实验组换液,用配制好的油酸继续培养24h,对照组换为含有1% BSA的完全培养基培养24h。油酸溶解在含1%BSA的完全培养基中,终浓度为2mmol/L,42℃,15min,以保证油酸充分溶解,之后用0.22μmol/L的滤膜过滤除菌后加入细胞,含1% BSA的完全培养基过滤除菌后作为对照使用[23, 26, 27]。 1.3.2 HepG2细胞脂肪变性鉴定
用油红O染色的方法进行HepG2细胞脂肪变性的鉴定。简单来说,将细胞用2%甲醛固定,之后用60%异丙醇洗涤,用新鲜配制的油红O溶液在37℃染色1h。最后,用蒸馏水洗涤并在光学显微镜下观察细胞内脂滴的形态[28]。 1.3.3 HepG2细胞核蛋白样品的制备
HepG2细胞核蛋白(NE)的提取使用Thermo公司的“NE-PER Nuclear and Cytoplasmic Extraction Reagents”试剂盒,按说明书提取。之后用Bradford法[29]测定核蛋白的浓度。 1.3.4 转录因子的富集
用富集分离内源转录因子及其复合物的方法[13]进行HepG2细胞的转录因子富集。简单来说,用Biotin标记的PCR引物,以构建好的pGEX4T2-catTFRE为模板,经PCR、琼脂糖凝胶电泳及胶回收后得到生物素标记的catTFRE。每组样品用4μg Biotin-TFRE 与80μl M280磁珠结合后,再与600μg的NE结合,洗去磁珠上非特异结合的蛋白质后,经过10% SDS-PAGE预分离和胶内酶解、肽段提取得到可以进行质谱分析的肽段样品,并将每组样品合并为3个组分进行干燥。 1.3.5 质谱鉴定和生物信息学分析
将每组样品的3个组分分别溶解于30μl溶液A中,12 000r/min,离心10min,取上清液进行第二次分离和质谱鉴定,每个组分上样量约为500ng。第二次分离为液-质连用质谱仪中的液相系统分离。溶液A:水-乙腈-甲酸(V∶V∶V = 98∶1.8∶0.2),溶液B:乙腈-水-甲酸(V∶V∶V = 98∶1.8∶0.2),pH 3。在液相分离的不同的洗脱时间,溶液A所占的比例A(A+B)为:95% (0min)、92%(5min)、77%(21min)、47%(55min)、37%(65min)、5%(75min),喷雾电压1.8kV,毛细管温度RT,碰撞能量为35%,碎裂模式:CID,一级采集质量范围:350~1 500Da。富集柱类型:自制,填料C18,填料直径5μm,柱内径100μm,长20mm,分离柱类型:自制,填料C18,填料直径3μm,柱内径 75μm,长120mm,流速:350nl/min。
搜索软件:Proteome Discoverer 1.4;搜索引擎:MASCOT;数据库:NCBI_ human Ref-sequence protein database (34 297种蛋白质,2013年7月更新)。一级误差:20ppm,二级误差:0.5Da。PSM(peptide-spectrum match)的假阳性率为1%的严格卡值或5%的卡值。
应用基于强度的绝对定量方法(intensity based absolute quantification,iBAQ),对鉴定到的蛋白质进行定量。其原理是利用每个蛋白质鉴定到的所有肽段的峰面积总和 (areas under the curve,AUCs)与该蛋白质理论酶解肽段数(n)的比值(AUCs/n)表征蛋白质的含量。依照两组间iBAQ 值之间的比值对蛋白质的表达量变化进行评定[30]。
对于catTFRE富集的转录因子,iBAQ值表征富集后的样品中每种蛋白质的含量。对于已注释的转录因子,认为不同组间,同种转录因子iBAQ值越大,DNA结合能力越强(既包括转录因子被活化导致DNA结合能力加强,也包括转录因子表达量增加引起的DNA结合能力加强的效果),反之亦然。
对于鉴定到的发生变化的转录因子,通过IPA(integrated pathway analysis)进行生物信息学分析。当Student t-检验P<0.05时,认为差异有统计学意义。 2 结 果 2.1 油酸诱导的HepG2细胞脂肪变性
油红O能使细胞内中性脂(主要是甘油三酯)着色[21]。因此,在实验组和对照组分别处理24h后,通过油红O染色的结果(图 1)可以明显的看出,2mmol/L的油酸使细胞内积聚了大量的甘油三酯。游离的油酸通过自由扩散或者在细胞膜表面载体的作用下进入细胞。进入细胞后的游离脂肪酸主要有三条去路:直接进入线粒体,通过氧化还原反应功能为细胞提供能量;与载脂蛋白耦合,形成极低密度脂蛋白分泌到胞外;或者经过酯化反应形成甘油三酯,并以脂滴的形式存在于细胞中[6]。这些由于饮食摄入过量脂类或者细胞内脂肪生成过于旺盛而造成的脂滴过量积累就导致了细胞发生脂肪病变。

|
| 图 1 HepG2细胞脂肪变性病理学鉴定 Fig. 1 Lipid droplets are accumulated in cells after oleic acid treatment Oil red O staining shows oleic acid treatment induces lipid accumulation in HepG2 cells |
对实验组和对照组的细胞核蛋白进行转录因子的富集后,用质谱鉴定对转录因子的富集情况[图 2(a)]。在转录组水平上对人类32种组织中转录因子深度覆盖的结果表明,每种组织中转录因子的数量为150~300种,肝脏的转录因子约为230种,约占肝脏所有基因的6%[12]。对于TFRE富集后的样品,采用SDS-PAGE进行预分离,经胶内酶解和肽段提取后进行质谱鉴定。每个样品分为3个组分进行质谱鉴定,以提高质谱的鉴定率。通过数据库搜索,共鉴定到1 290种蛋白质,其中有208种转录因子。所富集到的转录因子包含了bZIP(basic leucine zipper family)、HLH(basic helix-loop-helix family)、HOMEO(homeobox family)、NR(nuclear hormone receptors family)、HMG-box(high mobility group box family)、FOX(forkhead box family)及ZNF(zinc finger family)等各家族的转录因子[图 2(b)]。另外,转录因子在细胞内的表达丰度极低(仅占细胞总蛋白质量的0.01%~0.001%)[13],在实验中,每组鉴定到的转录因子的总量(iBAQ值)约占每组蛋白质总量的10%。 TFRE确实对转录因子有偏好地进行了有效富集。

|
| 图 2 TFRE pull-down/MS富集并鉴定转录因子流程及转录因子富集情况Fig. 2 Outline of TFRE pull-down/MS strategy and the enrichment of Transcription Factors by TFRE in HepG2 cells (a)Outline of TFRE pull-down/MS strategy (b)TFRE combines all families of TFs with preference |
对照组鉴定到转录因子170种,重复试验间共同鉴定到136种;实验组鉴定到转录因子190种,重复实验间共同鉴定到150种,实验重复性较好(图 3)。对照组与实验组共鉴定到208种转录因子,其中DNA结合活性加强(重复实验间iBAQ比值为0.5~2,且实验组与对照组的iBAQ比值大于2)的有67种,DNA结合活性减弱的(重复实验间iBAQ比值介于0.5~2,且实验组与对照组的iBAQ比值小于0.5)有34种。
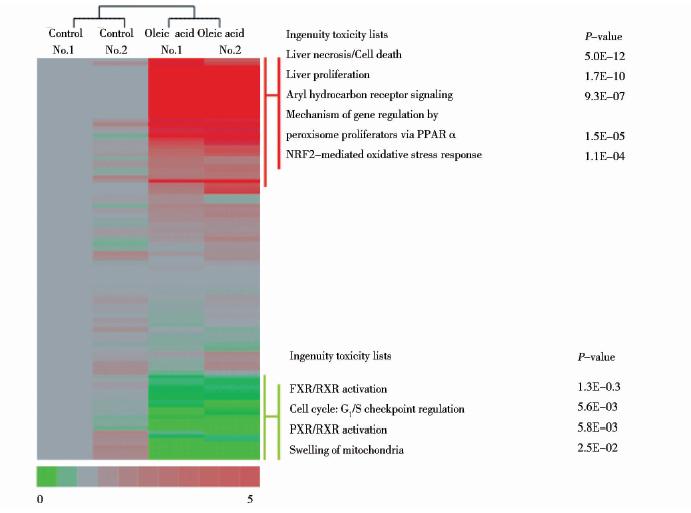
|
| 图 3 油酸诱导HepG2细胞脂肪变性后转录因子变化情况及IPA细胞毒性分析Fig. 3 The changes after oleic acid induced steatosis in HepG2 cells and the toxicity analysis by IPA |
在这些变化的转录因子中(表 1)可以看到MLX、MLXIPL(ChREBP)与DNA结合的数量是减少的,这与其他脂肪酸的影响是一致的。MLX、MLXIPL是被糖类物质激活的,从而促进糖酵解过程,将多余的糖类转化为脂肪酸。脂肪酸的摄入,减弱了这一过程,同时也减弱了对脂肪合成相关的酶FAS、ACC、SCD-1及GPAT的活化[17]。另外,NF-κB1与DNA结合的数量则是增加的。NF-κB1是NF-κB(有NF-κB1、P52、REL及P65组成)的DNA结合亚基,它与DNA结合活性的加强激活了NF-κB信号通路,促进了炎症反应的发生,这与饱和脂肪酸在非乙醇性脂肪肝患者中的影响是一致的[19],而且NF-κB活性的加强被认为是造成胰岛素耐受的原因之一[31, 32]。
 |
首先,为了明确油酸对HepG2细胞引起的毒性作用,利用IPA分别对DNA结合活性加强和减弱的转录因子进行了分析(图 3)。DNA结合活性加强的转录因子主要体现了油酸处理后加速了细胞的死亡及增殖过程;激活了NRF2介导的氧化应激反应;促进了依赖PPARα激活的基因的表达。在肝脏中,PPARα激活的基因往往与脂类的代谢和脂肪细胞的分化相关。NRF2介导的氧化应激反应的激活则是由于NFE2L2(NRF2)与抗氧化应答元件(DNA)结合活性加强(表 1),激活了谷胱甘肽巯基转移酶、细胞色素P450等抗氧化酶,从而缓解氧化损伤的途径。DNA结合活性减弱的转录因子则主要表现了与糖类、脂类代谢相关的途径的激活以及对细胞周期的调控(图 3)。
为了全面了解这些变化的转录因子(表 1)所起的作用,利用IPA对这些变化的转录因子的生物功能进行分析。在IPA中是通过倍数变化值(fold change)的正负来区分蛋白质、基因的上下调变化。因此,对于DNA结合活性加强的转录因子,其倍数变化以组间的iBAQ ratio表示;对于DNA结合活性减弱的转录因子,则定义fold change为(1/iBAQ ratio)。经分析,发现油酸导致的HepG2细胞脂肪变性促进了包括基因表达、细胞存活、细胞分化、细胞增殖等多种生物过程的发生[图 4(a)]。转录因子最直接的作用就是与RNA聚合酶Ⅱ一起,作用于靶基因的启动子区,控制下游基因的表达。例如,属于ATF/CREB家族的ATF1、ATF2、CREB1和CREB3L2,它们与CRE(cAMP-responsive element)结合活性的增加(表 1),促进了一系列基因的表达[33]。在油酸处理后的HepG2细胞中,由于FOXO1和FOXO3的DNA结合能力减弱,使其无法激活MYC的拮抗剂,MYC与DNA的结合能加强,加速了肿瘤的发展(progression of tumor)[34]。另外,在细胞处于异常环境,如大量的氧自由基存在的氧化压力或者细胞内存在大量的未折叠蛋白质时,XBP1在细胞存活与死亡的抉择中起到了关键作用[35]。XBP1[36, 37]、 HMGB1[38, 39]等促进细胞存活的转录因子的激活,以及NFAT5[40]活性的减弱,都促进了细胞的存活。在对这些变化的转录因子分析过程中还发现,油酸的摄入加速了G1/S检验点进行、促进DNA的复制,从而促进细胞周期的进程。通过这一系列过程,可减缓细胞脂肪变性带来不利影响,促进细胞的存活[图 4(b)]。
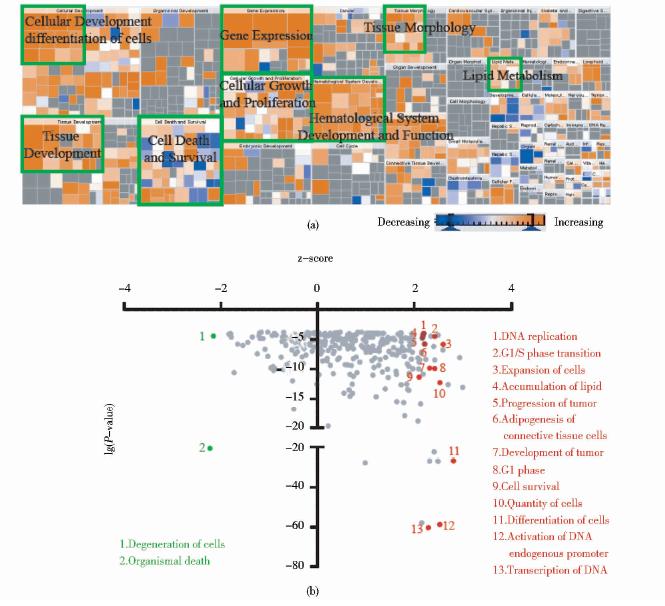
|
| 图 4 DNA结合活性变化的转录因子生物功能IPA分析Fig. 4 Biological function analysis of the altered TFs via IPA (a) The functional classification of altered TFs,the size means -lg(P-value) and p-values of all functions are less than 0.05; The brown means increase of functions and the blue means decrease (b) Cellular pathways responded to oleic acid treatment and the red plots means increased and the green means decreased |
多不饱和脂肪酸是人体必需的营养成分之一,除了为机体提供能量以外,也是细胞内膜结构的重要组成成分,同时也通过结合包括核受体在内的多种转录因子,直接或间接的调控基因的表达[20]。不同的脂肪酸对代谢、癌症的作用也越来越成为人们研究的热点。其中ω-3多不饱和脂肪酸在抑制炎症反应[18]、抑制肿瘤发生[41, 42]及促进肿瘤细胞凋亡方面表现突出,其在细胞内的作用已被广泛研究。
通过这些在油酸处理后变化的转录因子可以看出,作为不饱和脂肪酸,油酸与ω-3多不饱和脂肪酸的作用相反,对 NF-κB介导的炎症反应起到了激活的作用,这与饱和脂肪酸的作用相似[19];对人肝癌HepG2细胞的存活、增殖也起到了促进作用;同时在HepG2细胞内虽然会引起氧化应激反应,但造成的氧化损伤并不明显,这一点又与饱和脂肪酸不同。
另外,在IPA分析这些变化的转录因子的过程中,发现与血管再生(angiogenesis)及造血系统发育(development of hematopoietic system)相关的转录因子明显促进了这些进程的发生。油酸是否像ω-3多不饱和脂肪酸一样具有降低心血管疾病的危险还需要在合适的模型中验证。同时,DNA结合活性变化的转录因子明显促进了结缔组织的分化和发育,这在肝脏脂肪变性继续发展成为非乙醇性脂肪肝、脂肪性肝炎、肝硬化及肝癌的过程中是否起到了促进的作用也需要在动物模型中进行进一步的验证。
总之,油酸的摄入会引起HepG2细胞脂肪变性的发生,也引起了一批转录因子DNA结合活性的变化。转录因子控制着机体几乎所有的过程,无论是细胞周期调控、器官发育[43]还是代谢的平衡。利用catTFRE进行富集后,经过质谱鉴定,我们鉴定到208种转录因子,第一次全面的展现了油酸摄入导致的HepG2细胞发生脂肪变性后转录因子DNA结合活性的变化情况。这些变化的转录因子反映出油酸诱导的HepG2细胞脂肪变性,增加了细胞内脂类的积累,影响了糖代谢与脂代谢的平衡,激活了NF-κB介导的炎症反应,引起细胞氧化损伤,但同时也激活了细胞的自我保护反应,通过促进细胞周期的进程、促进细胞增殖、激活NRF2介导的氧化应激反应等维持细胞的生存。
| [1] | Evans R M,Barish G D, Wang Y X. PPARs and the complex journey to obesity. Nature Medicine,2004,10(4):355-361. |
| [2] | Schaffer J E. Lipotoxicity: when tissues overeat. Current Opinion in Lipidology,2003,14(3):281-287. |
| [3] | Adams L A,Angulo PLindor K D. Nonalcoholic fatty liver disease. Canadian Medical Association Journal,2005,172(7):899-905. |
| [4] | Adams L A,Lymp J F,St Sauver J, et al. The natural history of nonalcoholic fatty liver disease: a population-based cohort study. Gastroenterology,2005,129(1):113-121. |
| [5] | Crabb D W,Galli A,Fischer M, et al. Molecular mechanisms of alcoholic fatty liver: role of peroxisome proliferator-activated receptor alpha. Alcohol,2004,34(1):35-38. |
| [6] | Cohen J C,Horton J D,Hobbs H H. Human fatty liver disease: old questions and new insights. Science,2011,332(6037):1519-1523. |
| [7] | Sunny N E,Parks E J,Browning J D, et al. Excessive hepatic mitochondrial TCA cycle and gluconeogenesis in humans with nonalcoholic fatty liver disease. Cell Metabolism,2011,14(6):804-810. |
| [8] | Guo Y,Darshi M,Ma Y, et al. Quantitative proteomic and functional analysis of liver mitochondria from high fat diet (HFD) diabetic mice. Molecular & Cellular Proteomics,2013,12(12):3744-3758. |
| [9] | Blake W L,Clarke S D. Suppression of rat hepatic fatty acid synthase and S14 gene transcription by dietary polyunsaturated fat. The Journal of Nutrition,1990,120(12):1727-1729. |
| [10] | Jump D B,Clarke S D. Regulation of gene expression by dietary fat. Annu Rev Nutr,1999,19(1):63-90. |
| [11] | Jump D B,Clarke S D,MacDougald O, et al. Polyunsaturated fatty acids inhibit S14 gene transcription in rat liver and cultured hepatocytes. Proceedings of the National Academy of Sciences of the United States of America,1993,90(18):8454-8458. |
| [12] | Vaquerizas J M,Kummerfeld S K,Teichmann S A, et al. A census of human transcription factors: function, expression and evolution. Nature Reviews Genetics,2009,10(4):252-263. |
| [13] | Ding C,Chan D W,Liu W, et al. Proteome-wide profiling of activated transcription factors with a concatenated tandem array of transcription factor response elements. Proceedings of the National Academy of Sciences of the United States of America,2013,110(17):6771-6776. |
| [14] | Jump D B. Fatty acid regulation of hepatic lipid metabolism. Current Opinion in Clinical Nutrition and Metabolic Care,2011,14(2):115-120. |
| [15] | Ren B,Thelen A P,Peters J M, et al. Polyunsaturated fatty acid suppression of hepatic fatty acid synthase and S14 gene expression does not require peroxisome proliferator-activated receptor alpha. The Journal of Biological Chemistry,1997,272(43):26827-26832. |
| [16] | Wang Y,Botolin D,Xu J, et al. Regulation of hepatic fatty acid elongase and desaturase expression in diabetes and obesity. Journal of Lipid Research,2006,47(9):2028-2041. |
| [17] | Postic C,Girard J. Contribution of de novo fatty acid synthesis to hepatic steatosis and insulin resistance: lessons from genetically engineered mice. The Journal of Clinical Investigation,2008,118(3):829-838. |
| [18] | Lee J Y,Plakidas A,Lee W H, et al. Differential modulation of toll-like receptors by fatty acids preferential inhibition by n-3 polyunsaturated fatty acids. Journal of Lipid Research,2003,44(3):479-486. |
| [19] | Ribeiro P S,Cortez-Pinto H,Sola S, et al. Hepatocyte apoptosis, expression of death receptors, and activation of NF-kappaB in the liver of nonalcoholic and alcoholic steatohepatitis patients. The American Journal of Gastroenterology,2004,99(9):1708-1717. |
| [20] | Jump D B,Tripathy S,Depner C M. Fatty acid regulated transcription factors in the liver. Annual Review of Nutrition,2013,33(4):249-249. |
| [21] | Gu L Y,Qiu L W,Chen X F, et al. Oleic acid induced hepatic steatosis is coupled with downregulation of aquaporin 3 and upregulation of aquaporin 9 via activation of p38 signaling. Hormone and Metabolic Research,2014,13(3):125-129. |
| [22] | Hwang Y J,Wi H R,Kim H R, et al. Pinus densiflora Sieb. et Zucc. alleviates lipogenesis and oxidative stress during oleic acid-induced steatosis in HepG2 cells. Nutrients,2014,6(7):2956-2972. |
| [23] | Holzer R G,Park E J,Li N, et al. Saturated fatty acids induce c-Src clustering within membrane subdomains, leading to JNK activation. Cell,2011,147(1):173-184. |
| [24] | Listenberger L L,Han X,Lewis S E, et al. Triglyceride accumulation protects against fatty acid-induced lipotoxicity. Proceedings of the National Academy of Sciences,2003,100(6):3077-3082. |
| [25] | Wu H T,Chen W,Cheng K C, et al. Oleic acid activates peroxisome proliferator-activated receptor δ to compensate insulin resistance in steatotic cells. The Journal of Nutritional Biochemistry,2012,23(10):1264-1270. |
| [26] | Chen Y,Billadello J J,Schneider D J. Identification and localization of a fatty acid response region in the human plasminogen activator inhibitor-1 gene. Arteriosclerosis, Thrombosis, and Vascular Biology,2000,20(12):2696-2701. |
| [27] | Byon C H,Hardy R W,Ren C, et al. Free fatty acids enhance breast cancer cell migration through plasminogen activator inhibitor-1 and SMAD4. Laboratory Investigation; A Journal of Technical Methods and Pathology,2009,89(11):1221-1228. |
| [28] | Reyes-Quiroz M E,Alba G,Saenz J, et al. Oleic acid modulates mRNA expression of liver X receptor (LXR) and its target genes ABCA1 and SREBP1c in human neutrophils. European Journal of Nutrition,2014,7(3):1-11. |
| [29] | Zor T,Selinger Z. Linearization of the bradford protein assay increases its sensitivity: theoretical and experimental studies. Analytical Biochemistry,1996,236(2):302-308. |
| [30] | Schwanhausser B,Busse D,Li N, et al. Global quantification of mammalian gene expression control. Nature,2011,473(7347):337-342. |
| [31] | Arkan M C,Hevener A L,Greten F R, et al. IKK-beta links inflammation to obesity-induced insulin resistance. Nature Medicine,2005,11(2):191-198. |
| [32] | Cai D,Yuan M,Frantz D F, et al. Local and systemic insulin resistance resulting from hepatic activation of IKK-beta and NF-kappaB. Nature Medicine,2005,11(2):183-190. |
| [33] | Dietze R,Konrad L,Shihan M, et al. Cardiac glycoside ouabain induces activation of ATF-1 and StAR expression by interacting with the alpha4 isoform of the sodium pump in Sertoli cells. Biochimica et Biophysica Acta,2013,1833 (3):511-519. |
| [34] | Gan B,Lim C,Chu G, et al. FoxOs enforce a progression checkpoint to constrain mTORC1-activated renal tumorigenesis. Cancer cell,2010,18(5):472-484. |
| [35] | Lu M,Lawrence D A,Marsters S, et al. Opposing unfolded-protein-response signals converge on death receptor 5 to control apoptosis. Science,2014,345(6192):98-101. |
| [36] | Kaufman R J, Cao S. Inositol‐requiring 1/X‐box‐binding protein 1 is a regulatory hub that links endoplasmic reticulum homeostasis with innate immunity and metabolism. EMBO Molecular Medicine,2010,2(6):189-192. |
| [37] | Verfaillie T,Salazar M,Velasco G, et al. Linking ER stress to autophagy: potential implications for cancer therapy. International Journal of Cell Biology,2010,7(4):352-356. |
| [38] | Klune J R,Dhupar R,Cardinal J, et al. HMGB1: endogenous danger signaling. Molecular Medicine,2008,14(7):476-476. |
| [39] | Ohmori H,Luo Y,Kuniyasu H. Non-histone nuclear factor HMGB1 as a therapeutic target in colorectal cancer. Expert Opinion on Therapeutic Targets,2011,15(2):183-193. |
| [40] | Andres-Hernando A,Lanaspa M A,Rivard C J, et al. Nucleoporin 88 (Nup88) is regulated by hypertonic stress in kidney cells to retain the transcription factor tonicity enhancer-binding protein (TonEBP) in the nucleus. Journal of Biological Chemistry,2008,283(36):25082-25090. |
| [41] | Hofmanova J,Vaculova A,Kozubik A. Polyunsaturated fatty acids sensitize human colon adenocarcinoma HT-29 cells to death receptor-mediated apoptosis. Cancer Letters,2005,218(1):33-41. |
| [42] | Schley P D,Brindley D N,Field C J. (n-3) PUFA alter raft lipid composition and decrease epidermal growth factor receptor levels in lipid rafts of human breast cancer cells. The Journal of Nutrition,2007,137(3):548-553. |
| [43] | Kadonaga J T. Regulation of RNA polymerase II transcription by sequence-specific DNA binding factors. Cell,2004,116(2):247-257. |
 2015, Vol. 35
2015, Vol. 35




