文章信息
- 高文, 高向东, 陆小冬, 徐晨
- GAO Wen, GAO Xiang-dong, LU Xiao-dong, XU Chen
- 蛋白质层析柱复性及工艺评价
- Liquid Chromatographic Refolding of Proteins and Process Evaluation
- 中国生物工程杂志, 2015, 35(3): 84-91
- China Biotechnology, 2015, 35(3): 84-91
- http://dx.doi.org/10.13523/j.cb.20150312
-
文章历史
- 收稿日期:2014-12-09
- 修回日期:2015-01-27
2. 北京三元基因工程有限公司 北京 102600
2. Beijing Tri-Prime Genetic Engineering Incorporated, Beijing 102600, China
以大肠杆菌为代表的原核表达系统,因其具有遗传背景清楚、稳定并高效表达、操作方便和发酵成本低等优点,一直是重组蛋白药物研发及生产的首选表达系统。大部分高表达的重组蛋白在大肠杆菌菌体内以包涵体的形式存在,需要通过包涵体变性后复性,才可获得具有活性、可溶的天然构象蛋白。一般认为,变性蛋白暴露的疏水区通过疏水相互作用逐渐被包埋至结构内部,直至折叠为能量最小化的构象[1]。折叠过程中会形成一些不稳定的中间体结构[2],这些结构不稳定并且由于分子间疏水相互作用极易产生聚集[3],同时也存在二硫键错配、脯氨酸异构化等问题[4],影响复性得率及活性回收率。选择何种方法复性并如何优化复性条件一直是重组蛋白质领域的研究热点。传统复性工艺如稀释法、透析法已较成熟地被应用于制药生产,但普遍存在复性率低、处理量大、工艺流程长等弊端。近20年来,大部分复性研究都将重点放在新技术开发上(表 1),这些方法中层析柱复性最受重视。层析柱复性不受蛋白浓度的限制,较好地控制复性过程(如变性剂的去除),并同时完成初步的分离纯化,具备规模放大的可操作性。
| 复性方法 | 报道时间 | 用于复性研究的蛋白质 | 文献 |
| 稀释法 | 1954 | 核糖核酸酶RNase | [5] |
| 透析法 | 1979 | 乙酰辅酶A | [6] |
| 反胶团法 | 1990 | RNase-A | [7] |
| 疏水相互作用层析 | 1991 | 干扰素-γ | [8] |
| 凝胶过滤层析 | 1994 | 人结直肠癌组织转录调节因子Ets-1蛋白/RNase-A/大肠杆菌整合宿主因子IHF | [9] |
| 离子交换层析 | 1994 | 乳头状瘤病毒16型E7MS2融合蛋白 | [10] |
| 亲和层析 | 1994 | 硫氰酸酶/乳酸脱氢酶/微管蛋白 | [11, 12] |
| 双水相法 | 1994 | 碳酸酐酶II | [13] |
| 高压法 | 1998 | 硫氰酸酶 | [14] |
| 扩张床吸附法 | 2001 | 重组人生长激素/谷胱甘肽S转移酶片段 | [15] |
| 沸石法 | 2003 | DNA聚合酶 | [16] |
稀释复性是通过稀释的方法降低变性剂浓度使蛋白复性,其优势在于操作简便,是目前应用最为广泛的一种复性方法(图 1)。高浓度蛋白的复性过程中易发生聚集,因此稀释复性往往采用高稀释倍数、多次稀释的方式降低蛋白损失,研究表明利用50倍体积、两次稀释复性IL-2可使复性得率达到52%[17],但由于操作体积较大、蛋白浓度较低,为后续纯化增加了难度。
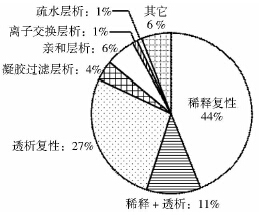
|
| 图 1 REFOLD数据库中不同复性方法应用比例的统计结果[17](http://refold.med.monash.edu.au/,总数据量1165,数据截止时间2012-01-09) Fig. 1 Analysis of data in REFOLD (http://refold.med.monash.edu.au/). The total amount of data is 1165(2012-01-09) and pie charts showing percentage breakdown of proteins entered in the database by refolding technique |
透析复性是一种利用透析膜内外的变性剂浓度差为驱动,降低透析袋内的变性剂浓度使蛋白复性的方法。为避免环境突变而引起聚集,一般采用多次透析的方式控制变性剂的去除速度,例如六次透析复性白介素IL-2可使得率达到50%[18],但整个透析过程耗时36h并消耗透析外液5L,相比于耗时24h并消耗100ml复性缓冲液的稀释复性方法,透析复性的工艺周期更长,试剂消耗量更大。
超滤复性不同于透析复性,以外加压力差作为驱动加速变性剂的去除,在高浓度蛋白条件下复性具有优势。Zhao等[19]采用恒体积超滤复性1.0mg/ml蛋白浓度的重组人粒细胞集落刺激因子rhG-CSF,得到51.0±2.6%的得率以及1.51±0.08×108IU/mg的比活,相比于同条件下的稀释复性方法分别提高了45%和200%以上。 2 柱层析复性
根据柱层析作用原理不同,可将柱层析分为两种:一种是分配型层析,即凝胶过滤层析;另一种是吸附型层析,即离子交换层析、亲和层析和疏水层析。 2.1 凝胶过滤层析复性
凝胶过滤层析复性方法将变性蛋白溶液通过一定孔径的凝胶固定相,不同大小的分子的流经体积不同,致使变性剂分子在柱中的停留时间长于蛋白分子,蛋白所处环境的变性剂浓度逐渐降低,自发地向热力学稳定[20]的构象状态复性。复性过程中蛋白构象逐渐变紧密,可以占有的孔体积增加,有效限制聚集,同时确保折叠的自主性而减少错误折叠的几率,而吸附型层析的介质对蛋白的较强吸附有可能会限制蛋白分子的柔韧性。有研究同时采用凝胶过滤层析(HPSEC)脲梯度洗脱和固定化金属离子亲和层析(Ni)两种方法复性重组人胰岛素原,得率分别为77.8%和56.8%,纯度分别为97.6%和93.7%,但仅凝胶过滤层析方法得到具有正确结构和活性的蛋白[21]。但由于高浓度变性蛋白在凝胶过滤层析柱顶端易产生聚集,限制复性规模。 2.2 离子交换层析复性
离子交换层析复性方法利用变性蛋白与固定相之间的静电吸附作用,将伸展的肽链吸附在介质上,在柱平衡或洗脱的过程中完成复性。Rane等[22]利用CM阳离子交换层析完成柱上复性、高盐洗脱骨形态发生蛋白单体BMP-2,得到26%的得率而纯度高于90%。离子交换层析复性处理量大且介质成本较低,可有效抑制聚集,同时静电作用以及离子环境有利于二硫键的形成,但静电作用可能会造成多点吸附,限制蛋白折叠的空间柔韧性。离子交换层析复性方法对变性条件有限制,例如变性的盐浓度过高会影响吸附效果,而盐酸胍会影响柱载量及分离效果。 2.3 亲和层析复性
亲和层析复性方法利用介质上配体与蛋白之间的特异性亲和作用,吸附变性蛋白并在柱上完成复性。亲和层析复性中应用最广泛的是固定化金属亲和层析复性,其吸附原理是基于蛋白组氨酸、半胱氨酸和色氨酸侧链与固定在介质上的过渡离子(如Cu2+、Ni2+、Zn2+等)的相互作用,目前主要针对具有多聚His标签的重组蛋白质的复性,Sharapova等[23]在α-甲胎蛋白的C末端引入7个His标签,利用Ni柱复性得到60%的得率和98%的纯度。固定化金属亲和层析复性方法不受强变性剂的影响,但其他变性添加剂如2-巯基乙醇、DTT等强还原剂、SDS等离子去污剂、甘氨酸或精氨酸会影响蛋白质的吸附,造成蛋白损失[24]。除固定化金属亲和层析复性以外,部分辅助复性添加剂如分子伴侣GroEL、二硫键形成蛋白A(DsbA)等[25],可固定在活化的凝胶介质上作为另一种亲和层析应用于蛋白质复性,有效提高复性效率以及添加剂的利用率。亲和层析复性方法的特异性强、得率高,但特殊配基的制备成本较高、容易失活,并且要求蛋白具有可识别的特殊结构,限制了它的应用。 2.4 疏水相互作用层析复性
疏水相互作用层析复性是利用蛋白疏水性氨基酸残基与固定相表面疏水配基之间的疏水相互作用,使蛋白疏水区被瞬间结合,降低蛋白聚集,有利于从疏水核心开始的折叠并在柱上完成复性。由于正确折叠与未折叠、错误折叠的结构疏水性不同,疏水层析复性可以得到高纯度的正确结构产物,规模放大仍可得到较高的得率和纯度。Wang等[26]利用疏水层析复性rhG-CSF,小规模实验可得到43%的得率和97%的纯度,而扩大规模至总蛋白量1.6g,其得率仍能达到36.9%且纯度为95.4%。由于蛋白质的复性是由一个涉及分子内疏水相互作用的自由能变化驱动的过程[27],因此固定相与蛋白分子的疏水结合可能会影响折叠过程中某些氨基酸序列间的疏水作用,从而导致错误的疏水区域暴露、形成错误折叠结构。疏水层析复性常采用高盐条件上样及平衡,因为高盐环境会增强疏水作用而有利于蛋白的吸附,但高盐环境同样会引起蛋白盐析、造成蛋白损失,因此疏水层析复性前需对蛋白的盐溶能力进行测试,避免堵塞层析柱。 3 复性策略 3.1 复性方法的选择
柱层析复性方法可以达到复性同时纯化的目的,而不同类型的柱层析复性对变性条件有不同的要求,下表 2总结了常用复性方法的特点,并以溶菌酶为例对不同复性方法的复性效果进行了比较。仅比较质量回收率,亲和层析复性和离子交换层析复性的效果最好,但这两种复性方法并不适用于常规变性方法溶解包涵体,即含有高浓度变性剂和强还原剂的变性蛋白。因此凝胶过滤层析复性方法一般联合稀释复性,离子交换层析和亲和层析复性方法可与稀释复性或透析复性配合使用,而疏水层析复性则可以直接结合常规方法变性的蛋白。复性方法的选择仍需参考目的蛋白的性质以及变性条件,同时可能还需考虑与后续纯化工艺设计的连贯性。
| 复性方法 | 条件 | 优点 | 缺点 | 文献报道1) | 质量 回收率 | 活性 回收率 | 文献 | |
| 稀释 | 低蛋白浓度 | 操作简便 | 易沉淀、得率低;体积大 | 4倍体积稀释 | 约50% | 80% | [28] | |
| 透析 | 低蛋白浓度;多次透析 | 控制折叠缓慢进行 | 易沉淀、得率低;耗时长 | 50倍体积透析液 | — | 约70% | [28] | |
| 凝胶过滤层析 | 低蛋白浓度 | 柱上不保留; 部分纯化 | 柱顶端易聚集 | Superdex 75 | 约70% | 84% | [29] | |
| 离子交换层析 | 无盐酸胍;低盐浓度 | 利于二硫键形成;部分纯化 | 多点吸附限制折叠 | SP Sepharose High Performance | 97% | 88% | [30] | |
| 亲和层析 | 含特殊结构; 无强还原剂 | 特异性强;部分纯化; 固定化分子可重复使用 | 介质成本高;配基易失活 | 固定化GroEL、DsbA/DsbC | 100% | — | [25] | |
| 疏水相互作用层析 | 高盐浓度吸附 | 抑制聚集;部分纯化; | 易错误折叠;需优化条件 | Poros PE | 85% | 86.30% | [31] | |
| 1)列举文献均为溶菌酶的复性研究,变复性样品为高纯度产品而非包涵体制备 1)The references mentioned in the table are researches in lysozyme refolding | ||||||||
蛋白的复性过程往往伴随着聚集、二硫键错配等竞争反应的发生,这些竞争反应是造成活性回收率低的直接原因。在复性工艺中使用不同类型的添加剂来进行辅助,可以有效抑制聚集、促进二硫键的形成,提高得率和活性回收率(表 3)。由于不同蛋白结构与性质的差异化,添加剂的种类和浓度对蛋白复性有不同程度的影响,因此对于添加剂的使用条件需要进行优化。Dechavanne等[32]通过高通量复性筛选,对复性添加剂非去污剂磺基甜菜碱NDSBs、乙醇胺、谷胱甘肽GSH/GSSG、Arg和DTT的浓度进行优化,将复性得率从3%提高到30%,纯度高于95%。
| 添加剂类型 | 常用添加剂 | 原理 | 文献 | |
| 小分子 | 变性剂 | 尿素、盐酸胍 | 低浓度增加蛋白结构柔韧性,干扰聚集 | [33, 34] |
| 氨基酸 | Arg | 胍基与色氨酸侧链反应;增加聚集体的溶解;加速氧化型折叠 | [33] | |
| 聚合物 | PEG | PEG结合熔球态中间体,限制聚集 | [34] | |
| 环糊精 | 配合去污剂使用,释放结合的蛋白 | [35] | ||
| 脂类 | 脂质体 | 结合蛋白疏水区,表面电荷和膜流动性促进折叠 | [36] | |
| 醇类 | 甘油、短链醇 | 增强疏水区结构的紧凑,促进折叠,不能抑制聚集和错误折叠 | [33] | |
| 糖 | 葡萄糖、蔗糖、海藻糖 | 稳定蛋白结构 | [33] | |
| 还原剂 | DTT | 游离巯基提供还原性环境 | [28] | |
| EDTA | 螯合金属离子,防止氧化 | [37] | ||
| 氧化还原对 | 半胱氨酸/胱氨酸、GSH/GSSG | 促进二硫键形成 | [34、38] | |
| 盐类离子 | Zn2+、Cu2+、Mg2+、SO42- | 增加离子强度,促进溶解;稳定折叠中间体 | [34、38] | |
| 表面活性剂 | 去污剂 | Triton X-100、CHAPS、CTAB | 与蛋白结合成复合物,降低分子间疏水作用 | [34] |
| 非去污剂 | NDSBs | 与还原剂协同作用 | [39] | |
| ATP依赖 | GroEL/GroES、DnaK、DnaJ | 结合未折叠和部分折叠肽链的疏水区 | [25] | |
| 分子伴侣 | 非ATP依赖 | DsbA、PDI | 催化二硫键的形成、重排 | [4、39] |
| PPI | 催化脯氨酸肽键的顺反异构反应 | [25] | ||
变性蛋白所处环境若发生突变,即短时间内变性剂浓度的大幅度变化,会迫使蛋白在短时间内形成更紧凑的结构,容易发生错误折叠和聚集[40]。对于吸附型柱层析复性方法,直接使用低浓度或无变性剂的缓冲液平衡或洗脱,蛋白产物中会同时存在错误折叠、聚集和正确复性三种结构,造成目的蛋白含量和活性损失。因此柱层析复性工艺中经常梯度降低变性剂浓度的方式平衡[22],使变性剂对蛋白结构的影响逐渐减弱,控制复性缓慢进行,提高目的蛋白得率和比活性。洗脱一般采用以下两种方式[41]:一、梯度降低变性剂浓度,蛋白在柱上完成复性后高盐洗脱;二、变性剂降低和盐(或咪唑)浓度增加的双梯度,使变性蛋白在吸附与解吸附的动态过程中完成复性。对于含有二硫键的蛋白,也常采用变性剂浓度和pH双梯度洗脱方式,pH的改变可以影响半胱氨酸附近的带电残基,促进二硫键的形成。Li等[30]采用SP离子交换层析方法复性溶菌酶,分别用无梯度、尿素梯度、pH梯度和尿素、pH双梯度四种不同的方式洗脱,产品的比活分别为2.5×104U/mg、3.2×104U/mg、3.5×104U/mg、4.3×104U/mg,因此双梯度洗脱方式的复性效果更好。 4 复性工艺评价
近十年以来,柱层析复性工艺被应用于重组蛋白药物的复性研究,而这种新的复性工艺的开发是否具有价值,取决于最终能否应用于规模化生产。一个新复性工艺的评价主要分三个方面(图 2):一、针对复性蛋白的定性分析,包括结构特性和生物学活性,以验证该工艺的有效性;二、针对复性效果的定量检测,以证明该工艺的优越性;三、针对规模放大能力的考察,以评价该工艺的适用性。
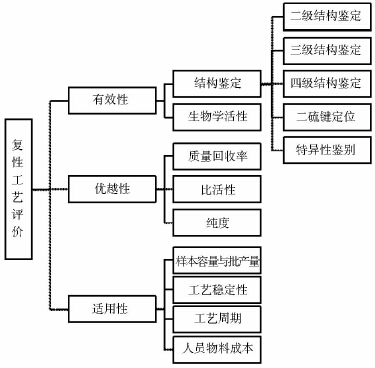
|
| 图 2 复性工艺评价流程图 Fig. 2 The refolding process evaluation procedures |
复性工艺的有效性主要考察蛋白复性结构的鉴定复性结构的分析方法有很多,根据检测目的不同可分为二级结构、三级结构和四级结构的鉴定,二硫键定位以及特异性鉴别(表 4)。
| 检测方法 | 检测目的 | 说明 | ||
| 紫外吸收 | 标准光谱法 | 二级结构和三级结构 | 色氨酸、酪氨酸等发色团结构具有不同的最大吸收波长和该波长下的摩尔吸收系数 | [42] |
| 差异光谱法 | 构象变化 | 推断蛋白在特定条件下的大致构象:芳香族残基是否暴露,处于极性还是非极性环境。 | [42] | |
| 圆二色性 | 远紫外 | 二级结构 | 180~240nm,肽键是主要发色团,估算二级结构(螺旋、折叠片和β转角)的含量。 | [22] |
| 近紫外 | 三级结构 | 240~340nm,色氨酸、酪氨酸和苯丙氨酸的芳香族残基 是主要的发色团。 | [43] | |
| 荧光光谱 | 荧光探针 | 二级结构和三级结构 | 内源性探针为色氨酸,外源性探针如硫代黄色素、1-苯氨基萘-8-磺酸。 | [44, 45] |
| 荧光偏振 | 四级结构和构象变化 | 与荧光探针结合用于测定疏水微区、多亚基蛋白质的缔合与解离。 | [42] | |
| 振动光谱 | 拉曼散射 | 二级结构 | 二级结构的定量估计;侧链构象。 | [43] |
| 红外吸收 | 二级结构 | 对于β片层的估计优于圆二色性 | [46] | |
| 高效液相色谱HPLC | 结构均一性 | 二体、寡聚体、聚集物、氧化氨基酸、脱氨基氨基酸以及其他微观结构不均一性。 | [38] | |
| 质谱MS | 不同层次结构和二硫键定位 | 折叠/去折叠三级构象,为NMR或X射线测定高分辨率的三级或四级结构提供依据。 | [21] | |
| 核磁共振NMR | 二级结构和三维结构 | 分子内动力学特性;模拟三维结构。 | [47] | |
| X射线衍射 | 三维结构 | 原子分辨率水平,构建完整3D模型。 | [48] | |
| 双向凝胶电泳 | 结构均一性和二硫键定位 | 第一向基于等电点不同,第二向按分子量不同;拖曳图像指示羟基化、二硫键等形成。 | [49] | |
| 免疫学分析 | ELISA | 特异性鉴别 | 重组蛋白作为抗原,复性构象的差异会导致其抗体的结合亲和力改变,可以较为准确的鉴定复性结构是否正确 | [45] |
| Western Blot | 特异性鉴别 | [18] | ||
复性工艺的优越性主要考察新工艺的质量回收率、比活性以及目的蛋白纯度,涉及总蛋白量、目的蛋白纯度、活性、目的蛋白含量以及纯度的检测。总蛋白含量测定的常用方法有Lowry法和Bradford法,目的蛋白纯度常用电泳或HPLC进行检测,用于目的蛋白含量的计算,也可以利用免疫学分析方法检测目的蛋白的含量。目的蛋白质量回收率的提高并不足以证明工艺的优越性,需参考比活性的变化。
某些重组蛋白的活性结构并不是单体形式,而是多聚体结构,如干扰素IFN-γ是由非晶体学二重轴连接的两个相同亚基构成的二聚体[50],因此在结构分析时需注意亚基和聚体的区别。重组人血管表皮生长因子VEGF165的活性结构也是二聚体结构,但不同于IFN-γ,VEGF165二聚体结构是借助二硫键联结形成,在亚基内以及两个亚基之间共形成16个二硫键[51],因此VEGF165的复性工艺评价不但要关注二聚体结构的含量,二硫键的定位也非常重要。 4.3 适用性评价
适用性评价主要是从工艺需求的角度考察复性工艺是否具备规模化生产的能力,例如样本容量(即层析复性工艺的最大载样量)与批产量,工艺稳定性,工艺周期的长短,以及成本问题包括人员、设备和物料的消耗。 5 展 望
到目前为止的研究成果都表明,层析柱复性法能有效抑制聚集,较好地控制复性缓冲环境的变化速度,并能分离变性剂和杂蛋白,得到较高的质量回收和活性回收。不同蛋白的复性工艺需要进行有针对性的优化,包括缓冲体系中的组分、pH及盐浓度等[36]。基础缓冲液组分的pKa直接关系到缓冲液的缓冲能力,而某些组分如Arg会影响蛋白复性的效果。对于含有二硫键的蛋白,由于半胱氨酸巯基的pKa为8.38,大部分复性都采用pH8~9.5[52, 53]的缓冲条件下进行,有利于二硫键与巯基之间的转换。而笔者进行了一系列有关疏水层析复性的缓冲条件筛选试验,发现pH在5.0~8.5范围内的疏水吸附效果最好,pH高于8.5时疏水作用降低而pH低于5.0时会增强蛋白的柱上保留。此外,硫酸铵等盐的加入会增强疏水层析的吸附效果,但高浓度的盐会造成蛋白的析出,因此是否引入缓冲体系外的盐以及盐的浓度需要根据质量回收和活性回收结果慎重考量。复性工艺的改进应避免引入具有潜在药理毒性的物质,并且需要考虑设计方案最终样品的后续工艺可行性,如缓冲环境的离子强度、添加剂等影响。
| [1] | Oliveberg M,Wolynes P G.The experimental survey of protein-folding energy landscape.Quarterly Reviews of Biophysics,2005,38(3):245-288. |
| [2] | Krishna M M, Lin Y, Mayne L,et al. Intimate view of a kinetic protein folding intermediate: residue-resolved structure, interactions, stability, folding and unfolding rates, homogeneity.Journal of Molecular Biology,2003,334(3):501-513. |
| [3] | Clark E D.Refolding of recombinant proteins.Current Opinion in Biotechnology,1998,9(2):157-163. |
| [4] | Hudson D A, Gannon S A,Thorpe C.Oxidative protein folding:From thiol–disulfide exchange reactions to the redox poise of the endoplasmic reticulum. Free Radical Biology and Medicine,2015,3:171-182. |
| [5] | Anfinsen C B,Redfield R R,Choate W L,et al.Studies on the gross structure,cross-linkages,and terminal sequences in ribonuclease.The Journal of Biological Chemistry,1954,207(1):201-210. |
| [6] | Frerman F E,Duncombe G R. Studies on the subunits of Escherichia coli coenzyme A transferase:reconstitution of an active enzyme.Biochimica et Biophysica Acta,1979,580(2):289-297. |
| [7] | Hagen A J,Hatton T A,Wang D I.Protein refolding in reversed micelles.Biotechnology and Bioengineering,1990,35(10):955-965. |
| [8] | 耿信笃,冯文科,常建华,等.用制备型高效疏水色谱复性和预分离重组人干扰素-gamma.高技术通讯,1991,1(7):1-4. Geng X D,Feng W K,Chang J H,et al.Refolding and pre-separation of recombinant human interferon-gamma using preparative high performance liquid chromatography. High Technology Letters,1991,1(7):1-4. |
| [9] | Werner M H,Clore G M,Gronenborn A M,et al.Refolding proteins by gel filtration chromatography.FEBS Letters,1994,345(2-3):125-130. |
| [10] | Suttnar J,Dyr J E,Hamsikova E,et al.Procedure for refolding and purification of recombinant proteins from Escherichia coli inclusion bodies using a strong anion exchanger.Jouranl of Chromatography B,1994,656(1):123-126. |
| [11] | Taguchi H,Makino Y,Yoshida M.Monomeric chaperonin-60 and its 50-kDa fragment posses the ability to interact with non-native proteins,to suppress aggregation,and to promote protein folding.The Journal of Biological Chemistry,1994,269(11):8529-8534. |
| [12] | Phadtare S,Fisher M T,Yarbrough L R. Refolding and release of tubulins by a functional immobilized groEL column. Biochimica et Biophysica Acta,1994,1208(1):189-192. |
| [13] | Forciniti D.Protein refolding using aqueous two-phase systems.Journal of Chromatography A,1994,668(1):95-100. |
| [14] | Gorovits B M,Horowitz P M.High hydrostatic pressure can reverse aggregation of protein folding intermediates and facilitate acquisition of native structure.Biochemistry,1998,37(17):6132-6135. |
| [15] | Cho T H,Ahn S J,Lee E K.Refolding of protein inclusion bodies directly from E.coli homogenate using expanded bed adsorption chromatography.Bioseparation,2001,10(4-5):189-196. |
| [16] | Chiku H,Matsui M,Murakami S,et al. Zeolites as new chromatographic carriers for proteins:easy recovery of proteins adsorbed on zeolites by polyethylene glycol.Analytical Biochemistry,2003,318(1):80-85. |
| [17] | Chow M K, Amin A A, Fulton K F, et al. The REFOLD database: a tool for the optimization of protein expression and refolding.Nucleic Acids Research,2006,34(Database issue):D207-D212. |
| [18] | Chen Z,Cui Y F,Leong Y A,et al.Efficient production of recombinant IL-21 proteins for pre-clinical studies by a two-step dilution refoldinong method.International Immunopharmacology,2013,16(3):376-381. |
| [19] | Zhao D W,Liu Y D,Wang Y J,et al.Membrane combined with hydrophilic macromolecules enhances protein refolding at high concentration.Process Biochemisty.2014,49(7):1129-1134. |
| [20] | Englander S W,Mayne L.The nature of protein folding pathways.Proceedings of the National Academy Sciences of the United States of America,2014,11(45):15873-15880. |
| [21] | Yuan J,Zhou H F,Yang Y C,et al.Refolding and simultaneous purification of recombinant human proinsulin from inclusion bodies on protein-folding liquid-chromatography columns.Biomedical Chromatography,2014,doi:10.1002/bmc.3358. |
| [22] | Rane A M,Jonnalagadda S,Li Z Y. On-column refolding of bone morphogenetic protein-2 using cation exchange resin.Protein Expression and Purification,2013,90(2):135-140. |
| [23] | Sharapova O A,Yurkova M S,Laurinavichyute D K,et al.Efficient refolding of a hydrophobic protein with multiple S-S bonds by on-resin immobilized metal affinity chromatography.Journal of Chromatography A,2011,1218(31):5115-5119. |
| [24] | Simpson R J.Purifying Proteins for Proteomics:A Laboratory Manual.New York:Cold Spring Harbor Laboratory Press,2004: 305,489-516. |
| [25] | Antonio-Perez A,Ramon-Luing L A,Ortega-Lopez J.Chromatographic refolding of rhodanese and lysozyme assisted by the GroEL apical domain,DsbA and DsbC immobilized on cellulose.Journal of Chromatography A,2012,1248:122-129. |
| [26] | Wang C Z,Geng X D.Refolding and purification of recombinant human granulocyte colony-stimulating factor using hydrophobic interaction chromatography at a large scale.Process Biochemistry,2012,47(12):2262-2266. |
| [27] | Englander S W,Mayne L.The nature of protein folding pathways.Proceedings of the National Academy Sciences of the United States of America,2014,11(45):15873-15880. |
| [28] | Chen Y C,Lin W Y,Wu K W,et al.Efficient lysozyme refolding at a high final concentration and a low dilution factor.Process Biochemistry,2012,47(12):1883-1888. |
| [29] | Wang S S,Chang C K,Peng M J,et al.Effect of glutathione redox system on lysozyme refolding in size exclusion chromatography.Food and Bioproducts Processing,2006,84(1):18-27. |
| [30] | Li M,Zhang G F,Su Z G.Dual gradient ion-exchange chromatography improved refolding yield of lysozyme.Journal of Chromatography A,2002,959(1-2):113-120. |
| [31] | Li J J,Liu Y D,Wang F W,et al.Hydrophobic interaction chromatography correctly refolding proteins assisted by glycerol and urea gradients.Journal of Chromatography A,2004,1061(2):193-199. |
| [32] | Dechavanne V,Barrillat N,Borlat F,et al. A high-throughput protein refolding screen in 96-well format combined with design of experiments to optimize the refolding conditions.Protein Expression and Purification,2011,75(2):192-203. |
| [33] | Yamaguchi H,Miyazaki M. Refolding techniques for recovering biologically active recombinant proteins from inclusion bodies.Biomolecules.2014,4(1):235-251. |
| [34] | Kuo M M,Nquyen P H,Jeon Y H,et al.MB109 as bioactive human bone morphogenetic protein-9 refolded and purified from E. coli inclusion bodies.Microbial Cell Factories.2014,13(1):29. |
| [35] | Han G J,Dong X Y,Sun Y. Purification effect of artificial chaperone in the refolding of recombinant ribonuclease A from inclusion bodies.Biochemical Engineering Journal.2013,77:15-19. |
| [36] | Kuboi R,Mawatari T,Yoshimoto M.Oxidative refolding of lysozyme assisted by negatively charged liposomes:relationship with lysozyme-mediated fusion of liposomes.Journal of Bioscience and Bioengineering.2000,90(1):14-19. |
| [37] | Ishida H,Garcia-Herrero A,Vogel H J.The periplasmic domain of Escherichia coli outer membrane protein A can undergo a localized temperature dependent structural transition.Biochimiaca et Biophysica Acta.2014,1838(12):3014-3024. |
| [38] | Xu J,Li J,Wu X,et al. Expression and refolding of bioactive α-bungarotoxin V31 in E. coli.Protein Expression and Purification.2015,doi:10.1016/j.pep. |
| [39] | Niforou K,Cheimonidou C,Trougakos I P.Molecular chaperones and proteostasis regulation during redox imbalance.Redox Biology.2014,2:323-332. |
| [40] | Tsumoto K,Ejima D,Kumagai I,et al.Practical considerations in refolding proteins from inclusion bodies.Protein Expression and Purification,2003,28(1):1-8. |
| [41] | Chen Y,Leong S S.Adsorptive refolding of a highly disulfide-bonded inclusion body protein using anion-exchange chromatography.Journal of Chromatography A.2009,1216(24):4877-4886. |
| [42] | 王静岩,朱胜庚,徐长法.生物化学.上册-3版.北京:高等教育出版社,2002:197-201. Wang J Y,Zhu S G,Xu C F.Biochemistry I.3rd ed.Beijing:Higher Education Press,2002:197-201. |
| [43] | Denslow N D,Wingfield P T,Rose K. Overview of the characterization of recombinant proteins.Current Protocols in Protein Science,2001,doi:10.1002/0471140864.ps0701s00. |
| [44] | Gull N,Mir M A,Khan J M,et al.Refolding of bovine serum albumin via artificial chaperone protocol using gemini surfactants.Journal of Colloid and Interface Science.2011,364(1):157-162. |
| [45] | Chura-Chambi R M,Nakajima E,Carvalho R R,et al.Refolding of the recombinant protein Sm29,a step toward the production of the vaccine candidate against schistosomiasis.Journal of Biotechnology,2013,168(4):511-519. |
| [46] | Linke T,Aspelund M T,Thompson C,et al. Development and scale-up of a commercial fed batch refolding process for an anti-CD22 two chain immunotoxin.Biotechnology Progress.2014,30(6):1380-1389. |
| [47] | Ogura K,Kobashigawa Y,Saio T,et al.Practical applications of hydrostatic pressure to refold proteins from inclusion bodies for NMR structural studies.Protein Engineering,Design & Selection.2013,25(6):409-416. |
| [48] | Barends T R,Foucar L,Botha S,et al. De novo protein crystal structure determination from X-ray free-electron laser data.Nature.2014,505(7482):244-247. |
| [49] | Gallagher S R. One-dimensional electrophoresis using nondenaturing conditions.Current Protocols in Protein Science,2001,doi:10.1002/0471142727.mb1002bs47. |
| [50] | Alsemgeest J,Old J M,Young L J.The macropod type 2 interferon gene shares important regulatory and functionally relevant regions with eutherian IFN-γ.Molecular Immunology,2015,63(2):297-304. |
| [51] | Pizarro S A,Gunson J,Field M J,et al.High-yield expression of human vascular endothelial growth factor VEGF165 in Escherichia coli and purification for therapeutic applications.Protein Expression and Purification,2010,72(2):184-193. |
| [52] | Coutard B,Danchin E G J,Oubelaid R,et al.Single pH buffer refolding screen for protein from inclusion bodies.Protein Expression and Purification.2012,82(2):352-359. |
| [53] | Dechavanne V,Barrillat N,Borlat F,et al.A high-throughput protein refolding screen in 96-well format combined with design of experiments to optimiza the refolding conditions.Protein Expression and Purification.2011,75(2):192-203. |
 2015, Vol. 35
2015, Vol. 35




