文章信息
- 申梁, 谭文杰
- SHEN Liang, TAN Wen-jie
- 冠状病毒反向遗传操作技术及其应用进展
- Progress on the Technique and its Application of Reverse Genetics for Coronaviruses
- 中国生物工程杂志, 2015, 35(2): 84-91
- China Biotechnology, 2015, 35(2): 84-91
- http://dx.doi.org/10.13523/j.cb.20150213
-
文章历史
- 收稿日期:2014-10-11
- 修回日期:2014-12-07
冠状病毒属于巢式病毒目(Nidovirales)、冠状病毒科、冠状病毒属,因在电子显微镜镜下观察到其病毒颗粒形似王冠而得名。国际病毒分类委员会(International Committee on Taxonomy of Viruses,ICTV)第九次报告中将冠状病毒分为α、β、γ以及新假定的δ共4个群[1]。其中α群冠状病毒主要包括人冠状病毒229E(HCoV-229E)、人冠状病毒NL63(HCoV-NL63)、犬冠状病毒(Canine coronavirus,CCoV)、猪流行性腹泻病毒(Porcine epidemic diarrhea virus,PEDV)、猪传染性胃肠炎病毒(Porcine transmissible gastroenteritis virus,TGEV)、猫传染性腹膜炎病毒(Feline infectious peritonitis virus,FIPV)以及一些蝙蝠类冠状病毒如长翼蝠冠状病毒HKU8(Miniopterus bat coronavirus HKU8,bat-CoV-HKU8)等。β群冠状病毒主要包括人冠状病毒OC43(HCoV-OC43)、HKU1(HCoV-HKU1)、SARS(Severe acute respiratory syndrome virus,SARS-CoV)以及新近发现的中东呼吸综合征冠状病毒(Middle east respiratory syndrome coronavirus,MERS-CoV)[2]等,还包括鼠肝炎病毒(Mouse hepatitis virus,MHV)和牛冠状病毒(Bovine coronavirus,BCoV)等。γ群冠状病毒主要包括禽传染性支气管炎病毒(Avian infectious bronchitis virus,IBV)和火鸡冠状病毒(Turkey coronavirus,TCoV)等。而新假定的δ群冠状病毒主要包括文鸟冠状病毒(Munia coronavirus,MuCoV)、鹦鹉冠状病毒(Parrot coronavirus,PaCoV)等。
冠状病毒粒子为多形态,略呈球形,成熟的冠状病毒直径为60~220 nm。病毒基因组为线性单股正链RNA,是迄今为止已发现的基因组最大的RNA病毒。病毒基因组全长在27~31 kb之间,基因组5′末端具有甲基化的帽状结构,其后是65~98个核苷酸的前导序列以及200~400个核苷酸的5′端非翻译区(Untranslated region,UTR),3′末端则包括200~500个核苷酸的3′端非翻译区(3′-UTR)和poly(A)尾巴(图 1)。其中5′端UTR和3′端UTR参与病毒基因组的转录和复制过程。病毒基因组RNA可作为mRNA,具有感染性。距5′端约2/3的基因组包含两个大的重叠开放阅读框(Open reading frame,ORF),分别是ORF1a和ORF1b,其中ORF1b的翻译依赖于一种特殊的核糖体移码翻译机制。这二者主要负责编码与病毒复制、转录相关的酶等非结构蛋白。余下约1/3的基因组负责编码刺突蛋白(Spike,S)、小膜蛋白(Small envelope membrane,E)、膜蛋白(Membrane,M)和核衣壳蛋白(Nucleocapsid,N)等主要结构蛋白,以及由嵌套在3′端基因组中的一系列亚基因组所编码的决定冠状病毒群特异性的蛋白[3,4]。
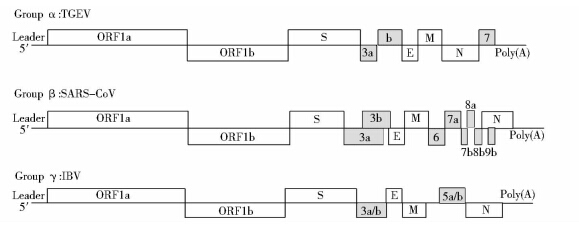
|
| 图 1 冠状病毒基因组结构示意图 Fig. 1 Schematic diagram of representative genomes from each of the coronavirus groups |
反向遗传学技术是通过构建病毒感染性分子克隆,在DNA水平上进行分子操作,从而研究病毒的结构与功能的方法。基本思路如下:首先,通过RT-PCR获得覆盖全长基因组的cDNA片段;其次,利用特殊的酶切位点将这些片段进行体外连接或依次克隆于携带启动子的载体,随后将此体外转录本或全长cDNA转染易感细胞,使其在细胞中完成病毒基因组的复制与装配,从而成功获得病毒。由于冠状病毒基因组较大,过去也没有合适的载体能够容纳如此庞大的基因组来获得病毒。因此冠状病毒基因组的研究一直局限于温度敏感突变株(Temperature sensitive mutants strain,Ts strain)[5]、缺陷病毒[6]以及利用靶向RNA同源重组技术构建的重组病毒,其中靶向RNA同源重组技术是最早用于冠状病毒反向遗传学研究的技术。后来随着细菌人工染色体(Bacterial Artificial Chromsome,BAC)、体外连接技术以及痘苗病毒载体的出现,冠状病毒反向遗传学研究获得了突飞猛进的发展。 2.1 靶向RNA同源重组技术
靶向RNA同源重组技术是最早建立的用于筛选获得重组冠状病毒的反向遗传操作技术。其原理是利用冠状病毒基因组对同源RNA序列具有极高同源重组效率的特点,将人工合成的覆盖冠状病毒基因组3′末端约10 kb大小的RNA电转已经感染了具有筛选能力的母本冠状病毒(Ts strain)细胞中,通过控制病毒培养温度,噬斑纯化等方式来获得重组病毒。利用该技术研究MHV温度敏感突变株时发现在MHV Ts株感染的细胞中,重组的频率可达到整个基因组的25%,并且重组频率从基因组5′末端到3′末端会逐渐增高[7,8,9]。
尽管靶向RNA同源重组技术在冠状病毒反向遗传学发展的进程中扮演着重要的角色,但是该技术存在很明显的缺点。比如不易对冠状病毒复制酶基因进行同源重组操作,筛选过程较为复杂等。因此,通过构建全长cDNA感染性克隆来获得冠状病毒是反向遗传学发展所必需的。 2.2 基于BAC载体的反向遗传学技术
最早有关冠状病毒全长cDNA感染性克隆的报道是在2000年,Almazán等[10]以BAC载体为基础构建出了TGEV的全长感染性克隆并且成功获得该病毒。构建策略是将TGEV全长cDNA 5′末端置于人类巨细胞病毒(Cytomegalovirus,CMV)启动子下,基因组cDNA 3′末端依次加入poly(A)尾巴、丁型肝炎核酶(Hepatitis delta virus,HDV)、牛生长激素BGH(Bovine growth hormone,BGH)终止信号,其中丁型肝炎核酶的作用是切割产生精确的病毒基因组3′末端。利用此载体,该研究者又在2006年成功获得了SARS-CoV[11]。随后,越来越多的冠状病毒利用BAC载体被成功筛选获得。如人冠状病毒OC43[12]、FIPV [13],以及最近爆发的MERS-CoV[14]。其中MERS-CoV病毒的获得与MERS-CoV疫情的爆发仅仅相隔4个月,这足以说明利用BAC作为冠状病毒载体的优越之处。另外,Pfefferle等[15]还对BAC载体进行了修饰,利用 T7启动子替换了CMV启动子,通过体外转录病毒基因组的方式同样成功地获得了SARS样冠状病毒Frankfurt-1株。
BAC载体是在大肠杆菌F质粒基础上构建的高容量、低拷贝载体。该载体用于筛选获得冠状病毒具有多种优点,不但能够装载较大的外源片段,在每个细菌细胞中仅存在1~2个拷贝,避免了冠状病毒中含有的毒性序列在拷贝数较高时导致基因组cDNA在质粒中出现不稳定现象;而且具有较高的转染效率和表达效率,使得病毒基因组RNA能在被转染细胞中迅速复制。最重要的是该载体操作方便,可在体外完成对病毒基因组的修饰和改造,加快了冠状病毒分子生物学研究的步伐。 2.3 基于体外连接的反向遗传操作技术
体外连接技术最早用于获得TGEV,是将覆盖TGEV全长基因组的cDNA亚克隆体外连接成完整的病毒全长cDNA[16]。其原理是在每个cDNA亚克隆两端分别引入BglI限制性内切酶位点,其切割序列为GCCNNNN↓NGGC。该酶是一种II型限制性内切酶,能够切割产生含有不同序列的3个碱基的黏性末端,只有黏性末端能够互补配对的cDNA片段才能连接成为完整的全长cDNA。体外连接的全长cDNA在5′末端有一个帽子结构,3′末端包含一个poly(A)尾巴,通过体外转录的方式合成病毒全长基因组RNA,然后与带有帽子结构的冠状病毒N基因转录物共同转染细胞获得病毒。虽然有研究报道利用该技术获得了重组病毒Bat-SARS-CoV(含有SARS-CoV RBD序列)[17]以及MERS-CoV[18]。但是利用BglI这种II型限制性内切酶的同时也带来了一些问题,比如需要在病毒全长基因组中引入一些无义突变甚至可能需要直接引入BglI识别位点来完成全长感染性克隆的体外连接。为了解决这一问题,Yount等[19]巧妙地利用IIS型限制性内切酶Esp31成功获得了MHV(图 2)。该限制性内切酶切割序列为CGTCTCN↓NNNN。与BglI限制性内切酶有所不同,该酶能在识别位点之外切开DNA,因此利用该酶不需要寻找合适的酶切位点即可随意操纵cDNA亚克隆的长度和位置。后来,这种“隐形克隆”策略也成功地运用于获得IBV[20,21]和HCoV-NL63[22]病毒。
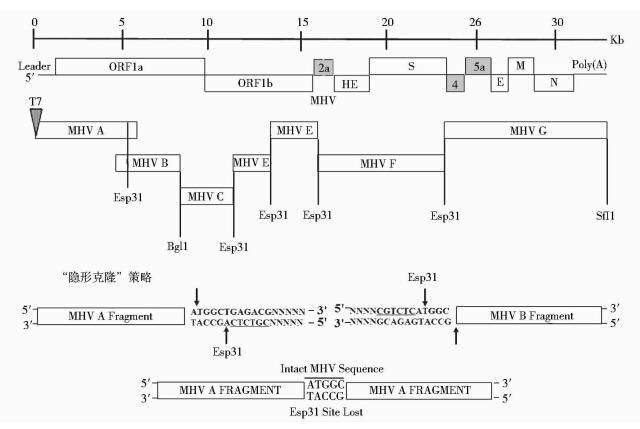
|
| 图 2 以体外连接方式构建小鼠肝炎病毒MHV感染性cDNA克隆 Fig. 2 Construction of a MHV infectious full-length cDNA clone by in vitro ligation |
通过体外连接cDNA亚克隆技术来获得冠状病毒具有一个明显的优点,即能够在最短的时间内获得新发病毒,不需要将所有片段分别连入载体来构建全长感染性克隆,并且便于在cDNA亚克隆上对病毒基因组任何位点进行操作。但是每次获得病毒都需要对所有亚克隆片段进行酶切、连接以及纯化全长cDNA,操作较为繁琐。 2.4 基于痘苗病毒载体的反向遗传操作技术
痘苗病毒是痘病毒科的典型成员,基因组大约187 kb,不具有感染性。痘苗病毒基因组中存在广泛的非必需区,可允许大片段外源基因的插入,且外源基因的插入不会影响到病毒复制,因此痘苗病毒载体是构建冠状病毒全长感染性克隆的理想选择。最早利用痘苗病毒载体获得了HCoV-229E[23],接着IBV[24]、MHV[25]、FIPV[26]、SARS-CoV[27]等冠状病毒也利用痘苗病毒载体相继获得。将冠状病毒全长cDNA克隆进痘苗病毒载体有两种方式,一种是构建出覆盖冠状病毒全长基因组的cDNA亚克隆,然后在体外连接成全长cDNA[全长cDNA两侧含有 T7启动子和poly(A)尾巴以及痘苗病毒特异DNA序列],接着将全长cDNA质粒转染痘苗病毒感染的细胞中,痘苗病毒DNA与质粒的同源序列发生同源重组,进而将外源基因整合入痘苗病毒的基因组中,最后利用体外转录本转染细胞来获得病毒;另一种是克隆载体,即由Merchlinsky等[28]构建的vNotI/tk载体,该载体含有单一的酶切位点,可用于外源片段的克隆。利用该载体可将体外连接获得的冠状病毒全长cDNA直接克隆至痘苗病毒基因组中,如Worm等[27]利用该载体获得了SAR样冠状病毒HKU-39849株。然而不管用哪种方式,N蛋白的表达均有利于冠状病毒的获得。 运用痘苗病毒作为冠状病毒感染性克隆的构建载体有两点优势[29]:一方面,痘苗病毒载体可以通过同源重组的方式插入体外连接的病毒全长cDNA,避免了构建携带部分基因组cDNA的中间载体过程。另一方面,可以利用痘苗病毒自身介导的高效的同源重组特性在细胞内对冠状病毒基因组cDNA进行修饰,修饰的原理主要是利用大肠杆菌鸟嘌呤磷酸核糖转移酶基因(guanine-phosphoribosyl transferase gene,gpt)所介导的阳性选择和阴性选择两次同源重组的过程。 3 反向遗传学技术在冠状病毒研究中的应用 3.1 在基因功能研究中的应用
冠状病毒反向遗传学研究技术的发展使得研究者可以通过病毒基因组的操作来快速定位病毒的致病基因、确定病毒的入侵机制和复制规律等。 3.1.1 致病基因的研究
Wathelet等[30]研究发现SARS-CoV的nsp1蛋白能够显著抑制IRF3和STAT1的磷酸化及表达,但是对STAT2、JAK1和TYK2的影响较小,利用反向遗传学技术突变nsp1上抑制干扰素的关键氨基酸后,虽然突变毒株与野生毒株在干扰素缺陷细胞中具有相似的复制动力学,但是在干扰素存在的情况下,突变毒株的复制明显被抑制,猜测nsp1是一个重要的毒力基因,在帮助SARS-CoV逃逸宿主天然免疫的过程中扮演着重要的角色。Frieman等[31]利用反向遗传学技术研究SARS-CoV小鼠适应株(MA15株)与其亲本毒株SARS-CoV(Urbani株)存在的6个氨基酸差异时发现:将Urbani株S蛋白和nsp9蛋白上的两个差异氨基酸同时突变为MA15株氨基酸序列后,该重组病毒对幼年BALB/c小鼠无致病性但对成年BALB/c小鼠具有致死性,证明了S蛋白和非结构蛋白nsp9与冠状病毒致病性相关。Pfefferle等[32]研究发现SARS-CoV(Frankfurt-1株)所编码的ORF7b蛋白基因中自然缺失的45个核苷酸对于该蛋白抑制I型干扰素和诱导细胞凋亡的功能无影响,但会造成病毒复制能力及对小鼠的致病性增强,证明了该附属蛋白与病毒致病性相关。而结构蛋白E比较特殊,缺失该蛋白后虽然对MHV和同群MERS-CoV的基因组复制都没有影响,但是只能获得MHV却不能获得具有感染性的MERS-CoV[14,33]。最近,Jimenez-Guardeo等[34]研究发现将SARS-CoV E蛋白的PDZ-binding motif(PDZ结合基序)删除后,激活促细胞活化蛋白激酶p38的能力和刺激机体产生炎性因子的能力与野生毒株相比明显下降,对小鼠肺部的致病性也明显降低,由此证明SARS-CoV E基因也是重要的致病基因。 3.1.2 病毒入侵机制的研究
Casais等[35]利用反向遗传学技术将致病性IBV(M41-CK株)的S基因替换到非致病性IBV(Beaudette株)后,所获得的嵌合病毒BeauR-M41组织嗜性发生了变化,证明了S蛋白是参与病毒入侵宿主细胞的主要受体识别蛋白。不同亚群的冠状病毒通过S蛋白与相应宿主细胞受体识别结合,进而入侵宿主。 3.1.3 病毒复制的研究
Almazán等[11]在 SARS-CoV(Urbani株)感染性克隆的基础上构建了该病毒的复制子,利用复制子研究发现参与病毒基因组RNA加工的核糖核酸外切酶基因(ExoN)、核糖核酸内切酶基因(NendoU)、2′-甲基转移酶基因(2′-O-MT)都是病毒基因组复制所必需的,缺失其中任何一个基因都会导致病毒基因组不能复制。Bouvet等[36]研究发现nsp10蛋白能够通过与nsp14蛋白的相互作用来调节nsp14蛋白的核酸外切酶活性,利用反向遗传学技术在nsp10中引入突变破坏其与nsp14的相互作用后发现,病毒复制能力下降且病毒噬斑变小。最近,Graham等[37]在缺失了nsp2基因后成功地获得了MHV,由此证明复制酶基因也具有一定可塑性。而针对附属蛋白,利用反向遗传学技术发现,它们往往不是病毒复制所必须,但是这些附属蛋白的缺失可能会引起病毒毒力减弱(易被机体免疫系统识别,持续感染能力下降等)[32,38]。 3.2 在研制新型疫苗中的作用
与传统疫苗相比,利用反向遗传学技术构建的标记疫苗具有显著的优点。首先,利用反向遗传学构建疫苗比以往传统的传代弱毒疫苗速度要快,在疫情爆发时可在短时间内获得相应疫苗;同时,在免疫动物后,还可以通过与之相配套的检测方法,将野毒感染和疫苗免疫区分开来。
DeDiego等[39]于2006年利用反向遗传学技术缺失了SARS样冠状病毒E蛋白的大部分序列,发现虽然E基因对于SARS样冠状病毒复制是非必需的,但E基因的缺失会使病毒对仓鼠的致病性减弱(肺部病变明显减轻,肺组织内病毒载量下降)。Netland等[40]、Fett等[41]在此研究理论的基础上删除了SARS样冠状病毒Urbani株及MA15株病毒完整的E基因,构建了能分别抵抗相应毒株攻击的候选疫苗。Cavanagh等[42]先将IBV(Beaudette株)的ORF3和ORF5缺失,又在此基础上将Beaudette株S基因替换为致病性IBV(M41株)S基因,将该重组病毒作为疫苗接种试验鸡,可诱导产生对抗M41株攻击的免疫保护。Ribes等[43]将冠状病毒MHV的S基因替换TGEV感染性克隆的S基因后,进一步将轮状病毒VP7基因插入TGEV病毒基因组,免疫小鼠后能对轮状病毒产生部分免疫保护,进一步说明冠状病毒具有作为载体用来构建二联苗的潜力。值得一提的是Graham等[44]创新性地将MAV核糖核酸外切酶基因(ExoN)失活后发现,突变病毒MHV-在感染严重联合免疫缺陷鼠(SCID mice)的过程中,基因组突变频率比正常毒株高15~20倍,但是ExoN基因能够维持稳定,而且对小鼠的致病性明显减弱。最近,Almazán等[14]又利用反向遗传学技术构建了缺失E基因的MERS-CoV感染性克隆,并与体外表达E蛋白的质粒共同转染Huh-7细胞,利用反式互补策略获得了仅能复制但不能增殖的MERS-CoV候选疫苗。 3.3 在筛选抗病毒药物中的应用
反向遗传学技术在筛选抗病毒抑制剂的过程中具有重要的作用,尤其是在建立高通量筛选方法中扮演着重要角色。对于致病性较低的病毒,如HCoV-NL63,则可以直接利用报告病毒来筛选抗病毒抑制剂;而对于高致病性的冠状病毒,如SARS-CoV,可以在含有报告基因的全长感染性克隆基础上进一步缺失结构蛋白而构建出复制子,通过复制子来进行抗病毒抑制剂筛选。Wang等[45]在缺失E基因的SARS-CoV感染性克隆的基础上,将EGFP基因替换了S基因,构建出了SARS-CoV复制子,并成功地利用丝氨酸蛋白酶抑制剂E-64D抑制了SARS-CoV的复制。Adedeji 等[46]发现化合物SSYA10-001能够通过抑制SARS-CoV解旋酶(nsp13)活性,进而抑制病毒复制。Carbajo-Lozoya等[47]则分别利用携带Rluc和EGFP基因的HCoV-229E病毒证明了抗生素FK506能够显著抑制该病毒的增殖。
与完整的报告病毒相比,利用复制子筛选病毒抑制剂具有一个明显的优势,即能够用来筛选高致病性病毒的抗病毒抑制剂。然而它也有缺陷:由于复制子缺失了结构蛋白,不能表达完整的基因,因此不能用来筛选那些可能对病毒吸附或者装配阶段具有抑制作用的抑制剂[48]。 4 结 语
反向遗传学技术为研究冠状病毒开辟了新途径,利用该技术可以了解病毒基因组的功能,可以对病毒复制及其致病机制进行探究,从而开发有效的抗病毒药物。此外,反向遗传学技术最具潜力的应用在于研发新型疫苗,可以通过删除毒力基因并同时引入分子标记在短时间内迅速研制出相应疫苗。虽然近年来分子病毒学发展迅速,但是对冠状病毒的研究仍不够深入。结构蛋白中M、E蛋白的研究还较少;从复制酶基因ORF1a和ORF1ab中切割下来的许多非结构蛋白功能还不太清楚;冠状病毒附属蛋白虽然是病毒复制非必需的,但是对于这些附属蛋白的具体功能研究还不够深入。
因此,未来冠状病毒的研究重点可能主要会集中在以下几个方面。冠状病毒编码的哪些蛋白能够抑制宿主的天然免疫以及这些蛋白作用的具体信号通路?冠状病毒自身编码蛋白间的相互作用及其对病毒自身复制的调控?能否研发出针对冠状病毒的广谱性药物?反向遗传学技术作为这些研究中的一种必不可少的工具,必定会发挥出巨大的作用!
| [1] | King A M, Adams M J, Carstens E B, et al.Virus taxonomy: classification and nomenclature of viruses: Ninth Report of the International Committee on Taxonomy of Viruses. San Diego, CA: Elsevier Academic Press, 2012. |
| [2] | Zaki A M, van Boheemen S, Bestebroer T M, et al. Isolation of a novel coronavirus from a man with pneumonia in Saudi Arabia. N Engl J Med, 2012, 367(19): 1814-1820. |
| [3] | Enjuanes L, Almazán F, Sola I, et al. Biochemical aspects of coronavirus replication and virus-host interaction. Annu Rev Microbiol, 2006, 60: 211-230. |
| [4] | Masters PS. The molecular biology of coronaviruses. Adv Virus Res, 2006, 48(6): 193-292. |
| [5] | Fu K, Baric R S. Map locations of mouse hepatitis virus temperature-sensitive mutants: confirmation of variable rates of recombination. J Virol, 1994, 68(11): 7458-7466. |
| [6] | Masters PS. Reverse genetics of the largest RNA viruses. Adv Virus Res, 1999, 53: 245-264. |
| [7] | Baric R S, Fu K S, Schaad M C, et al. Establishing a genetic recombination map for MHV-A59 complementation groups. Virology, 1990, 177(2): 646-656. |
| [8] | Kusters J G, Jager E J, Niesters H G, et al. Sequence evidence for RNA recombination in field isolates of avian coronavirus infectious bronchitis virus. Vaccine, 1990, 8(6): 605-608. |
| [9] | Makino S, Keck J G, Stohlman S A, et al. High-frequency RNA recombination of murine coronaviruses. J Virol, 1986, 57(3): 729-737. |
| [10] | Almazán F, Gonzalez J M, Penzes Z, et al. Engineering the largest RNA virus genome as an infectious bacterial artificial chromosome. Proc Natl Acad Sci USA, 2000, 97(10): 5516-5521. |
| [11] | Almazán F, DeDiego M L, Galan C, et al. Construction of a SARS-CoV infectious cDNA clone and a replicon to study coronavirus RNA synthesis. J Virol, 2006, 80(21): 10900-10906. |
| [12] | St-Jean J R, Desforges M, Almazán F, et al. Recovery of a neurovirulent human coronavirus OC43 from an infectious cDNA clone. J Virol, 2006, 80(7): 3670-3674. |
| [13] | Balint A, Farsang A, Zadori Z, et al. Molecular characterization of feline infectious peritonitis virus Strain DF-2 and studies of the role of ORF3 abc in viral cell tropism. J Virol, 2012, 86(11): 6258-6267. |
| [14] | Almazán F, DeDiego M L, Sola I, et al. Engineering a replication-competent, propagation-defective Middle East respiratory syndrome coronavirus as a vaccine candidate. mBio, 2013, 4(5): e00650-13. |
| [15] | Pfefferle S, Krahling V, Ditt V, et al. Reverse genetic characterization of the natural genomic deletion in SARS-coronavirus strain Frankfurt-1 open reading frame 7b reveals an attenuating function of the 7b protein in vitro and in vivo. Virol J, 2009, 6: 131. |
| [16] | Yount B, Curtis K M, Baric R S. Strategy for systematic assembly of large RNA and DNA genomes: transmissible gastroenteritis virus model. J Virol, 2000, 74(22): 10600-10611. |
| [17] | Becker M M, Graham R L, Donaldson E F, et al. Synthetic recombinant bat SARS-like coronavirus is infectious in cultured cells and in mice. Proc Natl Acad Sci USA, 2008, 105(50): 19944-19949. |
| [18] | Scobey T, Yount B L, Sims A C, et al. Reverse genetics with a full-length infectious cDNA of the Middle East respiratory syndrome coronavirus. Proc Natl Acad Sci USA, 2013, 110(40): 16157-16162. |
| [19] | Yount B, Denison M R, Weiss S R, et al. Systematic assembly of a full-length infectious cDNA of mouse hepatitis virus strain A59. J Virol, 2002, 76(21): 11065-11078. |
| [20] | Fang S, Chen B, Tay F P, et al. An arginine-to-proline mutationin a domain with undefined functions within the helicase protein (Nsp13) is lethal to the coronavirus infectious bronchitis virus in cultured cells. Virology, 2007, 358(1): 136-147. |
| [21] | Tan Y W, Fang S, Fan H, et al. Amino acid residues critical for RNA-binding in the N-terminal domain of the nucleocapsid protein are essential determinants for the infectivity of coronavirus in cultured cells. Nucleic Acids Res, 2006, 34(17): 4816-4825. |
| [22] | Donaldson E F, Yount B, Sims A C, et al. Systematic assembly of a full-length infectious clone of human coronavirus NL63. J Virol, 2008, 82(23): 11948-11957. |
| [23] | Thiel V, Herold J, Schelle B, et al. Infectious RNA transcribed in vitro from a cDNA copy of the human coronavirus genome cloned in vaccinia virus. J Gen Virol, 2001, 82(6): 1273-1281. |
| [24] | Casais R, Thiel V, Siddell S G, et al. Reverse genetics system for the avian coronavirus infectious bronchitis virus. J Virol, 2001, 75(24): 12359-12369. |
| [25] | Coley S E, Lavi E, Sawicki S G, et al. Recombinant mouse hepatitis virus strain A59 from cloned, full-length cDNA replicates to high titers in vitro and is fully pathogenic in vivo. J Virol, 2005, 79(5): 3097-3106. |
| [26] | Teke G, Hofmann-Lehmann R, Stallkamp I, et al. Genome organization and reverse genetic analysis of a type I feline coronavirus. J Virol, 2008, 82(4): 1851-1859. |
| [27] | van den Worm S H, Eriksson K K, Zevenhoven J C, et al. Reverse genetics of SARS-related coronavirus using vaccinia virus-based recombination. PLoS ONE, 2012, 7(3): e32857. |
| [28] | Merchlinsky M, Moss B. Introduction of foreign DNA into the vaccinia virus genome by in vitro ligation: recombination-independent selectable cloning vectors. Virology, 1992, 190(1): 522-526. |
| [29] | Carroll M W, Moss B. Poxviruses as expression vectors. Curr Opin Biotechnol, 1997, 8(5): 573-577. |
| [30] | Wathelet M G, Orr M, Frieman M B, et al. Severe acute respiratory syndrome coronavirus evades antiviral signaling: role of nsp1 and rational design of an attenuated strain. J Virol, 2007, 81(21): 11620-11633. |
| [31] | Frieman M, Yount B, Agnihothram S, et al. Molecular determinants of severe acute respiratory syndrome coronavirus pathogenesis and virulence in young and aged mouse models of human disease. J Virol, 2012, 86(2): 884-897. |
| [32] | Pfefferle S, Krähling V, Ditt V, et al. Reverse genetic characterization of the natural genomic deletion in SARS-Coronavirus strain Frankfurt-1 open reading frame 7b reveals an attenuating function of the 7b protein in vitro and in vivo. Virol J, 2009, 6: 131. |
| [33] | Kuo L, Masters PS. The small envelope protein E is not essential for murine coronavirus replication. J Virol, 2003, 77(8): 4597-4608. |
| [34] | Jimenez-Guardeño J M, Nieto-Torres J L, DeDiego M L, et al. The PDZ-binding motif of severe acute respiratory syndrome coronavirus envelope protein is a determinant of viral pathogenesis. PLoS Pathog, 2014, 10(8): e1004320. |
| [35] | Casais R, Dove B, Cavanagh D, et al. Recombinant avian infectious bronchitis virus expressing a heterologous spike gene demonstrates that the spike protein is a determinant of cell tropism. J Virol, 2003, 77(16): 9084-9089. |
| [36] | Bouvet M, Lugari A, Posthuma C C, et al. Coronavirus Nsp10, a critical co-factor for activation of multiple replicative enzymes. J Biol Chem, 2014, 289(37): 25783-25796. |
| [37] | Graham R L, Sims A C, Brockway S M, et al. The nsp2 replicase proteins of murine hepatitis virus and severe acute respiratory syndrome coronavirus are dispensable for viral replication. J Virol, 2005, 79(21): 13399-13411. |
| [38] | Cruz J L, Sola I, Becares M, et al. Coronavirus gene 7 counteracts host defenses and modulates virus virulence. PLoS Pathog, 2011, 7(6): e1002090. |
| [39] | DeDiego M L, Alvarez E, Almazán F, et al. A severe acute respiratory syndrome coronavirus that lacks the E gene is attenuated in vitro and in vivo.J Virol, 2007, 81(4):1701-1713. |
| [40] | Netland J, DeDiego M L, Zhao J, et al. Immunization with an attenuated severe acute respiratory syndrome coronavirus deleted in E protein protects against lethal respiratory disease. Virology, 2010, 399(1): 120-128. |
| [41] | Fett C, DeDiego M L, Regla-Nava J A, et al. Complete protection against severe acute respiratory syndrome coronavirus-mediated lethal respiratory disease in aged mice by immunization with a mouse-adapted virus lacking E protein. J Virol, 2013, 87(12): 6551-6559. |
| [42] | Cavanagh D, Casais R, Armesto M, et al. Manipulation of the infectious bronchitis coronavirus genome for vaccine development and analysis of the accessory proteins. Vaccine, 2007, 25(30): 5558-5562. |
| [43] | Ribes J M, Ortego J, Ceriani J, et al. Transmissible gastroenteritis virus (TGEV)-based vectors with engineered murine tropism express the rotavirus VP7 protein and immunize mice against rotavirus. Virology, 2011, 410(1): 107-118. |
| [44] | Graham R L, Becker M M, Eckerle L D, et al. A live, impaired-fidelity coronavirus vaccine protects in an aged, immunocompromised mouse model of lethal disease. Nat Med, 2012, 18(12): 1820-1826. |
| [45] | Wang J M, Wang L F, Shi Z L. Construction of a non-infectious SARS coronavirus replicon for application in drug screening and analysis of viral protein function. Biochem Biophys Res Commun, 2008, 374(1): 138-142. |
| [46] | Adedeji A O, Singh K, Calcaterra N E, et al. Severe acute respiratory syndrome coronavirus replication inhibitor that interferes with the nucleic acid unwinding of the viral helicase. Antimicrob Agents Chemother, 2012, 56(9): 4718-4728. |
| [47] | Carbajo-Lozoya J, Müller M A, Kallies S, et al. Replication of human coronaviruses SARS-CoV, HCoV-NL63 and HCoV-229E is inhibited by the drug FK506. Virus Res, 2012, 165(1): 112-117. |
| [48] | Kilianski A, Baker S C. Cell-based antiviral screening against coronaviruses: developing virus-specific and broad-spectrum inhibitors. Antiviral Res, 2014, 101: 105-112. |
 2015, Vol. 35
2015, Vol. 35




