文章信息
- 徐雪丽, 张伟, 刘艳, 谢丽萍, 胡又佳
- XU Xue-li, ZHANG Wei, LIU Yan, XIE Li-ping, HU You-jia
- 头孢菌素C酰化酶突变位点的研究
- Study on Mutations of Cephalosporin C Acylase
- 中国生物工程杂志, 2015, 35(2): 59-65
- China Biotechnology, 2015, 35(2): 59-65
- http://dx.doi.org/10.13523/j.cb.20150209
-
文章历史
- 收稿日期:2014-11-11
- 修回日期:2014-12-19
2. 上海医药工业研究院 上海 200437
2. Shanghai Institute of Pharmaceutical Industry, Shanghai 200437, China
由于头孢菌素类抗生素在治疗中、重症感染时,疗效显著且安全,所以临床上对它的需求量很大。7-氨基头孢烷酸(7-amino cephalosporanic acid,7-ACA)是医药工业生产半合成头孢菌素的重要中间体,一般由头孢菌素C(cephalosporin C CPC)通过化学法[1]或生物酶法裂解脱去侧链分子制备得到。生物酶法又可细分为两步酶法[2]和一步酶法[3]。
经过多年的发展,两步酶法生产7-ACA的生产工艺已经趋于成熟,并在工业上广泛应用。但还存在着一些不足,如:酶的制备相对复杂;反应过程中依然需要通入氧气,产生的副产物H2O2会造成产物分解和酶的失活;两个酶的最适催化条件不一致造成催化效率降低等[4]。因此,一些研究者将目光投向了一步酶法生产7-ACA。一步酶法(图 1)是利用头孢菌素C酰化酶(Cephalosporin C acylase)催化CPC使其直接脱去侧链(D-α-氨基己二酸)从而生成7-ACA的生产工艺。由于野生菌来源的CPC酰化酶的活性较低,不能满足工业催化的要求,人们希望通过基因工程及酶分子改造来提高它的催化效率。
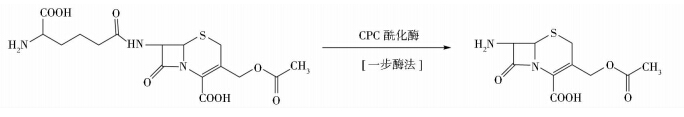
|
| 图 1 一步酶法生产7-ACA Fig. 1 One-step enzymatic conversion of CPC to 7-ACA |
1991年,Aramori等[5]克隆了来自Pseudomonas diminuta N176以及Pseudomonas diminuta V22中的CPC酰化酶的基因,并在大肠杆菌中分别进行了表达。 Saito等[6]进一步对N176来源的头孢菌素酰化酶进行了多处定点突变,这些突变体对戊二酰基-7-氨基头孢烷酸(GL-7-ACA)的活力都有所下降,其中突变酶M164L对CPC的活力提高了22%,M269Y和M269F的突变使其对CPC的比活提高了1.5和1.7倍[7]。Pollegioni等[8]通过对源于Pseudomonas diminuta N176的CPC酰化酶进行定向进化并结合定点突变,使A215Y/H296S/ H309S突变体的CPC/GL-7-ACA活力提高了约100倍,并表现出了比GL-7-ACA更高的CPC酶活,实现了底物特异性的转移,即获得了一种真正意义上的CPC酰化酶。Shin等[9]以提高酶对CPC的专一性为目的,对来源于Pseudomonas sp.SE83的酶进行了一系列的定点突变,最终得到的突变体S12:V122α A/G139αS/F58βN/ I75βT/I176βV/S471βL对CPC的比活为野生型的8.5倍(CPC酰化酶成熟后会发生自我切割,形成α、β亚基)。任羽等[10]对S12在不同原核系统中的表达情况进行了研究,发现pET-S12[BL21(DE3)]体系表达的酶的活力是pBC-S12(DH5α)体系表达的酶的15倍。本实验室在前期的工作中,就对Pseudomonas diminuta N176来源的基因进行了密码子优化,得到了ecs基因,再经易错PCR,筛选出的ECS50酶活力比ECS酶提高了35%[11]。
重叠延伸PCR技术[12]是使用具有互补末端的引物,使PCR产物形成了重叠链,从而在随后的扩增反应中通过重叠链的延伸,将不同来源的扩增片段重叠拼接起来。由于该技术操作简单,耗时短,成本低廉,且突变效率高,所以被广泛用于产生突变体分子,杂合基因及构建突变体库。王华等[13]就利用此技术构建了β-环糊精葡糖基转移酶基因定点突变的原核表达载体,成功地在该基因上插入了两个突变位点Y127F,R254F。
突变体S12是在1条序列上对6个位点同时进行突变,但这几个位点对酶活力提高所做的贡献是否一致,是否仅有其中某几个位点的突变就能达到同样的效果仍然未知。本实验利用重叠延伸PCR技术,在ecs50基因的基础上,针对这6个位点进行单一的突变,分析各个突变点对酶活力提高的作用。 1 材料与方法 1.1 材 料 1.1.1 菌株与质粒
克隆宿主菌E.coli DH5α、表达宿主菌E.coli BL21(DE3)、大肠杆菌表达载体pET-28a(+)、含ecs50基因的DH5α出发菌为本实验室保存。 1.1.2 酶和试剂
高保真酶Prime STAR Max Premix、限制性内切酶EcoRⅠ和SalⅠ,10×H Buffer,T4 DNA Ligase均购自TaKaRa公司;AxyPrep DNA凝胶回收试剂盒购自上海百赛生物;Ni-NTA树脂购自上海申能博彩公司;其他试剂均为分析纯。 1.1.3 仪器和标品
HPLC色谱仪为安捷伦科技有限公司。
CPC标准品和7-ACA标准品均由山西威奇达药业有限公司提供。 1.2 方 法 1.2.1 引物合成
根据突变体S12[9]的6个突变位点V122αA/G139αS/F58βN/I75βT/I176βV/S471βL,利用Vector NTI分别设计了6对突变位点的引物,突变位点分别位于ecs50基因的122,140,297,314,415,710位,其中FM和RM是用于引入突变氨基酸的引物序列(FM为正向序列,RM为反向序列),同时合成用于全长PCR的5′和3′引物。引物序列如表 1所示。
| Primer name | primer sequence(5′→3′) |
| ecs5′ | GCGGAATTCATGACCATGGCAGCTAATACGGATC |
| ecs3′ | ACTGTCGACTCAGTGGTGGTGGTGGTGGTG |
| FM122 | CCTACGCTGCCGGGGTGAACGCCTTC |
| RM122 | GAAGGCGTTCACCCCGGCAGCGTAGG |
| FM140 | GGAATACAGCCTGCTTGGTGCGGAACCG |
| RM140 | CGGTTCCGCACCAAGCAGGCTGTATTCC |
| FM297 | GGGCTTCCCAAGCAATGCCCATAACGGTAA |
| RM297 | TTACCGTTATGGGCATTGCTTGGGAAGCCC |
| FM314 | GCGCTTTTATGGATACCCACGATCTTTACCTGGAACAG |
| RM314 | CTGTTCCAGGTAAAGATCGTGGGTATCCATAAAAGCGC |
| FM415 | GGGTTGGTCGATCACAACCTCGTCGCC |
| RM415 | GGCGACGAGGTTGTGATCGACCAACCC |
| FM710 | GGCGCCTTATTACGGTACGTGTTCGATGTCGG |
| RM710 | CCGACATCGAACACGTACCGTAATAAGGCGCC |
| The underline sequences are the restriction endonuclease sites | |
以含ecs50基因的大肠杆菌DH5α的DNA为模板,ecs5′与RM混合成上游片段PCR的引物,FM与ecs3′混合成下游片段PCR的引物。利用高保真酶Prime STAR Max Premix进行突变位点上下游目的片段的PCR反应。PCR反应体系包括:模板2μl,上下游引物各1μl,酶25μl,加ddH2O至50μl。PCR反应条件:98℃预变性10s;98℃变性10s,55℃退火5s,72℃延伸速率约5s/kb,30个循环;72℃延伸10min。
用1%琼脂糖凝胶电泳检测PCR产物,分别切下各目的片段的胶条,采用速冻-快速离心法[14]进行回收:于-80℃冷冻30min,再于4℃、12 000g离心10min,吸取含目的片段的上清液。
重叠PCR时,取利用速冻-离心法所收集的含有每个突变位点的上下游PCR产物上清液各1μl混合,作为模板,其他条件同前,用1%的琼脂糖凝胶电泳检测PCR产物,切下目的片段胶条,用DNA试剂盒回收全长PCR产物。 1.2.3 突变目的片段的体外重组
将回收的各PCR全长片段用EcoRⅠ和SalⅠ进行双酶切,与经相同酶切的pET-28a(+)载体在T4连接酶作用下进行过夜连接,再转化大肠杆菌DH5α感受态细胞,涂布于含50μg/ml卡那霉素的LB平板上,37℃过夜培养。挑取单克隆培养,经质粒PCR和质粒双酶切验证为是目的转化子后,送上海睿迪生物科技有限公司测序。 1.2.4 CPC酰化酶的表达和纯化
将测序正确的突变体质粒转化至大肠杆菌E.coli BL21(DE3)感受态细胞,方法同转化DH5α感受态细胞。经验证得到正确的表达菌,对其进行诱导表达:将含有转化了重组表达载体的E.coli BL21(DE3)菌株接入含有50 μg/ml Kan 的LB 液体培养基中,37℃,220 r/min振荡培养过夜。第二天,按1%接种量将过夜培养的重组菌转接入装有250 ml LB培养基(Kan 终浓度50μg/ml)的750 ml摇瓶,37℃,220 r/min振荡培养至OD600=0.6后,加入IPTG(终浓度0.6mmol/L),于25度200r/min,培养20h。利用SDS电泳检测目的蛋白是否表达。 对各突变体的可溶蛋白利用金属螯合亲和层析法(参考申能博彩公司的使用说明),进行纯化,收集目的蛋白。 1.2.5 CPC酰化酶活性检测
对纯化获得的各个突变体进行酶活检测,CPC转化的反应体系:5mg/ml CPC钠盐(终浓度),0.5mg/ml纯酶(终浓度),以0.1 mol/L pH8.0的磷酸钠缓冲液补足至1ml。反应条件:120 r/min,37℃反应1h。用HPLC检测产物7-ACA的生成量,采用大连依利特公司Hypersil 2ODS,C18(5μm,4.6×250mm)柱,流动相为甲醇:0.2%(w/v)磷酸二氢钠(5∶95),检测波长为254nm,流速0.8ml/min,柱温为40℃,进样量20μl。 2 结 果 2.1 重叠延伸PCR
6个位点的突变是通过分别进行突变位点上下游PCR(图 2),产物回收后混合作为模板,再进行全长PCR(图 3),都得到了大小正确的目的片段。(仅以415位点为代表)
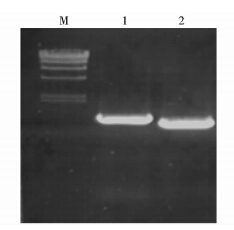
|
| 图 2 415位点上下游PCR Fig. 2 Up and downstream PCR of 415 M:HindⅢ digest;1:Upstream PCR products; 2:Downstream PCR products |
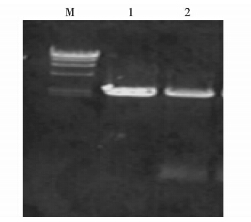
|
| 图 3 415全长PCR Fig. 3 Full-length PCR of 415 M:HindⅢ digest; 1:Positive control; 2:Full-length PCR products |
将全长片段与pET-28a(+)进行双酶切,分别回收后,连接,转化DH5α,挑取单菌落进行质粒PCR验证(图 4)和质粒双酶切验证(图 5),都得到了正确大小的条带:pET-28a(+)质粒5369bp,ecs50全长2349bp。再将验证正确的单菌落送测序,比对后,仅有目的位点发生突变(仅以415位点为代表)。我们用同样的方法获得了其他突变体,命名为122、140、 297、314、415、710(图 6)。

|
| 图 4 单菌落质粒PCR Fig. 4 Plasmid PCR of single colony M:HindⅢ digest; 1:Colony1; 2: Colony 2; 3: Colony 3; 4: Colony 4; 5: Positive control |
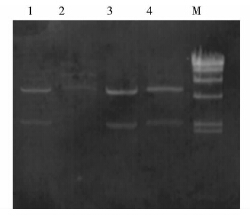
|
| 图 5 质粒双酶切 Fig. 5 Plasmid double digestion 1: Plasmid 1; 2: Plasmid 2; 3: Plasmid 3; 4: Plasmid 4; M:HindⅢ digest |
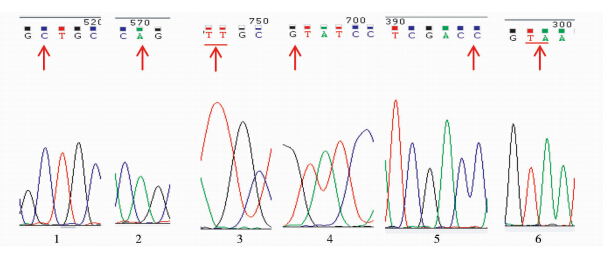
|
| 图 6 各突变序列突变位点测序峰图 Fig. 6 Sequence figures of mutation sites 1:122;2:140;3:297;4:314;5:415;6:710 |
500ml诱导表达培养物离心获得的菌体,按照天然金属螯合亲和层析法纯化带His-tag标签的可溶蛋白的方法纯化各突变体的可溶蛋白,各取20μl用于SDS-PAGE电泳检测(图 7),可见都有目的蛋白,两个亚基的大小正确,α亚基30.6kDa,β亚基58.3kDa。利用Brandford法测蛋白浓度,计算纯化的可溶蛋白量分别是:ecs50:5.6mg; 122:7.0mg; 140:4.4g; 297:0.5mg; 314:6mg; 415:4.2mg; 710:1mg。
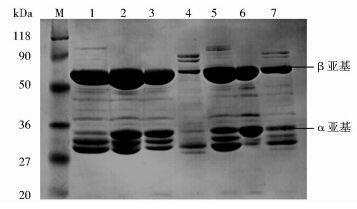
|
| 图 7 各突变体蛋白电泳 Fig. 7 The electrophoresis of each mutant protein M:Protein Marker;1:ecs50;2:122;3:140;4:297;5:314;6:415;7:710 |
利用上述纯化的酶进行活性检测,反应液经HPLC检测7-ACA的生成量,重复3次。酶活单位定义为在37℃,pH8.0时,每分钟生成1μmol的7-ACA所需的酶量。以出发菌的比活为100%,对比各个突变体酶活性变化,可见122的活性比ECS50高,其他的活性均不如ECS50(图 8)。122的比活达到106U/mg,ECS50的比活为86 U/mg。
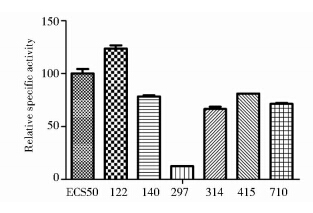
|
| 图 8 各突变体的相对比活力 Fig. 8 Relative specific activity of the mutants |
利用一步酶法直接将CPC转化成7-ACA具有诱人的前景,近年来,随着蛋白质工程的研究,CPC酰化酶的活性也有所提高。安明等[15]通过人工设计,对CPC酰化酶基因分两段进行了全局优化设计,包括了密码子替换、鸟嘌呤和胞嘧啶含量调整以及酶切位点修改等,获得目的基因,克隆并诱导表达,最适条件下的酶活力为2956U/L。Conti等[16]结合结构学知识,半理性设计,计算方法学和进化学分析等,得到的突变体H57βS-H70βS-L154βY具有很高的转化率,可能适合于工业化应用。
本实验所用的模板基因ecs50是来源于Pseudomonas sp. N176的经易错PCR获得的180位氨基酸发生突变的基因[11],本文利用重叠延伸PCR技术,顺利地得到了一系列定点突变体,也证明了重叠延伸PCR在定点突变上的有效性和可行性,本实验所用的方法可以为其他定点突变提供一定的借鉴。而且180位点氨基酸突变后,酶的活性高于最初的酶,可见180位点对该酶的活性影响很大,也就是说180有可能是该酶的活性位点。
Pollegioni等[8]对Pseudomonas sp. N176的CPC酰化酶的定向进化进行了详细的研究。通过易错PCR,发现了突变体F270M,A215Y的活性是未突变前的两倍;又通过定点饱和突变发现突变体H296S,H309S的活性也提高了2~3倍;再对不同的突变体进行不同的组合,发现A215Y-H296S-H309S对CPC的特异性显著提高。同时,文中还指出了靠近活性中心的一些位点,包括了H57β,V68β,T69β,H70β等。
N176和SE83的氨基酸序列存在90%以上的同源性,它们的催化机理也类似[17]。本研究在ECS50基础上对S12中的6个突变位点分别进行单一突变,发现只有V122A的活性有所提高,其他的活性都降低,其中F297N几乎没有活性。对比本研究所用的ECS50与专利报道的S12的序列,其DNA同源性为75.5%,氨基酸同源性为91.3%。但是S12序列在α亚基末端加入终止密码子,β亚基前面再加入起始密码子,使得这两个亚基可以分开表达,而不需要在形成总蛋白后经过自身的两次切割,断裂中间的连接肽来得到α、β亚基,而本研究中的ECS50序列并未做此改造。利用在线蛋白结构预测软件SWISS-MODEL进行ECS50蛋白空间结构的同源模拟,发现122和140处于α螺旋中,297和314处于β折叠中,415和710则位于无规卷曲中。从Xiao等[18]利用Discovery studio 2.1对SE83 acyⅡ进行的空间结构模拟中,也可以看出,122和140离活性中心Sβ1较远,处于底物进入通道上,其他4个位点都离活性中心Sβ1较近;另外还存在三个其他活性位点H57β,V68β,H70β,这与N176来源的酰化酶是一样的。122位由缬氨酸变成丙氨酸后,由于丙氨酸侧链比缬氨酸侧链所占空间小,这可能使得底物更容易进入酶的活性中心。而原先较大的缬氨酸侧链会限制周围其他氨基酸残基的侧链空间取向,变成丙氨酸后可能使酶蛋白处于更利于底物-酶结合的构象状态。因此122位点突变后,酶的活性提高了23.26%。140位由甘氨酸变成丝氨酸后,有利于形成氢键,但是由于140位离蛋白的活性中心较远,对稳定CPC-酶复合物所起的作用不大,所以其突变后对活性变化的影响不大。同样,314,415和710这三个位置的氨基酸单独对其中一个进行突变,虽然氨基酸残基侧链所占的空间会变小,但由此引起的酶活变化也不是很大。而靠近活性中心的297位由苯丙氨酸突变为天冬酰胺后,其侧链的空间取向可能不利于底物与酶的稳定结合,从而使酶活明显降低。也可能是297与296位点距离近,297的氨基酸发生突变后,会直接影响到296位在酶促反应中起作用,导致酶活下降。另外参考文献[16]中构建的系统进化树中也指出297位氨基酸是该类酶的保守氨基酸,对其进行突变很可能会造成酶活降低。而且从图 4的蛋白电泳图可以看出,F297N的可溶蛋白量很少,从侧面证明了297位氨基酸对于该酶活性影响很大。
另外,Satito等对N176的改造,发现M269Y和M269F的突变分别使CPC的比活提高了1.5和1.7倍[7],Wang等[19]对H309进行了不同的突变,得到的H309L的活性与未突变前相近,H309S,H309V的活性均不如原始酶,说明突变的氨基酸不同,酶活性也不同。而且,笔者在实验中试着将F297和S710变成其它的氨基酸,得到了突变体F297C、S710A,虽然它们的活性没有多大变化,但在相同培养条件下,可溶蛋白的表达量明显提高。因此可以考虑对122、140、297、314、415、710等位点进行定点饱和突变,或许从中可以发现酶活提高或者表达量提高的突变体。
虽然单独进行某个位点氨基酸突变引起的酶活变化不是很大,但是几个位点同时突变后,可能会有加强效应,进而使酶的活性得到提高,如S12对6个位点组合突变后所得到的酶的活性远高于原始酶;Xiao等[18]发现突变体H57βA/H70βY不存产物抑制现象,突变体H57βA/H70βY/I176βN的催化效率为原始酶的3.26倍;Li等[20]发现H57βS/H70βS/Mα165S的催化效率反而不如H57βS/H70βS。所以不同突变的不同组合,得到的酶的特性是不一样的,可能会提高酶的活性,或是降低反应中底物、产物的抑制效应,或是影响酶与底物之间氢键的形成,那么在后续改造中,可以利用DNA Shuffling技术,将122,140,297,314,415和710这些突变体作为模板进行改造,一次即可得到不同形式的突变体组合,可能会从中发现酶活更高的突变体。
| [1] | Fechtig B, Peter H, Bickel H, et al. Concerning the preparation of 7-amino-cephalosporanic acid. Helvetica Chimica Acta, 1968, 51(5):1108-1119. |
| [2] | Conlon H D, Baqai J, Baker K, et al. Two-step immobilized enzyme conversion of cephalosporin C to 7-aminocephalosporanic acid. Biotechnology and Bioengineering, 1995, 46(6):510-513. |
| [3] | Fritz-Wolf K, Koller K P, Lange G, et al. Structure-based prediction of modifications in glutarylamidase to allow single-step enzymatic production of 7-aminocephalosporanic acid from cephalosporin C. Protein Science, 2002, 11(1):92-103. |
| [4] | Lopez-Gallego F, Batencor L, Hidalgo A, et al. One-pot conversion of cephalosporin C to 7-aminocephalosporanic acid in the absence of hydrogen peroxide. Advanced Synthesis & Catalysis, 2005, 347(14):1804-1810. |
| [5] | Aramori I,Fukagawa M,Tsumura M,et al.Isolation of soil strains producing new cephalesporin acylases.Journal of Fermentation and Bioengineering,1991,72(4):227-231. |
| [6] | Saito Y, Fujimura T, Ishii Y,et al. Saito Y F, Ishii Y,et al. Oxidative modification of acephalosporin C acylase from Pseudomonas strain N176 and site-directed mutagenesis of the gene. Applied and Environment Microbiology, 1996, 62(8):2919-2925. |
| [7] | Ishii Y,Satio Y, Fujimura T,et al.High-level production, chemical modification and site-directed mutagenesis of a cephalosporin C acylase from Pseudomonas strain N176. European Journal of Biochemistry, 1995,6, 230(2):773-778. |
| [8] | Pollegioni L, Lorenzi S, Rosini E, et al. Evolution of an acylase active on cephalosporin C. Protein Science, 2005, 14(12):3064-3076. |
| [9] | Shin Y C, Jeon J Y, Jung K H, et al. Cephalosporin C acylase mutant and method for preparing 7-ACA using same.US Patent 0,207,519,2007. |
| [10] | 任羽,张建安, 朱玉山,等. 头孢菌素C 酰化酶S12 在不同原核系统中的表达. 科技创新导报, 2012,12:226-228. Ren Y, Zhang J A, Zhu Y S,et al. Expression of cephalosporin C acylase S12 in different prokaryotic systems. Science and Technology Innovation Herald, 2012,12:226-228. |
| [11] | 王颖秋,郑林冲,谢丽萍等. 基于易错 PCR 的头孢菌素 C 酰化酶的定向进化. 中国医药工业杂志, 2013, 44(4):344-347. Wang Y Q, Zheng L Q, Xie L P,et al. Directed evolution of cephalosporin C acylase activity by Error-prone PCR.Chinese Journal of Pharmaceuticals, 2013, 44(4):344-347. |
| [12] | Horton R M, Cai Z L, Ho S N, et al. Gene splicing by overlap extension: tailor-made genes using the polymerase chain reaction. BioTechniques, 1990, 8(5):528-535. |
| [13] | 王华,文一, 于寒松,等. 重叠延伸PCR技术构建β-环糊精葡糖基转移酶基因的定点突变原核表达载体. 食品工业科技, 2013, 19(34):145-151. Wang H,Wen Y, Yu H S,et al. Site-directed mutagenesis of β-CGTase and expression vector construction by overlap extension PCR. Science and Technology of Food Industry, 2013, 19(34):145-151. |
| [14] | 王 春,陈琳玲, 许灿新,等. 简便快速的PCR产物回收方法. 南华大学学报, 2005,33(1):109-111. Wang C,Chen L L,Xu C X,et al. Comparison of several methods of extracting PCR products from Agarose Gel. Journal of Nanhua University(Medical Edition), 2005,33(1):109-111. |
| [15] | 安明,于慧敏,罗晖,等. CPC酰化酶基因的人工合成与重组表达. 清华大学学报(自然科学版), 2008, 9:1499-1503. An M, Yu H M,Luo H,et al. Artificial synthesis and expression of the CPC acylase gene in recombinant E. coli.Journal of Tsinghua Univesity(Science and Technology), 2008, 9:1499-1503. |
| [16] | Conti G, Pollegioni L, Molla G, et al. Strategic manipulation of an industrial biocatalyst--evolution of a cephalosporin C acylase. The FEBS Journal, 2014, 281(10):2443-2455. |
| [17] | Li Y, Chen J, Jiang W, et al. In vivo post-translational processing and subunit reconstitution of cephalosporin acylase from Pseudomonas sp. 130. European Journal of Biochemistry/FEBS, 1999, 262(3):713-719. |
| [18] | Xiao Y, Huo X, Qian Y, et al. Engineering of a CPC acylase using a facile pH indicator assay. Journal of Industrial Microbiology & Biotechnology, 2014,41(11):1617-1625. |
| [19] | Wang Y, Yu H, Song W, et al. Overexpression of synthesized cephalosporin C acylase containing mutations in the substrate transport tunnel. Journal of Bioscience and Bioengineering, 2012, 113(1):36-41. |
| [20] | Li Q, Huang X, Zhu Y. Evaluation of active designs of cephalosporin C acylase by molecular dynamics simulation and molecular docking. Journal of Molecular Modeling, 2014, 20(7):2314. |
 2015, Vol. 35
2015, Vol. 35




