扩展功能
文章信息
- 饶华祥, 于娟, 李寿江, 宋秀平, 栗冬梅
- RAO Hua-xiang, YU Juan, LI Shou-jiang, SONG Xiu-ping, LI Dong-mei
- 青藏高原麦秀国家森林公园小型哺乳动物巴尔通体基因多态性研究
- Gene polymorphisms of Bartonella species in small mammals in Maixiu National Forest Park in the Qinghai-Tibet Plateau, China
- 中国媒介生物学及控制杂志, 2021, 32(4): 398-403
- Chin J Vector Biol & Control, 2021, 32(4): 398-403
- 10.11853/j.issn.1003.8280.2021.04.003
-
文章历史
- 收稿日期: 2021-03-02
2 长治医学院基础医学部, 山西 长治 046000;
3 青海省疾病预防控制中心, 青海西宁 810007;
4 中国疾病预防控制中心传染病预防控制所, 传染病预防控制 国家重点实验室, 感染性疾病诊治协同创新中心, 北京 102206
2 Department of Basic Medical Sciences, Changzhi Medical College, Changzhi, Shanxi 046000, China;
3 Qinghai Center for Disease Control and Prevention, Xining, Qinghai 810007, China;
4 State Key Laboratory of Infectious Disease Prevention and Control, Collaborative Innovation Center for Diagnosis and Treatment of Infectious Diseases, National Institute for Communicable Disease Control and Prevention, Chinese Center for DiseaseControl and Prevention, Beijing 102206, China
巴尔通体属于变形菌纲、α亚纲、根瘤菌目、巴尔通体科、巴尔通体属,是一群革兰染色阴性,营养条件要求较高的细胞内寄生需氧杆菌。目前已经被确认的巴尔通体多达30余种,部分种及亚种可感染多种哺乳动物并导致人类巴尔通体病[1]。如杆菌样巴尔通体(Bartonella bacilliformis)[2]、五日热巴尔通体(B. quintana)[3]、汉赛巴尔通体(B. henselae)[4]、格拉汉姆巴尔通体(B. grahamii)[5]、伊丽莎白巴尔通体(B. elizabethae)[6]、文森巴尔通体伯格霍夫亚种(B. vinsonii subsp. berkhoffii)[7]可致人心内膜炎、心肌炎、视神经炎、脑膜炎、脾肿大、淋巴结病和神经系统疾病等。研究表明巴尔通体在啮齿动物中的感染率可高达70%,啮齿动物被认为是多种巴尔通体的天然宿主[8]。
麦秀国家森林公园(东经101°46'~102°04',北纬35°8'~35°21')位于青海省东南部的黄南藏族自治州泽库县境内,是三江源国家公园生态保护和建设的核心区,属原始森林自然风景区,境内野生动物资源丰富,尤其是啮齿类动物种类众多[9]。随着当地旅游业的快速发展,外来旅游人员与野外小型哺乳动物及媒介生物接触的机会大大增加,自然疫源性疾病感染风险较大。调查小型哺乳动物病原体携带情况,可为当地旅游业开发风险评估提供科学依据。本文调查分析了该地区小型哺乳动物巴尔通体流行情况及遗传多样性,现将结果报告如下。
1 材料与方法 1.1 标本采集采用夹夜法捕获小型哺乳动物,每天傍晚在林区布放中号鼠夹100夹,每5 m布放1夹,以生花生米为诱饵,共布放5夜,次晨收取鼠夹,对捕获的小型哺乳动物进行种属和性别鉴定后,用75%的乙醇溶液进行体表消毒,无菌操作取肝、脾组织,分置于1.5 ml灭菌冻存管后液氮保存。
1.2 巴尔通体分离培养每个样本取30 mg组织加入200 μl灭菌胰蛋白酶大豆肉汤(BD Biosciences,USA)进行研磨,接种在2个含5%去纤维羊血的胰蛋白酶大豆琼脂培养基的培养皿上(BD Biosciences,USA),置于37 ℃、5% CO2培养箱中分离培养。每日观察其生长情况并记录,挑选可疑菌落进行后续实验。
1.3 DNA提取、PCR扩增和DNA测序按照文献[10]方法提取DNA,采用PCR方法扩增巴尔通体枸橼酸合酶基因(citrate synthase gene,gltA)。PCR反应体系为50 μl,包含25 μl 2 × TransTaq-T PCR SuperMix(北京擎科新业生物技术有限公司),22 μl H2O,1 μl引物(10 μmol/L),1 μl DNA模板。所用引物为BhCS781.p:5'-GGGGACCAGCTCATGGTGG-3';BhCS1137.n:5'-AATGCAAAAAGAACAGTAAACA-3',扩增长度379 bp。PCR反应条件:94 ℃预变性5 min;94 ℃变性30 s,53 ℃退火30 s,72 ℃延伸20 s,30个循环;最后72 ℃总延伸7 min。PCR产物经1%琼脂糖凝胶电泳后凝胶成像仪观察结果。
1.4 遗传进化分析将gltA扩增阳性产物交由北京擎科新业生物技术有限公司直接测序,利用BLAST对所测核酸序列与GenBank中已报道的巴尔通体物种序列进行核苷酸序列同源性分析。利用Clustal W软件程序进行序列比对,用MEGA 7.0软件构建系统发育树,选择与巴尔通体亲缘性较近的布鲁氏菌(Brucella abortus)作为外群。
1.5 遗传多样性分析通过DnaSP 5.10软件分析核苷酸序列的多态性,包括多态位点数(s)、单倍型数(H)、位点核苷酸多样度(π)、平均核苷酸差异数(k)和单倍型多态率(Hd);用滑动窗口间隔25 bp 1次,1次分析100 bp用于发现目的基因序列中哪一段的π最高;进行Tajima’s D检验以鉴定目的基因序列在进化过程中是否遵循中性进化模型。
1.6 统计学分析采用SPSS 22.0软件对数据进行录入分析,不同种类、不同性别组间阳性率比较采用Fisher确切概率法,肝和脾组织培养阳性率比较采用配对χ2检验,检验水准α=0.05。
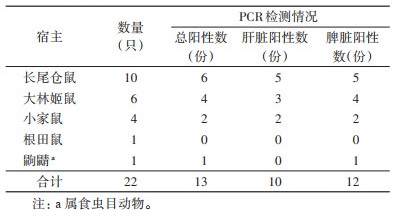
2 结果 2.1 采样情况本次调查共捕获21只啮齿动物,分属于2科4属,包括长尾仓鼠(Cricetulus longicaudatus,CL)10只,大林姬鼠(Apodemus speciosus,AS)6只,小家鼠(Mus musculus,MM)4只,根田鼠(Microtus oeconomus,MO)1只和食虫目鼩鼱(Sorex araneus,SA)1只。经性别鉴定,雌性13只,雄性9只。
2.2 巴尔通体分离培养将44份肝脏和脾脏组织进行巴尔通体分离培养,共培养出可疑阳性克隆22份,将可疑克隆进行gltA基因扩增,证实均为巴尔通体。共有13只小型哺乳动物巴尔通体阳性(表 1),总阳性率为59.09%,不同种属间阳性率差异无统计学意义(P=0.860)。雌性动物阳性11只,雄性动物阳性2只,不同性别间阳性率差异有统计学意义(P=0.007)。22份阳性标本中肝、脾组织双阳性9份,肝脏组织单阳性1份,脾脏组织单阳性3份,肝、脾组织培养阳性率差异无统计学意义(45.45% vs 54.55%,P=0.625)。
|
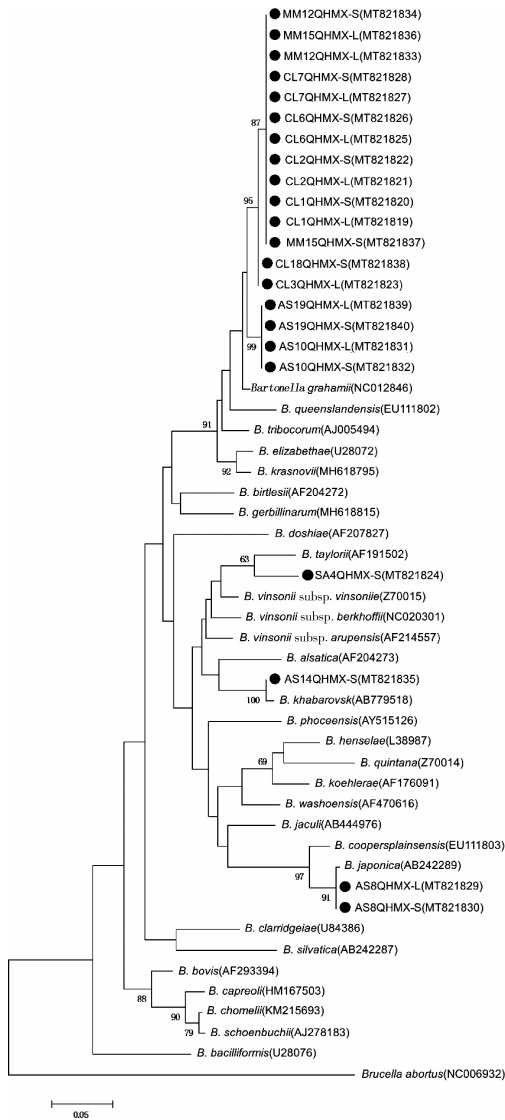
经测序共获得22个巴尔通体gltA基因的核苷酸序列(提交GenBank,序列号:MT821819-MT821840)。采用最大似然法(Maximum-likelihood method,ML)、邻接法(Neighbor-joining,NJ)和最小进化法(Minimum-evolution,ME)进行系统进化分析,结果一致。最终选择ML法,基于核苷酸Kimura 2-parameter模型,以1 000次重复计算Bootstrap值构建系统进化树,结果显示9份肝、脾组织双阳性标本序列比对完全一致,未发现多重感染现象,共获得13株巴尔通体菌。10株为格拉汉姆巴尔通体(同源性为97.10%~100%),其中6株来源于长尾仓鼠,2株来源于小家鼠,2株来源于大林姬鼠;1株为泰勒巴尔通体(同源性为94.68%),来源于鼩鼱;1株为哈巴罗夫斯克巴尔通体(同源性为99.41%),来源于大林姬鼠;1株为日本巴尔通体(同源性为99.70%),来源于大林姬鼠。见图 1。
|
| 注:●表示本研究中分离到的巴尔通体菌株。 图 1 基于gltA基因构建的22株巴尔通体分离株系统发育树 Figure 1 Phylogenetic trees of 22 Bartonella isolates constructed based on the gltA gene |
| |
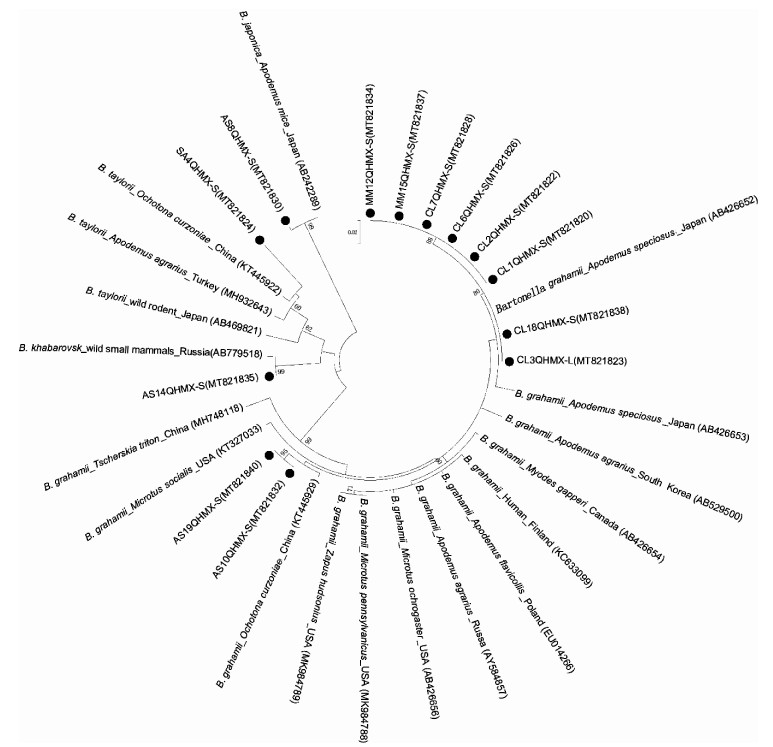
从GenBank中筛选截止2020年12月底前已提交的格拉汉姆巴尔通体、泰勒巴尔通体、哈巴罗夫斯克巴尔通体和日本巴尔通体菌株的gltA基因序列(筛选的序列要求信息完整,包括有具体的宿主动物名称、分离地点等),按照同时期同地区报道序列只随机筛选1株有代表性菌株的原则,共筛选出18株参考菌株,将本次调查最终确认的13株巴尔通体gltA基因序列与GenBank中不同来源的参考菌株序列进行溯源分析,结果显示格拉汉姆巴尔通体作为优势菌株主要聚为2簇,长尾仓鼠和小家鼠分离株与日本大林姬鼠分离株(AB426652、AB426653)聚为一簇,而大林姬鼠分离株与当地高原鼠兔(Ochotona curzoniae)分离株(KT445929)聚为一簇。哈巴罗夫斯克巴尔通体分离株与俄罗斯小型哺乳动物中的分离株(AB779518)比较接近,泰勒巴尔通体与当地高原鼠兔分离株(KT445922)较为接近,日本巴尔通体分离株与日本姬鼠分离株(AB242289)较为接近。见图 2。
|
| 注:●表示本研究中分离到的巴尔通体菌株。 图 2 13株巴尔通体分离株溯源分析 Figure 2 Traceability analysis of 13 Bartonella isolates |
| |
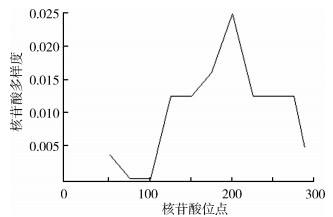
将10株格拉汉姆巴尔通体的gltA基因序列进行遗传多样性分析,共发现8个多态位点(s=8,均是二态),产生了3种单倍型(H=3)。根据10条序列分析得到Hd为0.622±0.138,k为3.200,π为0.010。用滑动窗口1次100 bp间隔25 bp分析,发现在152~251 bp间的片段多样度最高(图 3)。对序列进行中性检验,经Tajima’s D检验,P=0.568。
|
| 图 3 gltA基因不同核苷酸位点的多样度 Figure 3 Diversity of different nucleotide loci in the gltA gene |
| |
麦秀国家森林公园作为一个原始森林自然风景区,地处三江源国家公园腹地,近年来旅游人数不断增多,研究该地区小型哺乳动物,特别是啮齿动物病原体携带情况,对评估当地自然疫源性疾病人群感染风险意义重大。巴尔通体作为一个新发现的古老病原体,侵入人体可引起几乎所有器官系统包括心脏、肝脏、脾脏、骨骼、骨髓、淋巴、肌肉、软组织及中枢神经系统等的严重并发症。野生啮齿类动物是巴尔通体的重要自然宿主,本次调查我们发现该地区小型哺乳动物(包含1只食虫目的鼩鼱)巴尔通体感染率为59.09%,远高于我们前期在该地区高原鼠兔中的检出率(16.67%)[10],也高于我国大部分地区的感染率[11]。本研究中,不同性别小型哺乳动物巴尔通体感染率差异显著,与目前大部分研究结果不同,也与我们前期在柴达木盆地开展的啮齿动物巴尔通体感染状况调查结果不一致[12-13],这可能与采集的样本量较少有关,有必要在该地区开展进一步的调查研究。通过肝脏和脾脏2种组织的培养,结果发现2种组织的阳性率差异无统计学意义,与之前的研究结果也一致[10]。
本研究中,我们共分离出了4种巴尔通体,包括格拉汉姆巴尔通体、泰勒巴尔通体、哈巴罗夫斯克巴尔通体和日本巴尔通体,其中格拉汉姆巴尔通体分离于长尾仓鼠、大林姬鼠和小家鼠,泰勒巴尔通体分离于鼩鼱,哈巴罗夫斯克巴尔通体和日本巴尔通体分离于大林姬鼠。在获得的13株巴尔通体序列中,有10株为格拉汉姆巴尔通体,提示格拉汉姆巴尔通体为该地区优势流行株。研究表明格拉汉姆巴尔通体是人类致病性的病原体,感染可以导致人类出现视神经炎[14]和猫抓病[5]等,前期我们在该地区的高原鼠兔中也检测出该菌株[10],应引起相关部门的高度重视,加强该地区野生动物巴尔通体的监测和人类疑似临床病例的调查。但是,本研究标本采集过少,还不足以代表当地小型哺乳动物的物种分布及病原体携带谱,在今后的监测工作中应加大监测力度。
通过基于gltA基因的溯源分析,发现该地区格拉汉姆巴尔通体分离株主要聚成2簇,长尾仓鼠和小家鼠分离株与日本大林姬鼠分离株聚为一簇,而大林姬鼠分离株与当地高原鼠兔分离株聚为一簇,提示啮齿动物携带巴尔通体的生物多样性,没有严格的宿主依赖性,其遗传进化规律还需进一步研究。通过对10株格拉汉姆巴尔通体的gltA基因序列进行遗传多样性分析,发现了8个多态位点,产生了3种单倍型,核苷酸多样度为0.010,其中在152~251 bp间的片段多样度最高,提示该地区格拉汉姆巴尔通体分离株具有遗传多样性,但这些多态位点存在的意义还需进一步研究。此外,从鼩鼱中分离出的泰勒巴尔通体分离株与GenBank中的泰勒巴尔通体株仅有94.68%的同源性,提示其可能为一个新的基因亚型[15],有待后期研究进一步确认。
利益冲突 无
| [1] |
Mogollon-Pasapera E, Otvos L Jr, Giordano A, et al. Bartonella: emerging pathogen or emerging awareness?[J]. Int J Infect Dis, 2009, 13(1): 3-8. DOI:10.1016/j.ijid.2008.04.002 |
| [2] |
Ulloa GM, Vásquez-Achaya F, Gomes C, et al. Molecular detection of Bartonella bacilliformis in Lutzomyia maranonensis in Cajamarca, Peru: A new potential vector of carrion's disease in Peru?[J]. Am J Trop Med Hyg, 2018, 99(5): 1229-1233. DOI:10.4269/ajtmh.18-0520 |
| [3] |
Mai BHA, Barbieri R, Chenal T, et al. Five millennia of Bartonella quintana bacteraemia[J]. PLoS One, 2020, 15(11): e0239526. DOI:10.1371/journal.pone.0239526 |
| [4] |
Ksiaa I, Abroug N, Mahmoud A, et al. Update on Bartonella neuroretinitis[J]. J Curr Ophthalmol, 2019, 31(3): 254-261. DOI:10.1016/j.joco.2019.03.005 |
| [5] |
Oksi J, Rantala S, Kilpinen S, et al. Cat scratch disease caused by Bartonella grahamii in an immunocompromised patient[J]. J Clin Microbiol, 2013, 51(8): 2781-2784. DOI:10.1128/JCM.00910-13 |
| [6] |
Tay ST, Kho KL, Wee WY, et al. Whole-genome sequence analysis and exploration of the zoonotic potential of a rat-borne Bartonella elizabethae[J]. Acta Trop, 2016, 155: 25-33. DOI:10.1016/j.actatropica.2015.11.019 |
| [7] |
Müller A, Soto F, Sepúlveda M, et al. Bartonella vinsonii subsp. berkhoffii and B. henselae in dogs[J]. Epidemiol Infect, 2018, 146(9): 1202-1204. DOI:10.1017/S0950268818001127 |
| [8] |
Gutiérrez R, Krasnov B, Morick D, et al. Bartonella infection in rodents and their flea ectoparasites: an overview[J]. Vector-Borne Zoonotic Dis, 2015, 15(1): 27-39. DOI:10.1089/vbz.2014.1606 |
| [9] |
李海龙, 马英, 魏有文, 等. 青海省啮齿类动物地理分布格局探讨[J]. 中国媒介生物学及控制杂志, 2013, 24(5): 418-421. Li HL, Ma Y, Wei YW, et al. Investigation of geographical distribution pattern of rodents in Qinghai province, China[J]. Chin J Vector Biol Control, 2013, 24(5): 418-421. DOI:10.11853/j.issn.1003.4692.2013.05.011 |
| [10] |
Rao HX, Yu J, Guo P, et al. Bartonella species detected in the Plateau Pikas (Ochotona curzoiae) from Qinghai Plateau in China[J]. Biomed Environ Sci, 2015, 28(9): 674-678. DOI:10.3967/bes2015.094 |
| [11] |
Liu QY, Eremeeva ME, Li DM. Bartonella and Bartonella infections in China: from the clinic to the laboratory[J]. Comp Immunol Microbiol Infect Dis, 2012, 35(2): 93-102. DOI:10.1016/j.cimid.2012.01.002 |
| [12] |
Rao HX, Li SJ, Lu L, et al. Genetic diversity of Bartonella species in small mammals in the Qaidam Basin, western China[J]. Sci Rep, 2021, 11(1): 1735. DOI:10.1038/s41598-021-81508-w |
| [13] |
Qin XR, Liu JW, Yu H, et al. Bartonella species detected in rodents from eastern China[J]. Vector Borne Zoonotic Dis, 2019, 19(11): 810-814. DOI:10.1089/vbz.2018.2410 |
| [14] |
Kerkhoff FT, Bergmans AM, van Der Zee A, et al. Demonstration of Bartonella grahamii DNA in ocular fluids of a patient with neuroretinitis[J]. J Clin Microbiol, 1999, 37(12): 4034-4038. DOI:10.1128/JCM.37.12.4034-4038.1999 |
| [15] |
Blazes DL, Mullins K, Smoak BL, et al. Novel Bartonella agent as cause of verruga peruana[J]. Emerg Infect Dis, 2013, 19(7): 1111-1114. DOI:10.3201/eid1907.121718 |
 2021, Vol. 32
2021, Vol. 32


