扩展功能
文章信息
- 李心玫, 孙梦涛, 詹雨娟, 张兰香, 李梦珠, 陶东东, 孙恩涛
- LI Xin-mei, SUN Meng-tao, ZHAN Yu-juan, ZHANG Lan-xiang, LI Meng-zhu, TAO Dong-dong, SUN En-tao
- 马六甲肉食螨与转开肉食螨的形态及分子鉴定
- Molecular and morphological identification of Cheyletus malaccensis and C. carnifex
- 中国媒介生物学及控制杂志, 2021, 32(2): 230-234
- Chin J Vector Biol & Control, 2021, 32(2): 230-234
- 10.11853/j.issn.1003.8280.2021.02.022
-
文章历史
- 收稿日期: 2020-06-04
肉食螨隶属于蜱螨亚纲(Acari),真螨总目(Acariformes),绒螨目(Trombidiformes)前气门亚目(Prostigmata),肉食螨总科(Cheyletoidea)[1]。Xia[2]根据Bochkov和Faind的分类系统,报道国内肉食螨有24属48种。肉食螨能捕食害嗜鳞螨(Lepidoglyphus destructor)、腐食酪螨(Tyrophagus putrescentiae)、粗脚粉螨(Acarus siro)和二斑叶螨(Tetranychus urticae)等害螨,是一类可用于生物防治的螨类[3]。但是正由于这一捕食性,使得肉食螨在储藏物中随猎物不断迁移的过程中会携带霉菌快速蔓延,导致储藏物霉变,从而造成经济损失。此外,肉食螨产生的排泄物、代谢产物等具有较强的变应原性,可引发多种变态反应疾病,是人类居住环境中可能的致痒因素之一,具有一定的医学研究价值[4-5]。
肉食螨体型微小,肉眼下难以从形态上进行区分,需借助光学显微镜观察外部细微特征从而加以辨别[6]。马六甲肉食螨(Cheyletus malaccensis)和转开肉食螨(C. carnifex)同为肉食螨属,表型特征相似,进行形态学鉴定时主要依据文字描述和线条示意图,李朝品[7]曾绘制马六甲肉食螨的示意图。但实际情况下的标本形态往往与示意图有所出入,仅依据文字描述和线条示意图难以准确进行形态学鉴定。且肉食螨还存在幼螨、异型雄螨[8]、特殊体色[1]及螨体残缺等情况,给形态学鉴定进一步带来困扰。分子标记不受发育阶段、个体形态差异和环境等因素影响,具有可操作性强、可重复性强等优点,被广泛用于物种鉴定[9-11]。
线粒体细胞色素C氧化酶亚基Ⅰ(COⅠ)基因的进化速率适中,在无脊椎动物的系统分类、种类鉴别和分子进化学研究中应用广泛[12-13]。Sakamoto等[13]使用COⅠ对Stigmaeopsis属的7个种成功进行分子鉴定和系统发育分析,Webster等[14]利用COⅠ基因对静粉螨(A. immobilis)、粗脚粉螨、小粗脚粉螨(A. farris)这3种近缘种成功地进行了分类鉴别。本研究拟将肉食螨形态学鉴别要点比较分析,并提供肉食螨在高倍镜下的照片,便于参考和理解。同时,扩增2种肉食螨的COⅠ基因片段序列,建立马六甲肉食螨和转开肉食螨的分子鉴定方法。
1 材料与方法 1.1 样本采集、分离和形态鉴定2018年5月至2019年7月,从安徽省部分地区的农户粮仓中采集过期面粉和黄豆粉等储藏物样本,低倍镜下初步鉴定并分离获得肉食螨。将所获肉食螨制成玻片标本,在高倍镜下进行进一步形态学鉴定,挑取部分已经形态鉴定过的马六甲肉食螨和转开肉食螨,75%乙醇溶液固定,用于DNA的提取。
1.2 DNA提取、PCR扩增、克隆和测序参照本实验室单只螨基因组DNA的提取方法[15]。提取完成后,将DNA样本置于冰箱-20 ℃环境中留存备用。引物由生工生物工程(上海)股份有限公司合成,引物序列:COⅠ F 5x-GGTCAACAAATCATAAAGATATTGG-3x;COⅠ R 5x-TAAACTTCAGGGTGACCAAAAAATCA-3x[16]。
扩增产物用1%琼脂糖凝胶电泳检测,与克隆载体连接,转化到已制备好的感受态细胞,挑取阳性克隆送至生工生物工程(上海)股份有限公司测序。
1.3 序列分析使用Clustal X(1.81)软件对测序结果进行多序列比对,去除引物后,将所获序列进行BLAST比对分析。从GenBank数据库中查找并下载已有马六甲肉食螨、转开肉食螨的COⅠ基因序列(表 2),使用MEGA X软件完成序列分析,将网真扇毛螨(Eucheyletia reticulata)作为外群,最大似然法(maximum likelihood,ML)构建系统发育树。
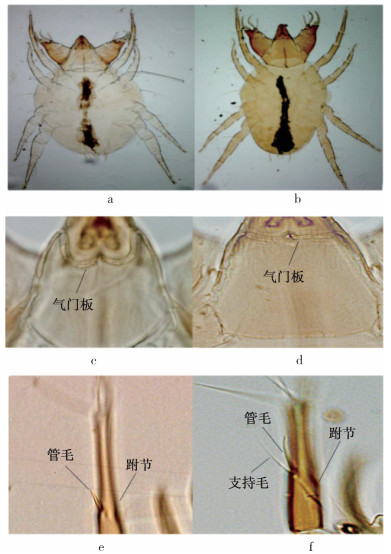
2 结果 2.1 形态学鉴定结果雌性马六甲肉食螨和转开肉食螨可通过整体外观、气门板形状、足Ⅰ跗节管毛的形态及与支持毛相对比例来区分[7](表 1、图 1)。雄性马六甲肉食螨和转开肉食螨可通过足Ⅰ跗节管毛的形态及与支持毛相对比例来进行区分。见表 1。

|
|
| 注:a马六甲肉食螨;b转开肉食螨;c马六甲肉食螨的气门板;d转开肉食螨的气门板;e马六甲肉食螨足Ⅰ跗节与管毛;f转开肉食螨足Ⅰ跗节、管毛与支持毛。 图 1 马六甲肉食螨和转开肉食螨的形态学差异(雌) Figure 1 Morphological differences between Cheyletus malaccensis and C. carnifex (female) |
| |
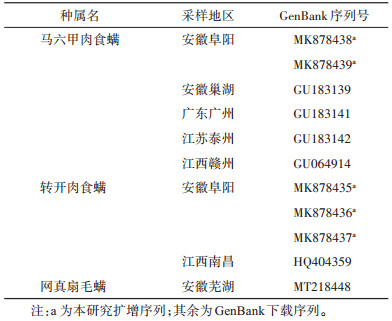
所获COⅠ基因序列长度均为709 bp,BLAST比对分析后,分子鉴定与形态学鉴定结果一致。结合从GenBank中搜索并下载国内已报道的马六甲肉食螨、转开肉食螨和网真扇毛螨的COⅠ基因片段序列(表 2),进行序列分析和系统发育分析。
|
使用MEGA X软件对本研究中的序列进行碱基组成分析得:A、T、C、G 4种碱基分别占比34.4%、36.5%、18.5%、10.6%,A+T为70.9%,C+G为29.1%,具有A+T偏向性。多序列比对未发现核苷酸位点缺失、插入以及终止密码子。马六甲肉食螨的5个地理种群间有696个保守位点,13个变异位点,5个简约信息位点;转开肉食螨的2个地理种群间有706个保守位点,3个变异位点。2种肉食螨间共检测到569个保守位点,140个变异位点以及132个简约信息位点。
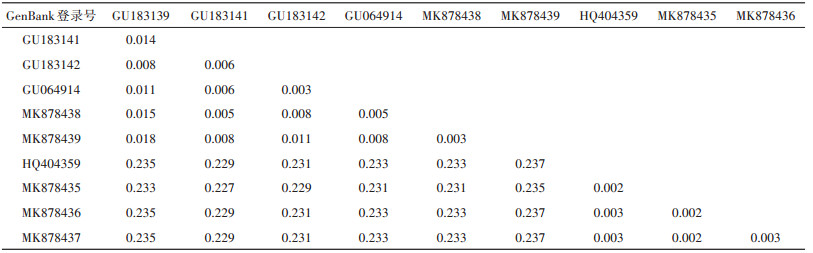
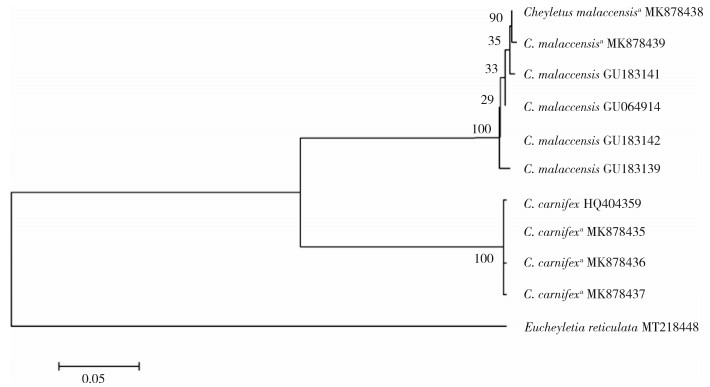
2.2.2 遗传距离及系统进化分析马六甲肉食螨和转开肉食螨基于COⅠ基因的种内遗传距离均 < 0.02,种间遗传距离为0.227~0.237(表 3)。从所构建的ML树上看,马六甲肉食螨和转开肉食螨各自聚为一支(图 2)。通过观察2个置信度为100的进化树分支,可明显区分不同的物种。
|
|
| 图 2 转开肉食螨与马六甲肉食螨线粒体细胞色素C氧化酶亚基Ⅰ基因的最大似然法进化树 Figure 2 Phylogenetic tree of mitochondrial cytochrome c oxidase subunit Ⅰ gene sequence of Cheyletus malaccensis and C. carnifex based on the maximum likelihood method |
| |
肉食螨能够捕食螨类和其他昆虫,在区域内达到一定内禀增长率的时候,可有效抑制该地区多种植食性螨虫和昆虫的数量[7],但肉食螨同样具有危害经济作物和人体健康的可能[4]。正确鉴定螨种是开展科学研究和应用防治的前提。马六甲肉食螨与转开肉食螨亲缘关系近,表型特征相似,现有文献对马六甲肉食螨的研究和形态描述较为详细,相关文献还提供了马六甲肉食螨的手绘线条形态示意图[7],但对转开肉食螨的研究较少。本研究主要将雌性马六甲肉食螨和转开肉食螨的外部形态特征进行比较,形态特征信息表与镜下实物照片相结合的形式,可更好地表示出2种肉食螨的形态特征差异。但形态鉴定常受限于肉食螨的性别以及发育阶段等情况[1, 8],鉴种结果的准确性难以得到保证。DNA分子标记不受限于以上情况,已被应用于许多物种的鉴定[16-17]。
本研究选用COⅠ基因作为分子标记,将实验所获COⅠ基因片段序列与GenBank中的已有物种序列进行BLAST比对,比对结果与形态鉴定结果一致。使用MEGA X软件对本研究中肉食螨的COⅠ基因序列进行分析,发现此段序列碱基组成有明显的AT偏向性。马六甲肉食螨的5个地理种群间检测到13个变异位点,转开肉食螨的2个地理种群间检测到3个变异位点,2种肉食螨COⅠ基因序列间共检测到140个变异位点、569个保守位点、132个简约信息位点,说明COⅠ基因具有丰富的遗传信息,且地理差异不明显。种内遗传距离均 < 0.02,种间遗传距离 > 0.02,种内种间遗传距离差异明显,说明COⅠ基因可作为鉴定马六甲肉食螨和转开肉食螨的分子标记[18]。ML树表明本实验所获COⅠ基因与下载自GenBank中的对应物种序列各自聚为一支,聚类结果进一步支持本研究形态鉴定的结果。综合对2种肉食螨COⅠ基因片段序列的遗传距离、变异位点和系统进化分析可知,COⅠ基因可作为分子标记鉴别马六甲肉食螨和转开肉食螨,分子鉴定与形态学鉴定结果相一致。分子鉴定方法的补充,使得对肉食螨近似种的鉴别不仅依赖于形态鉴定,2种方法相互验证,可进一步提高鉴种结果的准确性。
利益冲突 无
| [1] |
Bochkov AV. Mites of the family Cheyletidae (Acari: Prostigmata): phylogeny, distribution, evolution and analysis of parasite-host relationship[J]. Parazitologiia, 2004, 38(2): 122-138. |
| [2] |
Xia B. A review of progress on the systematics and biology of the family Cheyletidae in China, with a checklist of the Chinese cheyletids[J]. Zoosymposia, 2010, 4(1): 158-164. DOI:10.11646/zoosymposia.4.1.11 |
| [3] |
Yang XQ, Ye QT, Xin TR, et al. Population genetic structure of Cheyletus malaccensis (Acari: Cheyletidae) in China based on mitochondrial COⅠand 12S rRNA genes[J]. Exp Appl Acarol, 2016, 69(2): 117-128. DOI:10.1007/s10493-016-0028-3 |
| [4] |
李朝品, 湛孝东, 孙恩涛, 等. 储藏中药材孳生肉食螨种类及其群落生态研究[J]. 中药材, 2013, 36(9): 1412-1416. Li CP, Zhan XD, Sun ET, et al. Investigation on species and community ecology of cheyletoid mites breeding in the stored traditional Chinese medicinal materials[J]. J Chin Med Mater, 2013, 36(9): 1412-1416. DOI:10.13863/j.issn1001-4454.2013.09.004 |
| [5] |
陈静, 王康, 南丹阳, 等. 芜湖市某高校学生宿舍床席螨类和昆虫孳生状况调查[J]. 中国血吸虫病防治杂志, 2016, 28(2): 151-155. Chen J, Wang K, Nan DY, et al. Investigation on mites and insects breeding status of bed mats in college dormitories in Wuhu city[J]. Chin J Schistosom Control, 2016, 28(2): 151-155. DOI:10.16250/j.32.1374.2015231 |
| [6] |
Beroiz B, Couso-Ferrer F, Ortego F, et al. Mite species identification in the production of allergenic extracts for clinical use and in environmental samples by ribosomal DNA amplification[J]. Med Vet Entomol, 2014, 28(3): 287-296. DOI:10.1111/mve.12052 |
| [7] |
李朝品. 医学节肢动物学[M]. 北京: 人民卫生出版社, 2009: 1266-1272. Li CP. Medical arthropodology[M]. Beijing: People's Medical Publishing House, 2009: 1266-1272. |
| [8] |
Corpuz-Raros LA. Twelve new species and one new record of Cheyletidae (Acari) from the Philippines[J]. Int J Acarol, 1998, 24(4): 259-290. DOI:10.1080/01647959808683594 |
| [9] |
易忠权, 夏伟, 赵盼雯, 等. DNA标记技术在螨类系统学研究中的应用[J]. 现代生物医学进展, 2017, 17(31): 6196-6200. Yi ZQ, Xia W, Zhao PW, et al. DNA markers in the systematic study of mites[J]. Progr Mod Biomed, 2017, 17(31): 6196-6200. DOI:10.13241/j.cnki.pmb.2017.31.046 |
| [10] |
邓洁, 郭浩杰, 黄少康, 等. 基于线粒体COⅠ基因的印度新曲厉螨的分子鉴定[J]. 环境昆虫学报, 2019, 41(2): 380-386. Deng J, Guo HJ, Huang SK, et al. Molecular identification of Neocypholaelaps indica Evans based on mitochondrial COⅠgene[J]. J Environ Entomol, 2019, 41(2): 380-386. DOI:10.3969/j.issn.1674-0858.2019.02.21 |
| [11] |
Li MN, Yang QQ, Shao HF, et al. Species identification and phylogenetic relationships among five species of psocids from Chinese herbal medicines[J]. J Stored Prod Res, 2020, 85: 101539. DOI:10.1016/j.jspr.2019.101539 |
| [12] |
Ernieenor FCL, Ernna G, Jafson AS, et al. PCR identification and phylogenetic analysis of the medically important dust mite Suidasia medanensis (Acari: Suidasiidae) in Malaysia[J]. Exp Appl Acarol, 2018, 76(1): 99-107. DOI:10.1007/s10493-018-0285-4 |
| [13] |
Sakamoto H, Matsuda T, Suzuki R, et al. Molecular identification of seven species of the genus Stigmaeopsis (Acari: Tetranychidae) and preliminary attempts to establish their phylogenetic relationship[J]. Syst Appl Acarol, 2017, 22(1): 91-101. DOI:10.11158/saa.22.1.10 |
| [14] |
Webster LMI, Thomas RH, McCormack GP. Molecular systematics of Acarus siro s. lat., a complex of stored food pests[J]. Mol Phylogenet Evol, 2004, 32(3): 817-822. DOI:10.1016/j.ympev.2004.04.005 |
| [15] |
郑凌霄, 尹灿灿, 王逸枭, 等. 基于DNA条形码技术的粉螨种类鉴定研究[J]. 中国媒介生物学及控制杂志, 2019, 30(2): 180-184. Zheng LX, Yin CC, Wang YX, et al. Identification of acaroid mite species based on DNA barcoding[J]. Chin J Vector Biol Control, 2019, 30(2): 180-184. DOI:10.11853/j.issn.1003.8280.2019.02.015 |
| [16] |
Yang B, Cai JL, Cheng XJ. Identification of astigmatid mites using ITS2 and COⅠregions[J]. Parasitol Res, 2011, 108(2): 497-503. DOI:10.1007/s00436-010-2153-y |
| [17] |
Palasio RGS, de Almeida Guimarães MC, Ohlweiler FP, et al. Molecular and morphological identification of Biomphalaria species from the state of São Paulo, Brazil[J]. ZooKeys, 2017(668): 11-32. DOI:10.3897/zookeys.668.10562 |
| [18] |
Hebert PDN, Cywinska A, Ball SL, et al. Biological identifications through DNA barcodes[J]. Proc Roy Soc B: Biol Sci, 2003, 270(1512): 313-321. DOI:10.1098/rspb.2002.2218 |
 2021, Vol. 32
2021, Vol. 32


