扩展功能
文章信息
- 兰晓宇, 鲁亮, 候芝林, 杜国义, 史献明, 崔耀仁, 刘冠纯, 陈永明, 康东梅, 郑楠, 任兴宇, 闫东
- LAN Xiao-yu, LU Liang, HOU Zhi-lin, DU Guo-yi, SHI Xian-ming, CUI Yao-ren, LIU Guan-chun, CHEN Yong-ming, KANG Dong-mei, ZHENG Nan, REN Xing-yu, YAN Dong
- DNA条形码技术在河北省鼠疫疫源地常见蚤种鉴定中的应用
- Application of DNA barcoding technique in identification of common flea species in plague foci of Hebei province, China
- 中国媒介生物学及控制杂志, 2020, 31(6): 662-666
- Chin J Vector Biol & Control, 2020, 31(6): 662-666
- 10.11853/j.issn.1003.8280.2020.06.007
-
文章历史
- 收稿日期: 2020-05-11
2 中国疾病预防控制中心传染病预防控制所, 传染病预防控制国家重点实验室, 北京 102206
2 State Key Laboratory of Infectious Diseases Prevention and Control, National Institute for Communicable Disease Control and Prevention, Chinese Center for Disease Control and Prevention
蚤类在鼠疫流行中起着特异性媒介的作用,是鼠疫疫源地的重要组成部分。目前,河北省共发现蚤类71种,隶属于6科35属[1],占全国蚤种类的11%。蚤类营体外寄生,体型小,在形态上高度特化,身体结构复杂,在日常工作中准确鉴别较为困难。在蚤类的分类鉴定上,传统的鉴定方法是利用光学显微镜观察其特异性的外部形态特征来进行分类,如雄蚤外生殖器构造、雌蚤的受精囊等。传统形态学鉴定对于形态特征相似的近缘种标本、完整性有所损坏的标本、蚤幼虫以及同属不同种的雌蚤标本的鉴定都有一定的困难,并且形态学鉴定需要长期的鉴定知识和经验积累。分子生物学技术的发展为蚤类分类提供了新的思路,利用DNA条形码技术,使用一段核酸序列作为分子标记进行扩增、测序及序列的比对分析,可以快速、准确地鉴定蚤的种类,是形态学分类研究的重要补充和发展。
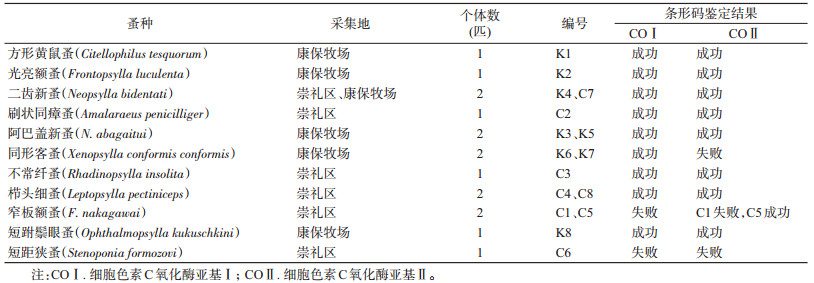
1 材料与方法 1.1 材料来源于张家口市康保牧场、崇礼区采集鼠体蚤及洞干蚤,存放在75%乙醇溶液中并进行编号。标本信息见表 1。
依据《中国蚤目志》[2],利用体视显微镜对采集的蚤类样本进行形态学初步鉴定,提取DNA后,将蚤类标本进行封片,再用光学显微镜进行正式的形态鉴定。经初检非透明标本和复检凭证标本,共鉴定蚤种11种,隶属于3总科4科9属。见表 1。
1.2.2 分子生物学鉴定 1.2.2.1 样品处理在体视镜下用解剖针和手术刀将蚤标本沿胸腹连接处切开,置于1.5 ml离心管中并进行编号。
1.2.2.2 蚤类DNA的提取根据QIAampDNA纯化试剂盒-Mini说明书操作步骤进行蚤类DNA的提取,并于-20 ℃保存,备用。
1.2.2.3 PCR扩增与序列测序PCR反应扩增体系为25 μl:Master Mix12.5 μl,正、反向引物各1μl,ddH2O 7.5 μl,模板DNA3 μl。PCR反应条件:COⅠ,96 ℃预变性5 min;95 ℃变性30 s,57 ℃退火30 s,72 ℃延伸1 min,20个循环;95 ℃变性30 s,54 ℃退火30 s,72 ℃延伸1 min,25个循环;4 ℃保存。COⅡ,95 ℃预变性3 min;94 ℃变性30 s,42 ℃退火30 s,72 ℃延伸15 s,37个循环;72 ℃延伸5 min,4 ℃保存。COⅠ基因引物序列为:LCO1490:5′-GGT CAA CAA ATC ATA AAG ATA TTG G-3′/HCO2198:5′-TAA ACT TCA GGG TGA CCA AAA AAT CA-3′[3],COⅡ基因引物序列为:COⅡ-F:5′-TTA AGT CTT CAA AAT A-3′/COⅡ-R:5′-CTA AAA TAG TAA TTA AAA-3′。扩增产物经1%琼脂糖凝胶电泳检测,选取目标条带清晰的样品,送北京擎科生物技术有限公司进行双向测序。
1.2.2.4 序列分析与系统树构建测定的序列应用DNAStar中的SeqMan软件进行正反链匹配校正与拼接。在美国国立生物技术信息中心(NCBI)上运行BLAST程序进行序列同源性比较,并下载同源性较高的数据库注册蚤种序列。通过MEGA 6.0软件,以邻接法(Neighbor-Joining,NJ)构建分子进化树,自检值设为1 000次。
1.2.2.5 标本封片将提取完DNA的蚤个体进行逐步脱水后,用加拿大树胶进行封片,干燥保存。
2 结果 2.1 基因扩增结果16份蚤类样本利用通用引物全部扩增出清晰的基因特异性片段,与COⅠ和COⅡ基因目标片段的长度相符。
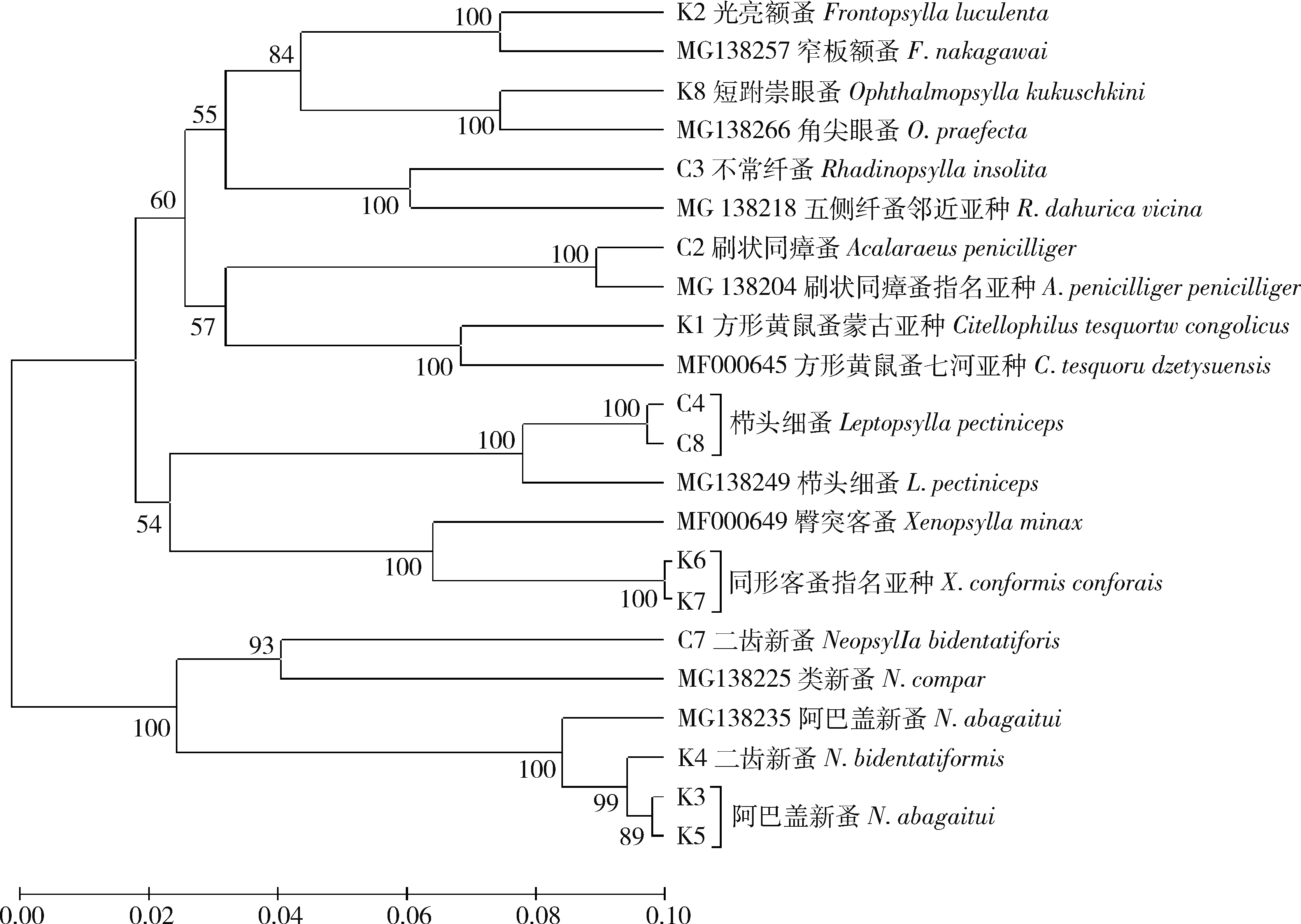
2.2 基因组成分析16份蚤类样本应用COⅠ基因引物进行扩增、测序,其中13份样本测序成功。构建分子进化树结果与形态学鉴定对比,12份样本均与形态学鉴定结果一致,1份样本结果与形态学有差异。见图 1。
|
| 图 1 13份蚤类样本COⅠ基因序列和参考序列的NJ系统进化树 Figure 1 Neighbor-joining phylogenetic tree of reference sequences and the CO Ⅰ sequences of l3 flea samples from the plague foci of Hebei province |
| |
16份蚤类样本应用COⅡ基因引物进行扩增,有12份样本测序成功。构建分子进化树结果与形态学鉴定对比,拼接成功的12份样本与形态学鉴定结果一致。见图 2。

|
| 图 2 12份蚤类样本COⅡ基因序列和参考序列的NJ系统进化树 Figure 2 Neighbor-joining phylogenetic tree of reference sequences and the COll sequences of 12 flea samples from the plague foci of Hebei province |
| |
河北省鼠疫自然疫源地位于河北省西北部冀蒙边际的康保县,疫源地面积约1 000 km2,是松辽平原达乌尔黄鼠(Spermophilus dauricus)鼠疫自然疫源地与内蒙古高原长爪沙鼠(Meriones unguiculatus)鼠疫自然疫源地的交错地带[4]。该鼠疫疫源地自判定以来,共发生5次动物间鼠疫疫情,分别为1971-1972、1994-1995、2002-2003、2005、2017-2018年[5],其中的3次疫情从媒介蚤中检出鼠疫耶尔森菌(鼠疫菌),即分别于1994年在方形黄鼠蚤蒙古亚种(Citellophilus tesquorum mongolicus)、2002年在秃病蚤蒙冀亚种(Nosopsyllus laeviceps kuzenkovi)和宽圆纤蚤(Rhadinopsylla rothschildi)、2005年在方形黄鼠蚤蒙古亚种和秃病蚤蒙冀亚种中检出鼠疫菌。赵智敏和郑建中[6]研究还表明,鼠体蚤、窝巢蚤染蚤率和蚤指数与动物间鼠疫发生明显相关,因此在动物鼠疫监测中,鼠疫宿主动物寄生蚤的分类鉴定和种群数量数据将直接影响鼠疫监测质量,对于鼠疫防治工作有重要意义。
从COⅠ基因进行的系统进化树结果来看,标本K4形态学鉴定结果为二齿新蚤(Neopsylla bidentatiformis),但是在分子进化树结构中却与二齿新蚤不在一个分支上,而与阿巴盖新蚤(N. abagaitui)聚在一个分支,提示在形态鉴定上可能有误。样本C5形态学鉴定为窄板额蚤(Frontopsylla nakagawai),在COⅠ基因测序中没有成功,但是在COⅡ基因分子进化树结构中与窄板额蚤、光亮额蚤(F. luculenta)聚在同一分支,提示C5应该为窄板额蚤,与传统形态学鉴定结果一致。针对上述鉴定结果不一致的情况,在今后的工作中,将加大样本采集量,进一步研究蚤类的种内形态变异,便于准确鉴定蚤种。
研究表明,COⅠ基因序列可以广泛应用于不同分类阶元层次上的分子系统学研究[7-8]。COⅡ基因在双翅目昆虫进化机制和系统重建研究中也是一种有效的分子标记[8-9]。根据COⅠ和COⅡ基因分子生物学鉴定和形态学鉴定结果对比可见,COⅠ和COⅡ基因在属间、属内种间的分类时,能够准确分类鉴定,但在解决亚科以上的高阶元分类分支关系时,能力较差。在蚤类鉴定中,COⅠ和COⅡ基因都可用于蚤种鉴定,选择任何一种基因都可以达到准确鉴定蚤种的目的。但在本次实验中,COⅠ和COⅡ基因扩增和测序,往往不能100%成功。因此,在条件允许的情况下,可以同时利用COⅠ和COⅡ基因片段进行蚤类分子鉴定,这样可以增加基因扩增和测序的成功率,提高鉴定的准确性,同时也可以积累2个基因的序列数据,能够为以后的蚤类分子鉴定技术提供可以利用的数据库。
自2003年Hebert等[10]提出DNA条形码这一概念开始,DNA条形码技术逐渐在生物分类学中开始应用,现已经广泛应用于植物、动物、微生物等很多领域。与传统的形态学鉴定方法相比,DNA条形码技术具有操作简便、快速、准确,具有高度的可重复性,使用方便,不受鉴定经验的限制等优势。它采用数字化形式,使样本鉴定过程能够实现自动化和标准化[11],是传统形态学鉴定方法的强有力补充。但是在实际工作中,DNA条形码技术受数据库序列数量、实验条件技术及经费等因素的限制,目前,我们还不能完全依赖于DNA条形码技术进行蚤类的分类鉴定。还是应该以形态学鉴定为主,结合分子鉴定结果,更好地克服传统形态学分类对于近缘种等鉴定的局限性,科学开展蚤类的分类鉴定工作。
| [1] |
王玉梅, 白万翔. 鼠疫防治与应急处理指南[M]. 北京: 中国社会出版社, 2007: 46-55. Wang YM, Bai WX. Guide to plague prevention and emergency management[M]. Beijing: China Society Press, 2007: 46-55. |
| [2] |
中国科学院中国动物志编辑委员会.. 中国动物志.昆虫纲.蚤目(上、下卷)[M]. 2版.. 北京: 科学出版社, 2007: 198-1878. EditorialCommittee of Zoology of China. Chinese Academy of Sciences. Fauna sinica-insecta, siphonaptera[M]. 2nd ed. Beijing: Science Press, 2007: 198-1878. |
| [3] |
马英. DNA条形码技术在青海省小型兽类及寄生蚤鉴定中的应用研究[D].北京: 中国疾病预防控制中心, 2010. Ma Y. Research on identification of small mammals and parasitic fleas by DNA barcoding in Qinghai province, China[D]. Beijing: Chinese Center for Disease Control and Prevention, 2010. |
| [4] |
陈永明, 闫东, 刘冠纯, 等. 河北省鼠疫自然疫源地长爪沙鼠体蚤多样性研究[J]. 中华卫生杀虫药械, 2018, 24(3): 225-257, 260. Chen YM, Yan D, Liu GC, et al. A diversity of body fleas on Meriones unguiculatus in plague natural epidemic foci of Hebei province[J]. Chin J Hyg Insect Equip, 2018, 24(3): 225-257, 260. DOI:10.19821/j.1671-2781.2018.03.011 |
| [5] |
杜国义, 闫东. 动物间鼠疫流行与主要宿主密度相关性研究[J]. 中国媒介生物学及控制杂志, 2019, 30(4): 442-443. Du GY, Yan D. A study of the correlation between plague epidemics among animals and the density of their hosts[J]. Chin J Vector Biol Control, 2019, 30(4): 442-443. DOI:10.11853/j.issn.1003.8280.2019.04.020 |
| [6] |
赵智敏, 郑建中. 山西省鼠疫流行病学调查结果[J]. 中国地方病学杂志, 2011, 30(4): 441-443. Zhao ZM, Zheng JZ. Epidemiological investigation results of plague in Shanxi province[J]. Chin J Endemiol, 2011, 30(4): 441-443. DOI:10.3760/cma.j.issn.1000-4955.2011.04.026 |
| [7] |
付景, 张迎春. 27种瓢虫mtDNA-COⅠ基因序列分析及系统发育研究(鞘翅目:瓢虫科)[J]. 昆虫分类学报, 2006, 28(3): 179-186. Fu J, Zhang YC. Sequence analysis of mtDNA-COⅠ gene and molecular phylogeny on twenty-seven species of Coccinellids (Coleoptera:Coccinellidae)[J]. Entomotaxonomia, 2006, 28(3): 179-186. DOI:10.3969/j.issn.1000-7482.2006.03.004 |
| [8] |
罗丹, 王安东, 尹小平, 等. 中哈边境阿拉山口口岸输入性猫栉首蚤指名亚种线粒体COⅠ和COⅡ基因序列分析[J]. 中国人兽共患病学报, 2016, 32(9): 789-792. Luo D, Wang AD, Yin XP, et al. COⅠ and COⅡ sequences analysis for an imported Siphonaptera sample of Ctenocephalides felis felis at Alataw pass, China-Kazakhstan[J]. Chin J Zoonoses, 2016, 32(9): 789-792. DOI:10.3969/j.issn.1002-2694.2016.09.005 |
| [9] |
黄朝晖, 王金福. 3种蚊虫COⅡ基因的克隆与序列分析[J]. 中国寄生虫学与寄生虫病杂志, 2001, 19(2): 90-92. Huang ZH, Wang JF. Cloning and sequencing of Cytochrome c Oxidase Ⅱ(COⅡ)gene of three species of mosquitoes[J]. Chin J Parasitol Parasit Dis, 2001, 19(2): 90-92. DOI:10.3969/j.issn.1000-7423.2001.02.008 |
| [10] |
Hebert PDN, Ratnasingham S, De Waard JR. Barcoding animal life:cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proc Biol Sci, 2003, 270(Suppl 1): S 96-99. DOI:10.1098/rsbl.2003.0025 |
| [11] |
何锴, 王文智, 李权, 等. DNA条形码技术在小型兽类鉴定中的探索:以甘肃莲花山为例[J]. 生物多样性, 2013, 21(2): 197-205. He K, Wang WZ, Li Q, et al. DNA barcoding in surveys of small mammal community:a case study in Lianhuashan, Gansu province, China[J]. Biodivers Sci, 2013, 21(2): 197-205. DOI:10.3724/SP.J.1003.2013.09160 |
 2020, Vol. 31
2020, Vol. 31