扩展功能
文章信息
- 周洁, 周晓君, 轩断断, 潘良源, 梁佳欣, 陈宗亮, 张萌萌, 苏容玉, 边晨缤, 董雨欣, 胡守奎, 牛莉娜
- ZHOU Jie, ZHOU Xiao-jun, XUAN Duan-duan, PAN Liang-yuan, LIANG Jia-xin, CHEN Zong-liang, ZHANG Meng-meng, SU Rong-yu, BIAN Chen-bin, DONG Yu-xin, HU Shou-kui, NIU Li-na
- 海南岛中部山区鼠类肠道菌群多样性研究
- Study on the diversity of intestinal microbiota of wild rodents from the central mountainous area of Hainan Island, China
- 中国媒介生物学及控制杂志, 2020, 31(5): 596-601
- Chin J Vector Biol & Control, 2020, 31(5): 596-601
- 10.11853/j.issn.1003.8280.2020.05.019
2 北京大学首钢医院, 北京 100144
2 Peking University Shougang Hospital
鼠类是野生动物的主要类群, 目前全世界鼠类有35个科, 389个属, 2700多种。由于其繁殖力强、传播途径多、与人类关系密切, 因此是许多已知病原体的储存宿主和传播媒介[1-2]。最新研究表明鼠类能携带200多种病原体,但目前已知的鼠传疾病仅57种, 包括细菌性疾病14种, 立克次体病5种, 其中许多属于自然疫源性热带传染病[3]。
海南岛位于我国最南部.面积约3.39万km2, 地处热带北缘,年平均气温为22~26℃, 年平均降雨量为1600 mm, 地理及生态环境独特。尤其以琼中和白沙县为代表的中部地区, 丛林植被繁茂, 啮齿类食物丰富, 适宜鼠类终年繁殖,鼠种繁多, 鼠密度高[4-5]。其中农田野栖鼠以黄毛鼠(Rattuslosea)、针毛鼠(Niniventer fulvescens)为主要鼠种.农舍家栖鼠以褐家鼠(R. noregicus)和黄胸鼠(R. taneumi)为主要鼠种。同时常年温润的气候导致虫媒种类多且密度大, 是多种鼠传自然疫源性热带传染病的高发区[6-10]。尤其是近年来伴随着城市化进程的加快、旅游观光业特别是探险等生态旅游的发展, 对自然疫源地的过度开发导致啮齿类动物生存环境恶化, 栖息范围突变, 病原体扩大的不确定因素增加, 更易导致新发鼠传疾病和再发传染病的再度暴发[11]。我国尚未开展过针对海南岛等热带地区鼠类携带菌群结构的研究, 作为我国唯一的热带海岛省份, 海南岛鼠类携带菌群结构如何, 是否携带已知病原体和未知新病原,都是亟待解决的问题。
高通量测序是近年来发展很快的--项DNA测序技术,该技术在短短数十年间, 从以Sanger法为代表的一-代测序技术发展到以罗氏454.Ilumina等为代表的二代测序技术[12-13]。在以16S/188 rRNA基因或ITS区域为目标序列的菌群多样性组成谱测序研究中, Ilumina二代测序平台能对单个或连续的两三个可变区序列进行测序分析, 从而获取“高分辨率”的微生物物种信息。目前该技术正逐步应用于菌群微生物组解析中[14-15]。
本研究选取海南岛中部山区典型地理生境野生鼠肠道粪便样本开展高通量测序分析, 明确海南岛中部山区鼠类肠道菌群结构的本底资料, 构建海南岛中部山区鼠类肠道病原菌谱, 为进一步研究鼠传热带传染病及发现未知新病原提供实验室依据, 为海南岛卫生部门预警新发突发鼠传热带传染病, 提前制定相应的热带病预防诊治应急预案提供理论依据, 同时也为我国热带地区应对、预防和控制未来可能发生的动物源新发传染病提供重要的信息资源。
1 材料与方法 1.1 实验材料2017年10月至2018年10月从海南岛中部地区不同地理生境捕获.经PCR检测鼠疫耶尔森菌(Yersinia pestis)为阴性的褐家鼠粪便样本共20份, 用于本课题的研究。无菌解剖取肠道粪便5~10g, 置于2ml甘油脑心脊液中,标记并迅速置于-70 C冻存。
1.2 基因组DNA的提取采用德国QIAGEN公司QLAamp Fast DNA Stool Mini Kit (51604)核酸提取试剂:盒, 提取鼠类粪便样本基因组DNA, 具体操作如下:(1)取180~220 mg粪便样本至2 ml离心管中,置于冰上。(2)加入1 ml inhibit EX Buffer, 连续涡旋1min或直至粪便样本完全均匀。(3)95C恒温水浴孵育5 min, 涡旋15s使其充分混匀。12 000Xg离心3 min, 得上清液。(4)将15 μl protein K加入新的1.5 ml离心管中。(5)将第4步得到的200μl上清液移入含有15 μl protein K的离心管中。(6)加入200 μl Buffer AL涡旋15s后70 C孵育10 min。(7)在裂解液中加人200μl无水乙醇, 涡旋混匀。(8)将步骤7中的600μl裂解液加入QLAamp吸附柱上, 离心1 min, 弃下方收集管, 将QLAamp吸附柱重新置于新的2 ml收集管中, 重复步骤(8), 直至所有的裂解液都被滤过。(9)向QIAamp吸附柱添加500μlBuffer AW1, 离心1 min, 将吸附柱置于新的2 ml收集管中。(10)向QLAamp吸附柱添加500 μl Buffer AW2, 离心3 min, 丢弃收集管中的滤液。(11)将QIAamp吸附柱置于新的2 ml收集管中, 离心3 min。(12)将QlAamp吸附柱转移到一个新的1.5 ml离心管, 加200 μl Buffer TAE在QIAamp膜上。室温静置1 min, 离心1min(14000×g)后滤出DNA。采用NanoDrop 2000超微量分光光度计(北京科誉兴业科技发展有限公司)测量核酸浓度, 提取基因组检查合格后将DNA产物-20 C保存备用。鼠类粪便基因组测序工作由北京诺禾致源生物信息有限公司采用Ther-mofisher的lonS5TMXL进行上机测序完成。
1.3 鼠类粪便样本16SrRNA基因扩增以提取的粪便样本总DNA为模板, 采用16S rRNA Ⅴ3/Ⅴ4区合成特定引物(每对引物带有不同barceode序列), 利用338F (5' -ACTCCTACGGGAGGCAGCA-3')和806R (5' -GGACTACHVGGGTWTCTAAT-3')引物进行PCR扩增。PCR扩增参数如下:94 C预变性5 min, 94 C变性30 s, 55 C退火30s, 72 C复性30 s, 25个循环; 72 C延伸10 min。采用生工生物工程(上海)股份有限公司DNA片段纯试剂盒纯化。文库构建完成后, 测序工作由北京诺禾致源生物信息有限公司采用二代测序平台llumina MiSeq测序技术进行测定。
1.4 数据处理及生物信息学分析对下机数据首先采用llumina Real-Time进行质控, 截除序列末端的低质量区域。然后对获得的双末端序列采用FLASH软件(Version 7.0)进行拼接, 采用QIME软件(Version 19.1)进行进一-步质控。然后对有效的序列信息开展物种分类分析和聚类相关分析。序列相似性>97%, 被聚为1个操作分类单元(operational taxonomie unit, OTU)。根据OTU聚类结果, 进行物种注释、丰度分析及Alpha多样性计算。其中菌群丰度主要由Ace指数和Chaol指数来显示,菌群多样性由Simpson指数和Shannon指数来表示。分别从门水平和属水平上对样本的物种丰度和菌群多样性进行统计。
2 结果采用llumina二代测序平台对16SrDNA序列Ⅴ3-Ⅴ4区进行双端测序, 利用QIIME等软件进行细菌群落结构及多样性分析。共得到原始数据1 684831条序列, 单个样本序列范围为63 877~ 89686, 经过各步质控后, 共得到1569543条有效序列, 单个样本序列范围为60 785~82 606, 平均每个样本78477条序列, 序列平均长度为434 bp。对样本进行聚类分析, 在97%的阈值聚类下, 平均每个样本产生937个OTUs(样本范围为540~1274)。丰富度指数: Ace指数平均值为990.58, 范围为662.52~1 313.97;Chaol指数平均值为994.78.范围为617.58~ 1 330.85。Shannon指数平均值为6.64, 范围为3.70~8.05;Simpson指数平均值为0.96, 范围为0.84~0.99, 每个样本的具体参数见表 1。对97%相似度的OTU采用mothur做稀释曲线, 当测序数量超过49600时, 各样本曲线逐渐趋于平缓, 同时Shannon曲线也趋于平坦。
2.1 门分类水平下菌群结构在门水平上排在前10位的包括厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、变形菌门(Proteobacteria)、疣微菌门(Verrucomicrobia)、柔壁菌门(Tenericutes)、蓝细菌门(Cyanobacteria)、放线菌门(Actinobacteria)、螺旋体门(Spirochaetes)、梭杆菌门(Fusobacteria)和Saccharibacteriad。从门分类水平分析海南岛鼠类肠道菌群, 其中厚壁菌门]所占比例最高, 占55.83%;其次为拟杆菌门占27.74%;随后依次为变形菌门占12.78%,疣微菌门占0.85%, 柔壁菌门占0.81%, 蓝细菌门占0.78%, Saccharibacteria占0.42%, 放线菌门占0.34%,螺旋体门占0.30%, 梭杆菌广占0.15%, 相对丰度见图 1。

|
| 图 1 20份鼠类粪便样本的肠道菌群在门水平.上的相对丰度 Figure 1 Relative abundance of intestinal microbiota from the 20 fecal samples of rodents at the phylum level |
| |
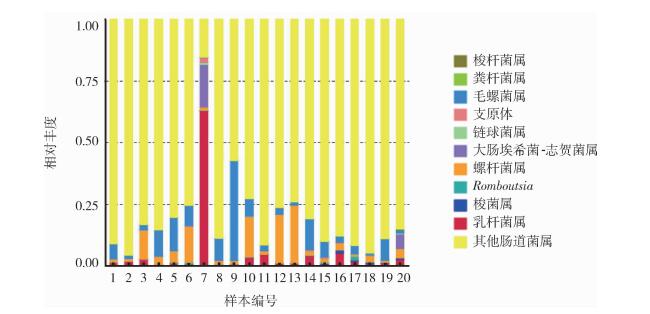
从属分类水平分析海南岛鼠类肠道菌群.排名前10种菌属包括乳杆菌属(Lactobacillus)、螺杆菌属(Helicobacter)、梭菌属(Clostridium)、大肠埃希菌一志贺菌属(Escherichia-Shigella).链球菌属(Streptococcus).毛螺菌属(Lachnospiraceae)、粪杆菌属(Faecalialea)、梭杆菌属(Fusobacterium)、支原体(Mycoplasma)和Rombousia。其中7.11和16号样本肠道菌群中最优势菌属为乳杆菌属, 尤其是7号样本, 乳杆菌属的相对丰度高达62.90%;3.6.10.12和13号样本肠道菌群中最优势菌属为螺杆菌属,其中13号样本螺杆菌的相对丰度达23.50%;4.5.8.9.14和19号样本肠道菌群中最优势菌属为毛螺菌属, 其中9号样本毛螺菌属的相对丰度高达40.40%; 20号样本肠道菌群复杂, 其中最优势菌属为大肠埃希菌-志贺菌属,相对丰度达5.80%, 见图 2。
|
| 图 2 20份鼠类类便样本肠道菌群在属水平上的相对丰度 Figure 2 Relative abundance of intestinal microbiota from the 20 fecal samples of rodents at the genus level |
| |
Alpha多样性是用来分析样本的微生物群落多样性的, 通常用Richness、Chaol、Shannon、Simpson、Dominance和Equitability等指数来评估。其中Richness、Chaol和Shannon 3个指数是常用的评估丰富度的指标, 数值越高表明样品包含的物种丰富度越高[16-18]。本研究中20个鼠类样本测序覆盖率都在99.40%以上, 表明测序结果对海南岛中部鼠类粪便样品中的细菌覆盖率高, 样本中序列未被测出的概率极低, 测序结果合适, 可满足鼠类肠道菌群多样性的分析需求。其中11号样本Shannon指数为8.05, 高出平均值1.41, 表明其肠道菌群多样性最高。7号样本Shannon指数为3.70, 肠道菌群多样性最低,可能的原因是7号样本的取样深度不够。综合表述, 20份褐家鼠样本肠道菌群中OTU平均值为937, Chaol指数平均值为994.78, Shannon指数平均值为6.64。张俊杰等[16]研究表明, 实验小鼠肠道菌群中OTU平均值为336, Chaol指数平均值为384, Shannon指数平均值为3.78。陈双双等[17]研究发现, 舍饲蓝狐肠道菌群中OTU平均值为509, Chaol指数平均值为595, Shannon指数平均值为5.27。与实验小鼠、舍饲蓝狐相比, 海南岛野生鼠的肠道菌群丰度与多样性更高。
从菌群结构分析, 海南岛鼠类肠道菌群中排名前5的优势菌门依次为厚壁菌门、拟杆菌门、变形菌门.疣微菌门和柔壁菌门。薄亭贝和王德华[18]对布氏田鼠(Lasiopodomys brandtii)的肠道菌群研究同样发现其肠道内主要的菌门包括拟杆菌门、厚壁菌门和变形菌门。此外,有学者对野生环境中狼的肠道菌群开展研究, 结果表明其优势菌门及比例依次为厚壁菌门(60.00%)、拟杆菌门(16.90%)、变形菌门(9.20%)、梭杆菌门(9.20%)和放线菌门(4.60%)[19]。对美洲獾的肠道菌群研究表明, 其优势菌门及比例依次为厚璧菌门(73.01%)、拟杆菌门(9.31%).变形菌门(7.44%)、放线菌门(3.08%)和疣微菌门(1. 66%)[20]。由此可见厚壁菌门和拟杆菌门是包括鼠类在内的多种动物肠道共同的优势菌门, 这主要与动物肠道的功能有关, 其中拟杆菌门细菌主要对类固醇、胆汁酸及多糖起代谢作用,如双歧杆菌(Bifidobacterium)作为益生菌, 能够帮助吸收多糖和合成蛋白质, 而厚壁菌门细菌如乳杆菌等主要是对碳水化合物及蛋白质水解发挥作用[21-22]。值得注意的是海南岛鼠类肠道菌群中疣微菌门仅次于3大优势菌门, 占到了0.85%, 这一点与野生环境的狼、豺的肠道菌群差异较大, 与美洲獾的肠道菌群相似, 同时也有研究表明.多纹黄鼠(Lctidomys tridecemlineatus)肠道(尤其是盲肠)中的优势菌门中的疣微菌门, 仅次于厚壁菌门和拟杆菌门, 位于第3位。表明海南野生褐家鼠和多纹黄鼠、美洲獾的肠道菌群有更多相似,与这些动物的生活习性和饮食有关。
从属水平分析表明, 海南岛中部山区鼠类肠道菌群结构复杂, 菌种丰富.不同个体间肠道菌群组成和多样性方面存在差异性, 主要包括乳杆菌属.螺杆菌属、梭菌属、大肠埃希菌-志贺菌属、链球菌属.毛螺菌属、粪杆菌属、梭杆菌属、支原体和Rombousia。值得注意的是3.6.10.12.13和18号样本肠道菌群中最优势菌属为螺杆菌属,其中13号样本螺杆菌的丰度高达23.50%, 螺杆菌属是近年来医学界关注的一一个重要菌属, 以幽门]螺杆菌为代表, 它们的生物学特征、致病潜能及其与人类疾病的关系是近年来研究的热点。而且大多数新螺杆菌属成员都是先在动物及禽类等体内分离出来的, 目前已发现多种螺杆菌能够定植在动物的肝脏、胃黏膜、下肠道, 如Helicobacter hepaticus、H. bilis、H. cinaedi. H. canis、H. Feilis及H. pullorum等[23-24]。2014年Hu等[25]从青藏高原旱獭体内分离到1株螺杆菌新种, 命名为喜马拉雅螺杆菌(Helicobacter himalayana sp. nov.), 其相关的致病性研究正在进行中。由此提示, 在后续研究中, 针对螺杆菌丰度高的这几个样本, 采用微生物培养组学的方法, 参照近缘细菌培养方法, 有导向地进行螺杆菌的分离培养和鉴定, 进一步开展致病性方面的相关研究。
本研究以海南岛中部地区野生鼠为研究对象,采用高通量测序的方法对鼠类肠道菌群开展研究,分析表明海南岛中部山区野生鼠肠道菌群结构复杂,菌种丰富, 有6个样本螺杆菌属丰度较高, 有进一步开展研究的意义和价值。该研究对建立完善的海南岛鼠类样本资源库和病原菌谱提供了本底资料, 并为进一步发现其携带的未知新病原体提供了重要的信息资源和实验室数据, 具有重要意义。
| [1] |
Chang ZR, Lu L, Mao DQ. Dynamics of rodent and rodent-borne disease during construction of the three gorges reservoir from 1997 to 2012[J]. Biomed Environ Sei, 2016, 29(3): 197-204. DOI:10.3967/bes2016.024 |
| [2] |
Yu JM, Li LL, Zhang CY, et al. A novel hepatovirus identified in wild woodchuck Marmota himalayana[J]. Sci Rep, 2016, 6: 22361. DOI:10.1038/srep22361 |
| [3] |
宝福凯. 热带医学与热带病的研究现状、挑战与展望[J]. 昆明医科大学学报, 2012(12): 1-3. F K. Research status, challenges and prospects of tropical medicine and tropical diseases[J]. J Kunming Med Univ, 2012(12): 1-3. DOI:10.3969/j.issn.1003--4706.2012.12.001 |
| [4] |
劳世军, 黄昌和, 王家豪, 等. 海南省鼠疫疫源地调查[J]. 中国热带医学, 2007, 7(9): 1530-1532. S J, Huang CH, Wang JH, et al. Survey of infectious foci of plague in Hainan province[J]. China Trop Med, 2007, 7(9): 1530-1532. DOI:10.3969/j.issn.1009--9727.2007.09.010 |
| [5] |
王弗望, 李鹏. 2014年海南省农区鼠害发生趋势分析及防控建议[J]. 园艺与种苗, 2013, 3(12): 36-38. F W, Li P. Occurence trend and control strategy of rodent damage in Hainan agricultural area[J]. Hortic Seed, 2013, 3(12): 36-38. DOI:10.3969/j.issn.2095--0896.2013.12.012 |
| [6] |
吴多荣, 朱雄, 黄会, 等. 海南地区类鼻疽伯克霍尔德菌PFGE分子分型及同源性分析[J]. 中国病原生物学杂志, 2014, 9(6): 516-518, 537. D R, Zhu X, Huang H, et al. Pulsed-field gel electrophoresis molecular typing and homology analysis of Burkholderia pseudomallei in Hainan province[J]. J Pathog Biol, 2014, 9(6): 516-518, 537. DOI:10.13350/j.cjpb.140610 |
| [7] |
Zhang CC, Yang HM, Li XW, et al. Molecular typing of pathogenie Leptospira serogroup icterohaemorrhagiae strains circulating in China during the past 50 years[J]. PLoS Negl Trop Dis, 2015, 9(5): e0003762. DOI:10.1371/journal.pntd.0003762 |
| [8] |
Malania L, Bai Y, Osikowicz LM, et al. Prevalence and diversity of Bartonella species in rodents from Georgia (Caucasus)[J]. Am J Trop Med Hyg, 2016, 95(2): 466-471. DOI:10.4269/ajtmh.16-0041 |
| [9] |
林碧瑚, 孙晓娟, 詹志农, 等. 海南岛立克次体血清学和病原学调查报告[J]. 海南医学院学报, 2003, 9(5): 257-260. B H, Sun XJ, Zhan ZN, et al. Investigation of serology and etiology of ricsttia on Hainan Island[J]. J Hainan Med Coll, 2003, 9(5): 257-260. DOI:10.3969/j.issn.1007-1237.2003.05.001 |
| [10] |
林碧瑚, 詹志农, 王华民, 等. 海南岛恙虫病立克次体研究[J]. 海南医学院学报, 2003, 9(5): 261-264. B H, Zhan ZN, Wang HM, et al. Investigation of rickettstia tsutsugamushi on Hainan Island[J]. J Hainan Med Coll, 2003, 9(5): 261-264. DOI:10.3969/jissn.1007-1237.2003.05.002 |
| [11] |
Beckley CS, Shaban s, Palmer GH, et al. Disaggregating tropical disease prevalence by climatic and vegetative zones within tropical west Africa[J]. PLoS One, 2016, 11(3): e0152560. DOI:10.1371/journal.pone.0152560 |
| [12] |
范忠原, 刘晗璐, 王峰, 等. 基于高通量测序技术研究水貂远端肠道细菌群落组成[J]. 微生物学通报, 2016, 43(1): 123-130. Z Y, Liu HL, Wang F, et al. Determination of bacterial community composition in the distal gut of mink (Mustela vison)by high-throughput sequencing[J]. Microbiol China, 2016, 43(1): 123-130. DOI:10.13344/j.microbiol.china.150173 |
| [13] |
石聪, 李世瑞, 李跑, 等. 基于高通量测序浏阳豆豉不同发酵阶段微生物多样性分析[J]. 食品与发酵工业, 2018, 44(2): 27-32, 39. Shi C, Li SR, Li P, et al. Analysis of microbial diversity in different fermented stages of Liuyang Douchi based on high throughput sequencing[J]. Food erment Ind, 2018, 44(2): 27-32, 39. DOI:10.13995/j.cnki.11-1802/ts.015773 |
| [14] |
曲巍, 张智, 马建章, 等. 高通量测序研究益生菌对小鼠肠道菌群的影响[J]. 食品科学, 2017, 38(1): 214-219. Qu W, Zhang Z, Ma JZ, et al. Effect of probiotics on gut microbiota in mice evaluated by high-throughput sequencing[J]. Food Sci, 2017, 38(1): 214-219. DOI:10.7506/spkx1002-6630-201701036 |
| [15] |
杨俊花, 赵志辉, 郭文博, 等. 应用llumina-MiSeq高通量测序技术分析脱氧雪腐镰刀菌烯醇对小鼠肠道菌群的影响[J]. 动物营养学报, 2017, 29(1): 158-167. J H, Zhao ZH, Guo WB, et al. Effects of deoxynivalenol on intestinal microbiota of mice analyzed by llumina-MiSeq high-throughput sequencing technology[J]. Chin J Anim Nutr, 2017, 29(1): 158-167. DOI:10.3969/j.issn.1006-267x.2017.01.018 |
| [16] |
张俊杰, 张淑静, 董瑞娟, 等. 高通量测序研究中药经方对正常小鼠肠道菌群的影响[J]. 世界中医药, 2019, 14(5): 1123-1132, 1138. J J, Zhang SJ, Dong RJ, et al. Effects of traditional Chinese medicine prescriptions on the intestinal flora of normal mice via high-throughput sequencing[J]. World Chin Med, 2019, 14(5): 1123-1132, 1138. DOI:10.3969/j.issn.1673-7202.2019.05.014 |
| [17] |
陈双双, 司华哲, 穆琳琳, 等. 高通量测序技术分析蓝狐肠道菌群多样性[J]. 动物营养学报, 2008, 30(10): 4071-4080. s S, Si HZ, Mu LL, et al. Analysis of intestinal microbiota diversity of blue foxes by high-throughput sequencing technology[J]. Chin J Anim Nutr, 2008, 30(10): 4071-4080. DOI:10.3969/j.issn.1006-267x.2018.10.032 |
| [18] |
薄亭贝, 王德华. 鼠肠道中的微生物王国[J]. 大自然, 2017(4): 24-25. T B, W ang DH. Microbial kingdom in rat intestines[J]. ChinaNat, 2017(4): 24-25. DOI:10.3969/j.issn.0255-7800.2017.04.007 |
| [19] |
Zhang HH, Chen L. Phylogenetic analysis of 16S rRNA gene sequences reveals distal gut bacterial diversity in wild wolves(Canis lupus)[J]. Mol Biol Rep, 2010, 37(8): 4013-4022. DOI:10.1007/s11033-010-0060-z |
| [20] |
Wu XY, Zhang HH, Chen J, et al. Comparison of the fecal microbiota of dholes high-throughput llumina sequencing of the Ⅴ3 - Ⅴ4 region of the 16S rRNA gene[J]. Appl Microbiol Biotechnol, 2016, 100(8): 3577-3586. DOI:10.1007/s00253-015-7257-y |
| [21] |
Backhed F, Ley RE, Sonnenburg JL, et al. Host-bacterial mutualism in the human intestine[J]. Science, 2005, 307(5717): 1915-1920. DOI:10.1126/science.1104816 |
| [22] |
Xu J, Bjursell MK, Himrod J, et al. A genomic view of the human-Bacteroides thetaiotaomicron symbiosis[J]. Science, 2003, 299(5615): 2074-2076. DOI:10.1126/science.1080029 |
| [23] |
Roth L, King JM, Tennant BC. Hepatic lesions in woodchucks (Marmota monax) seronegative for woodchuck hepatitis virus[J]. J Wildl Dis, 1991, 27(2): 281-287. DOI:10.7589/0090-3558--27.2.281 |
| [24] |
Fox JG, Shen ZL, Xu SL, et al. Helicobacter marmotae sp. nov.isolated from livers of woodchucks and intestines of cats[J]. J.Clin Microbiol, 2002, 40(7): 2513-2519. DOI:10.1128/JCM.40.7.2513-2519.2002 |
| [25] |
Hu SK, Jin D, Lu S. Helicobacter himalayensis sp. nov.isolated from gastric mucosa of Marmota himalayana[J]. Int JSyst Evol Microbiol, 2015, 65(6): 1719-1725. DOI:10.1099/ijs.0.000163 |
 2020, Vol. 31
2020, Vol. 31


