扩展功能
文章信息
- 康央, 李庆多, 苗娇娇, 宋秀平, 徐爱玲, 张雯, 栗冬梅
- KANG Yang, LI Qing-duo, MIAO Jiao-jiao, SONG Xiu-ping, XU Ai-ling, ZHANG Wen, LI Dong-mei
- 应用微生物数据分析云平台筛选巴尔通体特异基因及设计探针
- Screening for Bartonella-specific genes and designing probes using Microbial Data Analysis Cloud Platform
- 中国媒介生物学及控制杂志, 2020, 31(5): 526-530
- Chin J Vector Biol & Control, 2020, 31(5): 526-530
- 10.11853/j.issn.1003.8280.2020.05.005
-
文章历史
- 收稿日期: 2020-04-20
2 天津市耀华中学, 天津 300040;
3 3山东第一医科大学, 山东 泰安 271016
2 Tianjin Yaohua Middle School;
3 Shandong First Medical University
特异基因是在生物体中进行特异性表达的基因,该基因的表达通常会赋子生物体特殊的形态结构特征和特异的生理功能,因此特异基因常被用于物种鉴定及功能研究等方面。目前查找特异基因的方法主要有2种:①文献法, 通过已发表文献中记录的特异基因序列编号查找; ②基因组比较法, 首先在美国国立生物技术信息中心(NCBI)或GenBank上下载该目标菌基因组序列及与目标菌分类学上相近的菌株基因组序列, 然后运用序列比对软件进行比对, 找出目标菌种的特有序列。上述2种方法被科研人员广泛应用于特异基因的筛选、致病机制、药物靶点和诊断等研究, 但操作费时费力,效率低, 检索结果不全面。
利用中国疾病预防控制中心传染病预防控制所的微生物数据分析云平台(https://analysis.mypathogen.org)中的Special_marker_gene工作流可以高通量筛选细菌的特异基因。本研究应用Special_marker_ gene查找仅在巴尔通体中存在、在其他细菌中不存在的基因, 基于这些候选基因筛选出巴尔通体属水平特异性引物和探针, 用于实时荧光定量PCR (fluorescence quantitative PCR, qPCR)检测巴尔通体, 可用于今后相关疾病的分子流行病学调查、宿主媒介携带病原体的检测与监测和临床诊断。
1 材料与方法 1.1 菌株、生物样品的核酸和基因组序列基因组DNA包括:汉赛巴尔通体(Bartonella henselae, Bh)、杆菌样巴尔通体(B. bacilliformis, Bb)、五日热巴尔通体(B.quintana, Bq)、格拉汉姆巴尔通体(B. grahamii, Bg)、文森巴尔通体伯格霍夫亚种(B. vinsonii subsp. berkhffii, Bvb)、伊丽莎白巴尔通体. (B. elizabethae, Be)、科勒巴尔通体(B. koehlerae, Bk)、克氏巴尔通体(B. clarridgeiae, Bc)、道志巴尔通体(B.doshiae, Bd)、文森巴尔通体文森亚种(B. vinsonii subsp. vinsonii, Bv)、文森巴尔通体阿鲁潘亚种(B. vinsonii subsp. arupensis, Bva)、特利波契巴尔通体(B.tribocorum, Bt)、根癌土壤杆菌(Agrobacterium tumefaciens, At)、牛氏布鲁氏菌(Brucella abortus, Ba)、金黄色葡萄球菌(Staphyloccocus aureus, Sa)、大肠埃希菌(Escherichia coli)、加氏乳杆菌(Lactobacillus gassei, Ig)、杀鲑气单胞菌(Aeromonas salmonicida, As)、长角血蜱. (Haemaphysalis longicornis,HI)、猫栉首蚤(Ctrnocephalides canis,Cc)、白纹伊蚊(Aedes albopictus, Aal).蝙蝠、鼠、猫、犬、猕猴和人, 均为中国疾病预防控制中心传染病预防控制所媒介生物控制室保存。本研究所用基因组序列信息见表 1。
qPCR仪CFX96(美国BioRad)和FAST7500(美国应用生物系统ABI公司)、全自动核酸提取仪AU1001(北京百泰克生物技术有限公司)。HR qPCR Master Mix(.上海辉睿生物科技有限公司)、2 × TransTaq®-T PCR SuperMix(+ dye)(北京全式金生物技术有限公司)、Wizard® Genomic DNA Purification Kit(北京普洛麦格生物技术有限公司)。
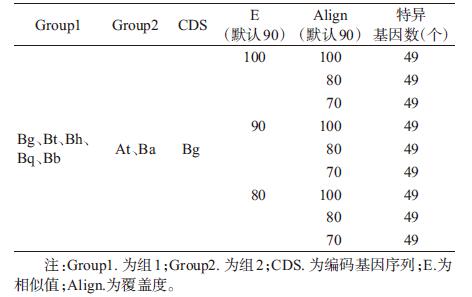
1.3 靶基因的筛选筛选步骤如下:第1步, 在NCBI的GenBank数据库(fp://ftp.ncbi.nlm.nih.gov/genbank/genomes/Bacteria)中下载分析所需要的细菌基因组序列和编码基因序列(codingsequences, CDS), .上传至云平台数据库; 第2步, 选择工作流中“Special_Marker_Gene"程序(图 1), 点击“运行”; 第. 3步, 进人工作流界面, 输人文件,Group1栏上传目标细菌的基因组序列Bg.Bt、Bh、Bb和Bq的基因组序列; Group2栏上传与目标细菌近源的细菌基因组序列: Ba和At基因组序列(Group1和Group2上传内容可以互换); CDS栏.上传Bg菌株基因编码序列作为参考基因组(表 2); 第4步, 设置参数Align(覆盖度)值分别为100.90和80, 系统默认值为90;设置参数E(相似度)值分别为100、80和70, 系统默认值为70, 点击“运行任务”。最后, 在“任务/报告中”下载结果报告。根据筛选结果编号在上传的CDS序列中找出特异基因序列。为进一步确认筛选基因的特异性, 可将筛选序列片段用NCBI-BLAST进行验证。
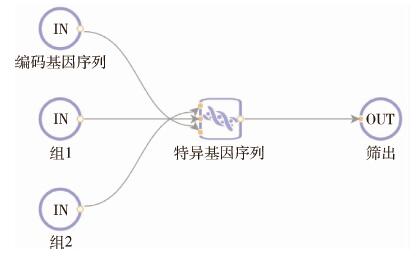
|
| 图 1 Special Marker Gene工作原理示意图 Figure 1 Schematic diagram of working principle of Special Marker Gene |
| |
将筛选所得序列用Beacon Designer软件[1-2)设计qPCR引物和TaqMan探针。用靶基因序列合成质粒标准品。引物、探针和质粒标准品合成与纯化由北京擎科新业生物技术有限公司完成。
1-4-2 通用性检测以12种巴尔通体基因组DNA为模板扩增, 检测引物和探针在巴尔通体属水平的通用性。
1.4.3 特异性检测以布鲁氏菌和根癌土壤杆菌等其他非巴尔通体属细菌及人、鼠、羊、猫和犬基因组DNA为模板, 检测引物和探针特异性。
1.4.4 敏感性检测将质粒标准品进行10倍系列稀释为3.47 × 100~7拷贝/反应, 以此为模板, 进行qPCR, 确定引物、探针的检测灵敏性。
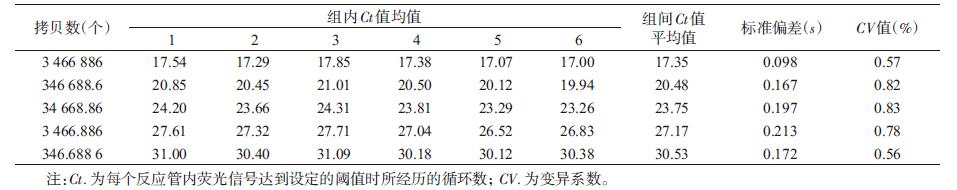
1.4.5 重复性检测以不同稀释度的质粒标准品为模板进行qPCR检测, 在同一-次PCR反应中,每个稀.释度标准品设置6个重复, 分析组内差异; 并且非同日重复上述实验6次, 分析组间差异。计算变异系数(cofficient of variation, CV), CV=标准偏差(s)平均数(x)。
1.4.6 扩增效率检验以质粒标准品为扩增模板, 测定初始浓度, 换算为拷贝数浓度(拷贝/μl)=(质量/相对分子质量) × 6.02 × 1023。质粒和含有重组子的阳性甘油菌分别保存于-20℃和-70℃。
标准曲线制备:将质粒标准品原液按10X系列稀释, 使浓度达到3.47 × 100~7拷贝/反应进行qPCR, 同时做6孔平行样。
根据6次重复性检测结果汇总建立引物探针的标准曲线, 确定引物探针的检测效率。
1.4.7 反应体系及参数以Bg标准菌株基因组DNA为模板, 反应体系20 μl:HR mix 10 μl, 正反向引物(10 μmol/L)各0.8 μl, 荧光标记探针(10 μmol/L) 0.8μl, DNA模板2 μl, 无菌水补齐。扩增程序: 94 C 5 min; 94℃ 15s, 60℃ 45s, 40个循环。
2 结果 2.1 靶基因筛选与引物设计不同参数设置得到的筛选结果一致, 均为49个特异基因, 以此作为巴尔通体检测靶标的候选基因库供进一-步筛选和验证。将此49个特异基因NCBI-BLAST后, 选择覆盖度和特异性水平高的6个基因序列设计引物和探针。见表 3。
以Bg基因组DNA为模板, 巴尔通体属通用qPCR探针ssrA-T[3]为阳性对照实验(表 4)。经预实验分析,以Bg基因组中的yfeC基因(ACS50472.1_ 79)为靶序列设计的Bar5引物和TaqMan探针可以扩增12种巴尔通体, 通用性较好(图 2)。
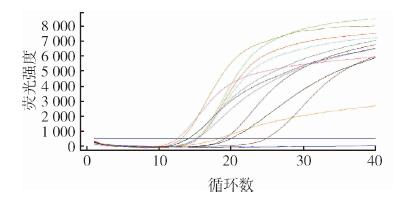
|
| 注:不同颜色扩增曲线为表1中不同种巴尔通体。 图 2 应用Bar5T探针qPCR扩增12种巴尔通体 Figure 2 Amplification of 12 Bartonella species by qPCR with Bar5T probe |
| |
将Bar5引物和探针序列在NCBI-BLAST中进行特异性搜索, 未检测到非特异性结合位点。
Bg菌株扩增为阳性, 检测布鲁氏菌等其他细菌和动物及人类核酸样品均未出现荧光信号, 扩增结果为阴性, 空白对照NTC(no template control)为阴性。
2.3 敏感性检测用10倍系列稀释的质粒标准品(3.47 × 100~7拷贝/反应)为模板, 引物和探针浓度均为400 nmol/L进行定量PCR, 当模板浓度为3.47 × 101拷贝/反应时,CFX96扩增反应的循环阈值(cycle threshold value, Ct值)为36, FAST7500扩增Ct值为34, 检测结果稳定, 为检出限, 图 3为CFX96扩增图。
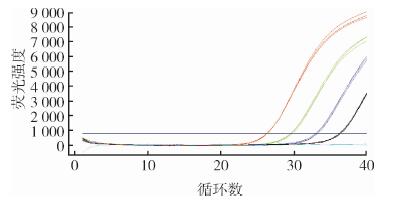
|
| 注:红、绿、蓝和黑色扩增曲线分别对应DNA模板浓度为3.47× 101~4. 图 3 Bar5T探针的敏感性实验扩增结果 Figure 3 Amplification results of sensitivity test of qPCR with Bar5T probe |
| |
质粒标准品在3.47X 102~6浓度范围时,组内孔间CV值在0.02%~2.27%之间, 组间重复测定CV值在0.56%~0.83%之间。见表 5。
扩增效率模板浓度之间呈现良好的线性关系(相关系数R2为1.00), 扩增效率E为101%。因此,依据标准曲线公式y=-3.30x + 38.86和Ct值可以较为准确的计算出样品中目的基因的拷贝数(图 4)。
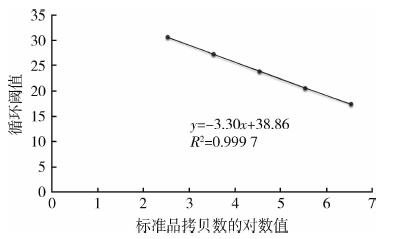
|
| 图 4 应用标准曲线检测PCR扩增效率 Figure 4 Determination of PCR amplification efficiency by standard curve |
| |
巴尔通体是革兰阴性小杆菌, 属于人类新发传染病病原体, 分类学位置在变形菌纲根瘤菌目巴尔通体科[4], 目前有35个种及3个亚种, 其中17种致病菌可引发猫抓病和心内膜炎等多种疾病。因此,建立灵敏、特异、快速的巴尔通体检测方法, 是解决临床诊断、流彳病学调查、疾病监测等重要前提。
为便于对巴尔通体的检测和监测展开调查, 利用该平台筛选特异基因, 设计引物和探针并进行qPCR实验验证。应用多种生物体核酸进行检测均无扩增信号,表明该检测序列的特异性良好。在敏感性检测中最低检测限为3.47X 10拷贝/反应,灵敏度较高, 能够满足临床诊断需要。经重复性评估实验, 得到组内和组间的CV值分别为0.02%~2.27%和0.56%~0.83%, 进一步说明此方法重复性良好,结果可靠。绘制标准曲线显示有良好的线性关系和扩增效率, 保证了结果的稳定性。因此, 本研究建立的实时荧光定量TaqMan探针法可为由巴尔通体引起的一系列疾病的早期临床诊断、疾病监测和筛查以及流行病学调查提供一种高效的实验室检测手段。
本研究利用微生物数据分析云平台中“Special_marker_gene"工作流筛选巴尔通体特异基因, 与运用MEGA 7.0软件传统生物信息学工具相比, 该平台具有以下优势:(1)操作简便快速。以巴尔通体为例, 从上传数据到检出特异序列只需约15 min, 而用MEGA 7.0软件至少需要30 min, 结果分析费时费力。该平台筛选结果以表格呈现, 筛选结果包含了特异序列的基因编号、名称、表达蛋白及位置等详细信息, 清晰直观, 便于用户进行后续筛选研究。(2)数据处理便捷。平台用户通过账号登陆, 可以建立个人常用数据库, 能随时随地在客户端进行数据处理和共享。通过平台日志记录, 确保实验数据的可追溯性, 使研究成果更具说服力。(3)数据分析能力增强。该平台可满足用户多任务同时处理及大数据量分析的需求, 有效提高了工作效率。当然, 该平台也存在一些不足, 如进行特异基因筛选时, Align和E2个参数值的改变对结果无影响。因此参数敏感性有待提高。
此外, 作为在线生物信息分析系统, 微生物数据分析云平台将数据库和多种生物信息工具进行整合开发, 为用户提供序列比对、进化树构建、数据.统计等功能, 该平台的出现有效填补了国内大型公共卫生生物数据库的空白[5], 目前在国内多用于基于序列比对的细菌分型鉴定的方法优化、微生物组间差异分析及预测微生物的致病性与流行潜力等方面[6-10], 本研究应用该平台成功查找并设计出巴尔通体特异性引物探针用于病原体检测, 为其他病原体同类研究提供操作范例, 也为应用该平台完成特异基因的搜索和评估应用提供相关经验。在国外尚未见该平台的推广利用。随着高通量测序时代的到来,微生物组学数据呈现暴发式增长, 大数据的管理与分析已成为必须, 该平台的构建将极大地促进生物信息学在微生物研究领域的应用, 为科学研究工作提供有力的支持与帮助。
| [1] |
谢甜, 罗陆婷, 尹庆庆, 等. 蚊寨卡病毒核酸检测体系的建立与初步应用[J]. 中国病原生物学杂志, 2017, 12(9): 811-817, 824. Xie T, Luo LT, Yin QQ, et al. Development of a system to detect the nucleic acids of Zika virus and its preliminary[J]. J Parasitic Biol, 2017, 12(9): 811-817, 824. DOI:10.13350/j.cjpb.170901 |
| [2] |
Chen QH, Huang JF, Fu WL, et al. Hybridization fluorescence microscopy of ethambutol-resistant embB gene mutation[J]. Chin J Nosocomiol, 2010, 20(11): 1509-1512. |
| [3] |
Diaz MH, Bai Y, Malania L, et al. Development of a novel genus-specific real-time PCR assay for detection and differentiation of Bartonella species and genotypes[J]. J Clin Microbiol, 2012, 50(5): 1645-1649. DOI:10.1128/JCM.06621-11 |
| [4] |
徐建国. 现场细菌学[M]. 北京: 科学出版社, 2011: 698-708. Xu JG. Site bacteriology[M]. Beijing: Science Press, 2011: 698-708. |
| [5] |
于伟文, 杜鹏程, 陈超, 等. 微生物在线生物信息分析系统的开发及应用[J]. 疾病监测, 2012, 27(4): 316-319. Yu WW, Du PC, Chen C, et al. Development and application of online bioinformatics analysis system for pathogenic microorganism[J]. Dis Surveill, 2012, 27(4): 316-319. DOI:10.3784/j.issn.1003-9961.2012.4.020 |
| [6] |
陈晨, 彭珂, 王海印, 等. 基于16S rDNA数据库的细菌在线分类鉴定平台的构建[J]. 疾病监测, 2013, 28(3): 236-240. Chen C, Peng K, Wang HY, et al. An online platform SSUDB:database of bacteria identification and classification with 16S rDNA[J]. Dis Surveill, 2013, 28(3): 236-240. DOI:10.3784/j.issn.1003-9961.2013.3.019 |
| [7] |
Zhang W, Du PC, Zheng H, et al. Whole-genome sequence comparison as a method for improving bacterial species definition[J]. J Gen Appl Microbiol, 2014, 60(2): 75-78. DOI:10.2323/jgam.60.75 |
| [8] |
Han N, Yu WW, Qiang YJ, et al. T4SP database 2.0:an improved database for type IV secretion systems in bacterial genomes with new online analysis tools[J]. Comput Math Methods Med, 2016, 9415459. DOI:10.1155/2016/9415459 |
| [9] |
Han N, Qiang YJ, Zhang W. ANItools web:a web tool for fast genome comparison within multiple bacterial strains[J]. Database, 2016, 2016: baw084. DOI:10.1093/database/baw084 |
| [10] |
Chen C, Zhang W, Zheng H, et al. Minimum core genome sequence typing of bacterial pathogens:a unified approach for clinical and public health microbiology[J]. J Clin Microbiol, 2013, 51(8): 2582-2591. DOI:10.1128/JCM.00535-13 |
 2020, Vol. 31
2020, Vol. 31