扩展功能
文章信息
- 姚丹丹, 姜洪雪, 隋晶晶, 冯志勇
- YAO Dan-dan, JIANG Hong-xue, SUI Jing-jing, FENG Zhi-yong
- 广东省江门市捕获鼠类的DNA条形码分析
- DNA barcoding analysis of rodents captured in Jiangmen, Guangdong province, China
- 中国媒介生物学及控制杂志, 2020, 31(3): 305-309
- Chin J Vector Biol & Control, 2020, 31(3): 305-309
- 10.11853/j.issn.1003.8280.2020.03.012
-
文章历史
- 收稿日期: 2019-12-25
DNA条形码技术是利用生物体基因组内一段短而标准的DNA序列作为标记来快速、准确和自动化鉴定物种的方法[1]。自2003年Hebert等[2]首次提出DNA条形码的概念以来, 至今已在鸟类、鱼类、昆虫、海洋生物、哺乳动物等多个生物类群的研究中得到了广泛应用[3-5]。目前用于条形码研究的分子标记基因主要是线粒体细胞色素C氧化酶亚基Ⅰ(COⅠ)[6], 线粒体细胞色素B(Cytb)和16S rRNA基因也常作为动物界物种鉴定的DNA条形码[7]。
鼠形动物DNA条形码的研究近年来发展较快。Lu等[8]采用DNA条形码技术研究了海南省啮齿动物的分类, 并对部分鼠种的分类地位进行了修订。Zhang等[9]基于线粒体Cytb和核基因对我国及越南地区鼠科白腹鼠属(Niviventer)的鼠类进行了种类界定和系统发育关系分析, 并通过该方法找到了更多的隐藏种。郭丽民等[10]对甘肃省鼠疫疫源地的啮齿动物进行了DNA条形码分析, 结果表明DNA条形码可用于啮齿动物标本的准确鉴定, 能更好地辅助研究啮齿类的分类及进化关系。此外, 该技术还被用于特殊环境中的啮齿动物鉴定、博物馆标本鉴定和检验检疫[11-12]。DNA条形码不受个体形态特征限制和个体发育阶段影响, 对于非成熟期、形态特征破损或残缺物种, 濒危珍稀动物的无创伤鉴别以及区分近缘种和隐存种等都具有绝对优势, 是形态学鉴定的有力补充[13]。本研究采用COⅠ基因对广东省江门市捕获的鼠类进行分类鉴定, 并与形态学鉴定结果进行比较, 为鼠形动物的多样性研究及鼠害防治积累基础资料。
1 材料与方法 1.1 样品的采集2017年12月在广东省江门市新会区野外农田捕获45只鼠, 根据鼠类的形态特征进行初步鉴定均为黄毛鼠(Rattus losea), 为进一步确定捕获鼠的种类, 分别取少许肝脏或肌肉保存于无水乙醇中, 编号JXS101-122、JXS201-223, 放置4 ℃冰箱备用。
1.2 鼠基因组DNA的提取采用MiniBEST Universal Genomic DNA Extraction Kit Ver.5.0提取鼠肝脏或肌肉总DNA, 4 ℃保存备用。
1.3 COⅠ基因的扩增与测序选用通用引物BatL5310:5′-CCTACTCRGCCATTTTACCTATG-3′和R6036R:5′-ACTTCTGGGTGTCCAAAGAATCA-3′由生工生物工程(上海)股份有限公司扩增线粒体COⅠ基因片段。PCR反应体系为:DNA模板1 μl, 引物各1 μl, Premix Taq 25 μl, 加ddH2O至50 μl, PCR反应条件:95 ℃预变性5 min;95 ℃变性30 s, 55 ℃退火30 s, 72 ℃延伸1 min, 35个循环;最后72 ℃延伸10 min。PCR产物经1%琼脂糖凝胶电泳检测, 送测序公司进行测序。Premix Taq、DNA Marker、上样缓冲液和Gold View核酸染料均购自宝生物工程(大连)有限公司。
1.4 COⅠ基因序列分析与系统发育树的构建采用Lasergene 5.0软件查看测序结果的峰图, 并对序列进行人工校正。同时将测定的序列在美国国立生物技术信息中心(NCBI)网站上进行BLAST核酸序列同源性检索, 以确保所获得的序列是目标序列。采用DnaSP 6.0软件分析变异位点数目, 并对序列进行单倍型分析[6]。从GenBank中选取与目的片段核苷酸序列同源性较高的序列, 运用MEGA 5.1软件构建Neighbor-Joining(NJ)系统发育树, 基于Kimura- 2-parameter(K2P)模型计算遗传距离。
2 结果 2.1 COⅠ基因扩增与假基因甄别45只鼠样本利用通用引物均成功扩增出750 bp左右的片段, 与目标片段的大小一致(图 1、2), 经过测序共获得45条COⅠ基因序列, 每条序列长度约710 bp, 为了避免核基因组中的假线粒体基因序列干扰, 对所有序列的开放阅读框进行确认, 未出现碱基缺失和插入现象, 能全部翻译成氨基酸序列。

|
| 注:M. 2 000 bp DNA Marker;1~22.分别代表样本JXS101~122。 图 1 样本编号JXS101-122细胞色素C氧化酶亚基Ⅰ基因的扩增结果 Figure 1 Amplification results of cytochrome c oxidase subunit I gene of sample JXS101-122 |
| |

|
| 注:M. 2 000 bp DNA Marker;1~23.分别代表样本JXS201~223。 图 2 样本编号JXS201-223细胞色素C氧化酶亚基Ⅰ基因的扩增结果 Figure 2 Amplification results of cytochrome c oxidase subunit I gene of sample JXS201-223 |
| |
将测序获得的序列与GenBank中其他鼠类的DNA条形码进行BLAST比对, 发现8只鼠样本存在外部形态鉴定错误, 样本编号JXS103、JXS105、JXS222、JXS215和JXS218 COⅠ基因序列与黄胸鼠(R. tanezmi, 序列号KY605369.1)相似度高达99.8%, 确认为黄胸鼠;样本编号为JXS116和JXS217与褐家鼠(R. norvegicus, 序列号KT763036.1)相似度达99.7%, 说明这2个样本实际应为褐家鼠幼体;样本编号JXS214与黑缘齿鼠(R. andamanensis, 序列号KC010294.1)的同源性达99.6%, 修订为黑缘齿鼠。
对45只鼠样本的COⅠ基因进行序列分析, 发现遗传相似度在89.5%~100%之间, 37只黄毛鼠样本遗传相似度在98.8%~100%之间, 5只黄胸鼠样本和2只褐家鼠样本的相似度均为100%, 黑缘齿鼠与黄毛鼠、黄胸鼠和褐家鼠的相似度分别为90.8%~91.2%、91.0%和89.5%。
2.3 DNA条形码分析对获得的45条序列进行比对分析发现有131个变异位点, 其中119个是系统发育信息位点, 单倍型分析结果显示有12个不同的单倍型。应用NJ邻接法构建系统发育树, 自检值设为1 000次, NJ树显示同一种鼠类的个体聚为一支, 支持率均为100%, 能清楚地区分不同鼠种(图 3)。
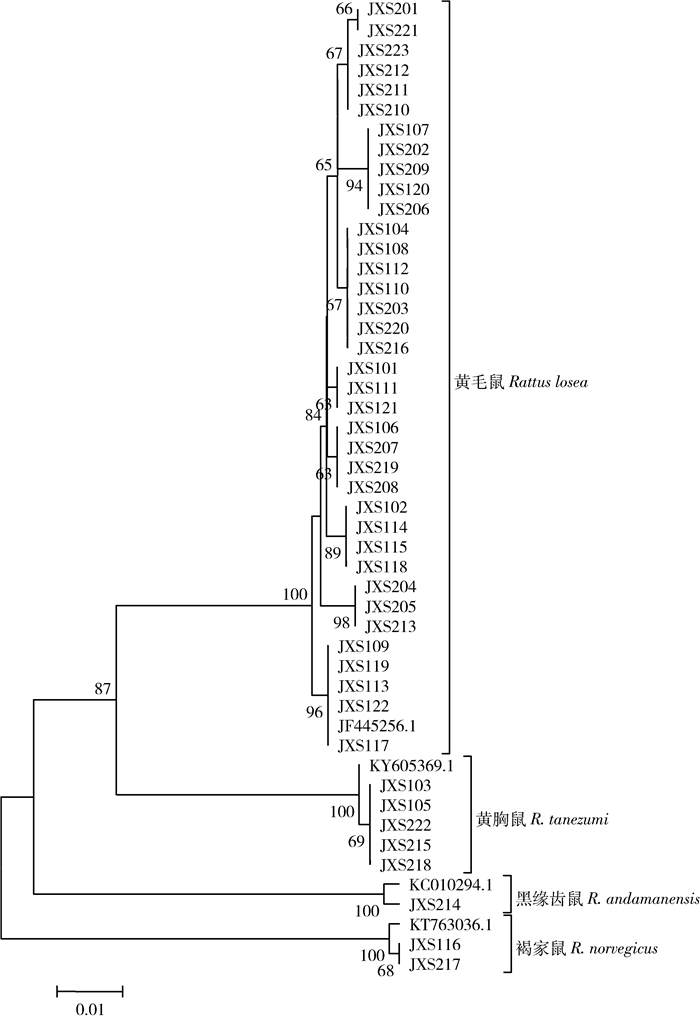
|
| 图 3 基于K2P模型构建的邻接法系统发育树 Figure 3 Neighbor-Joining phylogenetic tree based on K2P model |
| |
利用双参数法分析不同个体间的遗传距离, 黄毛鼠种内遗传距离为0~1.2%, 样本JXS214与黑缘齿鼠(KC010294.1)的遗传距离为0.5%;不同鼠种间遗传距离为7.1%~11.6%, 平均遗传距离(10.1±0.7)%, 种间平均遗传距离为种内平均遗传距离的16.8倍, 种间遗传距离最小的为黄毛鼠与黄胸鼠, 是种内平均遗传距离的11.8倍, 最大的为褐家鼠与黑缘齿鼠。
3 讨论对广东省江门市新会区农田捕获的45只鼠样本的分析结果显示, 8只鼠样本的DNA条形码鉴定结果与形态学鉴定存在差异, 经广东省农业科学院植物保护研究所冯志勇研究员复核, 其中5只鼠重新鉴定为黄胸鼠, 另外2只鼠重新鉴定为幼年褐家鼠, 另有1只鼠因COⅠ基因序列与黑缘齿鼠(KC010294.1)的同源性高达99.6%、遗传距离<1%而被鉴定为黑缘齿鼠。Hebert等[14]研究发现, 同属各种COⅠ序列的平均差异程度为11.3%, 种内的COⅠ序列差异程度基本<2%, 绝大多数<1%, 本研究结果与此结论相符。
黄毛鼠、黄胸鼠和褐家鼠为广东省农区常见鼠种, 黑缘齿鼠[15]为广东省偶见种, 又名锡金家鼠, 属于啮齿目鼠科大鼠属, 因其形态和生态与黄毛鼠、黄胸鼠有相似之处在以往常被误判为其他近缘种, 近年来随着分子检测技术的发展, 该鼠种陆续在我国广东、云南、海南省等地发现。Chen等[16]曾在广东省增城市农田捕获到黑缘齿鼠, 并研究了该鼠异性求偶的叫声特征。Liu等[17]研究发现该鼠种最显著的形态特征是腹毛为白色。关于黑缘齿鼠的分类地位, Lu等[8]认为屋顶鼠海南亚种(R. rattus hainanicus)和屋顶鼠滇西亚种(R. rattus sladeni)与黑缘齿鼠是同一种鼠。由于该鼠的研究报道很少且不容易捕到, 具体的分类特征如头骨、牙齿等和进化关系有待后续跟踪研究。
DNA条形码可以精确地获得物种的遗传信息, 其应用前景非常广阔, 尤其是在缺乏完整标本、甚至仅有粪便或毛发的情况下, 优点十分突出[18]。与传统的物种鉴定方法相比, 该方法简便快捷, 可操作性强, 准确性和可重复性高, 无需丰富的鉴定经验, 同时便于新物种的发现, 加快分类学科的发展。但是仅依靠DNA序列比对进行分子鉴定仍然存在一定的风险, 主要是因为GenBank收录的条形码基因序列尚不完整, 许多物种还无法通过DNA条形码准确鉴定;其次, 提交到GenBank中的一部分序列可能未经过分类学家的鉴定, 因此其物种分类地位可能不准确[18];此外, 不同类群内物种间的遗传距离可能不同, 而现有的物种判断阈值可能会对物种多样性造成一定的误判[19]。Ermakov等[20]对欧亚地区松鼠的研究结果显示, 12种松鼠能够通过DNA条形码进行准确鉴定, 4种需结合形态特征进行鉴定, 其中短尾黄鼠(Spermophilus brevicauda)和大黄鼠(S. major)由于线粒体DNA遗传渗入和杂交种间遗传距离<1%, 如果按照条形码种间和种内遗传距离的鉴别标准, 这2种鼠很容易被误判为同一种鼠。因此, 为保证物种鉴定的准确性, 应将形态鉴定和分子生物学技术有机地结合起来, 同时采用多基因标记技术, 从而快速准确地鉴别物种。
| [1] |
董石, 尹玲, 杨天天. DNA条形码在动植物检测中的应用[J]. 国土与自然资源研究, 2019(1): 81-82. Dong S, Yin L, Yang TT. Application of DNA barcode in animal and plant detection[J]. Terr Nat Resour Study, 2019(1): 81-82. DOI:10.16202/j.cnki.tnrs.2019.01.021 |
| [2] |
Hebert PDN, Cywinska A, Ball SL, et al. Biological identifications through DNA barcodes[J]. Proc Roy Soc B, 2003, 270(1512): 313-321. DOI:10.1098/rspb.2002.2218 |
| [3] |
Schäffer S, Zachos FE, Koblmüller S. Opening the treasure chest:a DNA-barcoding primer set for most higher taxa of central European birds and mammals from museum collections[J]. PLoS One, 2017, 12(3): e0174449. DOI:10.1371/journal.pone.0174449 |
| [4] |
Proshek B, Dupuis JR, Engberg A, et al. Genetic evaluation of the evolutionary distinctness of a federally endangered butterfly, Lange's Metalmark[J]. BMC Evol Biol, 2015, 15: 73. DOI:10.1186/s12862-015-0354-9 |
| [5] |
Gong SH, Ding YF, Wang Y, et al. Advances in DNA barcoding of toxic marine organisms[J]. Int J Mol Sci, 2018, 19(10): 2931. DOI:10.3390/ijms19102931 |
| [6] |
李争光, 丛林, 王大伟, 等. 黑龙江地区农田害鼠发生特点及DNA条形码鉴定技术的应用[J]. 植物保护, 2018, 44(6): 145-151, 167. Li ZG, Cong L, Wang DW, et al. Species identification of main pest rodents by DNA barcoding and their occurrence characteristics from croplands in Heilongjiang province[J]. Plant Prot, 2018, 44(6): 145-151, 167. DOI:10.16688/j.zwbh.2018001 |
| [7] |
Nicolas V, Schaeffer B, Missoup AD, et al. Assessment of three mitochondrial genes (16S, Cytb, COⅠ) for identifying species in the praomyini tribe (Rodentia:Muridae)[J]. PLoS One, 2012, 7(5): e36586. DOI:10.1371/journal.pone.0036586 |
| [8] |
Lu L, Chesters D, Zhang W, et al. Small mammal investigation in spotted fever focus with DNA-barcoding and taxonomic implications on rodents species from Hainan of China[J]. PLoS One, 2012, 7(8): e43479. DOI:10.1371/journal.pone.0043479 |
| [9] |
Zhang B, He K, Wan T, et al. Multi-locus phylogeny using topotype specimens sheds light on the systematics of Niviventer (Rodentia, Muridae) in China[J]. BMC Evol Biol, 2016, 6(1): 261. DOI:10.1186/s12862-016-0832-8 |
| [10] |
郭丽民, 席进孝, 盖永志, 等. 甘肃省鼠疫疫源地啮齿动物DNA条形码分析[J]. 中国媒介生物学及控制杂志, 2019, 30(2): 176-179. Guo LM, Xi JX, Gai YZ, et al. Application of DNA barcoding in the identification of rodent species in plague foci in Gansu province, China[J]. Chin J Vector Biol Control, 2019, 30(2): 176-179. DOI:10.11853/j.issn.1003.8280.2019.02.014 |
| [11] |
陈利伟, 赵志亮, 邱德义, 等. DNA条形码技术在兰州中川机场鼠类鉴定中的应用[J]. 中国国境卫生检疫杂志, 2018, 41(2): 102-105. Chen LW, Zhao ZL, Qiu DY, et al. Application of DNA barcoding in classification of rodents at Lanzhou Zhongchuan airport area[J]. Chin J Front Health Quar, 2018, 41(2): 102-105. DOI:10.16408/j.1004-9770.2018.02.008 |
| [12] |
Ndiaye A, Tatard C, Stanley W, et al. Taxonomic hypotheses regarding the genus Gerbillus (Rodentia, Muridae, Gerbillinae) based on molecular analyses of museum specimens[J]. ZooKeys, 2016, 566: 145-155. DOI:10.3897/zookeys.566.7317 |
| [13] |
金圣浩, 鲁亮, 闫东, 等. DNA条形码技术在河北省坝上地区夜行鼠鉴定中的应用[J]. 中国媒介生物学及控制杂志, 2015, 26(1): 55-57. Jin SH, Lu L, Yan D, et al. Application of DNA barcoding technique in identification of nocturnal rodent species in Bashang area of Hebei province, China[J]. Chin J Vector Biol Control, 2015, 26(1): 55-57. DOI:10.11853/j.issn.1003.4692.2015.01.014 |
| [14] |
Hebert PDN, Ratnasingham S, de Waard JR. Barcoding animal life:cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proc Roy Sci B, 2003, 270(S1): S96-99. DOI:10.1098/rsbl.2003.0025 |
| [15] |
蒋志刚, 马勇, 吴毅, 等. 中国哺乳动物多样性[J]. 生物多样性, 2015, 23(3): 351-364. Jiang ZG, Ma Y, Wu Y, et al. China's mammalian diversity[J]. Biodivers Sci, 2015, 23(3): 351-364. DOI:10.17520/biods.2014202 |
| [16] |
Chen Y, Su QQ, Qin J, et al. Call divergence in three sympatric Rattus species[J]. J Acoust Soc Am, 2017, 142(1): 29-34. DOI:10.1121/1.4990022 |
| [17] |
Liu SY, He K, Chen SD, et al. How many species of Apodemus and Rattus occur in China? a survey based on mitochondrial cytb and morphological analyses[J]. Zool Res, 2018, 39(5): 309-320. DOI:10.24272/j.issn.2095-8137.2018.053 |
| [18] |
何锴, 王文智, 李权, 等. DNA条形码技术在小型兽类鉴定中的探索:以甘肃莲花山为例[J]. 生物多样性, 2013, 21(2): 197-205. He K, Wang WZ, Li Q, et al. DNA barcoding in surveys of small mammal community:a case study in Lianhuashan, Gansu province, China[J]. Biodivers Sci, 2013, 21(2): 197-205. DOI:10.3724/SP.J.1003.2013.09160 |
| [19] |
马英, 李海龙, 鲁亮, 等. DNA条形码技术在青海海东地区小型兽类鉴定中的应用[J]. 生物多样性, 2012, 20(2): 193-198. Ma Y, Li HL, Lu L, et al. Application for identification of small mammals by DNA barcoding in Haidong area, Qinghai province, China[J]. Biodivers Sci, 2012, 20(2): 193-198. DOI:10.3724/SP.J.1003.2012.06188 |
| [20] |
Ermakov OA, Simonov E, Surin VL, et al. Implications of hybridization, NUMTs, and overlooked diversity for DNA barcoding of Eurasian ground squirrels[J]. PLoS One, 2015, 10(1): e0117201. DOI:10.1371/journal.pone.0117201 |
 2020, Vol. 31
2020, Vol. 31


