扩展功能
文章信息
- 朱晗婷, 吴美丽, 周鹰, 吴冰, 崔玉宝
- ZHU Han-ting, WU Mei-li, ZHOU Ying, WU Bing, CUI Yu-bao
- 粉尘螨长链脂肪酸转运蛋白基因克隆及序列分析
- Cloning and sequencing of the long-chain fatty acid transporter gene of Dermatophagoides farinae
- 中国媒介生物学及控制杂志, 2020, 31(2): 169-174
- Chin J Vector Biol & Control, 2020, 31(2): 169-174
- 10.11853/j.issn.1003.8280.2020.02.010
-
文章历史
- 收稿日期: 2019-10-17
- 网络出版时间: 2020-03-10 16:11
2 无锡市儿童医院, 江苏 无锡 214023
2 Wuxi Children's Hospital
目前,超过50%的过敏性疾病是由家庭尘螨(house dust mites,HDM)引起,这是一种常见的吸入性过敏原[1],可引发包括过敏性哮喘、过敏性鼻炎和过敏性皮炎等过敏性疾病,影响了30%~40%的世界人口,并且部分引发的过敏性疾病甚至会威胁生命[2]。尘螨的肠道、粪便、表皮和分泌物等均含有过敏原[3-4],目前已命名共有37个HDM过敏原组。室内尘螨尤其是屋尘螨(Dermatophagoides pteronyssinus)和粉尘螨(D. farinae),在引起过敏性疾病中占90%[5],2006年2月至2007年3月,我们采用皮肤点刺试验对我国西部、东部、西南和南部沿海17座城市6 304例哮喘或/和鼻炎门诊患者的大规模调查表明,粉尘螨阳性率为59.00%、屋尘螨阳性率为57.60%、热带无爪螨(Blomia tropicalis)阳性率为40.70%,分别占测试过敏原的第1、2、3位[6]。说明尘螨是引起哮喘的最重要过敏原。
粉尘螨属疥螨目(Sarcoptiformes)、羽螨总科(Analgoidea)、无气门股(Astigmata)、麦食螨科(Pyroglyphidae),是我国室内优势螨种之一。本课题组在前期工作中对粉尘螨进行了转录组测序,基因注释发现1个基因片段序列与长链脂肪酸转运蛋白(long-chain fatty acid transport protein,FATP)具有高度相似性。本文用反转录-聚合酶链式反应(RT-PCR)技术对该序列进行全长扩增并通过构建质粒进行测序验证,并用生物信息学软件预测其理化性质。
1 材料与方法 1.1 材料BamHΙ酶、XhoΙ酶、RNAiso Plus(Code.9108 & 9109)、High Fidelity Prime ScriptTMRT- PCR Kit(Code DR027A)、Tks Gflex DNA Polymerase(Code.R060A)、DNA Purification Kit Ver.2.0(Code. 9762)、MiniBEST Agarose Gel DNA Extraction Kit Ver.4.0(Code. 9762)、大肠埃希菌感受态细胞JM109(Escherichia coli JM109 Competent Cell)(Code.9052)均购自宝生物工程(大连)有限公司。In-FusionⓇ HD Cloning Kit(Clontech Code No. 639633)购自Clontech公司。pET28a(+) Simple Victor (Kit Lot No. N72770)由Novagen公司生产,其他试剂均为国产分析纯。
1.2 方法首先以粉尘螨总RNA为模板,根据转录组测序结果设计并合成引物,利用RT-PCR技术获得该基因全长,并在合成的片段两侧添加BamHΙ/XhoΙ酶切位点,与pET28a(+)载体连接,将连接产物热转化至大肠埃希菌感受态细胞JM109中,涂布平板,37 ℃过夜培养,挑取阳性克隆菌,对重组质粒测序,然后用生物信息学软件预测其理化性质。
1.2.1 粉尘螨总RNA提取挑取尘螨制备匀浆,使用RNAiso Plus(Code.9108 & 9109)按照说明书具体步骤提取螨虫Total RNA。
1.2.2 反转录以Total RNA为模板,使用3'-Full RACE Core Set with PrimeScriptTM RTase(Code No. 6106)进行反转录试验合成cDNA,同时设立M-MLV(-)对照。取Eppendorf管1支,依次加入总RNA 1 μl、各10 mmol/L dNTP Mixture 1 μl、20 μmol/L Random 6 mers 1 μl和RNase Free dH2O 4.50 μl,70 ℃水浴10 min后,立即置冰上放置2 min,然后加入5×Prime Script RT Buffer 2 μl、40 U/μl RNase Inhibitor 0.25 μl、200 U/μl Reverse Transcriptase M-MLV (RNase H-) 0.50 μl、RNase Free dH2O 7.50 μl,水浴30 ℃ 10 min,42 ℃ 60 min,70 ℃ 15 min。
1.2.3 PCR扩增全长目的基因根据目的基因编码区,由宝生物工程(大连)有限公司合成如下两段引物分别为F/R和F1/R1。正向引物F:5'-AATGGGT CGCGGATCCATGGGTGCTGGTAT GGGTAT-3'和反向引物R:5'-GGTGGTGGTGCTCGAGCTAAAACC GTATCG TACCA T-3'。正向引物F1:5'-TGCAAG TACCTGGAGCATGT-3'和反向引物R1:5'-CGTTT CGACTTCTGTTGTCG-3'。分别使用TaKaRa Tks Gflex DNA Polymerase(Code.R060A)进行PCR扩增,PCR反应总体积50 μl,具体组成如下:上述反转录反应液2 μl,2×Gflex PCR Buffer(Mg2+,dNTP plus)25 μl,Tks Gflex DNA Polymerase (1.25 U/μl) 1 μl、20 pmol/μl正向引物F及反向引物R各1 μl、dH2O 20 μl。反应条件:94℃变性1 min后,98℃ 10 s、60℃ 15 s条件下进行30个循环,68 ℃ 1 min延伸,最终获取PCR产物。取此PCR产物进行二次PCR,反应体积50 μl,组成如下:上述PCR产物1 μl、2×Gflex PCR Buffer(Mg2+,dNTP plus)25 μl、Tks Gflex DNA Polymerase (1.25 U/μl)1 μl、正向引物F (20 pmol/μl)1 μl、反向引物R1(20 pmol/μl)1 μl、dH2O 20 μl。反应条件:94 ℃变性1 min后,98 ℃ 10 s、60 ℃ 15 s条件下进行30个循环,68 ℃ 1 min延伸。取5 μl PCR产物上样电泳,观察目的条带。使用TaKaRa MiniBEST Agarose Gel DNA Extraction Kit Ver.4.0(Code.9762)切胶回收上述PCR产物获得目的片段FATP。
1.2.4 克隆质粒的构建及鉴定测序用BamHⅠ和HindⅢ对pET28a(+)载体进行酶切,反应体积50 μl,组成如下:pET-28a(+) 10 μl、10×K Buffer 5 μl、15 U/μl BamHⅠ1.50 μl、15 U/μl Hind Ⅲ 1.50 μl、dH2O 32 μl,反应条件:37℃ 16 h,取5 μl进行1%琼脂糖凝胶电泳,切胶回收大片段,使用In-FusionⓇHD Cloning Kit(Clontech Code.639633),将FATP PCR产物和pET28a(+)DNA连接,连接产物命名为pET28a(+)-FATP,连接体系及条件如下:(50 ng/μl)pET28a(+)DNA 1 μl、(50 ng/μl)FATP PCR产物2 μl、5×In-Fusion HD Enzyme Premix 2 μl、dH2O 5 μl,50 ℃ 15 min。将连接产物取1 μl热转化至大肠埃希菌感受态细胞JM109中,涂布平板,37 ℃过夜培养,挑取阳性克隆植菌,提取质粒命名为pET28a(+)-FATP,委托宝生物工程(大连)有限公司对上述质粒测序。
1.3 生物信息学分析利用Expasy Web(http://www.expasy.org/)服务器上的翻译工具确定粉尘螨FATP的氨基酸序列;其理化性质由expasy在线网址中的ProtParam tool(https://web.expasy.org/protparam/)和ProtScale(https://web.expasy.org/protscale/)预测。SingaIP 5.0()进行FATP基因信号肽序列分析。TMpred软件进行跨膜区分析,用GOR4进行氨基酸二级结构分析。在美国国立生物技术信息中心(NCBI)搜索螨及昆虫脂肪酸转运蛋白序列,Clustal Omega(https://www.ebi.ac.uk/Tools/msa/clustalo/)在线分析其保守及半保守氨基酸序列区并利用MEGA-X软件中的Neighbor-Joining (NJ)分析粉尘螨FATP与其他螨类及昆虫FATP的同源性。
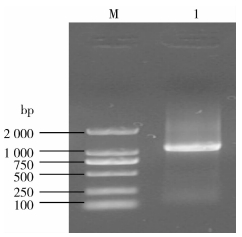
2 结果 2.1 核酸序列克隆测序从粉尘螨中分离总RNA,用RT-PCR从粉尘螨总RNA中扩增全长基因片段,构建原核表达质粒pET28a(+)-FATP,随后进行测序分析。经2次扩增的PCR产物经琼脂糖凝胶电泳见1条约为1 071 bp条带,与理论值相符(图 1)。将回收的PCR产物克隆到pET28a(+)质粒中转化于大肠埃希菌感受态细胞JM109中。选择阳性克隆质粒,进行序列验证,结果与预测结果一致。该基因全长由起始密码子ATG到终止密码子TAG结止,其对应翻译的编码氨基酸序列见图 2。
|
| 注:M. DL-2000 DNA标准品;1.粉尘螨脂肪酸转运蛋白RT-PCR产物。 图 1 粉尘螨脂肪酸转运蛋白反转录-聚合酶链式反应产物琼脂糖凝胶电泳结果 Figure 1 Agarose gel electrophoresis of RT-PCR products of fatty acid transport protein from Dermatophagoides farinae |
| |

|
| 图 2 粉尘螨脂肪酸转运蛋白的核苷酸测序结果及其推导的氨基酸序列 Figure 2 Nucleotide and deduced amino acid sequences of fatty acid transport protein of Dermatophagoides farinae |
| |
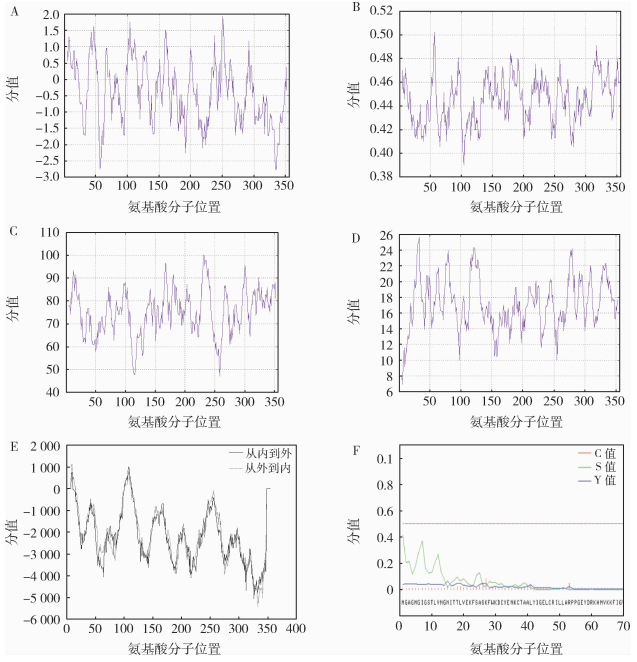
从起始密码子ATG到终止密码子TAG共1 071 bp,(G+C)为31.84%,溶解温度为74.60 ℃。利用Expasy在线软件对该核酸序列翻译分析发现,该基因编码由356个氨基酸组成的蛋白质,相对分子质量为41 085.20,理论等电点为8.57,在体外半衰期为30 h;该蛋白不稳定系数是25.76,性质稳定。其平均亲水性(grand average of hydropathicity,GRAVY)为-0.39,脂肪指数(aliphatic index)为77.72。平均柔软指数(average flexibility index)示第57位氨基酸处最高为0.50,第104位氨基酸处最低为0.39。TMpred软件分析发现该编码蛋白存2处较明显的跨膜区段,一个是由外向内的跨膜区位于1~20位氨基酸,一个是由内向外的跨膜区在100~116位氨基酸。信号肽分析发现不存在信号肽结构,见图 3。GOR4软件分析发现该蛋白属于混合型蛋白,其中α-螺旋(由91个氨基酸组成,占25.56%)、延伸主链(包含82个氨基酸,占23.04%)和无规则卷曲(由183个氨基酸组成,占51.40%),见图 4。将推导出的氨基酸序列进行结构域(http://www.ebi.ac.uk/InterProscan)在线分析,发现该蛋白存在一磷酸腺苷(AMP)结合位点。
|
| 注:A.亲水性分析(ProtScale软件);B.平均柔性指数分析(ProtScale软件);C.相对可变性分析(ProtScale软件);D.折射率分析(ProtScale软件);E.拓扑分析(TMpred软件);F.信号肽预测(SignaIP 5.0软件)。 图 3 粉尘螨脂肪酸转运蛋白氨基酸序列生物信息学分析 Figure 3 Bioinformatics analysis of amino acid sequence of fatty acid transport protein of Dermatophagoides farinae |
| |

|
| 注:h. α-螺旋;e.延伸主链;c.无规则卷曲;分析软件为GOR4。 图 4 粉尘螨脂肪酸转运蛋白二级结构分析结果 Figure 4 Secondary structure analysis of fatty acid transport protein of Dermatophagoides farinae |
| |
将粉尘螨的FATP序列输入到NCBI网站(http://www.ncbi.nlm.nih.gov/)检索找出大红斑蝶(Danaus plexippus)、二斑叶螨(Tetranychus urticae)、沙雷果蝇(Drosophila serrata)、巴氏果蝇(D. busckii)、粉尘螨、家蚕(Bombyx mori)、屋尘螨、疥螨(Sarcoptes scabiei)的FATP蛋白序列,并通过Clustal Omega在线比对与粉尘螨FATP序列的相似性,分析发现他们之间存在54个保守氨基酸、25个半保守氨基酸,构建分子进化树发现粉尘螨FATP与家蚕FATP构成一簇,并与屋尘螨FATP 1同源性较近,见图 5。

|
| 注:Der f.粉尘螨;Bom m.家蚕;Der p.屋尘螨;Sar s.疥螨;Dro b.巴氏果蝇;Dro s.沙雷果蝇;Dan p.大红斑蝶;Tet u.二斑叶螨。 图 5 粉尘螨脂肪酸转运蛋白与其他螨以及昆虫类同源氨基酸序列的分子进化树 Figure 5 Phylogentic tree derived from fatty acid transport protein of Dermatophagoides farinae and its homologous amino acid sequences of other mites and insect species |
| |
长链脂肪酸(long chain fatty acids,LCFAs)是细胞代谢重要的能量来源,LCFAs需要通过细胞膜上的如脂肪酸转位酶、LCFAs辅酶CoA合成酶和脂肪酸转运蛋白家族(fatty acid transport proteins,FATPs)等蛋白介导转运系统才能通过细胞膜进入胞内。其中FATPs又称溶质运载蛋白家族27(solutecarrier family 27,Slc27)[7-8],最初由Schaffer和Lodish[9]通过克隆表达技术在小鼠的3T3-L1脂肪细胞cDNA文库中发现。FATPs作为一种多功能载体蛋白,具有参与脂肪酸的转运及调节细胞内脂肪酸代谢的作用[10-13]。目前已发现在人类及鼠的基因组FATPs家族中已确定的有6种类型(FATP1-6),在哺乳动物中具有不同的组织定位及功能[14-16]。但关于尘螨脂肪酸转运蛋白基因及蛋白研究甚少,在尘螨体内具体作用机制不明。
本研究采用RT-PCR成功获得粉尘螨FATP蛋白的编码基因。生物信息学分析发现粉尘螨FATP的基因片段由1 071个碱基对组成,编码由356个氨基酸组成的蛋白质,该蛋白分子质量为41 085.20,理论等电点为8.57。分析预测序列表明它是一个稳定的具有一定亲水性的蛋白质。进一步分析发现该蛋白存在2个跨膜区域,不存在信号肽结构。二级结构是一般的三维结构蛋白质局部片段的形式,二级结构FATP由α-螺旋(25.56%)、延伸主链(23.04%)和无规则卷曲(51.40%)组成。将推导出的氨基酸序列输入interProcan中发现该蛋白存在AMP合酶结合位点。通过对FATPs家族的氨基酸序列分析发现FATPs高度保守的300个氨基酸长度序列的起始端有1个AMP结合位点序列的存在,而许多酶催化的反应都涉及到蛋白的AMP结合位点和腺嘌呤核苷三磷酸(ATP)依赖性的AMP结合,并且AMP结合区域对与FATP介导的脂肪酸转运非常重要[17],与LCFAs的吸收和结合有关[18-19]。通过对不同物种的FATPs全长序列与其他含有AMP结合位点的蛋白序列比对,分析结果指出FATP与其他各种酶都不具有同源性。本文前期预测的粉尘螨FATP的氨基酸只有少部分在胞外,大部分在胞内,并且存在AMP结合位点,为此后进一步研究尘螨体内脂肪酸转运代谢机制奠定基础。
利用Clustal Omega软件在线分析与粉尘螨长链脂肪酸转运蛋白的保守氨基酸、半保守氨基酸序列等,并利用MEGA-X软件构建分子进化树,结果发现粉尘螨FATP与家蚕同源性较近,与屋尘螨脂肪酸转运蛋白1亲缘性相对较近,其他则相对较远,推测脂肪酸转运机制可能类似于脂肪酸转运蛋白家族中1的功能。
综上所述,本研究成功获取了粉尘螨脂肪酸转运蛋白序列,生物信息学分析对于制备生产重组物,可为后续尘螨体内脂肪酸代谢生理机制研究及开发尘螨控制措施奠定基础。
| [1] |
Chan TF, Ji KM, Yim AKY, et al. The draft genome, transcriptome, and microbiome of Dermatophagoides farinae reveal a broad spectrum of dust mite allergens[J]. J Allergy Clin Immunol, 2015, 135(2): 539-548. DOI:10.1016/j.jaci.2014.09.031 |
| [2] |
Patel S, Meher BR. A review on emerging frontiers of house dust mite and cockroach allergy research[J]. Allergol Immunopathol (Madr), 2016, 44(6): 580-593. DOI:10.1016/j.aller.2015.11.001 |
| [3] |
Thomas WR, Hales BJ, Smith WA. House dust mite allergens in asthma and allergy[J]. Trends Mol Med, 2010, 16(7): 321-328. DOI:10.1016/j.molmed.2010.04.008 |
| [4] |
Banerjee S, Resch Y, Chen KW, et al. Der p 11 is a major allergen for house dust mite-allergic patients suffering from atopic dermatitis[J]. J Invest Dermatol, 2015, 135(1): 102-109. DOI:10.1038/jid.2014.271 |
| [5] |
Moingeon P. Progress in the development of specific immunotherapies for house dust mite allergies[J]. Exp Rev Vacc, 2014, 13(12): 1463-1473. DOI:10.1586/14760584.2014.948861 |
| [6] |
Li J, Sun B, Huang Y, et al. A multicentre study assessing the prevalence of sensitizations in patients with asthma and/or rhinitis in China[J]. Allergy, 2009, 64(7): 1083-1092. DOI:10.1111/j.1398-9995.2009.01967 |
| [7] |
Anderson CM, Stahl A. SLC27 fatty acid transport proteins[J]. Mol Aspects Med, 2013, 34(2/3): 516-528. DOI:10.1016/j.mam.2012.07.010 |
| [8] |
Kazantzis M, Stahl A. Fatty acid transport proteins, implications in physiology and disease[J]. Biochim Biophys Acta, 2012, 1821(5): 852-857. DOI:10.1016/j.bbalip.2011.09.010 |
| [9] |
Schaffer JE, Lodish HF. Expression cloning and characterization of a novel adipocyte long chain fatty acid transport protein[J]. Cell, 1994, 79(3): 427-436. DOI:10.1016/0092-8674(94)90252-6 |
| [10] |
Chiu CY, Chang TC, Liu SH, et al. The regulatory effects of fish oil and chitosan on hepatic lipogenic signals in high-fat diet-induced obese rats[J]. J Food Drug Anal, 2017, 25(4): 919-930. DOI:10.1016/j.jfda.2016.11.015 |
| [11] |
Rodríguez-Cruz M, González RS, Maldonado J, et al. The effect of gestational age on expression of genes involved in uptake, trafficking and synthesis of fatty acids in the rat placenta[J]. Gene, 2016, 591(2): 403-410. DOI:10.1016/j.gene.2016.06.034 |
| [12] |
SeeBle J, Liebisch G, Schmitz G, et al. Palmitate activation by fatty acid transport protein 4 as a model system for hepatocellular apoptosis and steatosis[J]. Biochim Biophys Acta, 2015, 1851(5): 549-565. DOI:10.1016/j.bbalip.2015.01.004 |
| [13] |
Dourlen P, Sujkowski A, Wessells R, et al. Fatty acid transport proteins in disease:new insights from invertebrate models[J]. Prog Lipid Res, 2015, 60: 30-40. DOI:10.1016/j.plipres.2015.08.001 |
| [14] |
Mishima T, Miner JH, Morizane M, et al. The expression and function of fatty acid transport protein-2 and -4 in the murine placenta[J]. PLoS One, 2011, 6(10): e25865. DOI:10.1371/journal.pone.0025865 |
| [15] |
Gallardo D, Amills M, Quintanilla R, et al. Mapping and tissue mRNA expression analysis of the pig solute carrier 27A (SLC27A) multigene family[J]. Gene, 2013, 515(1): 220-223. DOI:10.1016/j.gene.2012.11.029 |
| [16] |
Black PN, Ahowesso C, Montefusco D, et al. Fatty acid transport proteins:Targeting FATP2 as a gatekeeper involved in the transport of exogenous fatty acids[J]. Medchemcomm, 2016, 7(4): 612-622. DOI:10.1039/C6MD00043F |
| [17] |
Hu XY, Wang YF, Sheikhahmadi A, et al. Effects of glucocorticoids on lipid metabolism and AMPK in broiler chickens'liver[J]. Comp Biochem Physiol B Biochem Mol Biol, 2019, 232: 23-30. DOI:10.1016/j.cbpb.2019.02.001 |
| [18] |
Stremmel W, Ehehalt R, Herrmann T, et al. Role of FATP in parenchymal cell fatty acid uptake[J]. Biochim Biophys Acta, 2003, 33: 81-87. DOI:10.1016/S1569-2558(03)33005-X |
| [19] |
Doege H, Stahl A. Protein-mediated fatty acid uptake:novel insights from in vivo models[J]. Physiology (Bethesda), 2006, 21(4): 259-268. DOI:10.1152/physiol.00014.2006 |
 2020, Vol. 31
2020, Vol. 31


