扩展功能
文章信息
- 栗冬梅, 梁艳林, 宋秀平, 朱彩英, 康央
- LI Dong-mei, LIANG Yan-lin, SONG Xiu-ping, ZHU Cai-ying, KANG Yang
- Chelex 100树脂快速提取动物组织基因组DNA方法的评估
- Evaluation of Chelex 100 resin method for rapid extraction of genomic DNA from animal tissues
- 中国媒介生物学及控制杂志, 2019, 30(3): 286-291
- Chin J Vector Biol & Control, 2019, 30(3): 286-291
- 10.11853/j.issn.1003.8280.2019.03.013
-
文章历史
- 收稿日期: 2019-02-11
- 网络出版时间: 2019-4-23 16:06
2 山东第一医科大学, 山东 泰安 271016;
3 山东大学生命科学学院, 山东 济南 250100
2 Shandong First Medical University;
3 School of Life Sciences, Shandong University
近年来,人类面临新发突发传染病流行、生物恐怖袭击和外来生物入侵严重威胁。禽流感、埃博拉、登革热和中东呼吸综合征等传染病接踵而至,扩散迅速,引发全球公共卫生危机。从已知到未知病原体,从宿主动物到传播媒介再到易感人群,加强病原微生物流行与变异监测及现场快速检测研究对于传染病预防和控制已势在必行[1]。对病原体的现场检测和连续监测,需考虑采用操作简单、成本低、对样品要求低的检测技术,现场检测最好是快速、无设备条件限制的方法。目前较为普及和实用的方法是对生物样品中的靶核酸分子的检测,包括单重或多重PCR/实时荧光定量PCR(quantitative real-time PCR,qPCR)、各种等温扩增技术[2]以及近年来新兴的生物传感器和微流控技术[3]。检测病原体的标识性核酸分子,对于生物组织样品的高效处理是前提和基本要求,简便快速、灵敏特异的检测方法首先取决于样品的处理方法。在众多核酸提取方法中,目前应用较多的是硅胶膜法、磁珠法,还有较传统的酚氯仿抽提法和十六烷基三甲基溴化铵(cetyltrimethylammonium bromide,CTAB)抽提法等,操作均需要温浴、转管、清洗和离心等步骤,所需仪器试剂较多,操作时间一般在40 min以上,操作步骤多、耗时长、样品需要量大和交叉污染是上述这些方法的主要问题。为筛选适用于采样现场、大量操作或病原体长期监测所用的核酸提取方法,本研究选择Chelex 100树脂作为基本试剂,采用不同处理方法提取动物组织核酸,通过观察核酸浓度、纯度和PCR/qPCR扩增结果对这一方法进行评估。Chelex 100是一种由苯乙烯-二乙烯基苯共聚体组成的化学螯合树脂,包含配对亚氨基二乙酸离子,作为螯合基团能够螯合多价金属离子,在碱性环境下煮沸可裂解细胞释放DNA,并通过结合Mg2+失活脱氧核糖核酸酶(Deoxyribonucleases,DNases),防止DNA降解。一般来说,煮沸后的Chelex 100-DNA混合物较为稳定,可在4 ℃保存3~4个月[4]。Chelex 100树脂在法医鉴定中应用较多,用于毛发、组织、骨骼、石蜡包埋组织以及血样痕量检材的DNA提取,所提模板用于下游的法医DNA分型。近年来,也有用于细菌、真菌和昆虫等其他生物DNA的提取,所提模板用于病原体检测和物种鉴定等[5-11]。
1 材料与方法 1.1 主要仪器设备与试剂CFX96荧光定量PCR仪(BioRad公司,美国)、SensoQuest Labcycler PCR仪(圣欧,德国)、全自动核酸提取仪AU1001(百泰克生物技术有限公司,北京)、超微量分光光度计NanoDrop 1000(Thermo公司,美国)、GelDoc XR+全自动凝胶成像系统(BioRad公司,美国)、G100高通量组织研磨仪(卡尤迪生物科技有限公司,北京)、MB-102恒温振荡金属浴(博日科技有限公司,杭州)、旋转混匀器TR-02U(捷美电子有限公司,苏州)。
Chelex 100树脂(Cat.1422832,BioRad公司,美国)、磁珠法组织基因组DNA提取试剂盒AU19014(百泰克生物技术有限公司,北京)、HR qPCR Master Mix(辉睿生物科技有限公司,上海)、2×TransTaq-T PCR SuperMix(全式金生物技术有限公司,北京)。
1.2 组织样品选择6份巴尔通体感染阳性、野外采集的啮齿动物肝组织样品,作为核酸处理方法的实验样品(表 1)。分离培养和qPCR检测结果均为阳性,判断为组织感染巴尔通体。结果判断方法分别为:应用gltA引物[12]扩增培养菌落的水煮DNA模板,电泳出现379 bp条带进行测序验证为巴尔通体培养阳性;应用qPCR检测肝组织核酸(磁珠法组织基因组DNA提取试剂盒AU19014提取),循环值(quantification cycle,Cq)≤35.00时,判断为肝组织巴尔通体核酸阳性。
剪取-80 ℃冻存的上述6份肝组织样品4~10 mg,按照试剂盒AU19014操作说明,应用全自动核酸提取仪AU1001提取全基因组DNA,测定浓度。
1.3.2 Chelex 100树脂法用灭菌去离子水配制5%的Chelex 100悬液,pH值在10.0~11.0之间。吸取Chelex 100悬液时,持续混匀以保证树脂颗粒均匀分布在溶液中,使用1 000 μl枪头,保证组织裂解时工作液含有5%的Chelex 100树脂,离心管内终容量为200 μl。
剪取-80 ℃冻存的上述6份肝组织样品4~6 mg/样品×24,分别用6种不同的处理方法(C1~C6)提取基因组DNA(表 2),每种方法做4个平行样。举例:样品ML44剪取24份组织,每份4~6 mg,每4份分别用于C1~C6。
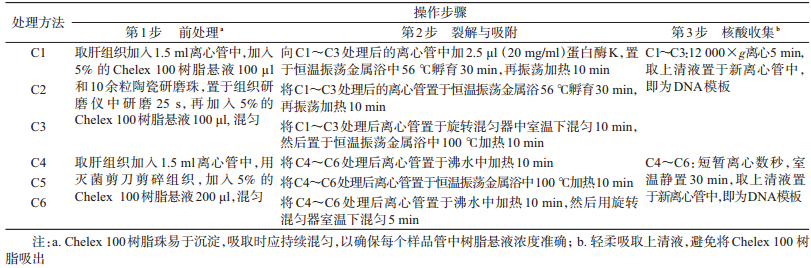
|
每份DNA模板取1 μl用Nano Drop-1000微量核酸浓度测定仪测定浓度,取4个平行样浓度和A260/A280比值的均值,与磁珠法提取的DNA进行比较。
1.4 引物、探针及扩增参数表 3中引物和探针分别用于扩增巴尔通体的tmRNA基因片段[13]和啮齿动物线粒体细胞色素C氧化酶亚基Ⅰ(COⅠ)的基因片段[14],由北京奥科鼎盛生物科技有限公司合成。
应用qPCR方法检测巴尔通体的扩增反应体系为20 μl,Taq DNA聚合酶和dNTP混合液(HR qPCR Master Mix)10 μl,探针0.4 μl(终浓度200 nmol/L),上、下游引物各0.8 μl(终浓度400 nmol/L),去离子水5 μl,样品DNA模板3 μl,做3个平行孔。阳性对照模板为质粒标准品1 μl。扩增反应参数:第1步,95 ℃变性5 min;第2步,95 ℃变性15 s,60 ℃退火45 s,40个循环。应用常规PCR方法扩增COⅠ基因片段的反应体系为25 μl,2×TransTaq-T PCR Super Mix加12.5 μl,上、下游引物各加1 μl (10 μmol/L),样品DNA模板加1 μl,去离子水补齐。扩增反应参数:第1步,95 ℃变性30 s;第2步,95 ℃变性30 s,54 ℃退火30 s,72 ℃延伸1 min,35个循环;第3步,72 ℃延伸3 min。上述扩增使用去离子水作为空白对照反应的模板。
1.5 电泳配制1.5%的琼脂糖凝胶,DNA模板和PCR产物上样量为1~3 μl,电压5 V/cm运行40 min。
1.6 统计学处理采用SPSS 24.0软件进行数据的正态分布Kolmogorov-Smirnov检验、方差齐性检验、t检验和Friedman M检验,P<0.05为差异有统计学意义。
2 结果 2.1 DNA模板的浓度和纯度测定结果用Chelex 100树脂C1~C6处理方法提取的样品ML44、ML51、ML70、ML102、ML108和ML150的DNA模板浓度在154.42~1 057.92 ng/μl之间,各样品的平均值在(203.93±36.27)~(769.86±256.41) ng/μl之间(表 4),各样品DNA模板浓度的变异系数(CV)值在6.21%~42.75%之间;磁珠法提取的DNA模板浓度分别为178.00、269.59、390.87、351.99、125.03和167.01 ng/μl,相较Chelex 100树脂法提取的DNA模板浓度略低,差异有统计学意义(t=6.021,P=0.002)。
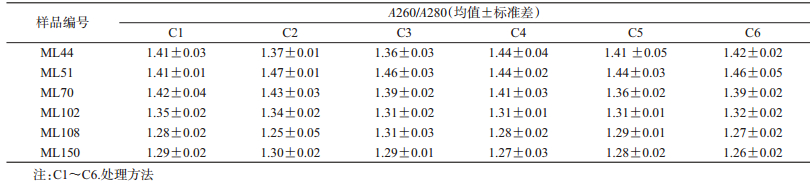
用Chelex 100树脂C1~C6处理方法提取的上述DNA模板A260/A280比值在1.25~1.51之间,各样品的平均值在(1.25±0.05)~(1.47±0.01)之间(表 5),CV值在0.73%~3.79%之间;磁珠法提取的DNA模板A260/A280比值在1.80~1.95之间,与Chelex 100树脂法差异有统计学意义(t=-23.029,P=0.000)。
|
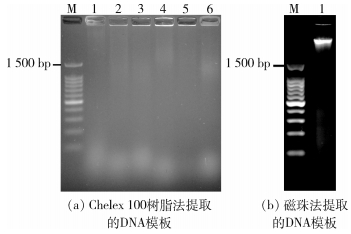
Chelex 100树脂法提取的DNA模板在电泳图中没有明显DNA条带,各泳道可见片状影像;磁珠法提取的DNA模板在电泳图中可见1条明亮的DNA条带(图 1)。
|
| 注:C1~C6.处理方法 图 1 DNA模板琼脂糖电泳结果 Figure 1 The agarose electrophoretograms of DNA templates |
| |
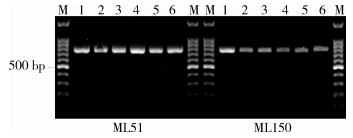
取样品ML51和ML150的C1~C6处理方法的COⅠ扩增产物电泳,扩增条带单一、清晰可见,长度符合目的基因片段,接近800 bp,见图 2。
|
| 注:M.Marker;1~6.C1~C6处理方法 图 2 COⅠ扩增产物琼脂糖电泳结果 Figure 2 The agarose electrophoretogram of PCR products of the COI gene |
| |
对Chelex 100树脂C1~C6处理方法提取的样品ML44、ML51、ML70、ML102、ML108和ML150的DNA模板进行qPCR扩增,各样品均可获得正常Cq值(表 6)。样品ML44~ML150的Cq平均值的极值分别为(27.94±0.38)~(28.86±0.46)、(29.68±0.31)~(31.11±1.27)、(27.36±0.42)~(33.24±0.79)、(31.97±0.58)~(34.16±1.41)、(28.25±0.62)~(31.00±2.81)和(29.45±1.05)~(30.70±0.77),CV值在1.37%~7.24%之间,C1~C6各处理方法之间差异有统计学意义(M=106.373,P=0.000),主要是C1和C2与其他处理方法之间有差别。磁珠法的DNA模板所得Cq值分别为28.90、24.20、29.63、28.91、28.43和26.42,略低于Chelex 100树脂法,差异有统计学意义(t=2.644,P=0.046)。

|
核酸分子检测方法已广泛应用于传染病病原体检测与监测中,特别是qPCR方法近年来发展迅速、快速普及,与常规PCR联合应用已成为实验室病原体检测的基本手段。虽然核酸扩增依赖于普通/定量PCR仪,但是目前已有可以用于现场检测的便携式仪器,这为开展现场即时检验(point-of-care testing,POCT)提供装备基础。高效的样品前处理是任何检测方法的前提和关键,简便、省时、省力和经济的核酸提取方法在长期、大规模监测工作中更易于开展实施。Chelex 100树脂法最早由Walsh等[4]用于DNA提取,目前已是法医实验室的常规方法,用于痕量检材的DNA提取,对样品要求较低,高度腐败、严重污染及暴露在极端恶劣环境中的样品均可应用Chelex 100树脂提取出DNA,操作简单方便、成本低,更有利于在野外现场和基层实验室推广使用。
本研究所用Chelex 100树脂为生物技术级,100~200目,适用于DNA提取。在本研究中,基于Chelex 100树脂采取了6种不同处理方法,区别在于前处理中组织的匀浆方式、裂解和吸附步骤中添加蛋白酶K与否及温浴方式和时间、最后核酸收集时是否需要离心。这些不同处理方法在其他研究中多有报道,特别是在提取不同性质的样品时,有些研究在裂解时加入蛋白酶K等裂解液[15-17]、增加纯化步骤[18],还有与自动化工作站联合使用简化操作方法等[19],用以提高提取效果。本研究的这些不同处理对DNA模板的浓度及qPCR的Cq值结果虽有差别,但不影响最终结果判断。Chelex 100树脂法所提DNA模板浓度普遍较磁珠法高,这与该法“一管式”提取、损失较少有关。A260/A280在1.8~1.9时,DNA纯度较高,若A260/A280<1.6,表明有蛋白等污染,Chelex 100树脂法所提DNA模板该值均<1.6。由于该方法没有纯化清洗步骤,所提DNA模板纯度不高在预期内,溶液中混有少量蛋白,所以A260/A280比值较低。常规PCR反应对模板DNA纯度要求不高,少量蛋白或其他杂质只要不抑制Taq DNA聚合酶活性即可,所以本研究中Chelex 100树脂法提取的DNA模板用于扩增COⅠ基因条带清晰,产物可用于测序鉴定物种。应用TaqMan探针法检测出全部样品中的巴尔通体,增加蛋白酶K处理和56 ℃温浴会降低Cq值,Chelex 100树脂法的Cq值高于磁珠法,表明组织中细菌DNA的充分释放会影响到检测的灵敏性,模板纯度会影响qPCR检测结果,对于一些在检测限附近的样品可能会受到限制,有关这方面影响在做其他组织和病原体时还需具体评估。
既往一些研究已总结出Chelex 100树脂提取DNA的优势[16],如步骤少、交叉污染少、检材用量少、可清除PCR抑制物和无毒性等。其他优势还有不需要特殊仪器,有加热设备即可,如电热炉/电磁炉,而其他方法最低配置是金属/水浴仪和离心机。该方法费用低廉,每份样品成本<1元,其他方法除传统的酚氯仿法成本较低,市售的各种试剂盒包括磁珠法和硅胶膜法等,均>5元。本研究中Chelex 100树脂提取核酸可以在15 min内完成,样品不需转管,可避免污染,简单快速,适用于常规监测和qPCR。在大规模调查、野外现场工作和长期监测中,综合考虑方法的可持续性、操作实施、设备要求和成本等因素,Chelex 100树脂法具有可行性,相关人员可根据调查和研究目的选择适当的核酸提取方法,以达到最佳效益。最后,提示应用Chelex 100树脂提取DNA时的注意事项:①样品用量不可过多;②树脂悬液配制时注意树脂珠要均匀分布;③要使用新鲜配置的树脂悬液;④吸上清液时不要带过树脂珠;⑤使用保存一段时间的DNA模板应再次离心。
| [1] |
Gao GF. From "A"IV to "Z"IKV:attacks from emerging and re-emerging pathogens[J]. Cell, 2018, 172(6): 1157-1159. DOI:10.1016/j.cell.2018.02.025 |
| [2] |
马丽敏. 核酸等温扩增技术研究进展[J]. 浙江预防医学, 2013, 25(1): 24-27. DOI:10.3969/j.issn.1007-0931.2013.01.007 |
| [3] |
王瑞丽, 卢晓, 孙中杰, 等. POCT技术在传染病病原检测中的应用[J]. 军事医学, 2016, 40(1): 70-73. DOI:10.3969/j.issn.1009-0754.2016.01.025 |
| [4] |
Walsh PS, Metzger DA, Higuchi R. Chelex 100 as a medium for simple extraction of DNA for PCR-based typing from forensic material[J]. Biotechniques, 1991, 10(4): 506-513. |
| [5] |
马芹, 王元智, 陆涛峰, 等. 棕背  性别的快速鉴定[J]. 中国比较医学杂志, 2018, 28(1): 96-99, 107. DOI:10.3969/j.issn.1671-7856.2018.01.017 性别的快速鉴定[J]. 中国比较医学杂志, 2018, 28(1): 96-99, 107. DOI:10.3969/j.issn.1671-7856.2018.01.017 |
| [6] |
胡晓红, 刘昕, 黄正根, 等. 应用于PCR及实时荧光定量PCR的细菌DNA提取方法[J]. 第三军医大学学报, 2006, 28(7): 734-735. DOI:10.3321/j.issn:1000-5404.2006.07.036 |
| [7] |
邹志文, 张素卿, 辛天蓉, 等. 一种改进的Chelex-100法提取单头螨DNA[J]. 南昌大学学报:理科版, 2011, 35(6): 564-567. DOI:10.3969/j.issn.1006-0464.2011.06.011 |
| [8] |
王宁, 都佳寅, 张卫军. 4种方法提取口腔致病菌DNA效果的比较[J]. 医学理论与实践, 2018, 31(9): 1253-1256. DOI:10.19381/j.issn.1001-7585.2018.09.003 |
| [9] |
柴海云, 张磊, 袁牧歌, 等. 一种简便、快速、高效适合PCR的真菌DNA提取方法[J]. 中国真菌学杂志, 2017, 12(6): 359-361, 367. DOI:10.3969/j.issn.1673-3827.2017.06.010 |
| [10] |
赵琳琳, 翟仙敦, 张振, 等. 基于28S rRNA基因序列对洛阳地区嗜尸性蝇类的分子鉴定[J]. 法医学杂志, 2018, 34(2): 114-119. DOI:10.3969/j.issn.1004-5619.2018.02.002 |
| [11] |
闫振天, 司风玲, 鲜鹏杰, 等. 基于Chelex-100和蛋白酶K的蚊虫足样本DNA提取技术[J]. 重庆师范大学学报:自然科学版, 2017, 34(5): 26-31. |
| [12] |
Norman AF, Regnery R, Jameson P, et al. Differentiation of Bartonella-like isolates at the species level by PCR-restriction fragment length polymorphism in the citrate synthase gene[J]. J Clin Microbiol, 1995, 33(7): 1797-1803. |
| [13] |
Diaz MH, Bai Y, Malania L, et al. Development of a novel genus-specific real-time PCR assay for detection and differentiation of Bartonella species and genotypes[J]. J Clin Microbiol, 2012, 50(5): 1645-1649. DOI:10.1128/JCM.06621-11 |
| [14] |
Robins JH, Hingston M, Matisoo-Smith E, et al. Identifying Rattus species using mitochondrial DNA[J]. Mol Ecol Notes, 2007, 7(5): 717-729. DOI:10.1111/j.1471-8286.2007.01752.x |
| [15] |
陈相波, 陈春, 沈金慧, 等. 利用改良的Chelex方法快速调查田间新蚜虫疠霉的流行[J]. 中国生物防治学报, 2014, 30(6): 731-735. |
| [16] |
欧阳刚, 胡文娟, 姚品芳, 等. PCR-RFLP中Chelex-100制备DNA模板的方法建立及其条件优化[J]. 中南民族大学学报:自然科学版, 2009, 28(1): 38-41. DOI:10.3969/j.issn.1672-4321.2009.01.010 |
| [17] |
Yang JL, Wang MS, Cheng AC, et al. A simple and rapid method for extracting bacterial DNA from intestinal microflora for ERIC-PCR detection[J]. World J Gastroenterol, 2008, 14(18): 2872-2876. DOI:10.3748/wjg.14.2872 |
| [18] |
Singh UA, Kumari M, Iyengar S. Method for improving the quality of genomic DNA obtained from minute quantities of tissue and blood samples using Chelex 100 resin[J]. Biol Proced Online, 2018, 20: 12. DOI:10.1186/s12575-018-0077-6 |
| [19] |
周如华, 孙溢华, 孙元鹏, 等. 自动化Chelex-100法在接触性检材DNA提取中的应用研究[J]. 刑事技术, 2017, 42(3): 206-208. DOI:10.16467/j.1008-3650.2017.03.009 |
 2019, Vol. 30
2019, Vol. 30




