扩展功能
文章信息
- 郭丽民, 席进孝, 盖永志, 吴斌, 王世明, 苗克军, 穆洮霞, 徐大琴
- GUO Li-min, XI Jin-xiao, GAI Yong-zhi, WU Bin, WANG Shi-ming, MIAO Ke-jun, MU Tao-xia, XU Da-qin
- 甘肃省鼠疫疫源地啮齿动物DNA条形码分析
- Application of DNA barcoding in the identification of rodent species in plague foci in Gansu province, China
- 中国媒介生物学及控制杂志, 2019, 30(2): 176-179
- Chin J Vector Biol & Control, 2019, 30(2): 176-179
- 10.11853/j.issn.1003.8280.2019.02.014
-
文章历史
- 收稿日期: 2018-10-15
- 网络出版时间: 2019-03-01 09:12
甘肃省地处黄河上游,地域分布从东南向西北呈一狭长地带,是青藏高原、内蒙古高原、黄土高原汇集的地区,境内地貌、气候、植被类型多样,多样性的自然景观孕育了种类繁多的啮齿动物。甘肃省啮齿动物区系组成研究始于20世纪50年代,共发现啮齿动物2目2亚目9科12亚科43属90种[1]。甘肃省存在两种类型鼠疫自然疫源地,即青藏高原喜马拉雅旱獭(Marmota himalayana)鼠疫自然疫源地和甘宁黄土高原阿拉善黄鼠(Spermophilus alaschamicus)鼠疫自然疫源地,疫源地面积占全省面积的19%。疫源地啮齿动物种类繁多,种类和分布基本清晰,共发现啮齿动物10科60余种。但是啮齿动物的区划缺乏全面深入的研究,存在同种异名现象,其中同种异名就达9种。种类的描述鉴定均以形态学特征为基础,易造成人为错误,影响鉴定结果。
DNA条形码技术是通过对一个标准目的基因DNA序列进行分析而对物种快速鉴定的技术[2-3]。在动物分类中最常用的基因序列是线粒体细胞色素C氧化酶亚基Ⅰ(COⅠ)序列[4-5]。本研究在甘肃省鼠疫疫源地及非疫源地多个地点采集啮齿动物,采用DNA条形码技术对标本线粒体COⅠ基因进行分析,准确鉴定种类,积累条形码数据,探索利用DNA条形码鉴别啮齿动物的可行性。
1 材料与方法 1.1 啮齿动物标本采集2017年在甘肃省鼠疫疫源地(阿克塞县、肃北县、玉门市、武威市、碌曲县、会宁县和平川区)及非疫源地(民勤和渭源县)采集啮齿动物标本。
1.2 形态学鉴定通过啮齿动物外部形态特征进行初步鉴定[6],根据外部形态无法确定的标本则需要制作剥制标本,然后测量头骨等相关数据,通过检索表进行鉴定。
1.3 DNA条形码扩增和分析 1.3.1 模板DNA提取使用北京天根生化科技有限公司基因组提取试剂盒提取啮齿动物肝脏DNA,-20 ℃保存备用。
1.3.2 PCR扩增与测序采用通用引物BatL5310(序列为5′-CCTACTCRGCCATTTTACCTATG-3′)和R6036R(序列为5′-ATCTCTGGGTGTCCAAAGAA TCA-3′)扩增COⅠ基因片段[7-8]。PCR反应扩增体系:10 ×缓冲液2.5 μl,dNTP 0.5 μl,正向和反向引物各0.5 μl,LA Taq DNA聚合酶0.15 μl,模板DNA1 μl,ddH2O 19.85 μl。PCR反应条件:95 ℃预变性5 min,95 ℃变性45 s,54 ℃退火1 min,72 ℃延伸1 min,35个循环,72 ℃延伸10 min。扩增产物经1%琼脂糖凝胶电泳检测,送兰州美伯生物有限公司进行双向测序。
1.3.3 DNA序列分析首先采用Chromas软件观察测序峰图质量,如果测序峰图质量很差不能准确判断碱基,则重新进行扩增和测序。然后采用Clustal X 2.1软件进行序列比对,将测定的序列在美国国立生物技术信息中心(NCBI)上进行BLAST同源性比对,选择形态鉴定符合或相似>95%的序列作为参照序列。运用MEGA 5.0软件计算各物种的遗传距离,采用邻接法(Neighbor- Joining,NJ)构建系统树[9-10]。
2 结果 2.1 形态学鉴定结果甘肃省鼠疫疫源地及非疫源地共采集啮齿动物164只。形态学鉴定15种,其中鼠科3种,分别为朝鲜姬鼠(Apodemus peninsulae)、北社鼠(Niviventer confucianus)和小家鼠(Mus musculus);跳鼠科2种,分别为三趾跳鼠(Dipus sagitta)和五趾跳鼠(Allactaga sibirica);仓鼠科4种,分别为长尾仓鼠(Cricetulus longicaudatus)、黑线仓鼠(C. barabensis)、大仓鼠(Tscherskia triton)和小毛足鼠(Phodopus roborovskii);松鼠科3种,分别为阿拉善黄鼠、花鼠(Eutamias sibiricus)、喜马拉雅旱獭;沙鼠科1种,为子午沙鼠(Meriones meridianus);鼢鼠科1种,为甘肃鼢鼠(Myospalax cansus);兔形目的鼠兔科1种,为甘肃鼠兔(Ochotona cansus),见表 1。

|
将测序获得的序列在NCBI上运行BLAST程序进行序列同源性比对,发现15个种的序列可在GenBank种找到一致的序列,与形态学鉴定结果相符合。发现同属各种间COⅠ序列差异度为14%~25%,种内序列差异度≤2%。序列比对结果见表 2。
全部物种间种内遗传距离为0~2.2%。种间遗传距离为14%~25%,种间遗传距离大于种内遗传距离。
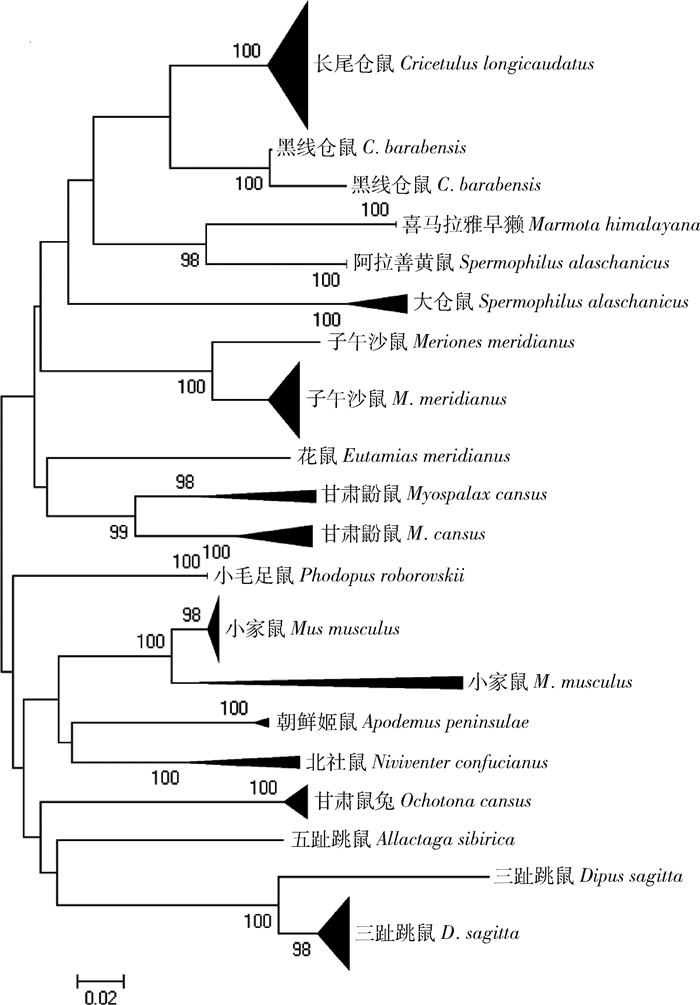
2.4 分子进化树构建全部COⅠ序列的NJ分子进化树可分为15个高度支持的分支,表明根据形态特征的分类基本准确,但是甘肃鼢鼠、小家鼠和三趾跳鼠分支间遗传距离数值基本处于种内遗传距离与种间遗传距离的交界处,见图 1。
|
| 注:箭头粗、细、长、短代表各种鼠类经过压缩的系统树。由于鼠类邻接系统树原图太大,故将样本量大的鼠类标本系统树在分支处进行压缩 图 1 基于线粒体细胞色素C氧化酶亚基Ⅰ(COⅠ)序列构建的鼠类邻接系统树 Figure 1 A neighbor-joining tree of rodents based on the mitochondrial COⅠsequence |
| |
本次标本采集区域以4种生境为主,即山地草原、荒漠草原、高山草原草甸和农田荒地。4种生境的啮齿动物构成中优势种不同,其他动物种类存在交叉。
对2017年采集的2目7科12属164只啮齿动物构建分子进化树,形成15个高度单一的分支,与形态学鉴定一致。阿拉善黄鼠是甘宁黄土高原鼠疫自然疫源地内的主要宿主动物,也是疫源地内的优势种。经过比对采集到的黄鼠COⅠ序列,鉴定为阿拉善黄鼠独立种,不是达乌尔黄鼠阿拉善亚种。采集的鼠兔经过比对鉴定均为甘肃鼠兔,未发现达乌尔鼠兔(Ochotona daurica)。而在进化树中,甘肃鼢鼠、小家鼠和三趾跳鼠分支间遗传距离数值基本处于种内遗传距离与种间遗传距离的交界处[11],是否为2个种尚不能确定。黑线仓鼠和子午沙鼠遗传距离则在DNA条形码技术实验研究的种间差异范围内,表明根据外部形态的结果可能存在2个种类。但是确定这些隐种的分类地位需要考虑更多的DNA条形码基因/片段,需增加标本收集量,进行综合分析才能作出科学判断。实验结果表明DNA条形码不同类群内物种间遗传距离可能不同,现有DNA条形码数据库的物种判断阈值可能会对物种多样性造成一定的误判[12-13]。
从进化树分析结果可见,同属和同种鼠均能够被明确地分入相应分支。说明DNA条形码COⅠ基因能够用于啮齿动物的分类鉴定,而且在标本不完整的情况下进行物种鉴定拥有绝对优势[14]。但GenBank数据库比对序列同源性<95%无法给予明确结果时,应采用多基因标记技术,使其包含更大的遗传信息[15]。综合考虑分类经验、动物发育时期、标本残缺等因素,啮齿动物鉴定应将外部形态和DNA条形码技术有机地结合起来,克服传统形态学鉴定中的错误,满足日后大量的以DNA条形码为基础的物种快速准确鉴定的需求。
| [1] |
花立民, 黄倩, 曹慧. 甘肃啮齿动物区系组成研究[J]. 草原与草坪, 2008(2): 46-51. DOI:10.3969/j.issn.1009-5500.2008.02.010 |
| [2] |
Witt JDS, Threloff DL, Hebert PDN. DNA barcoding reveals extraordinary cryptic diversity in an amphipod genus:implications for desert spring conservation[J]. Mol Ecol, 2006, 15(10): 3073-3082. DOI:10.1111/j.1365-294X.2006.02999.x |
| [3] |
Hebert PDN, Ratnasingham S, de Waard JR. Barcoding animal life:cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proc Biol Sci, 2003, 270 Suppl 1: S96-99. DOI:10.1098/rsbl.2003.0025 |
| [4] |
马英, 李海龙, 鲁亮, 等. DNA条形码技术在青海海东地区小型兽类鉴定中的应用[J]. 生物多样性, 2012, 20(2): 193-198. DOI:10.3724/SP.J.1003.2012.06188 |
| [5] |
张涛, 宁文艳, 吴建华, 等. 宁夏啮齿动物DNA条形码分析[J]. 中国人兽共患病学报, 2014, 30(12): 1234-1238. DOI:10.3969/cjz.j.issn.1002-2694.2014.12.014 |
| [6] |
郑智民, 姜志宽, 陈安国. 啮齿动物学[M]. 上海: 上海交通大学出版社, 2008: 57-74.
|
| [7] |
Kimura M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences[J]. J Mol Evol, 1980, 16(2): 111-120. DOI:10.1007/BF01731581 |
| [8] |
闫东, 鲁亮, 金圣浩, 等. DNA条形码技术在河北省鼠疫疫源地鼠种鉴定中的应用[J]. 中国媒介生物学及控制杂志, 2014, 25(1): 21-23. DOI:10.11853/j.issn.1003.4692.2014.01.006 |
| [9] |
Ivanova NV, Clare EL, Borisenko AV.DNA barcoding in mammals[M]//Kress WJ, Erickson DL.DNA Barcodes: Methods and Protocols.Totowa NJ: Humana Press, 2012: 153-182.
|
| [10] |
张涛, 段恒璐, 白学礼, 等. 宁夏六盘山地区小型兽类DNA条形码分析[J]. 中国媒介生物学及控制杂志, 2016, 27(4): 330-332. DOI:10.11853/j.issn.1003.8280.2016.04.004 |
| [11] |
鲁亮, 蒋卫, 刘蓉蓉, 等. 中国-哈萨克斯坦边境地区啮齿动物多样性调查[J]. 中国媒介生物学及控制杂志, 2016, 27(4): 317-320. DOI:10.11853/j.issn.1003.8280.2016.04.001 |
| [12] |
王祖郧, 李超. 青海鼠疫[M]. 北京: 人民卫生出版社, 2016: 56-61.
|
| [13] |
安翠红, 陈宝宝, 孙养信, 等. DNA条形码技术在鼠疫疫区啮齿动物残体鉴定中的应用[J]. 中华地方病学杂志, 2014, 33(5): 504-507. DOI:10.3760/cma.j.issn.2095-4255.2014.05.009 |
| [14] |
金圣浩, 鲁亮, 闫东, 等. DNA条形码技术在河北省坝上地区夜行鼠鉴定中的应用[J]. 中国媒介生物学及控制杂志, 2015, 26(1): 55-57. DOI:10.11853/j.issn.1003.4692.2015.01.014 |
| [15] |
程佳月, 王丽华, 彭克美, 等. 国际生命条形码计划-DNA Barcoding[J]. 中国畜牧兽医, 2009, 36(8): 49-53. |
 2019, Vol. 30
2019, Vol. 30


