扩展功能
文章信息
- 权力, 江爱民, 卢彦宗, 朱耐伟, 朱勇喆
- QUAN Li, JIANG Ai-min, LU Yan-zong, ZHU Nai-wei, ZHU Yong-zhe
- 应用CRISPR/Cas9基因编辑系统敲除小窝蛋白1基因的神经细胞构建与功能鉴定
- Constructing and functional analysis of knocking out CAV-1 gene by CRISPR/Cas9 system in neuron cells
- 中国媒介生物学及控制杂志, 2018, 29(5): 453-457
- Chin J Vector Biol & Control, 2018, 29(5): 453-457
- 10.11853/j.issn.1003.8280.2018.05.008
-
文章历史
- 收稿日期: 2018-06-02
- 网络出版时间: 2018-08-03 17:13
2 第二军医大学学员旅学员1队, 上海 200433;
3 第二军医大学学员旅学员9队, 上海 200433
2 Company1, Cadet's Brigade, Second Military Medical University;
3 Company 9, Cadet's Brigade, Second Military Medical University
流行性乙型脑炎(乙脑)病毒(Japanese encephalitis virus,JEV),又称日本脑炎病毒,是一种蚊媒病毒。三带喙库蚊(Culex tritaeniorhynchus)是JEV的主要传播媒介,可引起乙脑,是一种人畜共患病。JEV具有神经细胞嗜性,能够感染脑实质并引起广泛病变,存活患者中大多存在神经系统后遗症[1]。据国家卫生和计划生育委员会统计,2017年我国共有1 147例确诊患者,死亡79例[2]。
小窝蛋白(caveolin,CAV),又称微囊蛋白,为小窝主要组成蛋白,属整合膜蛋白[3]。目前研究表明,JEV主要通过小窝蛋白1(caveolin-1,CAV-1)介导的内吞作用入侵人神经细胞[4]。为进一步研究其机制,需对CAV-1基因进行敲除。传统的基因沉默主要采用RNA干扰(RNA interference,RNAi),通过引入外源性的小干扰RNA(small interfering RNA,siRNA)或短发夹RNA(short hairpin RNA,shRNA)介导靶基因沉默,但该法敲除效率低,作用时间短,存在脱靶效应,且无法完全去除基因功能[5-7]。因此,迫切需要一种高效的CAV-1敲除方法用以研究JEV致病机制。
近年来,成簇规律间隔的短回文重复序列(clustered regulatory interspaced short palindromic repeats,CRISPR)相关蛋白9(CRISPR-associated protein 9,Cas9)系统可对基因组进行简便和高效的编辑[7]。因此本实验通过CRISPR/Cas9技术对SH-SY5Y细胞株的CAV-1基因进行敲除,从而研究它对JEV感染性的影响,为后续进一步研究JEV的细胞入侵机制提供可靠的实验平台和技术支持。
1 材料与方法 1.1 材料人神经母细胞瘤SH-SY5Y细胞由本实验室保存。JEV病毒株SA14由俞永新教授惠赠。PX459质粒购自美国Addgene公司;Trans1T1感受态购自全式金公司;限制性内切酶BbsⅠ、T4 DNA连接酶和T4多聚核苷酸激酶购自美国NEB公司;质粒提取试剂盒购自德国Qiagen公司;Lipofectamine 3000转染试剂及羊抗兔Alexa Fluor 488荧光二抗购自美国Invitrogen公司;二盐酸嘌呤霉素购自美国Selleck公司;鼠抗人GAPDH单克隆抗体购自美国Proteintech公司;兔抗人CAV-1单克隆抗体购自美国Sigma公司;兔抗JEV NS3抗体购自美国GeneTex公司;DAPI购自瑞士Roche公司;DMEM培养基、细胞培养用青链霉素及胎牛血清为美国Gibco公司产品;质粒测序及寡核苷酸链合成由捷瑞生物公司完成。
1.2 针对人源CAV-1基因的sgRNA靶点选择及寡核苷酸链合成根据CRISPR/Cas9靶点设计原则,选择CAV-1基因的第1个外显子区(expressed region 1,EXON1)和第2个外显子区(expressed region 2,EXON2)为靶位点,使用http://crispr-mit.edu/网站分别设计2个EXON的sgRNA序列,选取其中分数较高的序列,并在正向引物的5′端添加CACC,反向引物的5′端添加AAAC,使其与BbsⅠ酶切后的载体形成黏性末端互补。
1.3 PX459-sgRNA真核表达质粒构建 1.3.1 sgRNA寡核苷酸单链退火形成双链取等量的正、反向引物,使其退火形成双链。反应体系:各取100 nmol正、反向引物sgRNA,1 μl 10×T4 Ligation Buffer,0.5 μl T4多聚核苷酸激酶加水至10 μl。反应程序:37 ℃ 30 min;95 ℃ 5 min;每分钟下降5 ℃,降至25 ℃。
1.3.2 PX459载体线性化使用BbsⅠ线性化PX459质粒,切胶回收。反应体系为:1 μg PX459载体,1 μl BbsⅠ,5 μl 10×NE Buffer,加水至50 μl。反应程序:37 ℃ 15 min。
1.3.3 连接体系线性化PX459载体50 ng,寡核苷酸双链产物150 ng,1 μl 10×T4 DNA连接酶缓冲液,1 μl T4 DNA连接酶,加水至10 μl。程序:25 ℃60 min。将产物转化Trans1-T1感受态,挑取单克隆进行摇菌,使用Qiagen质粒提取试剂盒提取质粒,进行酶切和测序验证。
1.4 细胞培养和细胞转染使用DMEM培养基(含10%胎牛血清)常规培养SH-SY5Y细胞。转染前12 h,将SH-SY5Y细胞接种至24孔板中培养,转染时细胞汇合度达到60%~70%为宜。将构建的PX459-CAV-1重组质粒与PX459载体按照Lipofectamine3000转染说明书转入细胞内,并在5%CO2、37 ℃培养箱中培养。
1.5 SH-SY5Y细胞CAV-1基因敲除稳定细胞株的单克隆筛选将PX459-CAV-1重组质粒转入SH-SY5Y细胞,48 h后更换含有2 μg/ml嘌呤霉素的DMEM培养基进行筛选。2 d后将筛选所得混合克隆取1/3细胞继续培养,余下2/3细胞提取蛋白,免疫印迹检测CAV-1的表达情况,确定效率最佳的重组质粒。利用稀释法对效率最佳的混合克隆进行单克隆筛选,将细胞接种到96孔板中(1个细胞/孔)。待细胞长满,移至24孔板中继续扩大培养,得到CAV-1敲除的SH-SY5Y细胞(SH-SY5Y-CAV-1-KO)。
1.6 免疫印迹将野生型SH-SY5Y(SH-SY5Y-WT)和筛选后得到的单克隆稳定株SH-SY5Y-CAV-1-KO分别接种至24孔板中培养。待细胞长满后,用PBS缓冲液洗涤细胞2次,加入100 μl RIPA裂解液,置于冰上裂解细胞,抽提细胞总蛋白,利用BCA蛋白测定试剂盒测定总蛋白浓度,经10%变性SDS-PAGE电泳后,用湿转化法将蛋白转移至聚偏二氟乙烯(PVDF)膜上,使用5%脱脂牛奶的TBST溶液封闭2 h,CAV-1抗体4 ℃孵育过夜(稀释倍数1:1 000),TBST洗涤3次,用辣根过氧化物酶标记的二抗孵育2 h(稀释倍数1:1 000),TBST再洗涤,ECL化学发光后获得目的条带。
1.7 JEV感染神经细胞在96孔板内接种SH-SY5Y-WT和SH-SY5Y-CAV-1-KO,使感染时细胞密度达到70%左右,每孔加入感染复数(multiplicity of infection,MOI)为1的JEV,37 ℃感染2 h,去除病毒液并加入100 μl DMEM培养基,36 h后进行免疫荧光检测。
1.8 免疫荧光检测CAV-1基因敲除神经细胞株对JEV的易感性JEV感染36 h后,加入甲醇,置于-20 ℃ 20 min进行固定和透膜。PBS洗涤3次。每孔加入3%BSA封闭1 h。将JEV NS3抗体用1%BSA稀释(稀释倍数1:1 000),室温孵育2 h。加入羊抗兔的Alexa Fluor488进行二抗孵育(稀释倍数1:1 000),避光孵育1 h。最后DAPI(稀释倍数1:10 000)进行细胞核染色,通过荧光显微镜随机取视野拍照,计算视野内的细胞个数。结果以感染率=阳性细胞个数/总细胞个数表示。
1.9 统计学处理通过SPSS 17.0软件对数据进行分析,数据采用x±s表示,两组间均数比较采用t检验。P < 0.05表示差异有统计学意义。
2 结果 2.1 sgRNA靶点的选择以及寡核苷酸序列设计针对CAV-1基因的EXON1和EXON2分别进行sgRNA设计。sgRNA正、反向引物见表 1。
利用限制性内切酶BbsⅠ对PX459-CAV-1-sgRNA-1和PX459-CAV-1-sgRNA-2进行酶切,结果见图 1A,连接成功的质粒不含有BbsⅠ酶切位点,因此不能被内切酶切割为线性。测序结果见图 1B,插入的sgRNA位置、方向及序列均正确,成功构建重组质粒。
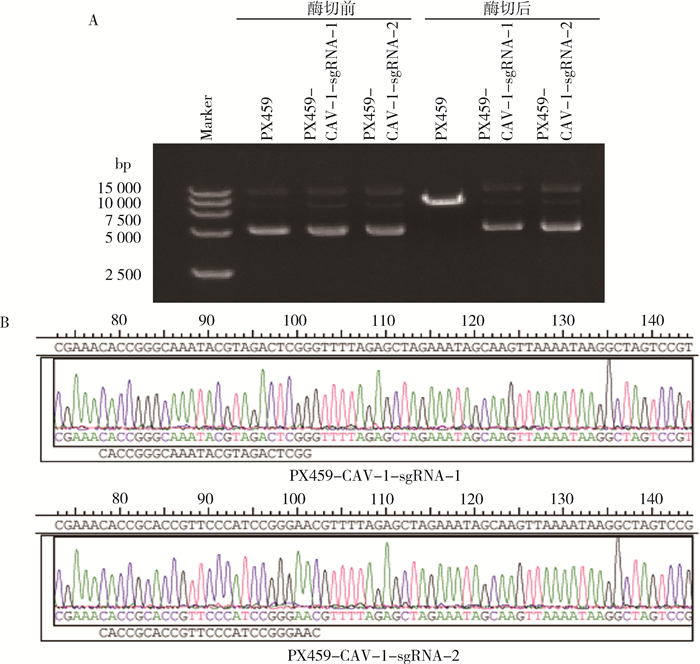
|
| 注:A. PX459-CAV-1-sgRNA重组质粒的酶切结果;B. PX459-CAV-1-sgRNA重组质粒的测序结果 图 1 PX459-CAV-1-sgRNA重组质粒的鉴定 Figure 1 The identification of PX459-CAV-1-sgRNA recombinant plasmid |
| |
将PX459-CAV-1-sgRNA-1和PX459-CAV-1-sgRNA-2分别转染细胞并加药筛选2 d后,裂解各孔混合克隆的细胞蛋白,免疫印迹检测重组质粒的敲除效率,确定PX459-CAV-1-sgRNA-1的敲除效率最高(图 2)。培养PX459-CAV-1-sgRNA-1重组质粒转染的细胞,最终得到单克隆细胞。SH-SY5Y-WT中的CAV-1正常表达,而SH-SY5Y-CAV-1-KO中的CAV-1没有表达(图 3),SH-SY5Y细胞CAV-1基因敲除稳定株成功构建。
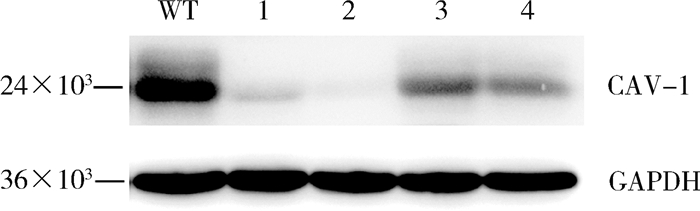
|
| 注:WT.野生型;1~2. PX459-CAV-1-sgRNA-1;3~4. PX459-CAV-1-sgRNA-2 图 2 免疫印迹对SH-SY5Y混合克隆细胞CAV-1表达的分析 Figure 2 The analysis of the expression level of CAV-1 in SH-SY5Y polyclonal cells by immunoblotting |
| |
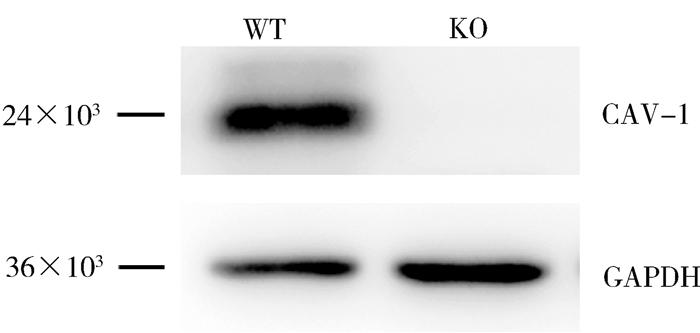
|
| 注:WT.野生型;KO. CAV-1基因敲除型 图 3 免疫印迹对SH-SY5Y单克隆细胞CAV-1表达的分析 Figure 3 The analysis of the expression level of CAV-1 in SH-SY5Y monoclonal cells by immunoblotting |
| |
将野生型SH-SY5Y细胞(WT)与CAV-1敲除细胞株(KO)以相同的条件进行感染,36 h后进行免疫荧光检测JEV的感染性。与WT相比,KO对JEV的感染性明显下降(P < 0.001),见图 4。
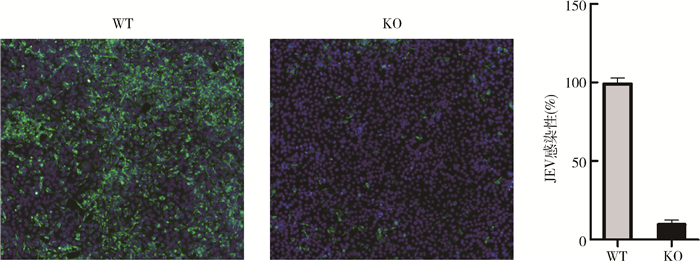
|
| 注:WT.野生型;KO. CAV-1基因敲除型 图 4 免疫荧光检测野生型与CAV-1敲除细胞株对JEV的感染性 Figure 4 The detection of JEV infection on wild type cells and CAV-1 knockout cells by immunofluorescence |
| |
病毒入侵细胞是病毒利用宿主细胞的某些成分,促进其在细胞内的移行。目前,已发现JEV可以利用多种内吞机制来入侵细胞[4, 8]。本课题组前期研究表明,CAV介导的内吞机制在JEV感染神经细胞中起主要作用。通过抑制CAV的表达有助于研究JEV的多种入侵途径,并且可为病毒的防治提供有力策略。然而采用RNAi只能在转录后水平下调CAV的表达,并不能完全抑制CAV的功能[5]。
CRISPR/Cas9作为一项全新的基因编辑技术,拥有快速、可靠、高特异性以及高敲除效率等特点。该技术由两部分构成,即CRISPR和Cas9核酸酶(Cas9)[9]。该系统原本是一种广泛存在于细菌和古生菌中的免疫机制,因其可特异结合并剪切DNA片段,有效抵御一系列外源性DNA侵染,因此近年来被广泛用于基因的定点修饰[10-12]。其利用与目的基因同源的sgRNA,特异性结合靶细胞基因组的DNA,形成杂合双链,之后Cas9可对该双链进行切割,使得此段DNA双链断裂(double-strand breaks,DSB)。DSB可诱导产生位点特异性核酸酶,通过错误倾向的非同源末端连接进行修复,该修复将在断裂位点造成插入或缺失,使得基因编辑变得简便并且高效[13]。
本研究将PX459质粒作为载体,分别以CAV-1基因的EXON1和EXON2为靶点设计了sgRNA,构建了PX459-CAV-1重组质粒,经酶切测序正确后,转染至SH-SY5Y细胞中,利用嘌呤霉素进行抗性筛选,最终筛选并获得CAV-1基因敲除稳定株。其中针对EXON1的PX459-CAV-1-sgRNA-1的敲除效率更高,而针对EXON2的PX459-CAV-1-sgRNA-2的敲除效率则较低,其可能是由于针对EXON2的sgRNA介导的Cas9核酸酶在靶点位置造成的突变并不能改变CAV-1蛋白的开放阅读框,不能达到完全敲除CAV-1基因的目的。故在设计sgRNA时,应尽量选取靠前的EXON,使其移码突变概率增加[10-11, 14]。同时检测了敲除细胞株与野生型对JEV感染性的差异,结果表明,CAV-1基因敲除后,JEV对神经细胞的感染性明显降低,进一步证明小窝蛋白介导的内吞对JEV感染神经细胞尤为重要。目前,JEV入侵细胞的分子机制尚未完全阐明,本研究为进一步探索CAV-1在JEV感染神经细胞过程中的分子机制提供了有效的细胞模型。
| [1] |
Misra UK, Kalita J. Overview:Japanese encephalitis[J]. Prog Neurobiol, 2010, 91(2): 108-120. DOI:10.1016/j.pneurobio.2010.01.008 |
| [2] |
国家卫生和计划生育委员会疾病预防控制局. 2017年全国法定传染病疫情概况[EB/OL]. (2018-02-26)[2018-03-08]. http://www.nhfpc.gov.cn/jkj/s3578/201802/de926bdb046749abb7b0a8e23d929104.shtml.
|
| [3] |
Schwencke C, Braun-Dullaeus RC, Wunderlich C, et al. Caveolae and caveolin in transmembrane signaling:implications for human disease[J]. Cardiovasc Res, 2006, 70(1): 42-49. DOI:10.1016/j.cardiores.2005.11.029 |
| [4] |
Xu QQ, Cao MM, Song HY, et al. Caveolin-1-mediated Japanese encephalitis virus entry requires a two-step regulation of actin reorganization[J]. Future Microbiol, 2016, 11(10): 1227-1248. DOI:10.2217/fmb-2016-0002 |
| [5] |
赵德根, 王建校, 刘旭, 等. 小干扰RNA的研究进展[J]. 现代生物医学进展, 2014, 14(4): 792-794. DOI:10.13241/j.cnki.pmb.2014.04.049 |
| [6] |
陈瑶, 刘青, 邢微微, 等. 基于CRISPR/Cas9基因编辑技术的RIP1稳定敲除细胞模型的构建及功能研究[J]. 军事医学, 2017, 41(2): 96-100, 105. DOI:10.7644/j.issn.1674-9960.2017.02.004 |
| [7] |
Morgens DW, Deans RM, Li A, et al. Systematic comparison of CRISPR/Cas9 and RNAi screens for essential genes[J]. Nat Biotechnol, 2016, 34(6): 634-636. DOI:10.1038/nbt.3567 |
| [8] |
Zhu YZ, Xu QQ, Wu DG, et al. Japanese encephalitis virus enters rat neuroblastoma cells via a pH-dependent, dynamin and caveola-mediated endocytosis pathway[J]. J Virol, 2012, 86(24): 13407-13422. DOI:10.1128/JVI.00903-12 |
| [9] |
Wang HX, Li MQ, Lee CM, et al. CRISPR/Cas9-based genome editing for disease modeling and therapy:Challenges and opportunities for nonviral delivery[J]. Chem Rev, 2017, 117(15): 9874-9906. DOI:10.1021/acs.chemrev.6b00799 |
| [10] |
Fu YF, Foden JA, Khayter C, et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells[J]. Nat Biotechnol, 2013, 31(9): 822-826. DOI:10.1038/nbt.2623 |
| [11] |
Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339(6121): 819-823. DOI:10.1126/science.1231143 |
| [12] |
Chen JS, Dagdas YS, Kleinstiver BP, et al. Enhanced proofreading governs CRISPR-Cas9 targeting accuracy[J]. Nature, 2017, 550(7676): 407-410. DOI:10.1038/nature24268 |
| [13] |
Paquet D, Kwart D, Chen A, et al. Efficient introduction of specific homozygous and heterozygous mutations using CRISPR/Cas9[J]. Nature, 2016, 533(7601): 125-129. DOI:10.1038/nature17664 |
| [14] |
卢利莎, 白杨, 刘鑫, 等. 利用CRISPR/Cas9技术构建敲除MEIS2基因的HEK293T人胚肾细胞系[J]. 中国细胞生物学学报, 2015, 37(4): 535-541. DOI:10.11844/cjcb.2015.04.0020 |
 2018, Vol. 29
2018, Vol. 29



