扩展功能
文章信息
- 王海峰, 周松, 李玉贵, 白雪薇, 孙睿, 胡乐乐
- WANG Hai-feng, ZHOU Song, LI Yu-gui, BAI Xue-wei, SUN Rui, HU Le-le
- 利用差异区段分型方法对沙鼠型鼠疫菌和鼠疫菌EV76疫苗株进行鉴定
- Identification of Yersinia pestis isolates from Mongolian gerbils and the plague vaccine via genotyping of different regions
- 中国媒介生物学及控制杂志, 2018, 29(5): 436-438
- Chin J Vector Biol & Control, 2018, 29(5): 436-438
- 10.11853/j.issn.1003.8280.2018.05.003
-
文章历史
- 收稿日期: 2018-06-17
- 网络出版时间: 2018-08-03 17:13
EV76为鼠疫疫苗株,其染色形态、培养特征、噬菌体裂解结果及动物实验与鼠疫耶尔森菌(鼠疫菌)野生分离株相同,在实际工作中,经常会因为实验室使用过菌苗而造成污染,使得依赖“四步检验”鉴定的疑似鼠疫菌被分离出来,而投入大量的人力财力进行疫情应急处理,如需进行一系列糖醇酵解、生化试验以及毒力与毒力因子测定试验,较长时间才能最终鉴定是否为鼠疫菌分离株。随着鼠疫菌全基因组序列的测定,比较基因组学研究发展迅速,已经建立起多种鼠疫菌基因分型方法,差异区段(different region,DFR)是指在同一物种里,某些菌株基因组中存在而在另一些菌株中缺失的基因片段。本研究采用DFR分型方法对沙鼠疫源地分离的沙鼠型鼠疫菌和鼠疫菌EV76疫苗株进行实验比对,可以在以后的工作中快速确认疑似菌株是否为鼠疫菌野生分离株。
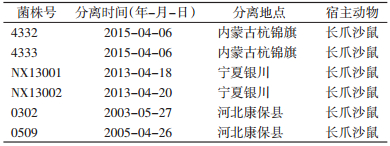
1 材料与方法 1.1 实验菌株实验所用菌株选取来自沙鼠疫源地不同地区的野生菌株和EV76疫苗株,其中宁夏回族自治区(宁夏)和内蒙古自治区(内蒙古)的鼠疫菌株DNA由中国CDC传染病预防控制所鼠疫室赠送,见表 1。
PCR扩增仪(杭州博日科技有限公司),高速台式离心机(Eppendorf 5424),水平电泳仪(北京市六一仪器厂),凝胶成像仪(上海基因科技有限公司)。DNA提取试剂盒购自Qiagen公司,PCR扩增试剂购自北京全式金生物技术有限公司,琼脂糖购自西班牙Biowest,阳性对照为62004和82009混合物由青海省地方病预防控制所赠送,阴性对照为去离子水。
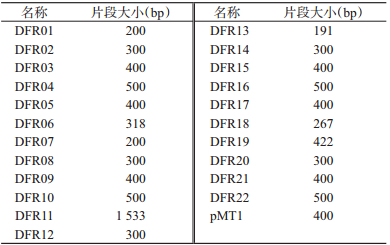
1.3 引物参照文献[1],引物选择22对DFR和pMT1,由生工生物工程有限公司合成,见表 2。
|
将鼠疫菌及其EV76疫苗株接种于赫氏培养基,28 ℃培养48 h后刮取菌苔于加有600 μl TE缓冲液的Eppendorf管中,100 ℃水浴20 min,离心半径8.4 cm,12 000 r/min离心10 min,收集上清液。将提取的DNA用去离子水稀释10倍后制备成PCR扩增用的模板。
1.4.2 PCR反应体系和条件① 反应体系:去离子水将提取的鼠疫菌DNA模板10倍稀释,采用25 μl反应体系,其中SuperMix混合液12.5 μl,正、反向引物各1.0 μl,模板1.0 μl,补水至25.0 μl;②反应条件:引物DFR1~17、DFR20~22、pMT1的扩增条件为94 ℃预变性3 min,94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸1 min,30个循环,72 ℃延伸5 min。引物DFR18和DFR19的退火条件为56 ℃ 30 s,其他条件同上。
1.4.3 电泳分析扩增产物用1.5%琼脂糖凝胶电泳,阳性对照采用62004和82009的混合物,阴性对照用去离子水。电泳条件为120 V,30~60 min。
1.4.4 结果判读在凝胶成像仪下读取结果,阳性对照扩增出全部条带时结果有效,标本出现目标条带者判为出现目标并与阳性对照一致的记为“1”,未出现目标条带者记为“0”。对模糊不清的条带重复实验进行验证。
2 结果 2.1 沙鼠型鼠疫菌的DFR分型结果对照DNA所有条带,经过多次重复实验,通过与参考文献[2]中鼠疫菌DFR位点的分布和基因型结果比对,此次所用分离自不同沙鼠疫源地的菌株DFR分型分别为内蒙古G11、宁夏G20和河北G17,见表 3。
对照DNA所有条带,经过多次重复实验,EV76疫苗株缺少DFR01、DFR02、DFR04、DFR10,见图 1。

|
| 注:1~23.分别为DFR1~23的鼠疫菌DNA电泳结果 图 1 鼠疫菌EV76疫苗株DFR分型PCR产物电泳结果 Figure 1 The electrophoretogram of the EV76 amplified with DFR1-23 and pMT1 |
| |
目前鼠疫菌检验的金标准依然是“四步检验”[3],再辅以血清学及PCR疫苗株实验进行验证,然而污染导致分离的EV76疫苗株和真正的野生株鼠疫菌无论是“四步检验”,还是血清和PCR都有着相同的结果,进一步的鉴定只能依靠比较繁琐费时的糖醇生化及毒力因子测定试验[4-5],而DFR分型技术建立在传统的PCR技术上,操作简便安全,结果可靠,更能迅速分辨出EV76疫苗株和野生株,避免投入不必要的人力、物力。
3.2 沙鼠型疫源地菌株的遗传进化基因缺失是细菌基因组进化的主要策略之一[6],DFR的获得与缺失受制于自然选择压力,推动了鼠疫菌基因组的简约进化,3个疫源地虽然同属内蒙古长爪沙鼠(Meriones unguiculatus)鼠疫疫源地,但是地理分布不同,生态环境不同使得鼠疫菌为适应疫源地的生态环境发生了基因的一些进化,其中宁夏的G20基因型菌株相比于内蒙古长爪沙鼠疫源地主要基因型G11,缺失了DFR12和DFR18,而分离自河北省鼠疫疫源地的G17基因型菌株相比于内蒙古的菌株缺失了DFR18,DFR12编码噬菌体蛋白,DFR18编码保守的功能未知蛋白[7-8]。
3.3 基因分型的意义近年来我国多数鼠疫自然疫源地的鼠间疫情活跃,沙鼠鼠疫疫源地也是持续活跃,几乎每年在动物间均有疫情发生,2017年冬季在内蒙古化德县与河北省康保县交界处先后分离出鼠疫菌,河北省鼠疫自然疫源地距北京约200 km,具有交通发达、人口密集、人流量大的特点,鉴于沙鼠疫源地的活跃趋势、鼠疫危害的严重性以及河北省鼠疫疫源地特殊的地理位置,对该疫源地的基因遗传特征进行深入研究分析,建立不同沙鼠鼠疫疫源地菌株基因分型数据库,可为今后的疫情调查、应急处理及追溯传染源等工作提供依据,同时也能为研究沙鼠鼠疫疫源地各菌株间亲缘关系和遗传进化特征提供数据。
| [1] |
杨晓艳, 魏柏青, 靳娟, 等. 中国鼠疫耶尔森菌差异区段分型及其地理分布特征[J]. 中华流行病学杂志, 2014, 35(8): 943-948. DOI:10.3760/cma.j.issn.0254-6450.2014.08.015 |
| [2] |
俞东征. 鼠疫动物流行病学[M]. 北京: 科学出版社, 2009: 276.
|
| [3] |
刘合智, 刘满福, 李玉贵. 河北省鼠疫自然疫源地内自然染疫蚤的研究[J]. 中国媒介生物学及控制杂志, 2005, 16(3): 206-208. DOI:10.3969/j.issn.1003-4692.2005.03.017 |
| [4] |
董国润, 王桂琴, 白晓英, 等. 115株鼠疫菌的生物学特性及流行病学研究[J]. 中国媒介生物学及控制杂志, 2001, 12(2): 112-114. DOI:10.3969/j.issn.1003-4692.2001.02.010 |
| [5] |
周冬生, 韩延平, 宋亚军, 等. 鼠疫耶尔森菌基因组进化与生态位适应研究[J]. 解放军医学杂志, 2004, 29(3): 204-210. DOI:10.3321/j.issn:0577-7402.2004.03.005 |
| [6] |
崔玉军, 宋亚军, 杨瑞馥. 鼠疫耶尔森菌的进化研究:从系统发育学到系统发育基因组学[J]. 中国科学:生命科学, 2013, 43(1): 23-30. DOI:10.1360/052012-424 |
| [7] |
杨瑞馥, 黄培堂. 鼠疫耶尔森菌比较和进化基因组学研究[J]. 解放军医学杂志, 2004, 29(3): 189-191. DOI:10.3321/j.issn:0577-7402.2004.03.001 |
| [8] |
Li YJ, Dai EH, Cui YJ, et al. Different region analysis for genotyping Yersinia pestis isolates from China[J]. PLoS One, 2008, 3(5): e2166. DOI:10.1371/journal.pone.0002166 |
 2018, Vol. 29
2018, Vol. 29