扩展功能
文章信息
- 陈辉莹, 林怡, 陈翰明, 刘义, 李凯利, 高景鹏, 陈学文, 蔡芳, 刘超, 杨新艳, 杨振洲, 彭恒, 马雅军
- CHEN Hui-ying, LIN Yi, CHEN Han-ming, LIU Yi, LI Kai-li, GAO Jing-peng, CHEN Xue-wen, CAI Fang, LIU Chao, YANG Xin-yan, YANG Zhen-zhou, PENG Heng, MA Ya-jun
- 海南省常见白蛉种特异分子特征的研究
- Molecular characteristics of common sandflies species (Diptera: Psychodidae) in Hainan province
- 中国媒介生物学及控制杂志, 2018, 29(1): 15-19
- Chin J Vector Biol & Control, 2018, 29(1): 15-19
- 10.11853/j.issn.1003.8280.2018.01.004
-
文章历史
- 收稿日期: 2017-10-30
- 网络出版时间: 2017-12-12 11:28
2 海口市疾病预防控制中心, 海口 571100;
3 解放军疾病预防控制中心, 北京 100071;
4 第二军医大学基础部病原生物学教研室, 上海 200433
2 Haikou Center for Disease Control and Prevention;
3 Center for Disease Control and Prevention of PLA;
4 Department of Medical Microbiology and Parasitology, Basic Medical College, Second Military Medical University
白蛉隶属双翅目(Diptera)、毛蠓科(Psychodidae)、白蛉亚科(Phlebotominae),全世界已记录8属900余种[1],是一类体型较小的吸血昆虫,部分种类可传播利什曼病、白蛉热和巴尔通体病等[2]。经多年研究,我国白蛉区系已基本阐明,北起内蒙古自治区和吉林省,南至海南省,东起沿海各省(直辖市、自治区),西至新疆维吾尔自治区,均有白蛉分布[3-6]。由于白蛉种群数量相对较少,不易捕获,且孳生地难以确定,海南省白蛉相关研究报道较少。1957年冷延家[7]在海南省发现方亮司蛉(Sergentomyia fanglianensis),2016年笔者在海南省三沙市发现贝氏司蛉(S. bailyi)分布[8]。传统的白蛉种类鉴定方法复杂,需要解剖白蛉观察其咽甲、口腔和受精囊等形态特征进行鉴别。随着分子生物学技术方法的迅速发展和广泛运用,利用分子特征进行白蛉分类鉴定成为可能。目前,许多研究者尝试利用多种分子标志鉴定白蛉,包括线粒体DNA(mitochondrial DNA,mtDNA)的细胞色素b(cytochromb,Cytb)、细胞色素C氧化酶亚基Ⅰ(COⅠ)及核糖体DNA(ribosomal DNA,rDNA)转录间隔区和18S编码区等序列[1, 9-14],澄清了一些混淆问题,使白蛉鉴别操作变得相对简单,而分子鉴别的前提是必须确定种特异的分子特征。
通过对采集自海南省海口市的白蛉进行形态特征鉴别,对应种类分析mtDNA Cytb基因部分片段序列,确定种特异的分子特征,初步了解了海口市白蛉的季节消长规律;并对种特异分子特征在海南省三沙市的白蛉鉴别进行了初步应用,丰富和更新了海南省白蛉的区系分布资料,为我国白蛉分子鉴定积累了数据。
1 材料与方法 1.1 调查地点2015年8月至2016年7月在海南省海口市选择不同的环境类型,即农村环境的演丰镇,城乡结合部的新琼村、周仁村和农垦卫校,及猪圈、鸡鸭圈或人房环境;2017年8月在海南省三沙市的永兴岛、石岛和赵述岛,选择宿舍周围的院落、废弃的猪圈等环境。
1.2 调查方法利用诱虫灯(MY-W-1型,广州市大四喜灭鼠除虫有限公司)每月至少1次进行通宵采集(18:00-08:00)。将捕获的样本带回实验室,解剖镜下根据外部形态分拣出白蛉并计数[3]。
1.3 形态鉴定取成蛉干燥标本检视其形态特征,分类鉴定至种。用75%乙醇完全浸润白蛉,晃动去除其体表的毛;用水冲洗后,在解剖镜下切下头部和尾部(腹部第8~10节),置10%KOH溶液中,37 ℃浸泡20 h;用水洗净后,置于解剖镜下,持针解剖头部的口腔、咽甲及雌蛉的受精囊,移至显微镜下观察并拍照,根据形态特征鉴定至种[3]。
1.4 mtDNA Cytb基因扩增与测序收集经形态鉴定的白蛉样本,将其解剖后的组织置60 μl DNAzol(Invitrogen,美国)中,按照说明书抽提基因组DNA。参照文献[15]合成引物,扩增mtDNA Cytb基因部分片段,序列如下:正向(CB1)5′-TAT GTA CTA CCA TGA GGA CAA ATA TC-3′,反向(CB3-R3A)5′-GCT AAT TAC TCC TCC TAA CTT ATT-3′。PCR反应体系:1×KOD FX Neo缓冲液、0.4 mmol/L dNTPs、0.3 μmol/L引物、0.5 U KOD FX Neo酶(TOYOBO,日本)和4.0 μl模板,总体积25 μl。反应条件:94 ℃ 2 min;94 ℃ 30 s,45 ℃ 30 s,72 ℃ 1 min,4个循环;94 ℃ 30 s,51 ℃ 30 s,72 ℃ 1 min,30个循环;72 ℃ 8 min。扩增产物经1%琼脂糖凝胶(1.5 μl/40 ml Goldview)电泳检测后,送至铂尚生物技术(上海)有限公司,应用四色荧光标记的双脱氧末端终止法,以正向引物进行测序。
1.5 mtDNA Cytb序列分析获得的序列用SeqMan检查峰图,应用MEGA 6.0软件包进行序列比对,结合形态鉴定结果,计算序列在个体间的遗传距离(p distance),确定白蛉的种特异序列。收集GenBank公布的我国白蛉与本研究蛉种的mtDNA Cytb基因序列,用MEGA 6.0软件计算种间遗传距离,应用最大似然法(Maximum Likelihood,ML)构建系统发育树,并进行1 000次置信值分析,建树前检验其最佳替换模型。
2 结果 2.1 白蛉的季节消长2015年8月至2016年7月,在海口市共捕获白蛉377只。其中2016年1-4月捕获的白蛉数量极少,5-6月呈快速上升趋势,白蛉密度在6月达到高峰,7-10月维持在稳定水平,11月再次上升,12月再次迅速下降,见图 1。

|
| 图 1 海口市白蛉季节消长曲线 Figure 1 Seasonal fluctuations of sandflies in Haikou city |
| |
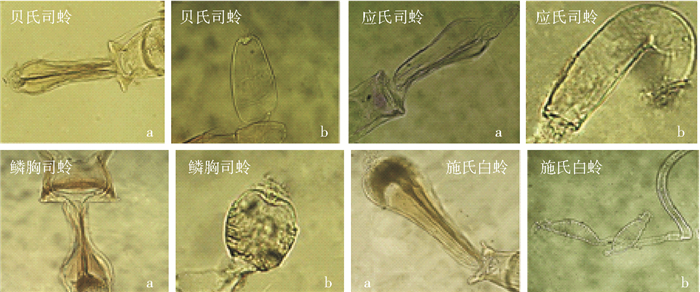
随机挑取58只雌性白蛉进行解剖和形态鉴定(图 2),检视其口腔、咽甲和受精囊特征,经鉴定隶属于2属4种,分别为贝氏司蛉36只、应氏司蛉(S. iyengari)3只、鳞胸司蛉(S. squamipleuris)4只和施氏白蛉(P. stantoni)15只。
|
| 注:a.口腔和咽甲;b.受精囊 图 2 海口市4种白蛉 Figure 2 Four sandfly species in Haikou city |
| |
共获得49条白蛉mtDNA Cytb基因序列,片段长度为410 bp,无插入和缺失位点,其中保守位点301个(73.41%),变异位点109个(26.59%),自裔位点1个,平均(G+C)含量为30.50%。鳞胸司蛉个体间序列保守(MF966766~MF966768)无任何差异,个体间遗传距离贝氏司蛉(MF966769~MF966788,MF966790~MF966799)为0~0.046,应氏司蛉(MF966751~MF966752)为0.012,施氏白蛉(MF966753~MF966765)为0.002~0.007。
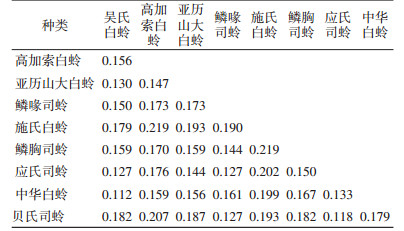
2.4 种间mtDNA Cytb基因序列差异从GenBank中下载已公布的我国分布的白蛉mtDNA Cytb序列,包括吴氏白蛉(P. wui,HQ704685.1)、高加索白蛉(P. caucasicus,FJ217390.1)、亚历山大白蛉(P. alexandri,KX024723.1)、中华白蛉(P. chinensis,MF966800)和鳞喙司蛉(S. squamirostris,MF966807),将这些序列与本研究获得的贝氏司蛉、应氏司蛉、鳞胸司蛉和施氏白蛉的Cytb基因部分序列进行比对,共2属9种。种间遗传距离为0.112~0.219,见表 1。应用GTR+G模型构建ML系统发育树,拓扑结构显示,中华白蛉与吴氏白蛉、鳞胸司蛉与鳞喙司蛉分别聚为一支,置信值大于50%,但其他分支置信值较低,可信度不高,司蛉属种类与白蛉属种类未各自聚类,见图 3。
 |

|
| 图 3 基于mtDNA Cytb序列的我国9种白蛉系统发育关系(ML法) Figure 3 The phylogenetic relationships of 9 sandflies based on mtDNA Cytb sequences (ML method) |
| |
对采自三沙市永兴岛、石岛和赵述岛的14只白蛉mtDNA Cytb基因片段序列进行分析发现,其个体间无任何差异,与采自海口市的贝氏司蛉mtDNA Cytb基因序列进行比对,差异范围为0~0.049,确认采自三沙市的白蛉均为贝氏司蛉。通过对本研究贝氏司蛉38个个体的mtDNA Cytb进行比对,显示存在19个变异位点,其中15个为固定差异,均存在于三沙群体与海口群体的样本中,海口群体的个体间差异为0~0.014。应用T92+G模型构建ML系统发育树,拓扑结构显示,可分为2个大支,分别对应海口与三沙群体的贝氏司蛉样本,见图 4。

|
| 图 4 基于mtDNA Cytb基因序列的海口市与三沙市贝氏司蛉群体的聚类关系(ML法) Figure 4 The clustering relationships of S. bailyi samples from Haikou and Sansha city based on mtDNA Cytb sequences (ML method) |
| |
我国白蛉区系研究主要集中在利什曼病流行的地区,目前其他地区的研究报道较少,且报道时间较早[4-6]。我国海南省曾经记载10种白蛉,即印地格蛉(Geassomyia indica)、中华白蛉、广西司蛉(S. kwangsiensis)、施氏白蛉、应氏司蛉、马来司蛉(S. malayensis)、鲍氏司蛉(S. barraudi)、贝氏司蛉、平原司蛉(S. campester)和方亮司蛉[5, 7],其中贝氏司蛉为优势种[16]。熊光华等[3]记载海南省分布的白蛉为后7种及鳞胸司蛉。本研究基于形态和分子特征进行分类,确定了4种白蛉,均为已报道的白蛉种类,但尚有7种白蛉未检测到,可能因采集的范围和环境不够广泛,或与生态环境变化有关。因相关报道较少,海南省白蛉种群是否出现变化,有待进一步调查证实。
海口市白蛉全年活动时间长,但种群密度较低,可能与海口市城市化程度越来越高,导致孳生地减少有关,也可能与海口市杀虫剂使用量较大有关[17]。因此,应进一步收集海南省其他地区的白蛉进行综合分析,从而更准确地评估海南省白蛉种群密度及其消长规律。海口市白蛉的季节消长规律与当地海洋性热带季风气候的温湿度变化有关,当地1-4月气温较低,不适宜白蛉活动,6月和11月白蛉密度较高,提示海口市白蛉每年至少可完成2个生活史周期。目前,虽无海南省白蛉传播疾病的直接证据,但本研究结果提示,在6月和11月应加强个人防护,避免白蛉叮咬吸血。
种类鉴定是生物学研究的基础,经典分类学通常依据个体外部形态特征,白蛉个体较小,主要依据咽甲、口腔和受精囊等特征进行鉴别,而这些结构需要有经验的专业人员解剖后于显微镜下观察,因此,白蛉的形态学鉴定比较困难;且部分蛉种的形态存在一定个体变异,为准确鉴定种类增加了难度[18-19]。mtDNA和rDNA部分片段序列具有种的特异性,近年来DNA提取技术日益成熟和简化,测序费用越来越低,故对白蛉进行分子分类已成为研究热点之一。mtDNA Cytb基因序列作为白蛉DNA条形码已用于多种白蛉的种类鉴别和群体遗传差异分析[1, 20-28]。本研究采用该基因序列对海南省常见白蛉进行分子分类,发现种内遗传差异最大的为贝氏司蛉(0.049),而种间遗传差异最小为吴氏白蛉与中华白蛉(0.112),种间遗传差异显著大于种内差异,提示mtDNA Cytb基因序列可作为区分上述白蛉的DNA条形码。同时,系统发育关系拓扑结构显示,相同属阶元种类并未聚在一支,可能因应用的蛉种代表性不够,或mtDNA Cytb基因序列在种上阶元的解析度不高,因此,需积累更多的白蛉种类数据进行深入分析。
有报道分析,贝氏司蛉是包含多个隐种的复合体[9],其外部形态已有分化,触角鞭节和叉形刺的长度有所不同[29-30]。在印度及斯里兰卡,存在贝氏司蛉A种与B种(或Ⅰ和Ⅱ),两种间的mtDNA COⅠ序列差异大于0.135[9, 14, 30],但未见mtDNA Cytb基因序列差异性。本研究共获得38条贝氏司蛉的mtDNA Cytb序列,构建的ML系统发育树白蛉分为海口群体和三沙群体,提示海南省贝氏司蛉不同群体已出现遗传分化,可能存在隐种,本研究获得的mtDNA-COⅠ序列数据也支持该结论(未发表数据)。但其形态特征是否存在差异需进一步观察记录。
综上所述,本研究通过收集海南省海口市及三沙市的白蛉,了解了海口市白蛉活动的初步规律,确定了可用于种类鉴别的分子特征;基于mtDNA Cytb分子特征分类的贝氏司蛉在我国可能存在隐种。
志谢: 中国CDC寄生虫病预防控制所金长发在本研究的标本形态鉴定中提供很大帮助,海口市CDC王晓花、林春燕和林忠岭,海南省CDC王善青、曾林海,解放军91922部队卫生防疫所詹道成,解放军91892部队医院凌晓敏、孙民民等协助采集现场标本,一并志谢| [1] |
Depaquit J. Molecular systematics applied to Phlebotomine sandflies:review and perspectives[J]. Infect Genet Evol, 2014, 28: 744-756. DOI:10.1016/j.meegid.2014.10.027 |
| [2] |
Service MW. Medical entomology for students[M]. 3rd ed. Liverpool: Cambridge University Press, 2004, 90-98.
|
| [3] |
熊光华, 金长发, 管立人. 中国的白蛉[M]. 北京: 科学出版社, 2016, 15-25.
|
| [4] |
杨曼尼, 马雅军. 我国白蛉的分类现状[J]. 国际医学寄生虫病杂志, 2008, 35(1): 46-49. |
| [5] |
Zhang LM, Leng YJ. Eighty-year research of Phlebotomine sandflies (Diptera:Psychodidae) in China (1915-1995). Ⅱ. Phlebotomine vectors of Leishmaniasis in China[J]. Parasite, 1997, 4(4): 299-306. DOI:10.1051/parasite/1997044299 |
| [6] |
管立人. 中国白蛉(双翅目:毛蛉科)调查研究工作的展望[J]. 中国寄生虫学与寄生虫病杂志, 2013, 31(4): 310-314. |
| [7] |
冷延家. 在海南岛继续发现的蛉种与新种白蛉:方亮白蛉Phlebotomus fanglianensis sp. nov.的记述[J]. 昆虫学报, 1964, 13(1): 118-128. |
| [8] |
杨明磊, 李逸明, 陈辉莹, 等. 我国西沙群岛吸血昆虫的种群动态和鼠形动物病原感染检测[J]. 第二军医大学学报, 2016, 37(3): 355-359. |
| [9] |
Kumar NP, Srinivasan R, Jambulingam P. DNA barcoding for identification of sand flies (Diptera:Psychodidae) in India[J]. Mol Ecol Resour, 2012, 12(3): 414-420. DOI:10.1111/men.2012.12.issue-3 |
| [10] |
de Souza Pinto I, das Chagas BD, Rodrigues AAF, et al. DNA barcoding of neotropical sand flies (Diptera, Psychodidae, Phlebotominae):species identification and discovery within Brazil[J]. PLoS One, 2015, 10(10): e0140636. DOI:10.1371/journal.pone.0140636 |
| [11] |
Polseela R, Jaturas N, Thanwisai A, et al. Towards monitoring the sandflies (Diptera:Psychodidae) of Thailand:DNA barcoding the sandflies of Wihan Cave, Uttaradit[J]. Mitochondrial DNA A DNA Mapp Seq Anal, 2016, 27(5): 3795-3801. |
| [12] |
Kocher A, Gantier JC, Gaborit P, et al. Vector soup:high-throughput identification of Neotropical phlebotomine sand flies using metabarcoding[J]. Mol Ecol Resour, 2017, 17(2): 172-182. DOI:10.1111/men.2017.17.issue-2 |
| [13] |
Dokianakis E, Tsirigotakis N, Christodoulou V, et al. DNA sequencing confirms PCR-RFLP identification of wild caught Larroussius sand flies from crete and cyprus[J]. Acta Trop, 2016, 164: 314-320. DOI:10.1016/j.actatropica.2016.09.003 |
| [14] |
Gajapathy K, Tharmasegaram T, Eswaramohan T, et al. DNA barcoding of Sri Lankan phlebotomine sand flies using cytochrome c oxidase subunitⅠ reveals the presence of cryptic species[J]. Acta Trop, 2016, 161: 1-7. DOI:10.1016/j.actatropica.2016.05.001 |
| [15] |
Esseghir S, Ready PD, Ben-Ismail R. Speciation of Phlebotomus sandflies of the subgenus Larroussius coincided with the late Miocene-Pliocene aridification of the Mediterranean subregion[J]. Biol J Linn Soc, 2000, 70(2): 189-219. DOI:10.1111/bij.2000.70.issue-2 |
| [16] |
冷廷家. 贝氏白蛉及其亚种分类的研究[J]. 暨南大学学报:自然科学版, 1980, 1(2): 23-30. |
| [17] |
高景鹏, 李凯利, 陈辉莹, 等. 菊酯类杀虫剂对海口市白纹伊蚊的效果测定[J]. 中国热带医学, 2017, 17(9): 867-870. |
| [18] |
Zhang L, Ma YJ, Xu JN. Genetic differentiation between sandfly populations of Phlebotomus chinensis and Phlebotomus sichuanensis (Diptera:Psychodidae) in China inferred by microsatellites[J]. Parasit Vectors, 2013, 6(1): 115. DOI:10.1186/1756-3305-6-115 |
| [19] |
Zhang L, Ma YJ. Identification of Phlebotomus chinensis (Diptera:Psychodidae) inferred by morphological characters and molecular markers[J]. Entomotaxonomia, 2012, 34(1): 71-80. |
| [20] |
Parvizi P, Naddaf SR, Alaeenovin E. Molecular typing and phylogenetic analysis of some species belonging to Phlebotomus (Larroussius) and Phlebotomus (Adlerius) subgenera (Diptera:psychodidae) from two locations in Iran[J]. Iran J Arthropod Borne Dis, 2010, 4(2): 1-10. |
| [21] |
Krüger A, Strüven L, Post RJ, et al. The sandflies (Diptera:Psychodidae, Phlebotominae) in military camps in northern Afghanistan (2007-2009), as identified by morphology and DNA "barcoding"[J]. Ann Trop Med Parasitol, 2011, 105(2): 163-176. DOI:10.1179/136485911X12899838683241 |
| [22] |
Berdjane-Brouk Z, Koné AK, Djimdé AA, et al. First detection of Leishmania major DNA in Sergentomyia (Spelaeomyia) darlingi from cutaneous leishmaniasis foci in Mali[J]. PLoS One, 2012, 7(1): e28266. DOI:10.1371/journal.pone.0028266 |
| [23] |
Gajapathy K, Peiris LB, Goodacre SL, et al. Molecular identification of potential leishmaniasis vector species within the Phlebotomus (Euphlebotomus) argentipes species complex in Sri Lanka[J]. Parasit Vectors, 2013, 6(1): 302. DOI:10.1186/1756-3305-6-302 |
| [24] |
Kasap OE, Vot'pka J, Alten B. The distribution of the Phlebotomus major complex (Diptera:Psychodidae) in Turkey[J]. Acta Trop, 2013, 127(3): 204-211. DOI:10.1016/j.actatropica.2013.05.001 |
| [25] |
Bounamous A, Lehrter V, Hadj-Henni L, et al. Limits of a rapid identification of common Mediterranean sandflies using polymerase chain reaction-restriction fragment length polymorphism[J]. Mem Inst Oswaldo Cruz, 2014, 109(4): 466-472. DOI:10.1590/0074-0276130584 |
| [26] |
Depaquit J, Randrianambinintsoa FJ, Jaouadi K, et al. Molecular and morphological systematics of the sandfly Sergentomyia (Sintonius) clydei Sinton, 1928 and questions about its record in the Seychelles[J]. Infect Genet Evol, 2014, 21: 41-53. DOI:10.1016/j.meegid.2013.10.016 |
| [27] |
Ebrahimi S, Bordbar A, Parvizi P. Genetic dynamics in the sand fly (Diptera:Psychodidae) nuclear and mitochondrial genotypes:evidence for vector adaptation at the border of Iran with Iraq[J]. Parasit Vectors, 2016, 9(1): 319. DOI:10.1186/s13071-016-1603-5 |
| [28] |
张丽, 马雅军. 基于线粒体DNA的我国中华白蛉群体遗传分化研究[J]. 中国热带医学, 2016, 16(10): 947-952. |
| [29] |
Yogeswari S, Srinivasan R. A note on variations in morphological features of the Phlebotomine sand fly Sergentomyia bailyi (Diptera:Psychodidae) in a population from Pondicherry UT, India[J]. J Med Entomol, 2016, 53(3): 712-716. DOI:10.1093/jme/tjv249 |
| [30] |
Tharmatha T, Gajapathy K, Ramasamy R, et al. Morphological and molecular identification of cryptic species in the Sergentomyia bailyi (Sinton, 1931) complex in Sri Lanka[J]. Bull Entomol Res, 2017, 107(1): 58-65. DOI:10.1017/S0007485316000626 |
 2018, Vol. 29
2018, Vol. 29


