扩展功能
文章信息
- 齐润姿, 王董, 普丽娅, 赵欣, 高雪萌, 马翔, 余斌, 张晓龙
- QI Run-zi, WANG Dong, PU Li-ya, ZHAO Xin, GAO Xue-meng, MA Xiang, YU Bin, ZHANG Xiao-long
- 应用细胞色素C氧化酶亚基Ⅰ和16S rRNA基因鉴定浙江口岸鼠类及其序列特征分析
- Identification of rodent species in Zhejiang port using cytochrome C oxidase subunitⅠ and 16S rRNA gene and analysis of sequence characteristics
- 中国媒介生物学及控制杂志, 2017, 28(2): 104-107, 116
- Chin J Vector Biol & Control, 2017, 28(2): 104-107, 116
- 10.11853/j.issn.1003.8280.2017.02.002
-
文章历史
- 收稿日期: 2016-10-24
- 网络出版时间: 2017-02-17 09:42
2 中国检验检疫科学研究院, 北京 100176;
3 温州出入境检验检疫局, 浙江 温州 325027
2 Chinese Academy of Inspection and Quarantine;
3 Wenzhou Entry-Exit Inspection and Quarantine Bureau
目前鼠类鉴定主要依靠传统形态学和分子鉴定手段。传统形态学鉴定比较经典但存在局限性,受鉴定者的经验、鉴定对象形态完整程度及性别、发育阶段等限制。分子鉴定则主要应用动物线粒体DNA(mitochondrial DNA,mtDNA)序列差异进行鉴定。由于mtDNA具有分子小且稳定、结构简单、进化速度快、母系遗传、检测方便等优点而成为有效的分子鉴定标记,被广泛应用。诸多研究运用线粒体细胞色素C氧化酶亚基Ⅰ(COⅠ)、细胞色素b(Cytb)、16S rRNA和D-loop等分子标记对不同地区、不同鼠种进行鉴定[1-6]。本研究则选择16S rRNA和COⅠ基因作为分子标记对浙江口岸捕获的鼠类进行鉴定及其序列特征分析。
1 材料与方法 1.1 标本采集于2015年4-10月,采用鼠笼法在浙江口岸捕获鼠类,全部解剖获取肝脏样品,保存于-80 ℃,用于提取基因组DNA。
1.2 DNA提取、扩增及测序用电动研磨棒将肝脏样品研磨至匀浆。按血液/细胞/组织基因组DNA提取试剂盒〔离心柱型试剂盒编号:DP304;天根生化科技(北京)有限公司生产〕说明书提取肝脏中的基因组DNA,并于-20 ℃保存备用。COⅠ基因扩增引物为BatL5310(5′-CCT ACT CRG CCA TTT TAC CTA TG-3′)和R6036R(5′-ACT TCT GGG TGT CCA AAG AAT CA-3′)[7];扩增条件:94 ℃热变性5 min,再以94 ℃ 30 s、55 ℃ 30 s、72 ℃ 1 min为条件循环35次,最后72 ℃退火10 min。16S rRNA基因扩增上游引物:5′-CGC CTG TTT AAC AA AAA CAT-3′ [8],下游引物:5′-AGA TCA CGT AGG ACT TTA AT-3′ [9];扩增条件:94 ℃预变性3 min;94 ℃ 30 s、50 ℃ 40 s,72 ℃ 1 min,循环35次;72 ℃延伸7 min。PCR反应体系为25 μl:2×TSINGKETM MasterMix 12.5 μl,灭菌蒸馏水9.5 μl,模板2 μl,上下游引物各0.5 μl。经琼脂糖凝胶电泳后,将扩增条带单一且清晰的扩增产物送北京擎科新业生物技术有限公司进行测序。
1.3 数据分析 1.3.1 序列矫正及同源性比对利用SeqMan软件进行序列拼接,同时结合Chromas软件读取测序峰图进行矫正,然后在GenBank上进行同源性比对,确定所得序列的准确性。同时下载GenBank中的鼠类COⅠ数据22条及16S rRNA数据13条,比对结果及其他数据见表 1。
利用Mega 5.05软件对所得序列片段进行组成分析,计算总序列的碱基变异位点、保守位点、转换与颠换的比率(Tv/Ts)以及各碱基(A、T、C、G)的平均含量。采用Mega 5.05软件基于Kimura-2-parameter(K2P)模型计算各分类单元间的遗传距离。
1.3.3 构建系统发育树运用Clustal X 2.1软件对数据进行多重比对,并人工修正比对结果。Paup4.0b 10和ModelTest 3.7软件结合使用确定最佳进化模型。采用邻接法(Neighbor-Joining,NJ)和贝叶斯法(Bayesian Inference,BI)(MrBayes 3.2)构建单基因系统发育树和联合基因系统发育树。
2 结果获得8条COⅠ序列和16S rRNA序列,通过比对确定为褐家鼠(Rattus norvegicus)和小家鼠(Musmusculus)序列各4条,COⅠ扩增长度(不含引物)均为702 bp,褐家鼠和小家鼠的16S rRNA基因长度分别为537和542 bp。
2.1 序列组成分析通过比对剪切结果,30条COⅠ序列片段总长度为620 bp,21条16S rRNA序列片段总长度为533 bp。COⅠ序列片段中保守位点430个,变异位点190个,变异率为30.64%;碱基(A+T)平均含量为57.60%,T含量最高,为29.50%;G含量最低,为17.00%。碱基频率均存在偏倚性。碱基替换分析显示,核苷酸替换主要出现在密码子第3位点上,为88.70%,第2位点最为保守,无变异。其总的Tv/Ts为1.72,转换大于颠换;转换以T、C间为主,颠换以A、T间为主。
16S rRNA序列片段中保守位点424个,变异位点109个,变异率为20.45%;碱基(A+T)平均含量61.10%;A含量最高,为33.50%;C含量最低,为19.20%。碱基频率均存在偏倚性。Tv/Ts为1.31,转换大于颠换;转换以T、C间为主,颠换以A、T间为主。
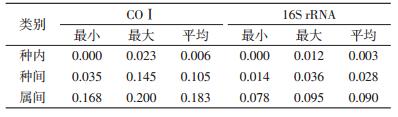
2.2 遗传距离分析COⅠ和16S rRNA基因的遗传距离为种内<种间<属间,遗传距离随分类阶元的上升而增加,无重叠且有一定的距离间隔(表 2),说明两种基因均可以作为鼠亚科分类鉴定的DNA分子标记。
|
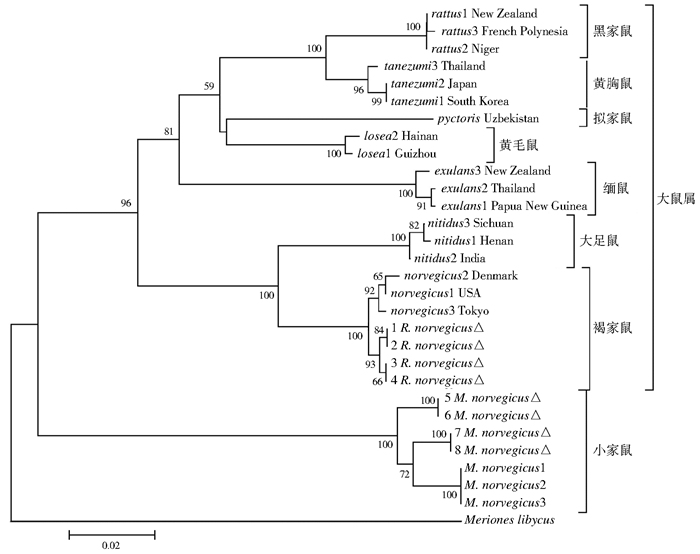
本研究内群数据为鼠亚科的大鼠属和小鼠属,故外群选择沙鼠亚科沙鼠属的红尾沙鼠(Meriones libycus)。基于部分COⅠ和16S rRNA基因以及两个基因的联合数据,分别采用K2P和GTR模型构建NJ树和BI树。结果显示,相同基因、不同方法构建的系统发育树拓扑结构基本一致(16S rRNA基因NJ树和BI树不同),仅后验概率和自展支持率不尽相同,BI法所构建的系统发育树后验概率普遍高于NJ法的自展支持率。
16S rRNA基因的NJ树和BI树仅在黄胸鼠(R. tanezumi)和黑家鼠(R. rattus)这一分支位置不同,NJ树中两个鼠种聚在一起,再与大鼠属其他种聚在一起,见图 1;BI树中其各自为一分支,为平行分支,其余拓扑结构完全一致。
|
| 注:△为浙江口岸数据,<50的自展支持率未显示。 图 1 基于COⅠ基因构建的NJ树 Figure 1 Neighbor-Joining tree based on COⅠ gene |
| |
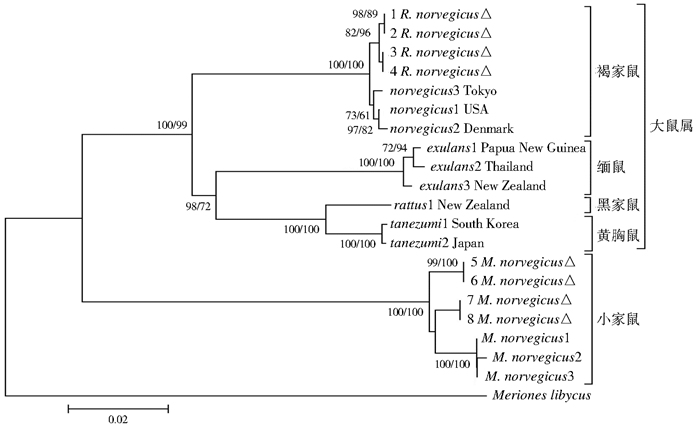
COⅠ基因的NJ树和BI树拓扑结构有微小差异,BI树中褐家鼠单独为一支,而NJ树中褐家鼠和大足鼠(R. nitidus)聚为一支;NJ树中,拟家鼠(R. pyctoris)和黄毛鼠(R. losea)聚为一支,节点自展支持率<50,见图 1。但在BI树中,其与黄胸鼠和黑家鼠聚为一大支,其余拓扑结构完全一致。两基因联合的NJ树和BI树拓扑结构除节点自展支持率不一致外,总体拓扑结构完全一致,见图 2。
|
| 注:△为浙江口岸数据;<50的自展支持率未显示;图中数值为后验概率/自展支持率。 图 2 16S rRNA和COⅠ基因联合构建的BI及NJ合一树 Figure 2 Neighbor-Joining and Bayesian Inference unity tree based on COⅠ and 16S rRNA gene |
| |
本研究捕获的褐家鼠和小家鼠均为浙江口岸常见鼠种[10-12]。2005-2006年浙江口岸以褐家鼠为优势种[10];而2013-2015年以小家鼠为主要鼠种,占22.5%。故本研究选取的鼠类在浙江口岸有一定代表性。
通过COⅠ氨基酸比对发现只有3个(含外群)氨基酸变异,而除去外群则仅有1个氨基酸位点变异,变异率为0.5%,而大鼠属和小鼠属的碱基变异率为27.4%,表明大鼠属和小鼠属的COⅠ基因在进化过程中以同义突变为主。从总体碱基变异率和平均遗传距离看,COⅠ基因的变异率大于16S rRNA基因,且COⅠ基因的种内、种间、属间的平均遗传距离均是16S rRNA基因的2~3倍,表明后者较前者保守。COⅠ基因种间的最小遗传距离是种内最大遗传距离的近1.5倍(表 1),中间有一定间隔,可较好地区分不同鼠种,而种间最大遗传距离则与属间最小遗传距离接近;反之,16S rRNA基因种间的最小遗传距离与种内最大遗传距离相近,种间最大遗传距离则与属间最小遗传距离间隔较远。说明16S rRNA基因较COⅠ基因更适用于高级阶元的分类研究,而COⅠ基因适合低级阶元的分类研究,16S rRNA基因的种类鉴别能力低于COⅠ基因,与Nicolas等[13]结论一致。
本研究中因数据库的16S rRNA基因数据少于COⅠ基因,故联合基因建树并未包含所有样本数据,但鼠种种类仍较全,因此,系统发育树间仍可比较,且所构建的6个系统发育树拓扑结构基本一致。对于16S rRNA基因BI树的平行分支,黑家鼠分支与黄胸鼠分支、大鼠属其余种类分支的遗传距离分别为0.014和0.029;黄胸鼠分支与大鼠属其余种类分支遗传距离为0.033,故比较合理的拓扑结构应为黑家鼠与黄胸鼠聚为一支,即16S rRNA基因的NJ树较为合理。同理,在COⅠ基因的NJ树中,褐家鼠分支与大足鼠分支的遗传距离最近,为0.059,应聚为一支,即COⅠ基因NJ树拓扑结构较为合理。因此,可利用分支间遗传距离判断系统发育树拓扑结构的合理性。16S rRNA单基因树与两基因联合树拓扑结构基本一致,两基因联合的BI树和NJ树拓扑结构完全一致。总体来说,联合基因构建的系统发育树的拓扑结构较单基因构建的稳定,6个系统发育树间分支结构差异不大,主要体现在种间分支结构不稳定,可能与所选基因包含的系统发育信息量有关,但种内的分支结构相对稳定。
目前,物种的鉴定方法均存在不足,如形态学鉴定中若发生趋同进化易导致鉴定错误,分子生物学各方法可能得不到统一结果等。故应将形态学、分子生物学、行为生态、染色体组型分析等信息结合进行物种鉴定,从而得到较为可靠的结果。
| [1] | 马英, 李海龙, 鲁亮, 等. DNA条形码技术在青海海东地区小型兽类鉴定中的应用[J]. 生物多样性, 2012, 20(2): 193–198. |
| [2] | 常子丽, 刘芳, 王建军, 等. DNA条形码鉴别内蒙古地区啮齿动物[J]. 生物技术通报, 2013, (8): 94–98. |
| [3] | 刘润吉, 张荣波, 郭天宇, 等. DNA条形码技术在贵州茂兰鼠类鉴定中的应用[J]. 中华卫生杀虫药械, 2014, 20(1): 59–62. |
| [4] | 胡群, 马思杰, 袭炯良. 4种DNA条形码在黄毛鼠种类鉴定中的比较[J]. 中国媒介生物学及控制杂志, 2015, 26(3): 286–289. |
| [5] | 庞博, 侯志军, 柴洪亮, 等. 基于传统形态分类学和DNA条形码技术确定东方田鼠在贺兰山的新分布[J]. 经济动物学报, 2015, 19(4): 195–201. |
| [6] | 骆星丹, 王董, 胡燕, 等. 三峡库区沿江口岸鼠类及其分子鉴定特征研究[J]. 中国国境卫生检疫杂志, 2015, 38(增刊1): 1–3. |
| [7] | Robins JH, Hingstion M, Matisoo-Smith E, et al. Identifying Rattus species using mitochondrial DNA[J]. Mol Ecol Notes, 2007, 7(5) : 717–729 .DOI:10.1111/men.2007.7.issue-5. |
| [8] | Palumbi SR, Martin AP, Romano SL, et al. The simple fool's guide to PCR[M]. Honolulu: University of Hawaii Press, 1991: 1-18. |
| [9] | Quérouil S, Hutterer R, Barrière P, et al. Phylogeny and evolution of African shrews(Mammalia:Soricidae) inferred from 16S rRNA sequences[J]. Mol Phylogenet Evol, 2001, 20(2) : 185–195 .DOI:10.1006/mpev.2001.0974. |
| [10] | 陈敏, 周健青, 颜小军. 浙江口岸2005-2006年鼠形动物及体表寄生虫调查[J]. 中国国境卫生检疫杂志, 2007, 30(3): 159–162. |
| [11] | 高雪萌, 陈建国, 刘峰. 2012-2013年温州七里港口岸医学媒介生物调查及分析[J]. 中国国境卫生检疫杂志, 2014, 37(4): 259–263. |
| [12] | 高雪萌, 陈建国, 谢建雄, 等. 2014年温州七里港口岸医学媒介生物调查[J]. 中国国境卫生检疫杂志, 2016, 39(1): 29–32. |
| [13] | Nicolas V, Schaeffer B, Missoup AD, et al. Assessment of three mitochondrial genes (16S, Cytb, COⅠ) for identifying species in the Praomyini tribe (Rodentia:Muridae)[J]. PLoS One, 2012, 7(5) : e36586.DOI:10.1371/journal.pone.0036586. |
 2017, Vol. 28
2017, Vol. 28