扩展功能
文章信息
- 单振菊, 邱德义, 岳巧云
- SHAN Zhen-ju, QIU De-yi, YUE Qiao-yun
- 一种具厚几丁质外壳微小节肢动物无损伤凭证标本高质量DNA模板的获取方法
- A method of obtaining high quality DNA templates from tiny thick-chitin arthropods without damaging morphology of the voucher specimens
- 中国媒介生物学及控制杂志, 2017, 28(1): 27-30
- Chin J Vector Biol & Control, 2017, 28(1): 27-30
- 10.11853/j.issn.1003.8280.2017.01.008
-
文章历史
- 收稿日期: 2016-09-01
- 网络出版时间: 2016-12-06 17:10
2 中山大学生命科学学院, 广东 广州 510275
2 College of Life Science Sun Yat-sen University
目前,对物种的分类和鉴定主要依靠形态学方法,传统的形态学鉴定方法存在一定的缺陷,如大多数医学媒介生物的形态鉴定是以成虫完整的形态特征为基础,而肢体残缺的虫体很难准确鉴定[1]。随着PCR技术的建立与完善,涌现出一系列分子生物学技术,这些新技术在物种鉴定方面己经广泛应用,很大程度上弥补了传统形态学鉴定的不足,其中DNA条形码技术就是此类技术中具有代表性的一种[2-3]。对于微小节肢动物,通常制备成玻片标本进行鉴定和保存[4],该方法专业性高,专家依赖性强,DNA条形码技术为这种小型节肢动物的鉴定提供了一个可行的方法。由于个体微小,常规模板DNA的获取需要对整个个体进行研磨,破坏了标本的形态,造成凭证标本不能保存,有时甚至需要将几个甚至几十个样品混为一组进行研磨,难以保证模板DNA来源的单一性,不利于后续的结果分析[5]。近年来,越来越多的学者在不断摸索针对微小节肢动物个体的DNA条形码获取方法,也有一些采取直接裂解无损伤凭证标本形态的方法[5-7],但对于具厚几丁质外壳的微小节肢动物个体没有效果,如土壤甲螨。因此,有必要研究一种针对具厚几丁质外壳微小节肢动物模板DNA的获取方法,以保证凭证标本的完整性。
几丁质酶是降解几丁质的糖苷酶,关于几丁质酶的研究主要集中在基因结构、其在植物病害防治中的作用或利用几丁质酶测酶活性等方面[8-10],未发现有利用几丁质酶消化微小节肢动物几丁质壳,用于基因组DNA提取的报道。本研究尝试用几丁质酶去消化微小节肢动物厚的几丁质外壳,以达到不研磨微小节肢动物个体,保证凭证标本的形态完整性并能获得高质量DNA模板的目的。
1 材料与方法 1.1 材料 1.1.1 甲螨的获得按文献[11]方法,于2016年4月20日采自中山出入境检验检疫局后山。
1.1.2 试剂0.05 mol/L pH值6.0磷酸盐缓冲液配制:0.05 mol/L NaH2PO4 87.7 ml,0.05 mol/L Na2HPO4 12.3 ml配制成100 ml的磷酸盐缓冲液。组织和血液DNA磁珠提取试剂盒(Magpure Tissue & Blood DNA KF Kit,Magen公司)。几丁质酶chitinase购于Sigma公司。
1.2 方法 1.2.1 甲螨几丁质外壳的消化综合几丁质酶厂家提供的产品说明书和相关资料[12]以及本研究的实际情况,最终确定在25 ℃,pH值 6.0条件下进行微小节肢动物厚几丁质外壳的消化。
用pH值6.0磷酸盐缓冲液将几丁质酶配制成浓度分别为0.01、0.10、1.00 mg/ml的溶液,分别处理4、8、24 h,每个浓度、每个处理时间进行6个重复。将甲螨分成4组,第1组6只,不用几丁质酶消化,直接进行模板DNA的提取,作为对照;其他3组各18只,将3组分别放入1.5 ml离心管中,分别加入100 μl各浓度的酶溶液,每组分别在25 ℃金属浴中消化4、8、24 h,取出6只,终止消化,备用。
1.2.2 DNA模板制备按照组织和血液DNA磁珠提取试剂盒说明书,稍加改进,手动提取模板DNA,具体如下:将上述用几丁质酶处理的甲螨样品取出,将单个甲螨分别放入1.5 ml离心管中,加入200 μl ATL裂解液和20 μl蛋白酶K,55 ℃ 裂解过夜;然后加入200 μl AL液,70 ℃温浴10 min;短暂离心,移上清液至EP管中,加入GW1洗液600 μl;13 400×g离心1 min,重复2次;再加入GW2洗液600 μl;13 400×g离心1 min,重复2次;晾干,加入50 μl AE溶解DNA,13 400×g,离心2 min,备用。
1.2.3 PCR扩增利用经典的DNA条形码片段的扩增效率作为模板质量的衡量标准,扩增引物[13]如下:正向引物Lco1490:5′-GGT CAA CAA ATC ATA AAG ATA TTG G-3′;反向引物Hco2198:5′-TAA ACT TCA GGG TGA CCA AAA AAT CA-3′; 引物由宝生物工程(大连)有限公司合成。PCR的反应体系及反应条件参照文献[14],PCR产物片段长度为710 bp。
1.2.4 测序及比对将PCR产物进行电泳分析;目的条带送上海立菲生物科技有限公司进行双向测序;测序所得结果与BOLD数据库进行比对,以校对扩增序列的正确性。
1.2.5 样本玻片标本的制作将提取完DNA的甲螨样本用100 ℃ 10%的NaOH溶液煮5~10 min,用双蒸水清洗3次,以除掉甲螨体表的杂质,将甲螨放在凹形载玻片上,滴上封片胶,用昆虫针调整甲螨肢体的姿势,使其接近自然,轻轻盖上盖玻片,均匀按压,赶出气泡,晾干后显微镜下观察并拍照。
另外,未用几丁质酶处理过的甲螨样本同样利用上述方法,制成玻片标本,用以对比处理前后样本形态的变化。
2 结果 2.1 未经几丁质酶消化的样本未经几丁质酶消化的甲螨样本,按1.2.2中的方法直接用裂解液裂解提取模板DNA,经PCR扩增及琼脂糖凝胶电泳分析,未扩增出目的条带,见图 1。

|
| 注: M. 标准分子质量; +. 阳性对照; -. 阴性对照(以水做模板); 1~6. 甲螨样本。 图 1 未经几丁质酶消化的甲螨样本PCR电泳结果 Figure 1 The PCR results of oribatid mites untreated with chitinase |
| |
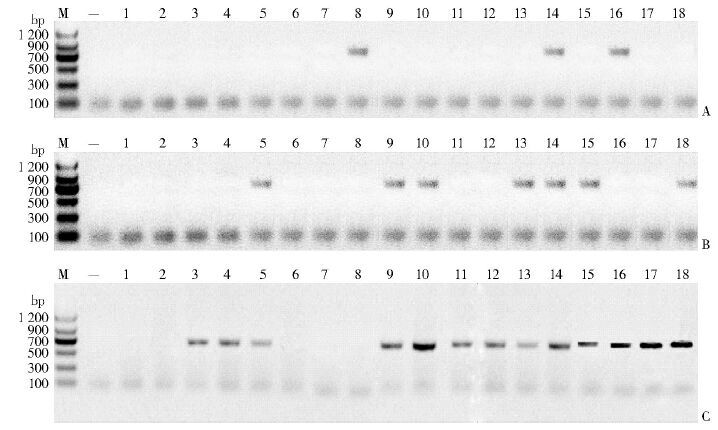
用0.01 mg/ml的酶溶液将甲螨样本分别消化4、8、24 h,每个处理时间6个重复,提取DNA模板,经PCR扩增、琼脂糖凝胶电泳后,消化4 h的甲螨样本未扩增出目的条带,消化8 h的只有1个样本扩增出目的条带,消化24 h的有2个样本扩增出目的条带,见图 2A。用0.10 mg/ml的酶溶液将甲螨样本分别消化4、8、24 h,每个处理时间进行6个重复,提取DNA模板,经PCR扩增、琼脂糖凝胶电泳,消化4、8、24 h 的甲螨样本分别有1、2、4个样本扩增出目的条带,见图 2B。用1.00 mg/ml的酶溶液将甲螨样本分别消化4、8、24 h,每个处理时间进行6个重复,提取DNA模板,经PCR扩增、琼脂糖凝胶电泳,消化4、8、24 h 的甲螨样本分别有3、4、6个样本扩增出目的条带,见图 2C。
|
| 注: M. 标准分子质量; -. 阴性对照(以水做模板); 1~6、7~12、13~18. 分别为消化4、8、24 h的甲螨样本。 图 2 用0.01、0.10、1.00 mg/ml几丁质酶消化不同时间的甲螨样本PCR电泳结果 Figure 2 The PCR results of oribatid mites digested with 0.01,0.10,1.00 mg/ml chitinase for different time |
| |
不同浓度几丁质酶,分别处理0、4、8、24 h,DNA条形码获取结果见表 1。

|
鉴于1.00 mg/ml几丁质酶消化24 h,所有的样本均能扩增出目的片段,为了节约试剂和时间成本,不再增加酶的浓度和处理时间。
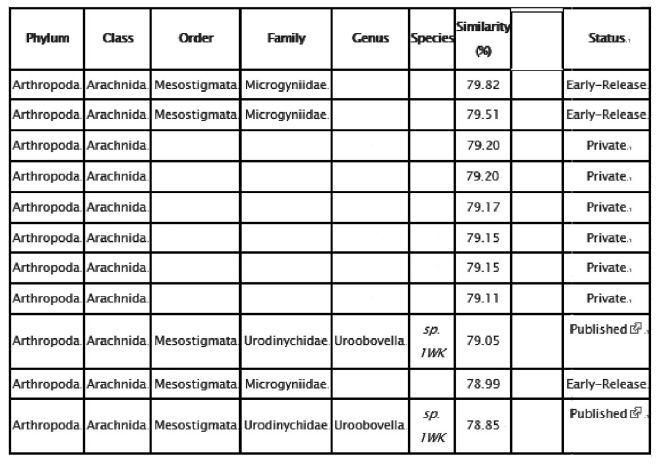
2.3 DNA条形码的序列分析为验证所获得的DNA条形码序列为目的种类,将扩增的PCR产物进行测序,并在BOLD(http://www.barcodinglife.org/)网站上进行比对,比对结果显示所获得的DNA条形码序列与中气门亚目(Mesostigmata)小雌螨科(Microgyniidae)同源性最高,与已知序列的同源性为79.82%(图 3)。由于数据库中缺少目的种类的DNA条形码数据,造成同源性不高,不能达到种类鉴定的目的,但该结果证明了本研究所获得的DNA条形码为正确序列,不是由于污染造成的假阳性扩增。
|
| 图 3 BOLD系统中的比对结果 Figure 3 Sequences alignment results in BOLD |
| |
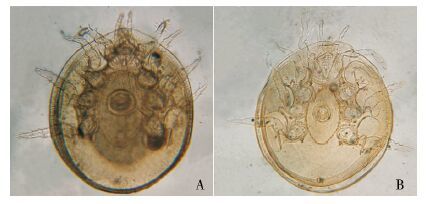
将未经几丁质酶处理的样本和处理后的样本制成玻片标本进行形态对比,结果显示,未经处理和处理后的样本形态无明显变化,均可显示主要的分类特征,可作为凭证标本保存,便于日后的查证,见图 4。
|
| 图 4 甲螨样本处理前(A)、后(B)的形态(×10) Figure 4 Morphology comparison of the oribatid before and after the digestion (×10) |
| |
DNA条形码技术作为一种分类鉴定的手段,越来越被重视,对于微小节肢动物而言,其难点在于高质量DNA模板的获得以及凭证标本的保存。以往获得微小节肢动物DNA模板,主要是通过研磨个体,结果造成标本的破坏,而不能制成凭证标本保存作为日后查阅的凭证。近几年来,为保证DNA条形码的凭证标本不被破坏,一些学者致力于不损害个体形态特征的DNA模板获取方法的研究,如高艳和卜云[7]研究出一种针对微小节肢动物无损形态的DNA提取方法,但这些方法不适用于具有厚几丁质外壳的微小节肢动物。本研究利用几丁质酶的消化作用,先将微小节肢动物的几丁质外壳消化,增加其通透性,然后无需研磨直接用裂解液裂解,获得高质量的DNA模板,且能保证个体的形态完整性,解决了具厚几丁质外壳微小节肢动物DNA模板提取过程中需要对个体进行研磨,导致形态被破坏,凭证标本不能完整保存的难题,同时也保证了DNA模板的单一性,提高了DNA条形码序列分析的准确性。
| [1] | 刘勇, 宋毓, 李晓宇. 基于线粒体COⅠ基因的DNA条形码技术在昆虫分子鉴定中的应用[J]. 植物检疫, 2010, 24(2): 46–50. |
| [2] | Stoeckle M. Taxonomy,DNA,and the bar code of life[J]. BioScience, 2003, 53(9) : 796–797 .DOI:10.1641/0006-3568(2003)053[0796:TDATBC]2.0.CO;2. |
| [3] | Hebert PDN, Cywinska A, Ball SL, et al. Biological identifications through DNA barcodes[J]. Proc Roy Soc B, 2003, 270(1512) : 313–321 .DOI:10.1098/rspb.2002.2218. |
| [4] | 陈琪, 姜玉新, 郭伟, 等. 3种常用封固剂制作螨标本的效果比较[J]. 中国媒介生物学及控制杂志, 2013, 24(5): 409–411. |
| [5] | 胡佳, 岳巧云, 邱德义, 等. 医学媒介生物微量组织直接扩增DNA条形码序列方法的研究[J]. 中国媒介生物学及控制杂志, 2014, 25(4): 297–300. |
| [6] | 温硕洋, 何晓芳. 一种适用于昆虫痕量DNA模板制备的方法[J]. 昆虫知识, 2003, 40(3): 276–279. |
| [7] | 高艳, 卜云. 一种改进的小型节肢动物无形态损伤的DNA提取方法[J]. 四川动物, 2014, 33(2): 216–220. |
| [8] | 张志忠, 吴菁华, 吕柳新, 等. 植物几丁质酶及其应用研究进展[J]. 福建农林大学学报:自然科学版, 2005, 34(4): 494–499. |
| [9] | 李瑶, 范晓军. 昆虫几丁质酶及其在害虫防治中的应用[J]. 应用昆虫学, 2011, 48(5): 1489–1494. |
| [10] | 魏巍, 贺淹才, 刘爱花. 几丁质酶酶活性测定方法的研究进展[J]. 工业催化, 2006, 14(增刊): 11–14. |
| [11] | 殷秀琴, 马祝阳. Tullgren法对土壤动物的分离效率[J]. 东北师大学报:自然科学版, 2002, 34(2): 84–91. |
| [12] | 谢晓兰, 曾炜, 高平章, 等. 黄粉虫几丁质酶的纯化及性质[J]. 泉州师范学院学报, 2012, 30(4): 56–61. |
| [13] | Mora C, Tittensor DP, Adl S, et al. How many species are there on earth and in the ocean[J]. PLoS Biol, 2011, 9(8) : e1001127.DOI:10.1371/journal.pbio.1001127. |
| [14] | 魏晓雅, 邱德义, 岳巧云, 等. 应用DNA条形码技术鉴定口岸截获的国内未见分布蚊种[J]. 检验检疫学刊, 2014, 24(6): 46–49. |
 2017, Vol. 28
2017, Vol. 28


