扩展功能
文章信息
- 黄鹏, 杨章女, 刘源, 姚苹苹, 胡建利, 王笑辰, 俞建家, 李军, 韩亚萍, 金柯, 杨龙, 张云, 岳明
- HUANG Peng, YANG Zhang-nyu, LIU Yuan, YAO Ping-ping, HU Jian-li, WANG Xiao-chen, YU Jian-jia, LI Jun, HAN Ya-ping, JIN Ke, YANG Long, ZHANG Yun, YUE Ming
- 东南沿海地区1980-2015年汉坦病毒分子特征及流行病学分析
- The molecular characteristics and epidemiological analysis of Hantavirus in southeast coastal area of China from 1980 to 2015
- 中国媒介生物学及控制杂志, 2017, 28(4): 354-358
- Chin J Vector Biol & Control, 2017, 28(4): 354-358
- 10.11853/j.issn.1003.8280.2017.04.012
-
文章历史
- 收稿日期: 2017-04-06
- 网络出版时间: 2017-06-12 16:52
2 浙江省疾病预防控制中心, 浙江省传染病疫苗与预防控制研究重点实验室, 杭州 310051;
3 南京医科大学第一附属医院, 南京 210029;
4 江苏省疾病预防控制中心, 南京 210009;
5 江苏省丹阳市人民医院, 江苏 丹阳 212300;
6 中国人民解放军南京军区军事医学研究所, 江苏 南京 210002
2 Zhejiang Center for Disease Control and Prevention;
3 The First Affiliated Hospital of Nanjing Medical University;
4 Jiangsu Center for Disease Control and Prevention;
5 Danyang People's Hospital;
6 Huadong Research Institute for Medicine and Biotechnics
汉坦病毒(Hantavirus,HV)与宿主动物之间是一种共进化共形成的特殊关系,病毒进化受到基因变异的压力,也受到从自然宿主到终宿主随机选择及自然环境的影响,因此出现了病毒抗原及基因的多样性,导致新疫区不断出现和疾病流行[1-3]。了解HV在自然界遗传进化过程,无论对该病毒的预防控制以及新发现的RNA病毒对分子遗传特性等病毒学理论研究均有极其重要的意义[4-7]。本研究结合肾综合征出血热(hemorrhagic fever with renal syndrome,HFRS)流行规律,对1980-2015年我国东南沿海地区不同地域、时间、宿主、媒介和患者中分离的HV部分M基因序列进行了分析,以了解其分子特征。
1 材料与方法 1.1 材料来源采用夹夜法捕鼠,将捕获鼠类进行分类鉴定、登记后取鼠肺,分装于冻存管置液氮罐中保存备用;利用直接荧光法筛选阳性HV标本。对于疑似HFRS患者血清,利用试剂盒检测IgM抗体和抗原片检测IgG抗体。
1.2 HV序列的测定HV阳性标本部分M、S片段基因的扩增及RNA提取、cDNA制备过程见文献[8]。
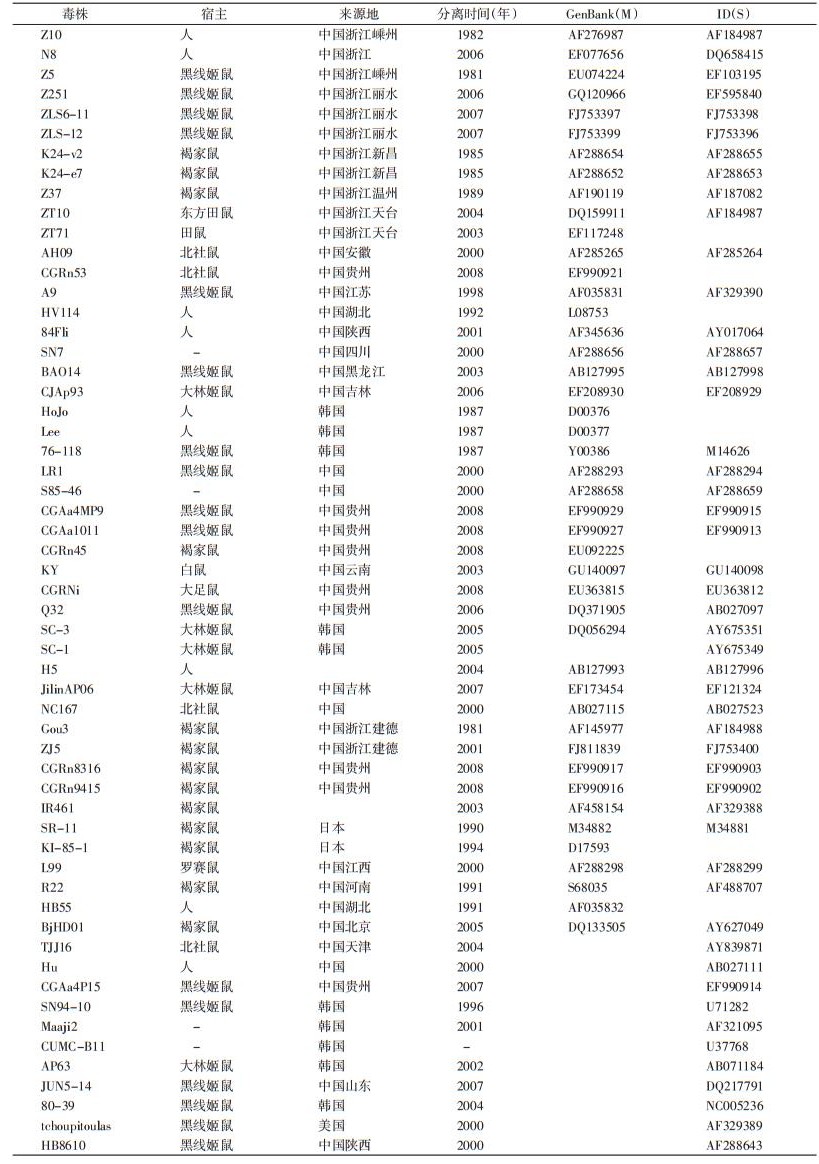
1.3 系统发育树构建委托生工生物工程(上海)股份有限公司完成核苷酸序列测定,得到HV部分M、S基因序列,结合GenBank数据库,获取汉滩型病毒(HTNV)和汉城型病毒(SEOV)的部分M基因序列以及部分S基因序列,应用Mega 4.0和DNAStar软件进行系统发育分析[9],以邻位相连法(NJ)构建系统发生树。此外,本研究使用的毒株信息见表 1。除了浙江省分离的序列是自己得到的,其余序列来自GenBank数据库,所有使用的序列均有GenBank号。
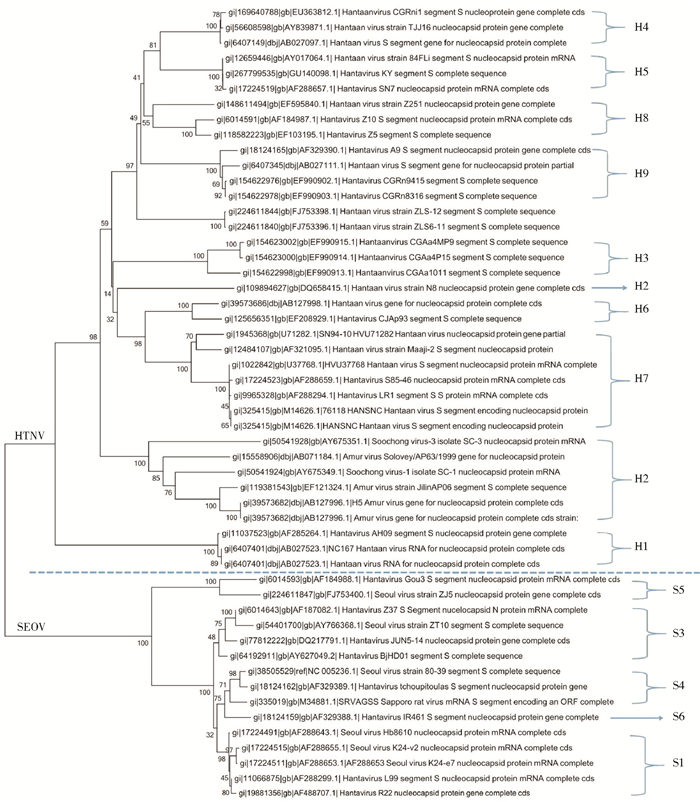
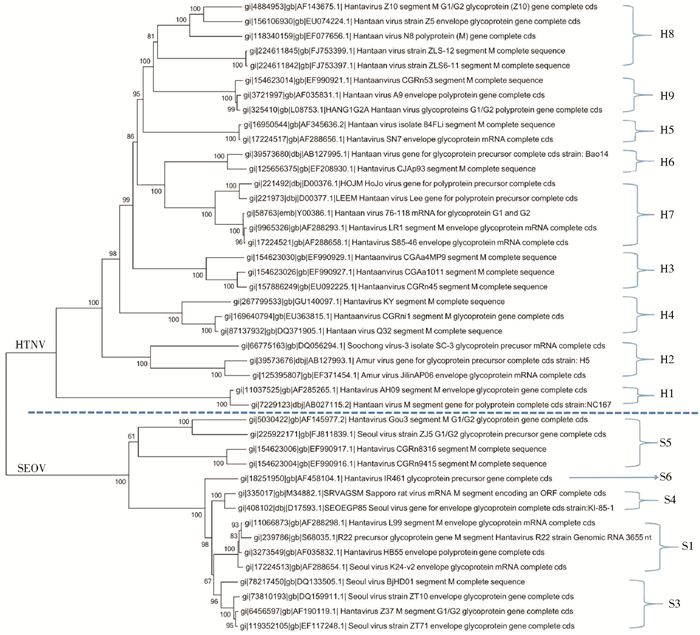
临床确诊的HFRS患者血清共657份,其中阳性标本547份,仅获得其中Z10和N8这2株的HV S、M片段序列,从GenBank中获得其他6株人源M片段序列,以及3株人源S片段序列,将这些序列与现有的鼠源病毒基因一起进行同源性分析,结果显示,无论是利用M还是S基因构建的进化树,HV主要分为HTN、SEO,其中HTN又可分为H1~H9分支,SEO有S1~S6分支,HFRS临床患者感染的病毒有HTN和SEO,其中Z10、N8均为HTN(图 1、2),无论是在M还是S构建的进化分析树上,Z10和N8均为HTN中的H8型。而其余的人源序列,84FLi在进化分析上显示为HTN中的H5型,HV114则为H9型,而韩国的2株Lee和JoHo则为H7;在S片段构建的进化树上,H5为H2型,HB55为S1型。
|
| 图 1 基于S片段构建的HV系统进化树 Figure 1 Phylogenetic tree based on S segment sequences of Hantavirus |
| |
|
| 图 2 基于M片段构建的HV系统进化树 Figure 2 Phylogenetic tree based on M segment sequences of Hantavirus |
| |
从2 025份鼠源性样本中筛选出阳性样本131份,阳性率为6.5%。对这些样本进行病毒cDNA合成以及PCR扩增,获得206个序列的鼠源样本测序结果,其中M片段119个,S片段87个。选取11株的M、S片段部分序列与公共基因数据库(GenBank)进行病毒基因进化分析(图 1、2)。SEOV S片段可各分为6个进化分支,东南沿海地区主要集中于1、3和5分支上。HTN HV基因有9个进化分支,但序列间的变化更加多样。HTN Z5、Z251属于分支H8;基于M片段的进化分析显示,ZLS6-11、ZLS-12与Z5属于同一分支,而基于S片段的系统发育则截然不同。SEO K24-e7、K24-v2(浙江省丽水)与R22(江西省)、L99(河南省)位于同一分支,即分支S1,ZT71、ZT10与Z37(浙江省温州台州地区)为同一分支,共属于分支S3;Gou3和ZJ5(浙江省建德)与其他SEOV株的亲缘关系较远,属于SEOV的独立分支S5。表明宿主与地区的进化分支有关,即具有宿主特异性和地区聚集性。SEOV主要来源于褐家鼠(Rattus norvegicus),HTNV主要来源于黑线姬鼠(Apodemus agrarius)。
3 讨论进化分析显示,东南沿海地区为HTN和SEO HV混合型疫区,病毒亲缘性主要表现为地域性和宿主差异,呈现高度的地理聚集现象。流行病学调查表明,近年HFRS疫区发病率有上升趋势,秋冬季为发病高峰,春季为小高峰,患者以男性青壮年农民为主,提示HFRS发病与患者参与农业生产劳动和传染源接触有关,疫区范围趋于稳定;浙江、江苏省疫情主要分布于河湖众多的平原及山坡丘陵地区,地势低、潮度高、杂草多,疫区可分为姬鼠型、家鼠型和混合型。福建省HFRS疫源地主要为家鼠型和混合型,后者主要分布于闽东北、闽西北地区;家鼠型疫源地分布于沿海丘陵地区及闽西、闽中的山地。疫区的地理景观分析表明,HFRS自然疫源地和疫区主要分布在以平原和丘陵地形为主的亚热带湿润地区,黑线姬鼠及褐家鼠是HV的主要宿主动物和传染源,是此类地理景观中的优势种,其分布广、数量多、带病毒率高,可相互感染,有助于宿主更替,对HFRS疫源地的维持具有重要作用,与本监测结果相符。
| [1] |
Xiong HR, Li Q, Chen W, et al. Specific humoral reaction of hemorrhagic fever with renal syndrome (HFRS) patients in China to recombinant nucleocapsid proteins from European hantaviruses[J]. Eur J Clin Microbiol Infect Dis, 2011, 30(5): 645-651. DOI:10.1007/s10096-010-1134-5 |
| [2] |
Schmaljohn C, Hjelle B. Hantaviruses:a global disease problem[J]. Emerg Infect Dis, 1997, 3(2): 95-104. DOI:10.3201/eid0302.970202 |
| [3] |
Klein SL, Calisher CH. Emergence and persistence of hantaviruses[M]//Childs JE, Mackenzie JS, Richt JA. Wildlife and Emerging Zoonotic Diseases:The Biology, Circumstances and Consequences of Cross-Species Transmission. Berlin Heidelberg:Springer, 2007:217-252.
|
| [4] |
Sch-nrich G, Rang A, Lütteke N, et al. Hantavirus-induced immunity in rodent reservoirs and humans[J]. Immunol Rev, 2008, 225(1): 163-189. DOI:10.1111/imr.2008.225.issue-1 |
| [5] |
Laine O, Joutsi-Korhonen L, M-kel-S, et al. Polymorphisms of PAI-1 and platelet GP Ia may associate with impairment of renal function and thrombocytopenia in Puumala hantavirus infection[J]. Thromb Res, 2012, 129(5): 611-615. |
| [6] |
Guo WP, Lin XD, Wang W, et al. Phylogeny and origins of hantaviruses harbored by bats, insectivores, and rodents[J]. PLoS Pathog, 2013, 9(2): e1003159. DOI:10.1371/journal.ppat.1003159 |
| [7] |
李婵, 姚苹苹, 朱函坪, 等. 东南沿海地区汉坦病毒的遗传进化分析[J]. 中国媒介生物学及控制杂志, 2015, 26(3): 275-278. DOI:10.11853/j.issn.1003.4692.2015.03.014 |
| [8] |
徐芳, 朱函坪, 姚苹苹, 等. 浙江省2008-2011年啮齿动物中汉坦病毒的分离及鉴定[J]. 中国媒介生物学及控制杂志, 2013, 24(4): 285-288. |
| [9] |
Tamura K, Stecher G, Peterson D, et al. Mega 6:molecular evolutionary genetics analysis Version 6.0[J]. Mol Biol Evol, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197 |
 2017, Vol. 28
2017, Vol. 28