扩展功能
文章信息
- 罗雪莲, 郑雅匀, 戴晓懿, 金东, 孙晖, 熊衍文
- LUO Xue-lian, ZHENG Ya-yun, DAI Xiao-yi, JIN Dong, SUN Hui, XIONG Yan-wen
- 青藏高原喜马拉雅旱獭粪便携带腹泻相关病毒的调查分析
- Investigation and sequence analysis of diarrhea related viruses in the feces of Marmota himalayana on the Qinghai-Tibet Plateau, China
- 中国媒介生物学及控制杂志, 2016, 27(4): 333-335
- Chin J Vector Biol & Control, 2016, 27(4): 333-335
- 10.11853/j.issn.1003.8280.2016.04.005
-
文章历史
- 收稿日期: 2016-03-02
- 网络出版时间: 2016-06-03 12:09
2 感染性疾病诊治协同创新中心, 浙江 杭州 310003
2 Collaborative Innovation Center for Diagnosis and Treatment of Infectious Diseases
随着严重急性呼吸综合征(SARS)、埃博拉出血热等新发传染病的不断出现,野生动物被认为是“病毒库”[1-2]。因此,掌握野生动物携带病毒种类,是评估野生动物来源人兽共患病潜在风险的重要基础,为预测和预警可能出现的传染病提供依据。喜马拉雅旱獭(Marmota himalayana)属啮齿目(Rodentia)松鼠科(Sciuridae)旱獭属(Marmota),是青藏高原特有种,主要以草为食物,偶食小型啮齿动物。随着人类活动范围扩大和生态环境变化,人与喜马拉雅旱獭的接触越发频繁。研究表明喜马拉雅旱獭是鼠疫的主要储存宿主,为青藏高原区域鼠疫预防的重点监控对象。然而,喜马拉雅旱獭是否携带与人类致病相关病毒,尚不清楚。本研究以喜马拉雅旱獭粪便为研究对象,展开腹泻相关病毒的调查。对喜马拉雅旱獭粪便中腹泻相关病毒进行序列分析,为全面了解喜马拉雅旱獭携带病毒谱系奠定基础。
1 材料与方法 1.1 标本来源于青海省玉树藏族自治州采集旱獭粪便96份,-80℃冰箱保存。
1.2 方法 1.2.1 标本处理将0.2 g或200μl粪便标本用PBS缓冲液制成10%粪便悬液,振荡3次,静置10 min,4 700×g离心5 min,取上清,-20℃待检。
1.2.2 核酸提取取140μl粪便悬液,参照Qigen公司QIAamp Viral RNA Mini Kit(Cat No. 52904)操作说明书进行。所得RNA用60μl无菌水溶解,置-80℃保存。
1.2.3 腹泻相关病毒的检测根据文献[3-4],确定旱獭粪便标本中待检测腹泻相关病毒为诺如病毒(Norovirus)、札如病毒(Sapovirus)、星状病毒(Astrovirus)、腺病毒(Adeovirus)、小双节RNA病毒(Picobirnavirus)以及嵴病毒(Kobuvirus)。根据各病毒的保守区设计兼并引物,见表 1。腺病毒检测以提取核酸为模板,PCR反应条件如下:94℃预变性5 min,35个循环(94℃变性30 s,55℃退火30 s,72℃延伸1 min),72℃终延伸7 min,4℃终止反应。其他病毒检测采用一步法RT-PCR。按TaKaRa公司PrimeScript one step RT?PCR Kit(Cat No. RR055A)的说明书进行。取PCR产物进行琼脂糖凝胶电泳鉴定,目的条带送测序公司(北京天一辉远生物科技有限公司)测序确认。
已获得的病毒核苷酸序列剔除两端引物序列后,在NCBI数据库利用Blast进行序列比对。以NCBI数据库已发布的相关病毒序列作为参考序列,利用BioEdit软件编辑核苷酸序列,利用PhyML 3.0软件构建系统进化树。
2 结果 2.1 喜马拉雅旱獭粪便中携带的腹泻相关病毒PCR或者一步法RT-PCR检测96份喜马拉雅旱獭粪便标本,检出星状病毒、小双节病毒和嵴病毒。其中星状病毒阳性34份,阳性率为35.4%,小双节病毒阳性42份,阳性率为43.8%,嵴病毒阳性5份,阳性率为5.2%,未检测出腺病毒、诺如病毒和札如病毒。
2.2 核苷酸序列同源性比对随机抽取星状病毒、小双节RNA病毒和嵴病毒阳性PCR产物各5份,进行序列测定,经Blast比对,喜马拉雅旱獭粪便中星状病毒与鼠星状病毒(GenBank:KC999691)核苷酸序列一致性为78%,氨基酸一致性为77%;小双节RNA病毒与猪小双节RNA病毒(GenBank:KC841459)的核苷酸一致性为77%,氨基酸一致性为84%;嵴病毒与鼠嵴病毒(GenBank:JF755427)的核苷酸序列一致性为93%,氨基酸序列一致性为91%。
2.3 遗传进化树分析 2.3.1 喜马拉雅旱獭中星状病毒进化树分析选择NCBI数据库中星状病毒科参考序列,与所测得喜马拉雅旱獭星状病毒核苷酸序列比对,利用PhyML 3.0软件构建系统进化树。喜马拉雅旱獭星状病毒均处于哺乳动物星状病毒簇内,且与鼠星状病毒JX544743、野猪星状病毒NC016896的亲缘关系较近,见图 1。
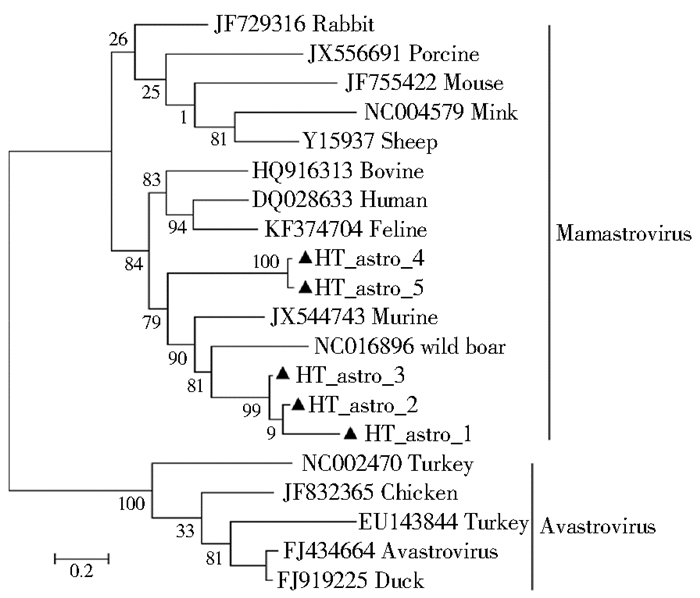
|
| 注:▲喜马拉雅旱獭中星状病毒序列。 图 1 基于418 bp喜马拉雅旱獭星状病毒RdRp基因构建的系统进化树 Figure 1 Phylogenetic tree based on 418 bp of RdRp genes of astroviruses in M. himalayana |
| |
选择参考序列为小双节RNA病毒科小双节RNA病毒属的病毒株。根据RdRp基因序列不同,将小双节RNA病毒分为GenogroupⅠ和GenogroupⅡ两大类。GenogroupⅠ中的代表株1-CHN-97为外群,而喜马拉雅旱獭粪便中的小双节RNA病毒属于GenogroupⅡ。小双节RNA病毒片段处于2个不同的分支,见图 2,说明小双节RNA病毒片段的序列变异较大,喜马拉雅旱獭粪便中小双节RNA病毒存在序列多样性。
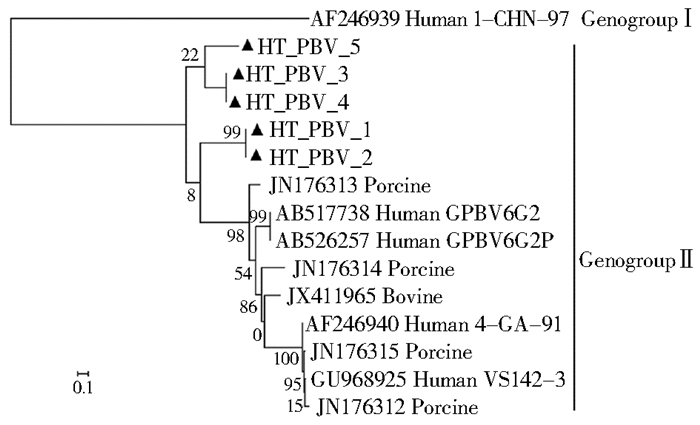
|
| 注:▲喜马拉雅旱獭小双节RNA病毒序列。 图 2 基于343 bp喜马拉雅旱獭小双节RNA病毒RdRp基因构建的系统进化树 Figure 2 Phylogenetic tree based on 343 bp of RdRP gene of picobirnaviruses in M. himalayana |
| |
嵴病毒属于小RNA病毒科嵴病毒属成员,喜马拉雅旱獭嵴病毒序列相同;喜马拉雅旱獭粪便的嵴病毒与鼠源嵴病毒亲缘关系较近,见图 3。
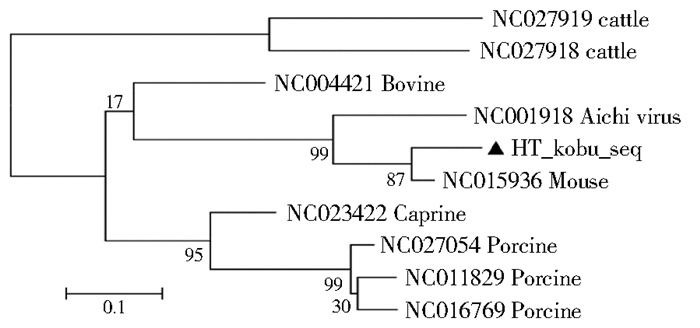
|
| 注:▲喜马拉雅旱獭嵴病毒序列。 图 3 基于418 bp喜马拉雅旱獭嵴病毒RdRp基因构建的系统进化树 Figure 3 Phylogenetic tree based on 418 bp of RdRp gene of kobuviruses in M. himalayana |
| |
随着人类活动范围的扩大及生态环境的变化,人类与野生动物的接触越发频繁,感染野生动物携带病原的风险大大增加。研究表明野生动物是许多烈性病毒的宿主[5-7],如蝙蝠是尼帕病毒(Nipah virus)、亨德拉病毒(Hendra virus)、冠状病毒(Coronavirus)的天然宿主[8];骆驼是中东呼吸系统综合征冠状病毒(MERS-CoV)的自然宿主[9]。因此,研究野生动物自身携带的病毒有利于野生动物源性疾病的监测和预警。
喜马拉雅旱獭是青藏高原特有种,是鼠疫耶尔森菌的自然宿主[10]。本研究以喜马拉雅旱獭为研究对象,利用兼并引物,调查喜马拉雅旱獭粪便中6种常见的腹泻相关病毒。结果表明,喜马拉雅旱獭粪便中存在星状病毒、小双节RNA病毒以及嵴病毒,以小双节RNA病毒阳性率最高。兼并引物未检测到腺病毒、札如病毒和诺如病毒,可能由于其基因组在保守区也存在较大差异,无法设计兼并,通过利用兼并引物检测病毒的传统方法可能不适用于对野生动物携带的未知病毒的筛查。据报道利用454高通量测序对蝙蝠粪便携带病毒进行研究,取得一定进展[11-12]。下一步,本课题组将利用高通量技术对喜马拉雅旱獭携带病毒进行全面研究。
序列分析表明,喜马拉雅旱獭粪便中星状病毒和小双节RNA病毒的序列存在变异,而嵴病毒序列非常保守。将这些序列与其参考序列分别进行进化分析,结果表明,喜马拉雅旱獭粪便中的星状病毒和嵴病毒与相对应的鼠源病毒在进化关系上最亲近,而小双节RNA病毒与猪源小双节RNA病毒在进化关系上最亲近,提示小双节RNA病毒可能出现跨种重组。由于所获得序列较短,无法全面地反映病毒的进化关系。因此,应进一步获得病毒全基因组序列。
本研究以青藏高原特有种——喜马拉雅旱獭为研究对象,首次调查其粪便携带腹泻相关病毒,为了解喜马拉雅旱獭携带病毒情况奠定了基础。
志谢: 样品采集过程中中国疾病预防控制中心传染病预防控制所疾控应急处卢珊处长及青海省玉树藏族自治州疾病预防控制中心人员给予帮助,特此志谢| [1] | Morse SS, Mazet JAK, Woolhouse M, et al. Prediction and prevention of the next pandemic zoonosis[J]. Lancet, 2012, 380 (9857) : 1956–1965 .DOI:10.1016/S0140-6736(12)61684-5. |
| [2] | 李永成, 孙贺廷, 刘枫. 野生动物在人兽共患病发生、传播中的作用[J]. 林业科学,2006,42 (5) :120–126. |
| [3] | Chen X, Zhang B, Yue H, et al. A novel astrovirus species in the gut of yaks with diarrhea in the Qinghai-Tibetan plateau, 2013[J]. J Gen Virol, 2015, 96 (12) : 3672–3680 .DOI:10.1099/jgv.0.000303. |
| [4] | 刘晓峰, 张扬, 张兰荣, 等. 北京市肠道门诊腹泻患者病原谱分析[J]. 中华传染病杂志,2015,33 (8) :460–464. |
| [5] | World Health Organization. Outbreak news. Ebola. democratic republic of Congo-update[J]. Wkly Epidemiol Rec, 2012, 87 (36) : 357. |
| [6] | Zaki AM, van Boheemen S, Bestebroer TM, et al. Isolation of a novel coronavirus from a man with pneumonia in Saudi Arabia[J]. N Engl J Med, 2012, 367 (19) : 1814–1820 .DOI:10.1056/NEJMoa1211721. |
| [7] | Mahalingam S, Herrero LJ, Playford EG, et al. Hendra virus: an emerging paramyxovirus in Australia[J]. Lancet Infect Dis, 2012, 12 (10) : 799–807 .DOI:10.1016/S1473-3099(12)70158-5. |
| [8] | Kohl C, Kurth A. European bats as carriers of viruses with zoonotic potential[J]. Viruses, 2014, 6 (8) : 3110–3128 .DOI:10.3390/v6083110. |
| [9] | Perera RA, Wang P, Gomaa MR, et al. Seroepidemiology for MERS coronavirus using microneutralisation and pseudoparticle virus neutralisation assays reveal a high prevalence of antibody in dromedary camels in Egypt, June 2013[J]. Euro Surveill, 2013, 18 (36) : 20574.DOI:10.2807/1560-7917.ES2013.18.36.20574. |
| [10] | 王成祥, 梁坤, 杨英中, 等. 2014年乌鲁木齐县鼠疫监测结果分析[J]. 疾病预防控制通报,2015,30 (3) :56–57. |
| [11] | Wu ZQ, Ren XW, Yang L, et al. Virome analysis for identification of novel mammalian viruses in bat species from Chinese provinces[J]. J Virol, 2012, 86 (20) : 10999–11012 .DOI:10.1128/JVI.01394-12. |
| [12] | Ge XY, Li Y, Yang XL, et al. Metagenomic analysis of viruses from bat fecal samples reveals many novel viruses in insectivorous bats in China[J]. J Virol, 2012, 86 (8) : 4620–4630 .DOI:10.1128/JVI.06671-11. |
 2016, Vol. 27
2016, Vol. 27



