扩展功能
文章信息
- 胡群, 邹春颖, 马思杰
- HU Qun, ZOU Chun-ying, MA Si-jie
- 沙粒病毒属四步法一致-简并杂合寡核苷酸引物实时荧光PCR检测体系的建立
- Development of CODEHOP 4-step program Real-time PCR to detect Arenaviruses in rodents
- 中国媒介生物学及控制杂志, 2016, 27(3): 248-252
- Chin J Vector Biol & Control, 2016, 27(3): 248-252
- 10.11853/j.issn.1003.8280.2016.03.009
-
文章历史
- 收稿日期: 2015-12-27
- 网络出版时间: 2016-04-14 12:00
沙粒病毒(Arenavirus)属于沙粒病毒科(Arenaviridae)沙粒病毒属(Arenavirus),为有包膜的单股负链RNA病毒,基因组由L和S两个节段组成,已知至少由23种病毒组成,新的沙粒病毒在不断的发现中[1-2]。主要引起的人类疾病有淋巴细胞性脉络丛脑膜炎(lymphocytic choriomeningitis,LCM)、拉沙热(Lassa hemorrhagic fever,LHF)、阿根廷出血热(Argentine hemorrhagic fever,AHF)和玻利维亚出血热(Bolivian hemorrhagic fever,BHF)等。本研究设计了一对沙粒病毒属的一致-简并引物,并将该引物与SYBR GreenⅠ实时荧光检测体系相结合,建立了沙粒病毒属一致-简并杂合寡核苷酸引物(consensus?degenerate hybrid oligonucleotide primer,CODEHOP)实时荧光PCR(Real-time PCR)检测体系,通过增加一个荧光采集温度步骤的方式,消除了简并引物较易产生非特异性扩增产物对检测结果的影响,方法操作简便、重复性好、灵敏度高,可实时对鼠类样品中可能携带的各种沙粒病毒进行排查。
1 材料与方法 1.1 主要试剂组织RNA提取试剂盒(QIAGEN RNeasy Mini Kit)购自德国Qiagen公司,一步法SYBR Real-time PCR试剂盒(One Step SYBR PrimeScriptTM Real-time PCR Kit Ⅱ)购自宝生物工程(大连)有限公司。
1.2 样品本实验室从宁波市捕获的鼠样品中分离的鼠肺样品,其中1份沙粒病毒属淋巴脉络丛脑膜炎病毒(LCMV)阳性样品,标识为MS1201作为阳性对照。狂犬病毒RNA、汉滩病毒RNA、汉城病毒RNA、基孔肯雅热病毒RNA、流行性乙型脑炎病毒RNA、柯萨奇病毒RNA、甲型H1N1流感病毒RNA由宁波市疾病预防控制中心提供。
1.3 病毒RNA提取参照Qiagen公司组织RNA提取试剂盒使用说明书方法提取病毒总RNA,用50 μl超纯水溶解,置-80 ℃保存备用。
1.4 引物设计下载GenBank中公布的22种沙粒病毒属病毒的N蛋白氨基酸序列,将氨基酸序列以FASTA格式保存,进行Block比对(http://blocks.fhcrc.org/blocks/codehop.html.),得到高度保守的连续氨基酸区域。使用CODEHOP引物搜索,根据简并度、得分、片段大小等综合因素筛选出1对合适的上下游引物。
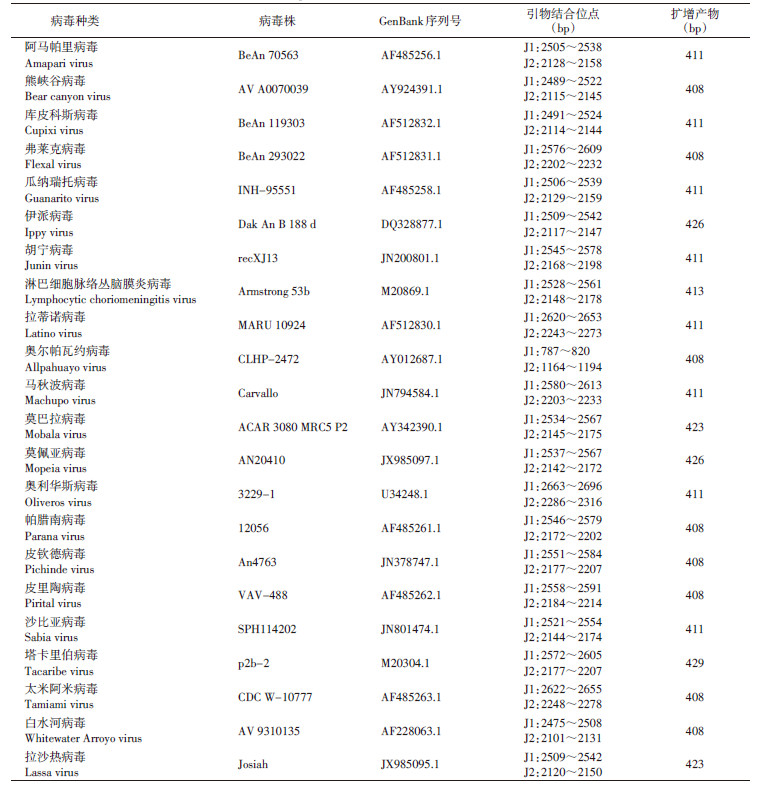
1.5 沙粒病毒引物结合位点和扩增序列片段大小分析从GenBank中下载22种沙粒病毒代表株的N蛋白基因组序列,用Mega 5.0软件分析不同病毒株引物可能的结合位点,并分析可获得的扩增产物大小。
1.6 Real-time PCR体系反应体系(20 μl),RNase Free Water 5.6 μl,2×One Step SYBR Real-time PCR Buffer 10 μl,PrimeScript 1 Step Enzyme Mix 0.8 μl,20 μmol/L上游引物0.8 μl,下游引物0.8 μl,RNA模板2 μl。常规三步法实时荧光反应程序:反转录反应45 ℃ 20 min,95 ℃ 10 min。扩增反应:变性94 ℃ 20 s,复性60 ℃ 30 s,延伸72 ℃ 35 s,共40个循环。四步法扩增反应程序改为变性94 ℃ 30 s,复性60 ℃ 30 s,延伸72 ℃ 30 s,荧光信号采集84 ℃ 5 s。
1.7 常规反应程序扩增曲线和熔解曲线分析以阳性鼠肺样品MS1201 RNA和6个阴性鼠肺样品RNA,纯水作为检测模板,以常规实时荧光反应程序进行反应,而后进行熔解曲线测定:94 ℃ 15 s,60 ℃ 60 s,之后温度上升,每隔1 ℃采集1次荧光信号,至97 ℃。
1.8 荧光信号采集温度优化为了减少非特异性扩增对实时荧光检测结果的影响,在常规实时荧光反应的基础上,增加1个荧光信号采集步骤,并将采集温度分别设为80、82、84、86 ℃进行检测效果比较,选择最优温度。
1.9 检测体系评价 1.9.1 特异性检验以阳性样品MS1201 RNA、汉滩病毒RNA、汉城病毒RNA、基孔肯雅热病毒RNA、柯萨奇病毒RNA、流行性乙型脑炎病毒RNA为模板,进行Real-time PCR扩增,分析扩增曲线和熔解曲线,并将扩增产物电泳后观察结果。
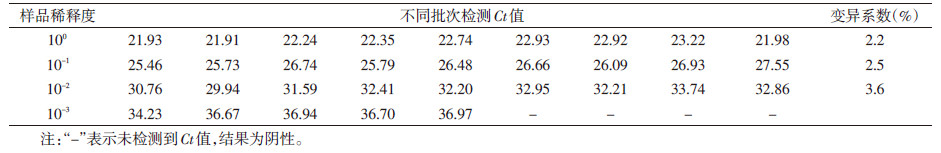
1.9.2 灵敏度和重复性评价将阳性样品MS1201 RNA,测定核酸浓度后,做10倍梯度稀释,10-1、10-2、10-3 3个梯度均平行3孔检测,连续检测3次,统计Ct值,计算变异系数,换算检测灵敏度。
1.9.3 盲样检测取实验室保存的6份沙粒病毒阳性鼠肺样品和6份阴性鼠肺样品,提取样品RNA后进行检测。
2 结 果 2.1 不同沙粒病毒引物结合位点和扩增序列片段大小分析经筛选获得1对引物J1:5′-CAT GTA TGA TTG ATG GAG GAA ATA TGY TNG ARA C-3′和 J2:5′-GAT CAT TTG GTG GTC CTT CAA TRT CNA NCC A-3′。将该引物与GenBank公布的22种沙粒病毒株进行比对,均存在引物结合位点,扩增产物大小为408~429 bp,见表 1。
|
由图 1可知,阳性样品MS1201具有明显的扩增曲线,Ct值为20.34,其余6个阴性RNA样品和水扩增曲线相对较低,Ct值分别为31.79、32.56、33.44、31.75、30.53、30.91和32.51。阳性样品MS1201 Tm峰值为86.84 ℃,阴性样品的Tm峰值为75.32~76.86 ℃。因此,沙粒病毒常规CODEHOP Real-time PCR存在较为明显的非特异性扩增,以Ct值<35作为阳性结果判定标准,6个阴性样品和水的结果均为假阳性。
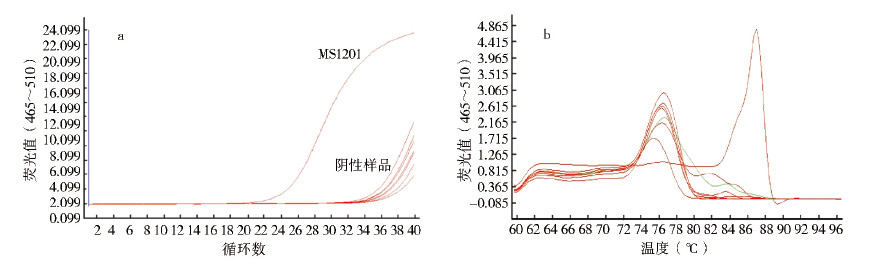
|
| 图 1 沙粒病毒常规三步法CODEHOP Real-time PCR扩增曲线(a)和熔解曲线(b) Figure 1 The amplification curves (a) and melting curves (b) of Arenaviruses conventional 3-step CODEHOP Real⁃time PCR |
| |
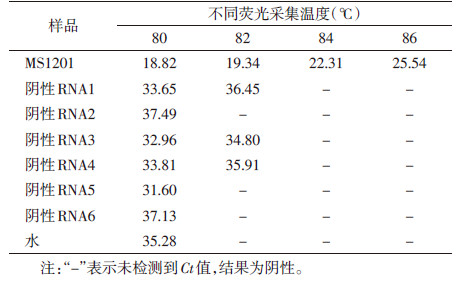
由表 2可见,将检测荧光采集温度设计在84 ℃和86 ℃时,可消除非特异性扩增对检测结果的影响,阳性样品的Ct值,随着荧光采集温度的上升而升高,考虑检测的灵敏度,沙粒病毒CODEHOP Real-time PCR检测体系的荧光采集温度以84 ℃最为合适。
|
从图 2可知,本反应仅阳性样品MS1201获得荧光扩增曲线,并且Ct值为22.95。其他作为特异性参考品的检测结果显示无荧光扩增曲线,可判断为阴性。
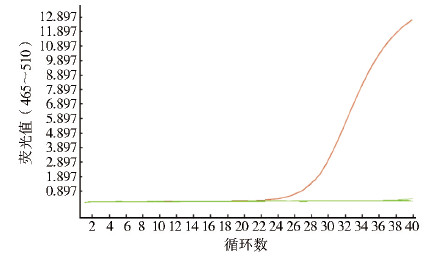
|
| 图 2 沙粒病毒四步法CODEHOP Real-time PCR 扩增曲线 Figure 2 The amplification curves of Arenaviruses 4-step CODEHOP Real-time PCR |
| |
经A值测定和换算,阳性样品RNA模板RNA浓度为24.80 ng/μl,从图 3和表 3可知,Real-time PCR方法可检测到10-3梯度稀释的样品,因此该方法的检测灵敏度为59.60 pg,3个梯度的Ct变异系数(CV)值均<5%。表明检测方法的重复性良好。
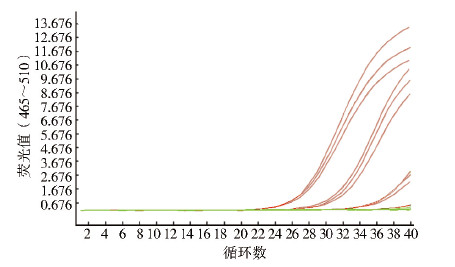
|
| 图 3 灵敏度和重复性实验结果 Figure 3 The results of sensitivity and reproducibility test |
| |
从图 4可知,6份沙粒病毒阳性鼠肺样品均获得特异性扩增曲线,6份阴性鼠肺样品均未获得扩增曲线,阳性样品和阴性样品检测结果的符合率均为100%。
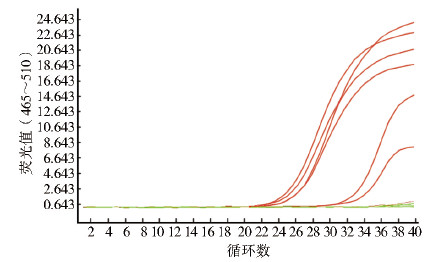
|
| 图 4 鼠肺盲样检测结果 Figure 4 The resutts of twelve blind rodent lung samples |
| |
鼠类是沙粒病毒的自然宿主,主要有LCMV,分布在欧美和亚洲,国内曾有少数病例;拉沙热病毒主要发生在西非,能引起发热、头痛、肌痛等症状,严重时有出血和休克,病死率为15%~25%;胡宁病毒、马秋波病毒、瓜纳瑞托病毒可引起人类出血性疾病,而且感染性极高[3-5]。建立沙粒病毒属的快速筛查方法并应用于鼠类样品中携带病原体的检测,有助于人感染沙粒病毒相关传染病的防控。
简并引物是根据病毒家族共有的蛋白质氨基酸的保守序列和氨基酸编码碱基的简并性原理设计的引物,从而能够实现某一病毒家族所有病毒的筛查。由于传统方法设计的简并引物很少考虑到物种的密码子偏爱性,简并度较高,同时传统简并引物的Tm值一般比较低,导致Real-time PCR 结果不理想,特异性不强,假阳性率也会增加[6-7]。Rose等[8]于1998年发明了CODEHOP设计方法,与传统方法设计的简并引物不同,利用CODEHOP方法设计的简并引物由两部分组成:3′端为根据3~4个保守氨基酸设计的核心简并区(3′-core region),长度只有9~12个碱基;5′端为非简并的一致性夹板结构(5′-consensus clamp region),是根据密码子物种偏向性设计的一致性序列,最大程度预测保守性氨基酸的编码序列,其长度取决于所需的退火温度。优点在于既减少了引物的简并度,又提高了引物的退火温度,保障PCR产物的特异性[9-11]。
沙粒病毒基因组RNA分为L和S大小两个节段,但并非等量出现在病毒粒子中。S节段通常较多,与L节段的摩尔比为2∶1,加之S节段中为核蛋白编码的N基因相对保守,因此常用于病毒的分类鉴定及进化研究。本研究以沙粒病毒共有的N蛋白氨基酸序列为依据,通过CODEHOP程序寻找保守区域,并筛选设计1对一致-简并引物。经分析,该对引物与GenBank中不同的沙粒病毒均匹配,因此认为该对引物可以用于各种沙粒病毒的检测。
实验室前期已经建立沙粒病毒属CODEHOP Real-time PCR检测体系,已证明该方法可以被有效地应用于快速筛查鼠类样品中携带各种沙粒病毒[12]。普通PCR方法由于其扩增产物需通过琼脂糖凝胶电泳后观察结果,检测操作步骤多,容易交叉污染,为了弥补该缺陷,本研究在沙粒病毒属CODEHOP 普通Real-time PCR检测体系中加入SYBR GreenⅠ荧光染料,构建Real-time PCR检测体系,该方法不仅保留了简并引物可对多种病毒进行筛查的特点,同时结合Real-time PCR在实时性、简便性和防污染性等方面的优势。
通过对三步法的常规Real-time PCR扩增曲线和熔解曲线,可以了解到沙粒病毒CODEHOP引物检测体系中存在着非特异性扩增。由于简并引物在PCR过程中较易形成引物二聚体,而引物二聚体会与SYBR GreenⅠ荧光染料相结合,从而产生非特异的荧光信号。从熔解曲线的Tm值分析可知特异性扩增的Tm值高于非特异性扩增的Tm值,因此本研究在Tm值之间选择一个温度值作为荧光信号采集的温度,研究结果表明,可以有效地去除非特异性扩增对检测结果的影响。
本研究建立了针对沙粒病毒属的CODEHOP Real-time PCR检测体系,该方法的检测灵敏度、方法的重复性好(CV值<5%),其他属种常见病毒的检测结果均为阴性,盲样结果准确性为100%,从样本处理到结果可在4 h内完成,适用于对鼠类携带沙粒病毒的日常检测和筛查。
| [1] | Hetzel U, Sironen T, Laurinmäki P, et al. Isolation, identification, and characterization of novel arenaviruses, the etiological agents of boid inclusion body disease[J]. J Virol, 2013, 87 (20) : 10918–10935 . |
| [2] | Kronmann KC, Nimo-Paintsil S, Guirguis F, et al. Two novel arenaviruses detected in pygmy mice, Ghana[J]. Emerg Infect Dis, 2013, 19 (11) : 1832–1835 . |
| [3] | Charrel RN, de Lamballerie X, Fulhorst CF. The Whitewater Arroyo virus: natural evidence for genetic recombination among Tacaribe serocomplex viruses (family Arenaviridae)[J]. Virology, 2001, 283 (2) : 161–166 . |
| [4] | Zeller H, Leitmeyer K, Santos CV, et al. Unknown disease in South Africa identified as arenavirus infection[J]. Euro Surveill, 2008, 13 (42) : 19008. |
| [5] | Kunz S, de la Torre JC. Novel antiviral strategies to combat human Arenavirus infections[J]. Curr Mol Med, 2005, 5 (8) : 735–751 . |
| [6] | 黄菁, 王少丽, 乔传令. 程序化设计简并引物与克隆小菜蛾酯酶基因[J]. 昆虫知识,2002,39 (6) :458–461. |
| [7] | Linhart C, Shamir R. The degenerate primer design problem[J]. Bioinformatics, 2002, 18 (Suppl 1) : S172–180 . |
| [8] | Rose TM, Schultz ER, Henikoff JG, et al. Consensus-degenerate hybrid oligonucleotide primers for amplification of distantly related sequences[J]. Nucleic Acids Res, 1998, 26 (7) : 1628–1635 . |
| [9] | Hermouet, Sutton CA, Rose TM, et al. Qualitative and quantitative analysis of human herpesviruses in chronic and acute B cell lymphocytic leukemia and in multiple myeloma[J]. Leukemia, 2003, 17 (1) : 185–195 . |
| [10] | Watanabe S, Ueda N, Iha K, et al. Detection of a new bat gammaherpesvirus in the Philippines[J]. Virus Genes, 2009, 39 (1) : 90–93 . |
| [11] | Zlateva KT, Crusio KM, Leontovich AM, et al. Design and validation of consensus-degenerate hybrid oligonucleotide primers for broad and sensitive detection of corona- and toroviruses[J]. J Virol Methods, 2011, 177 (2) : 174–183 . |
| [12] | 胡群, 马思杰, 裘炯良, 等. 鼠类感染沙粒病毒CODEHOP RT-PCR检测方法的建立[J]. 中国病原生物学杂志,2013,8 (12) :1074–1077. |
 2016, Vol. 27
2016, Vol. 27