扩展功能
文章信息
- 姜亚运, 鲁亮, 宋秀平, 岳玉娟, 王君, 刘起勇, 李锦春, 栗冬梅
- JIANG Ya-yun, LU Liang, SONG Xiu-ping, YUE Yu-juan, WANG Jun, LIU Qi-yong, LI Jin-chun, LI Dong-mei
- 宁夏不同栖息环境啮齿动物巴尔通体感染状况调查
- Investigation of the Bartonella infection in wild rodent populations in Ningxia Hui Autonomous Region, China
- 中国媒介生物学及控制杂志, 2016, 27(3): 235-240
- Chin J Vector Biol & Control, 2016, 27(3): 235-240
- 10.11853/j.issn.1003.8280.2016.03.006
-
文章历史
- 收稿日期: 2016-03-02
- 网络出版时间: 2016-04-14 12:00
2 中国疾病预防控制中心传染病预防控制所, 传染病预防控制国家重点实验室, 感染性疾病诊治协同创新中心, 北京 102206
2 State Key Laboratory of Infectious Diseases Prevention and Control, National Institute for Communicable Disease Control and Prevention, Chinese Center for Disease Control and Prevention, Collaborative Innovation Center for Diagnosis and Treatment of Infectious Diseases
巴尔通体(Bartonella)是一种寄生于哺乳动物红细胞内的革兰阴性菌,呈全球性分布。目前已经被确认的巴尔通体达30余种,是人畜共患病病原菌[1]。自1999年云南省首次从鼠群分离出巴尔通体,国内逐步开展了该病原菌在啮齿动物中携带情况的调查[2]。国内外研究表明一些巴尔通体以啮齿动物为宿主,该菌在小型哺乳类动物中的感染率一般在50%左右[3-8],包含14个已命名的种类[9]。
宁夏地区有7例猫抓病(CSD)误诊报道,被误诊为淋巴结炎3例,淋巴结结核2例,淋巴瘤和淋巴结反应性增生各1例。诊断猫抓病需结合临床病史及肿大淋巴结CSD的组织病理分析[10]。宁夏回族自治区属于温带大陆性半干旱气候,地形地貌复杂多样,自然景观丰富,啮齿动物分布广泛、种类多样。啮齿动物携带巴尔通体,传播给人类的概率较大。为了解巴尔通体在该地区啮齿动物中的感染情况,于2013-2015年在宁夏地区选择多个采样点,并根据不同栖息环境诱捕啮齿动物进行调查。
1 材料与方法 1.1 材料 1.1.1 试剂和仪器胰酶大豆琼脂培养基(TSA)和胰酶大豆肉汤培养基(TSB)购自美国BD公司;2×TransTaq-T PCR SuperMix及EasyPure Bacteria DNA Kit核酸提取试剂盒(北京全式金生物技术有限公司);引物由北京天一辉远生物科技有限公司合成;Thermo 371型CO2培养箱购自美国THEMO公司;LabCycler PCR仪购自德国SensoQuest公司。
1.1.2 核酸序列同源性比较及系统发育分析利用gltA、16S rRNA基因、ftsZ和rpoB基因进行PCR扩增。引物分别为BhCS.781.p-BhCS.1137.n[11],扩增产物为379 bp;fD-rD[1],扩增产物为1 400~1 500 bp;Bfp1和Bfp2[12],扩增产物为896 bp;1400F和2300R[13],扩增产物为866 bp。
1.2 采样地点及方法2013年5-6月和2015年4-5月,在宁夏回族自治区自北向南在贺兰山、银川市、中卫市和固原市4个地区,选择月亮山、火石寨、南华山、贺兰山苏峪口、禅塔山、临河镇、月牙湖乡、沙塘镇、二龙河、老龙潭、野荷谷、水沟林场、叠叠沟和六盘山14个采样点。用夹夜法捕获啮齿动物,所有啮齿动物分类鉴定,无菌操作取肝和脾,置于2.0 ml冻存管中,-40 ℃保存。记录每只啮齿动物的采集时间、地点、生境、种类和雌雄等信息。
1.3 分离培养及菌株的初步鉴定每份样品取约25 mg组织,用灭菌玻璃研磨器研碎后加入600 μl TSB混匀,吸取0.1~0.2 ml平铺于2块含5%脱纤维羊血的TSA上,置于35 ℃、5% CO2的培养箱中,每天观察其生长情况并记录,培养20 d[14]。形态观察初步鉴定为疑似巴尔通体菌株后挑取单个菌落传代培养。纯化分离1~3次后,收集纯菌培养物于1 ml含30%甘油的胰酶大豆肉汤冻存管中,冻存于-70 ℃冰箱保存。并挑取单个菌落悬浮于100 μl ddH2O中,95 ℃加热10 min ,6 000×g离心5 min,取上清液作为粗制DNA模板用于PCR鉴定。
1.4 核酸提取、PCR扩增和测序挑取菌落,收集于含1 ml PBS(pH值8.0)的1.5 ml离心管中,采用核酸提取试剂盒,按照操作手册提取DNA。对gltA、ftsZ、rpoB和16S rRNA基因片段进行扩增测序。PCR反应体系20 μl,模板DNA加1 μl,10 μmol/L引物各1 μl,2×TransTaq-T PCR SuperMix 10 μl,加ddH2O 7~20 μl。gltA扩增程序为94 ℃ 3 min 预变性,94 ℃ 30 s;50 ℃ 30 s;72 ℃ 1 min,共30个循环,72 ℃延伸5 min 结束。16S rRNA、ftsZ和rpoB扩增的退火温度分别为48、55和50 ℃。取5 μl 扩增产物用Goldview染色后于1.5%的琼脂糖凝胶经110 V、40 min 电泳,设分子质量标准(Marker,100 bp Ladder)、阴性对照(用去离子水代替核酸模板)和阳性对照(对照菌株的DNA)。使用凝胶成像仪观察并照像。
1.5 序列分析及数据分析将扩增gltA、16S rRNA基因、ftsZ和rpoB所测得序列,分别与GenBank中巴尔通体种类的相同基因进行进化关系分析,用Chromas软件查看及核对核酸序列的峰形图,应用MUSCLE比对序列,基于Mega 6.06软件串联4个基因片段,应用邻接法,选择Kimura-2参数模型构建系统发育树,以布鲁氏菌(Brucella abortus)作为外群[15-16]。置信度用自展法检验,1 000次循环。同源序列参考NCBI GenBank的巴尔通体属物种对应的基因。
1.6 统计学处理运用SPSS 19.0软件,利用χ2检验分析不同栖息环境和啮齿动物种属间的巴尔通体带菌率差异。P<0.05为差异有统计学意义。
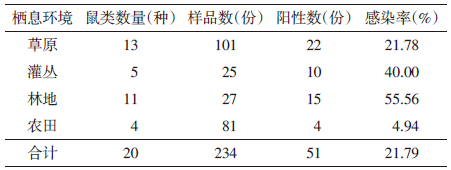
2 结 果 2.1 啮齿动物捕获情况共捕获啮齿动物234只。其中草原栖息环境共捕获啮齿动物9属13种101只;灌丛栖息环境共捕获25只,隶属3属5种;林地栖息环境共捕获27只,隶属6属10种;农田栖息环境共捕获81只,隶属4属4种。其中捕获的黑线姬鼠(Apodemus agrarius)数量最多(45只),其次为大林姬鼠(Apodemus peninsulae,33只),见表 1。
从234只啮齿动物组织样本中分离疑似菌落51株,总感染率为21.79%,见表 1。培养过程中,巴尔通体菌落最早可出现于接种后第3天。观察到微小的圆形凸起菌落,呈灰白色、边缘光滑或粗糙;培养时间延长,经接种环挑起后可见培养基生长处留下圆形凹陷[17]。不同采样点啮齿动物巴尔通体的分布情况见图 1。

|
| 图 1 宁夏地区各采样点啮齿动物中巴尔通体的分布及物种构成 Figure 1 The distribution and constitute of Bartonella in rodents at sampling points in Ningxia Hui Autonomous Region, China |
| |
宁夏地区主要以草原、灌丛、林地和农田4种栖息环境为主,其中每种栖息环境捕获的啮齿动物巴尔通体感染率分别为21.78%(22/101)、40.00%(10/25)、55.56%(15/27)和4.94%(4/81)(表 2)。经χ2检验,不同栖息环境的啮齿动物巴尔通体感染率差异有统计学意义(χ2=35.042,P=0.000);其中,农田与草原、灌丛和林地差异均有统计学意义(χ2=10.450、20.487、35.787,P<0.05);草原与林地两种栖息环境间差异有统计学意义(χ2=11.825,P=0.001);不同种啮齿动物间巴尔通体感染率差异无统计学意义(χ2=21.743,P=0.297),见表 2。
 |
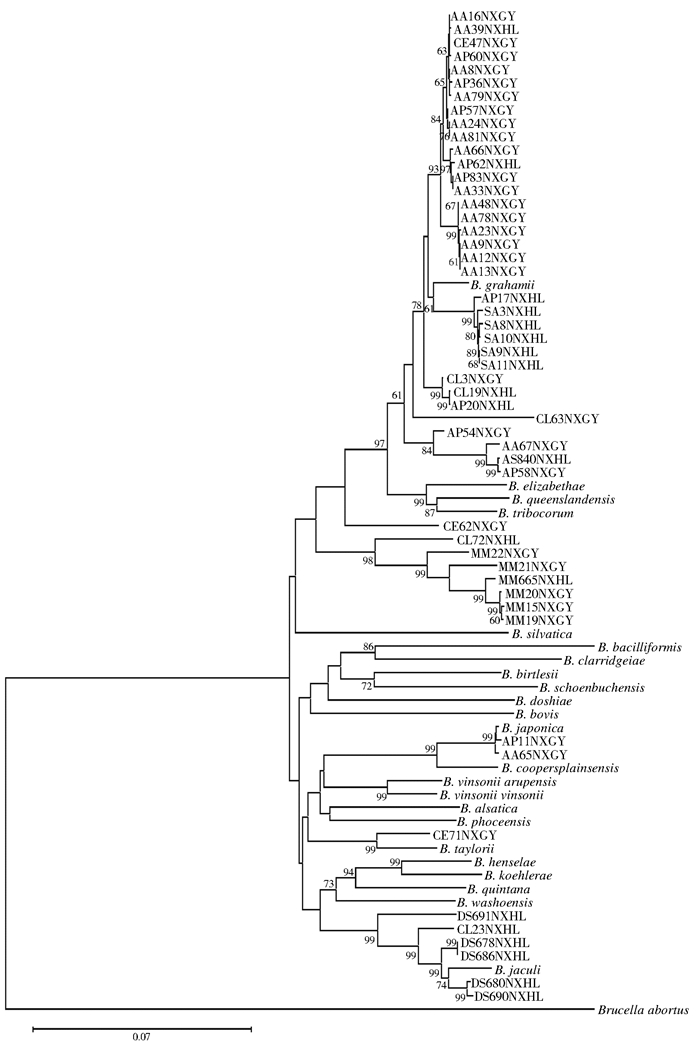
经NCBI Blast序列比对,菌株SA3NXHL、SA8NXHL、SA9NXHL、AA8NXGY、AA9NXGY、AA12NXGY、AP17NXHL、AP20NXHL、AP36NXGY、CL3NXGY、CL63NXGY、CL19NXHL、CE47NXGY、DS678NXHL和AS840NXHL等与格拉汉姆巴尔通体的同源性高达99%;菌株DS680NXHL、DS686NXHL、DS690NXHL、DS691NXHL和CL23NXHL与非洲跳鼠巴尔通体的同源性高达99%;菌株CE71NXGY和CE62NXGY与泰勒巴尔通体的同源性高达97%;菌株AP11NXGY和AA65NXGY与日本巴尔通体的同源性高达99%;菌株CL72NXHL与伊丽莎白巴尔通体的同源性高达99%;菌株MM15NXGY、MM19NXGY、MM20NXGY、MM21NXGY、MM22NXGY和MM665NXHL与B. pachyuromydis的最大相似值为92%,目前未确定种名。16S rRNA基因在巴尔通体分类中无法完全区别不同种[18],因此基于gltA(326 bp)、ftsZ(788 bp)和rpoB(824 bp)3对基因片段序列的串联分析(总长1 938 bp),51株巴尔通体,属5个已知巴尔通体。进一步探索宁夏地区啮齿动物中新分离的巴尔通体与已知巴尔通体间的关系,由系统发育树可看出(图 2),分离的巴尔通体菌株6株与B. grahamii聚为一个分支;菌株DS680NXHL和DS690NXHL与B. jaculi聚为一个分支;菌株CE71NXGY与B. taylorii聚为一个分支;菌株AP11NXGY和AA65NXGY与B. japonica聚为一个分支。
|
| 图 2 基于邻接法与Kimura-2参数模型连接序列片段(gltA、 ftsZ和rpoB)的巴尔通体系统发育关系 Figure 2 Phylogenetic relationships of Bartonella isolates constructed based on Neighbor?Joining with a Kimura 2-parameter model for concatenated sequence fragments (gltA, ftsZ and rpoB) |
| |
巴尔通体宿主动物广泛,除猫、犬等与人类关系密切外,野生啮齿动物是巴尔通体最大的宿主群。我国最早且报道最多的巴尔通体相关研究为啮齿类动物的感染状况,国内部分地区对啮齿动物巴尔通体自然感染状况的调查结果均显示平均感染率为4.44%~44.30%[3-8],美国部分地区为42.70%和英国部分地区高达62.20%[19]。巴尔通体在啮齿动物中的感染率较高,地区差异性较大。
近年来PCR和序列测定技术为临床采样检测病原体提供高效便捷的手段,广泛应用于巴尔通体的实验室检测。已发现30种巴尔通体种及亚种,至少有14种被人类接触而发病。目前,国内啮齿动物感染巴尔通体,可直接提取动物的肝或脾脏的DNA用于PCR检测[1]。将动物的肝、脾或血液进行接种培养,分离巴尔通体菌株,提取菌株DNA并进行PCR检测。由于巴尔通体的分离培养条件较为苛刻,且提取组织的DNA、核酸浓度受试剂质量影响较大。不同途径检测对结果的影响,有待进一步研究。
本研究分离的51株巴尔通体,基于gltA、ftsZ和rpoB基因的核酸片段串联,与标准菌株进行进化分析,与B. grahamii和B. japonica亲缘关系较近,已证实该种巴尔通体能导致人发热、猫抓病、视神经炎、菌血症、淋巴结炎和心内膜炎等[19-20]。宁夏地区医疗人员应对该病原体可能导致患者不明原因发热予以重视,有重要的公共卫生学意义。
| [1] | Li DM, Hou Y, Song XP, et al. High prevalence and genetic heterogeneity of rodent-borne Bartonella species on Heixiazi Island,China[J]. Appl Environ Microbiol, 2015, 81 (23) : 7981–7992 . |
| [2] | 白瑛, KosoyMY, MaupinGO, 等. 首次证实巴尔通体在我国云南鼠群中流行[J]. 中国人兽共患病杂志,2002,18 (3) :5–9. |
| [3] | 栗冬梅, 俞东征, 刘起勇, 等. 云南省不同环境鼠形动物巴尔通体感染情况的研究[J]. 中华流行病学杂志,2004,25 (11) :934–937. |
| [4] | 叶曦, 李国伟, 姚美琳, 等. 福建省沿海地区鼠形动物巴尔通体感染状况调查及基因类型研究[J]. 中华流行病学杂志,2009,30 (10) :989–992. |
| [5] | 林国华, 颜翠兰. 龙海市鼠形动物巴尔通体感染调查[J]. 中国人兽共患病学报,2010,26 (10) :989. |
| [6] | 宋秀平, 刘起勇, 鲁亮, 等. 海南省小型兽类巴尔通体的分离培养和序列分析[J]. 中国媒介生物学及控制杂志,2010,21 (2) :131–133. |
| [7] | 左双燕. 我国黑龙江林区鼠型动物巴尔通体感染调查与分离鉴定[D]. 长沙:中南大学,2012. |
| [8] | 凌锋, 丁丰, 龚震宇, 等. 浙江省啮齿动物巴尔通体感染状况调查[J]. 中国媒介生物学及控制杂志,2014,25 (1) :24–27. |
| [9] | Kosoy MY, Regnery RL, Tzianabos T, et al. Distribution, diversity, and host specificity of Bartonella in rodents from the Southeastern United States[J]. Am J Trop Med Hyg, 1997, 57 (5) : 578–588 . |
| [10] | 张久存, 李莺. 7例猫抓病误诊分析[J]. 宁夏医学院学报,2008,30 (5) :679–680. |
| [11] | Norman AF, Regnery R, Jameson P, et al. Differentiation of Bartonella-like isolates at the species level by PCR-restriction fragment length polymorphism in the citrate synthase gene[J]. J Clin Microbiol, 1995, 33 (7) : 1797–1803 . |
| [12] | Zeaiter Z, Liang ZX, Raoult D. Genetic classification and differentiation of Bartonella species based on comparison of partial ftsZ gene sequences[J]. J Clin Microbiol, 2002, 40 (10) : 3641–3647 . |
| [13] | Renesto P, Gouvernet J, Drancourt M, et al. Use of rpoB gene analysis for detection and identification of Bartonella species[J]. J Clin Microbiol, 2001, 39 (2) : 430–437 . |
| [14] | 栗冬梅, 苗志刚, 宋秀平, 等. 巴尔通体液体培养条件简化及生长曲线观察[J]. 微生物学通报,2012,39 (11) :1695–1702. |
| [15] | Tamura K, Stecher G, Peterson D, et al. MEGA6: molecular evolutionary genetics analysis version 6.0[J]. Mol Biol Evol, 2013, 30 (12) : 2725–2729 . |
| [16] | 栗冬梅, 刘起勇, 宋秀平, 等. 中国家猫克氏巴尔通体分离株生物学及分子特征分析[J]. 微生物学报,2009,49 (4) :429–437. |
| [17] | 杨发莲, 白鹤鸣, 杨慧, 等. 巴尔通体分离培养及生物学特性研究[J]. 中国热带医学,2007,7 (11) :2001–2004. |
| [18] | 黄起翡, 李平航, 危玲. 探讨基于不同基因设计的巴尔通体特异引物[J]. 中国媒介生物学及控制杂志,2014,25 (3) :284–288. |
| [19] | Breitschwerdt EB, Kordick DL. Bartonella infection in animals: carriership, reservoir potential, pathogenicity, and zoonotic potential for human infection[J]. Clin Microbiol Rev, 2000, 13 (3) : 428–438 . |
| [20] | Oksi J, Rantala S, Kilpinen S, et al. Cat scratch disease caused by Bartonella grahamii in an immunocompromised patient[J]. J Clin Microbiol, 2013, 51 (8) : 2781–2784 . |
 2016, Vol. 27
2016, Vol. 27



