扩展功能
文章信息
- 胡亚萍, 刘小波, 桑少伟, 陈斌, 刘起勇
- HU Ya-ping, LIU Xiao-bo, SANG Shao-wei, CHEN Bin, LIU Qi-yong
- 多位点序列分型技术在沃尔巴克氏体基因分型中的研究进展
- Application of multilocus sequence typing on Wolbachia genotyping
- 中国媒介生物学及控制杂志, 2016, 27(2): 197-201
- Chin J Vector Biol & Control, 2016, 27(2): 197-201
- 10.11853/j.issn.1003.8280.2016.02.028
-
文章历史
- 收稿日期: 2015-10-28
2 中国疾病预防控制中心传染病预防控制所, 世界卫生组织媒介生物监测与管理合作中心, 传染病预防控制国家重点实验室, 北京 102206
2 WHO Collaborating Centre for Vector Surveillance and Management, State Key Laboratory of Infectious iseases Prevention and Control, National Institute for Communicable Disease Control and Prevention, Chinese Center for Disease Control and Prevention
1924年,Hertig和Wolbach[1]在尖音库蚊(Culex pipiens)的生殖细胞中发现沃尔巴克氏体(Wolbachia),后被命名为wolbachiapipientis[2]。沃尔巴克氏体是广泛分布于节肢动物和线虫体内的胞内共生菌,隶属变形菌纲α亚群、立克次体科、沃尔巴克氏属。研究表明约40%的节肢动物及至少65%的昆虫种类自然携带沃尔巴克氏体[3, 4]。该共生菌可经宿主卵细胞垂直传播,可通过多种方式参与节肢动物宿主的生殖调控,包括胞质不亲和(CI)、孤雌生殖(PI)、雌性化以及杀雄等行为[5, 6, 7];其中,CI是节肢动物中常见的表型[6],即在同一物种内,感染沃尔巴克氏体的雄虫与未感染沃尔巴克氏体的雌虫或感染不同株系沃尔巴克氏体的雌虫交配产生的子代不能正常发育而导致早期胚胎死亡现象。目前,沃尔巴克氏体因其在生物学、生态学、进化上对宿主的影响及其在害虫与病媒生物防治上的应用潜力被广泛关注[7, 8]。
1 沃尔巴克氏体分型及系统发育研究概况对沃尔巴克氏体准确分型是所有与沃尔巴克氏体相关研究发展的基础。传统的细菌分型是基于细胞着色表现出的表型、细胞培养以及生化测定分析。这些方法只能用于可体外培养的微生物分型,而沃尔巴克氏体和其他多数立克次氏体均无法在细胞外离体培养,因此对沃尔巴克氏体的研究还主要依靠电子显微技术及现代分子生物学技术。
早期对沃尔巴克氏体的分类研究主要依靠DAPI(4’,6-diamidino-2-phenylindole)染色和电镜观察。随着分子生物学的发展,人们开始利用16S rDNA、 ftsZ、wsp等基因对沃尔巴克氏体进行分型及系统发育研究。基于16S rDNA序列的系统发育分析发现,沃尔巴克氏体菌属于同一单元,并根据该序列约2%的差异将沃尔巴克氏体属分为A、B两个主要分支[9]。后来,人们发现细胞分裂蛋白基因(ftsZ)进化速率更快,可以更细微地揭示Wolbachia品系间差异,根据ftsZ序列构建的进化树将沃尔巴克氏体分为A、B、C、D共4组,寄生在昆虫体内主要为A组和B组,并发现两大分支在5.8千万~6.7千万年前已经发生分歧[10]。随着研究的深入,人们发现细菌表皮蛋白(wsp)基因进化速度比16S rDNA和ftsZ等基因快;基于wsp基因序列2.5%的差异,wsp基因建立的系统进化树将沃尔巴克氏体分为4个组(A~D),同时又将主要感染昆虫及甲壳类动物的A、B两组细分为12个亚组,其中A组包含Mel、AlbA、Mors、Uni、Riv、Haw、Pap和Aus 8个亚组,B组分为Con、Dei、Ori和Pip 4个亚组[11]。随着研究发展,A组中加入了Ha、Eva、Riy和Kue 4个亚组,B组中加入了Vul、For、CauB、Ori、Sib、Kay和Div 7个亚组[12, 13, 14]。曾有研究表明,28.1%的蚊虫天然感染沃尔巴克氏体,分出的12个亚组中有9个在蚊虫中发现,其中,白纹伊蚊(Aedes albopictus)大多天然感染AblA和Pip两个亚组沃尔巴克氏体[15, 16]。
随着研究的深入,更多如groEL(热休克蛋白)基因、gltA(柠檬酸合成酶)基因以及dnaA(复制起始蛋白)被用于沃尔巴克氏体分型及系统发育研究,人们将ftsZ、groEL、gltA及dnaA 4个基因序列应用到该细菌的系统发育分析中,此方法可将沃尔巴克氏体分为A~H 8个组,见 图 1[17],其中G组分支存在争议,后有研究表明G组系A和B组菌株重组的结果[18]。后来,Ros等[19]根据gltA、ftsZ和groEL 3个基因进行系统发育分析,将沃尔巴克氏体分为13个大组(图 2)。综合5个基因序列16S rRNA、ftsZ、gltA、coxA、groEL的系统发育分析,在羽管螨体内发现两个新的沃尔巴克氏体大组,因此,目前沃尔巴克氏体有A~Q共16个组,见图 3[20]。节肢动物体内沃尔巴克氏体的系统发育研究主要是基于wsp基因序列差异。然而有研究表明沃尔巴克氏体的wsp 基因重组率较高,用此基因对沃尔巴克氏体进行基因分型会造成聚类混乱的问题,不能准确反映沃尔巴克氏体进化的真实情况。Baldo等[21]首次将MLST技术用来克服wsp基因的重组问题,为沃尔巴克氏体的比较分析提供新途径。
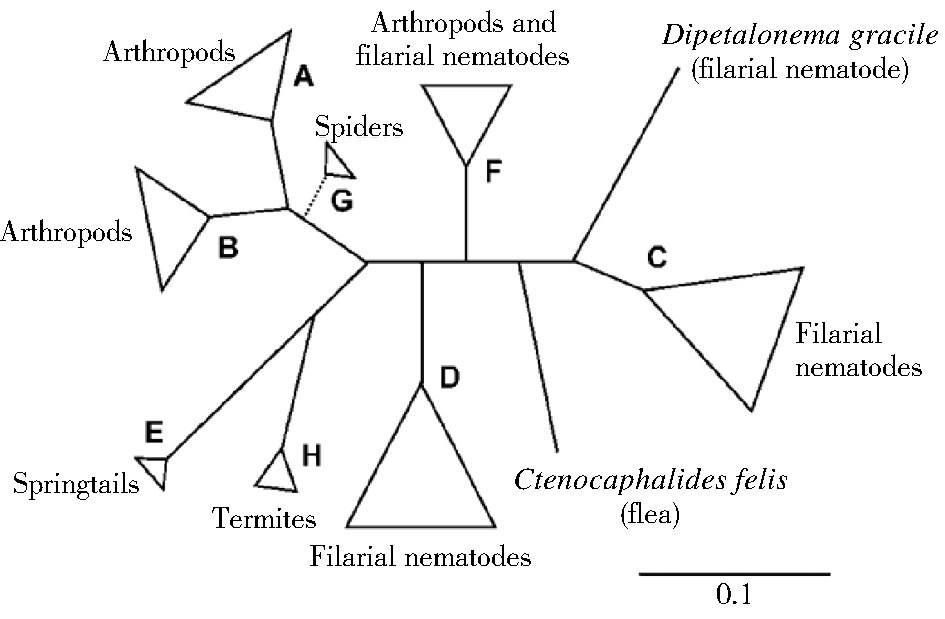
|
| 图 1 基于ftsZ、groEL、gltA和dnaA基因的 Wolbachia系统发育树 Figure 1 Schematic diagram of W. pipientis phylogeny based on the genes ftsZ, groEL, gltA, and dnaA |

|
| 图 2 基于gltA、ftsZ和 groEL基因的Wolbachia系统发育树 Figure 2 Schematic diagram of W. pipientis phylogeny based on the genes gltA, ftsZ, and groEL |
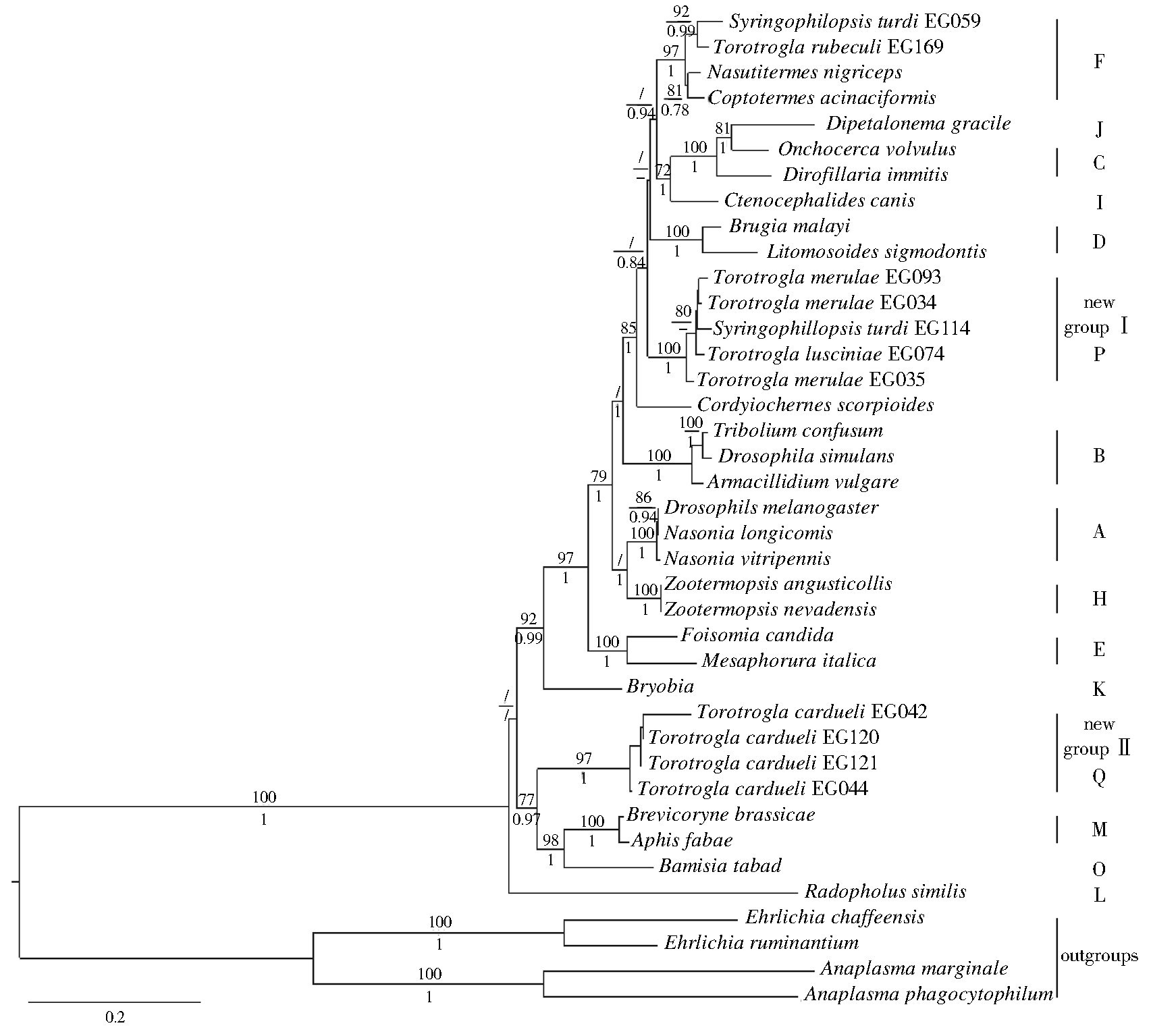
|
| 图 3 基于16S rRNA、ftsZ、gltA、coxA和groEL基因的Wolbachia系统发育树 Figure 3 Schematic diagram of W. pipientis phylogeny based on the genes 16S rRNA, ftsZ, gltA, coxA, and groEL |
MLST是一种基于管家基因核苷酸序列多态性的分子分型技术,由Maiden等[22]于1998年在分析脑膜炎奈瑟菌(Neisseria meningitidis)时首次提出。随后其广泛应用于流行病细菌的分型。该方法一般选定5~10个管家基因为研究对象,首先分析每个管家基因片段中长度为400~600 bp的保守序列,并与MLST数据库中的管家基因进行比对,每个序列会根据其发现的时间顺序给一个编号,即确定每个管家基因的等位基因型(allele type);然后,将每组等位基因型按特定次序的排列组合最后赋予一个菌的序列型(sequence type,ST)[23],这样得到的每个ST 均代表一组单独的核苷酸序列信息,以此用于对每株细菌进行标识与分型。MLST技术的关键在于其将各等位基因核苷酸序列转换为数值型的标识,以用于存储与分析,为核苷酸序列提供了系统的命名方案与通用的解释标准。
MLST数据分析有两条思路[24],第一,经过MLST数据库的比对,鉴定等位基因组合与ST型的差异。该类型的数据为数值型,分析工具通过比较各等位基因型与ST型的差异绘制系统发育树。若基因重组导致各菌株间ST型差异,每个等位基因重组概率相同,不考虑具体涉及改变的核苷酸数目,此时可以选择经典的聚类分析算法进行数据分析,以研究各等位基因型与ST型之间的差异,如非加权组算术平均法、BURST(based upon related sequence types)算法与eBURST算法(enhanced based upon related sequence types)等。第二,直接分析等位基因序列,借助多序列联配工具,在核苷酸水平绘制系统发育树,如果基因突变造成物种各菌株间ST型的差异,需对核苷酸序列进行差异分析,如相邻连接法、最小进化法。这两种思路分别适合不同的情形,各有利弊,主要分析待研究物种的基因是否存在基因重组。
3 MLST技术在沃尔巴克氏体基因分型中的研究目前,由Baldo等[21]提出沃尔巴克氏体的MLST分析是基于gatB(编码天冬氨酸/谷氨酸tRNA氨基转移酶B亚基蛋白)、coxA(编码呼吸链细胞色素氧化酶接触反应亚基蛋白)、hcpA(编码一种功能未确定的蛋白,此蛋白的氨基酸长度及序列与多数细菌高度保守序列十分相似)、 ftsZ(一种与细胞周期相关的基因)、 fbpA(编码沃尔巴克氏体果糖二磷酸醛缩酶)共5个基因的序列比对,其中各管家基因序列、等位基因型与序列型均存储于PubMLST数据库中(http://pubmlst.org/wolbachia/)。目前,该沃尔巴克氏体MLST方案包含菌株数为1 769个,ST型数为459个。其中,对于每个MLST等位基因位型而言,gatB共有246个,coxA共228个,hcpA共274个,最少的ftsZ共209个,最多的fbpA共415个。与wsp基因相比,目前已得到的MLST数据资源并不是十分丰富,研究人员在使用MLST方法的同时通常会结合wsp基因作为参考,所以在MLST数据库中同时建立了wsp基因数据库。该WSP(wMel locus WD_1063)分型系统是基于wsp基因中4个编码氨基酸序列的高突变区域(HVRs)进行分析,该方案目前共有wsp位点700个、HVR1位点240个、HVR2位点271个、HVR3位点271个、HVR4最多共306个。MLST方法可以使用无限的数字来标识沃尔巴克氏体各等位基因型及ST型,但等位基因型与ST型仅为数值型的标识符,彼此无具体的关系,不会因加入新数据而破坏已有数据之间的联系;每个ST型代表某些等位基因型和各等位基因序列的总和,例如,沃尔巴克氏体每个ST都是代表长度为2 079 bp的特异性核苷酸序列,其长度为gatB-coxA-hcpA-ftsZ-fbpA 5个等位基因片段长度的总和。
MLST数据分析目的在于借助多种系统发育分析算法寻找各ST型之间的进化关系,研究亲缘关系较近的ST型所属同类克隆复合体的特点及共同祖先[25]。最初MLST技术对沃尔巴克氏体进行分型是为克服单个基因发生重组造成聚类混乱,目前沃尔巴克氏体MLST数据分析主要通过各等位基因型与ST型之间的差异进行系统发育分析,用于菌株之间的进化关系及基因改变模型等研究。
宿主可同时感染多种型别的沃尔巴克氏体,由数据统计结果可知,MLST技术多用于单感染状态的沃尔巴克氏体分型研究,极少数用于双重及多重感染状态,见图 4(http://pubmlst.org/tmp/BIGSdb_3085_1441186976_35510_host_ infection_status.png)。已有统计结果显示MLST技术主要应用于A、B和F 3个大组的分型研究以A大组研究最多,见图 5(http://pubmlst.org/tmp/BIGSdb_3085_1441186976_35510_supergroup.png)。MLST数据库中共有已公布的530种菌株,包括感染于白纹伊蚊、尖音库蚊、致倦库蚊(Culex pipiens quinquefasciatus)、常型曼蚊(Mansonia uniformis)、非洲曼蚊(M. africana)、伯姆伊蚊(Ae. bromeliae)、金属伊蚊(Ae. metallicus)共7种蚊虫宿主体内的8种菌株Cpip_B、Cqui_B、Mafr_B、Muni_51_B、Muni_104_B、Amet-A、Abro_A和Aalb_A[26],其中伊蚊体内Amet-A、Abro_A两个菌株的ST号均为304,Aalb_A的ST为 2,未见白纹伊蚊体内常见菌株Pip亚组的MLST分型研究报道。
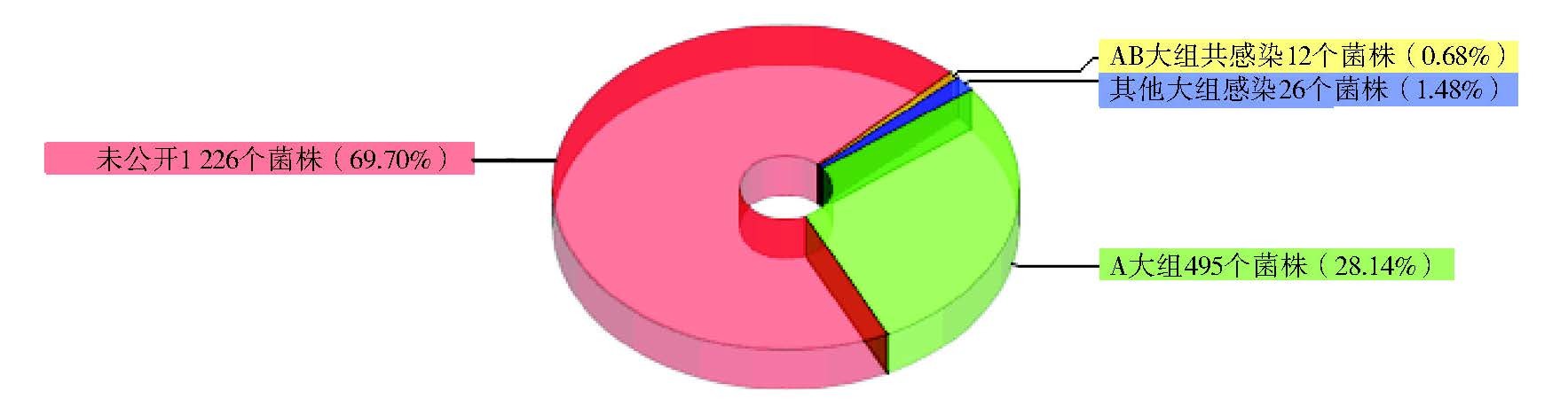
|
| 图 4 沃尔巴克氏体的MLST数据库中宿主感染情况 Figure 4 Host infection status by Wolbachia in studies by MLST |
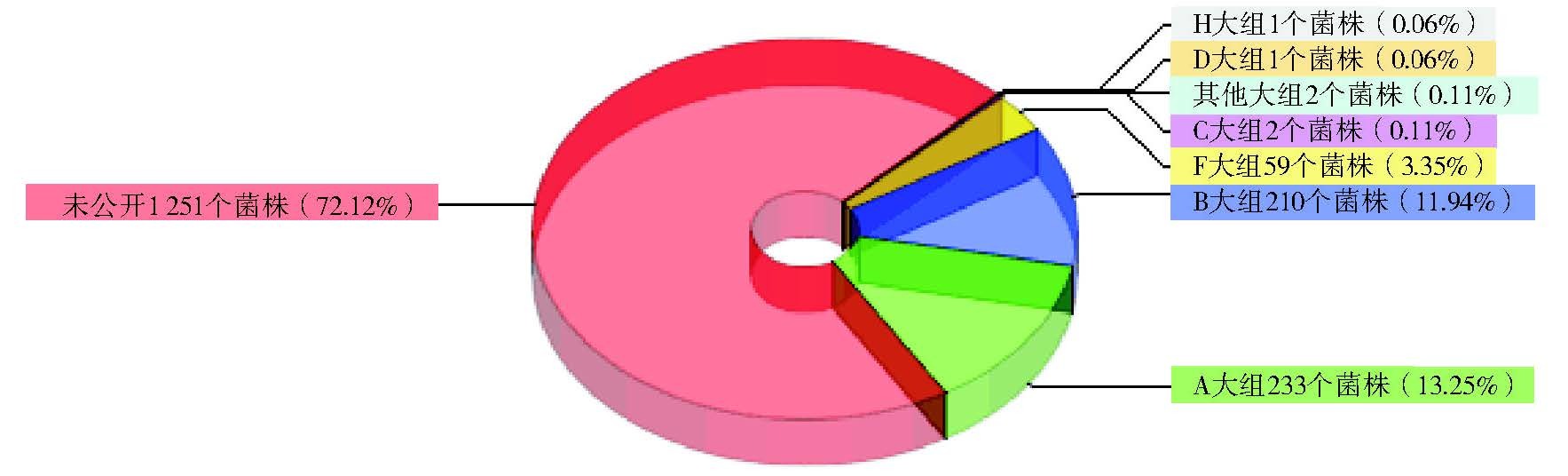
|
| 图 5 沃尔巴克氏体的MLST数据库中各Wolbachia所属大组 Figure 5 Supergroup of Wolbachia in studies by MLST |
沃尔巴克氏体已有分型表明A和B大组广布于节肢动物类群,可诱导CI,参与调控宿主的生殖系统[9]。然而,并非所有A和B大组的沃尔巴克氏体品系均能引起宿主的CI表型,例如,在感染拟果蝇(Drosophila simulans)的A大组菌株wRi、wHa和wAu中,wRi、wHa可诱导CI,而wAu却不能诱导CI,即不操纵宿主的生殖系统;另外,不同的沃尔巴克氏体诱导的CI强度不同,wRi感染的拟果蝇具有强的CI水平(胚胎死亡率较高),而wHa和wNo仅诱导弱的CI,这与沃尔巴克氏体品系及其感染的宿主遗传背景有关[27];同样,不同沃尔巴克氏体菌株可能拥有同一个ST编号,但会诱导不同宿主表型,如窃蝇金小蜂(Muscidifurax uniraptor)体内的Muni_A菌株和金小蜂(Nasonia vitripennis)体内的Nvit_A_12.1菌株ST编号均为ST-23,但Muni_A能诱导宿主孤雌生殖而Nvit_A_12.1则诱导宿主产生胞质不亲和,故基于MLST方法应结合表型分析沃尔巴克氏体菌株特性[28]。另外,与其他多个基因联合分析一样,MLST技术用于沃尔巴克氏体的分型,及确认某些线虫体内沃尔巴克氏体感染情况,例如,检测指状腹腔丝虫体内有无沃尔巴克氏体细菌的共生[29]。
4 MLST技术在蚊媒生物防治中的应用及展望目前,沃尔巴克氏体株型在蚊媒防控研究工作中具有重大作用。人工转染的wMelPop-CLA菌株能缩短新宿主埃及伊蚊(Ae. aegypti)寿命并降低病原菌的感染率[30, 31];澳大利亚将可阻断登革热传播的wMel菌株成功感染埃及伊蚊种群,随后释放携带wMel的埃及伊蚊,实验发现wMel感染的埃及伊蚊在自然界能稳定存在,且保持对登革热病毒的抗性[32]。目前,巴西、哥伦比亚、印度尼西亚、越南等国家均在进行或已完成基于沃尔巴克氏体的蚊媒生物防治项目[33, 34]。利用沃尔巴克氏体防控登革热的实际需求愈加凸显沃尔巴克氏体进行分型的重要性,首先,相似种类蚊虫之间的沃尔巴克氏体更易融合;其次,进行合适的沃尔巴克氏体菌株转染时,主要基于其对原有宿主表型的影响,例如CI;再次,评估野生蚊虫体内沃尔巴克氏体菌株情况有助于理解新转染的沃尔巴克氏体菌株对野生感染沃尔巴克氏体蚊虫的选择作用;最后,现场释放试验后期研究蚊虫体内沃尔巴克氏体菌株之间的进化关系利于评估该生物防治项目的有效性。
随着测序技术的发展,MLST技术可在多种物种体内的沃尔巴克氏体分型中应用,信息技术及互联网的发展与成熟,使沃尔巴克氏体MLST数据库不断地丰富完善。借助生物信息学分析工具,MLST可较好地应用于各菌株之间的进化关系、基因改变模型及种群生物学方面。因此,MLST分型方法将成为沃尔巴克氏体分型的最佳选择。
| [1] | Hertig M, Wolbach SB. Studies on rickettsia-like micro-organisms in insects[J]. J Med Res,1924,44(3):329-374. |
| [2] | Hertig M. The rickettsia, Wolbachia pipientis (gen. et sp. n.) and associated inclusions of the mosquito, Culex pipiens[J].Parasitology,1936,28(4):453-486. |
| [3] | Hilgenboecker K, Hammerstein P, Schlattmann P, et al. How many species are infected with Wolbachia?-a statistical analysis of current data[J]. FEMS Microbiol Lett,2008,281(2):215-220. |
| [4] | Zug R,Hammerstein P. Still a host of hosts for Wolbachia:analysis of recent data suggests that 40% of terrestrial arthropod species are infected[J]. PLoS One,2012,7(6):e38544. |
| [5] | Werren JH. Biology of Wolbachia[J]. Annu Rev Entomol,1997,42(1):587-609. |
| [6] | Stouthamer R, Breeuwer JAJ, Hurst GDD. Wolbachia pipientis:Microbial manipulator of arthropod reproduction[J]. Annu Rev Microbiol,1999,53(1):71-102. |
| [7] | Werren JH,Baldo L,Clark ME. Wolbachia:master manipulators of invertebrate biology[J]. Nat Rev Microbiol,2008,6(10):741-751. |
| [8] | Hoffmann AA, Montgomery BL,Popovici J, et al. Successful establishment of Wolbachia in Aedes populations to suppress dengue transmission[J]. Nature,2011,476(7361):454-457. |
| [9] | Werren JH, Windsor D, Guo LR. Distribution of Wolbachia among neotropical arthropods[J]. Proc R Soc B,1995,262(1364):197-204. |
| [10] | Van Meer MMM,Van Kan FJPM,Stouthamer R. Spacer 2 region and 5S rDNA variation of Wolbachia strains involved in cytoplasmic incompatibility or sex-ratio distortion in arthropods[J]. Lett Appl Microbiol,1999,28(1):17-22. |
| [11] | Zhou WG, Rousset F, O'Neill S. Phylogeny and PCR-based classification of Wolbachia strains using wsp gene sequences[J].Proc R Soc B,1998,265(1395):509-515. |
| [12] | Van Meer MMM,Witteveldt J, Stouthamer R. Phylogeny of the arthropod endosymbiont Wolbachia based on the wsp gene[J].Insect Mol Biol,1999,8(3):399-408. |
| [13] | Sakamoto H,Ishikawa Y,Sasaki T,et al. Transinfection reveals the crucial importance of Wolbachia genotypes in determining the type of reproductive alteration in the host[J]. Genet Res,2005,85(3):205-210. |
| [14] | Cordaux R,Michel-Salzat A,Frelon-Raimond M,et al. Evidence for a new feminizing Wolbachia strain in the isopod Armadillidium vulgare:evolutionary implications[J]. Heredity,2004,93(1):78-84. |
| [15] | Kittayapong P,Baisley KJ,Baimai V, et al. Distribution and diversity of Wolbachia infections in Southeast Asian mosquitoes (Diptera:Culicidae)[J]. J Med Entomol,2000,37(3):340-345. |
| [16] | Pidiyar VJ, Jangid K, Patole MS, et al. Detection and phylogenetic affiliation of Wolbachia sp. from Indian mosquitoes Culex quinquefasciatus and Aedes albopictus[J]. Curr Sci,2003,84(8):1136-1139. |
| [17] | Lo N,Paraskevopoulos C,Bourtzis K,et al. Taxonomic status of the intracellular bacterium Wolbachia pipientis[J]. Int J Syst Evol Microbiol,2007,57(3):654-657. |
| [18] | Baldo L,Werren JH. Revisiting Wolbachia supergroup typing based on WSP:spurious lineages and discordance with MLST[J]. Curr Microbiol,2007,55(1):81-87. |
| [19] | Ros VID,Fleming VM,Feil EJ,et al. How diverse is the genus Wolbachia? Multiple-gene sequencing reveals a putatively new Wolbachia supergroup recovered from spider mites (Acari:Tetranychidae)[J]. Appl Environ Microbiol,2009,75(4):1036-1043. |
| [20] | Glowska E,Dragun-Damian A,Dabert M,et al. New Wolbachia supergroups detected in quill mites (Acari:Syringophilidae)[J]. Infect Genet Evol,2015,30:140-146. |
| [21] | Baldo L, Hotopp JCD, Jolley KA, et al. Multilocus sequence typing system for the endosymbiont Wolbachia pipientis[J]. Appl Environ Microbiol,2006,72(11):7098-7110. |
| [22] | Maiden MCJ, Bygraves JA, Feil E, et al. Multilocus sequence typing:a portable approach to the identification of clones within populations of pathogenic microorganisms[J]. Proc Nat Acad Sci USA,1998,95(6):3140-3145. |
| [23] | Zarrilli R,Giannouli M,Rocco F,et al. Genome sequences of three Acinetobacter baumannii strains assigned to the multilocus sequence typing genotypes ST2,ST25,and ST78[J]. J Bacteriol,2011,193(9):2359-2360. |
| [24] | Cooper JE, Feil EJ. Multilocus sequence typing-what is resolved?[J]. Trends Microbiol,2004,12(8):373-377. |
| [25] | 阮陟. 中国碳青霉烯耐药鲍曼不动杆菌多位点序列分型及OXA酚分布研究[D]. 杭州:浙江大学,2012. |
| [26] | Osei-Poku J, Han C, Mbogo CM, et al. Identification of Wolbachia strains in mosquito disease vectors[J]. PLoS One,2012,7(11):e49922. |
| [27] | Clark ME,Veneti Z,Bourtzis K,et al. Wolbachia distribution and cytoplasmic incompatibility during sperm development:the cyst as the basic cellular unit of CI expression[J]. Mech Dev,2003,120(2):185-198. |
| [28] | Hu HY, Li ZX. A novel Wolbachia strain from the rice moth Corcyra cephalonica induces reproductive incompatibility in the whitefly Bemisia tabaci:sequence typing combined with phenotypic evidence[J]. Environ Microbiol Rep,2015,7(3):508-515. |
| [29] | Voronin D,Abeykoon AMLL,Gunawardene YIS,et al. Absence of Wolbachia endobacteria in Sri Lankan isolates of the nematode parasite of animals Setaria digitata[J]. Vet Parasitol,2015,207(3/4):350-354. |
| [30] | McMeniman CJ,Lane RV,Cass BN,et al. Stable introduction of a life-shortening Wolbachia infection into the mosquito Aedes aegypti[J]. Science,2009,323(5910):141-144. |
| [31] | Moreira LA,Iturbe-Ormaetxe I,Jeffery JA,et al. A Wolbachia symbiont in Aedes aegypti limits infection with dengue, Chikungunya, and Plasmodium[J]. Cell,2009,139(7):1268-1278. |
| [32] | Frentiu FD, Zakir T, Walker T, et al. Limited Dengue virus replication in field-collected Aedes aegypti mosquitoes infected with Wolbachia[J]. PLoS Negl Trop Dis,2014,8(2):e2688. |
| [33] | Nguyen TH,Le Nguyen H,Nguyen TY,et al. Field evaluation of the establishment potential of wmelpop Wolbachia in Australia and Vietnam for dengue control[J]. Parasit Vectors,2015,8:563. |
| [34] | Dutra HLC,dos Santos LMB,Caragata EP,et al. From Lab to Field:the influence of Urban landscapes on the invasive potential of Wolbachia in Brazilian Aedes aegypti mosquitoes[J]. PLoS Negl Trop Dis,2015,9(4):e0003689. |
 2016, Vol. 27
2016, Vol. 27


