文章信息
- 孙炳欣, 孙宇, 沈博, 吴静
- SUN Bing-xin, SUN Yu, SHEN Bo, WU Jing
- 长春市鼠类携带汉坦病毒的基因型分析
- Genotype analysis of Hantavirus carried by the rodents in Changchun city
- 中国媒介生物学及控制杂志, 2015, 26(6): 600-604
- Chin J Vector Biol & Control, 2015, 26(6): 600-604
- 10.11853/j.issn.1003.4692.2015.06.016
-
文章历史
- 收稿日期: 2015-08-19
2 吉林省疾病预防控制中心
2 Jilin Center for Disease Control and Prevention
肾综合征出血热(HFRS)是由汉坦病毒(Hantavirus,HV)感染引起的一种自然疫源性疾病[1]。HV属于布尼亚病毒科(Bunyavidae)汉坦病毒属的成员,基因组均由3个片段(L、M、S)构成,属于负链单股分节段的RNA,到目前为止,HV至少有23种不同的基因型,非正式命名的基因型有28种[2, 3]。在这些亚型中,引起人类HFRS的 HV基因型,如汉滩病毒(Hantaan virus,HTNV)、汉城病毒 (Seoul virus,SEOV)、多布拉法-贝尔格莱德病毒(Dobrava?Beigrade,DOBV)、普马拉病毒(Puumala virus,PUUV)主要分布在亚洲和欧洲,我国主要以HTNV和SEOV为主[4, 5]。
HV是通过啮齿动物和食虫动物传播的[6]。通常是人接触了带病毒动物的分泌物(粪便、唾液或尿液)或吸入了分泌物所形成的气溶胶而感染。HV具有宿主动物特异性,其基因型与宿主动物种类密切相关,并与宿主动物存在共进化的关系[7]。我国是受HV危害最为严重的国家,HFRS病例数占全世界病例数的90%以上[8]。吉林省是HFRS老疫区,自2005年起,该省的疫源地由单纯的姬鼠型疫区发展到以家鼠型为主的混合型疫区[9]。为进一步了解长春市HV的流行情况,分析了该地区鼠中HV的自然感染情况,并首次进行HV基因的扩增和分析,现将结果报告如下。
1 材料与方法 1.1 标本来源在长春地区捕捉鼠类。于无菌条件下解剖,采集鼠肺组织保存到冻存管,登记编号,使用液氮罐储运,-80 ℃冰箱保存。
1.2 方法 1.2.1 免疫荧光法检测肺组织病毒抗原使用冷冻切片机(Thermo公司生产,HM525型)将鼠肺冷冻切成厚度为4~6 μm薄片,冷丙酮固定10 min,用蒸馏水冲洗3次,吹干,每孔滴加10 μl荧光素标记的单克隆抗体(HV单克隆荧光抗体由第四军医大学生产,工作浓度1:10,分为SEOV型、HTNV型和共同型),置湿盒内37 ℃水浴30 min,取出,用PBS洗液振荡洗涤3次,再使用蒸馏水漂洗1次,吹干,在组织上滴加缓冲甘油,盖上玻片封片,使用荧光显微镜(OLYMPUS公司生产,CX41RF型)观察结果。
结果判定:特异性免疫荧光呈黄绿色颗粒,分布在鼠肺组织的上皮细胞浆中,正常组织细胞呈暗红色。若镜下见到鼠肺组织的上皮细胞浆内有颗粒状黄绿色荧光即可判断为HV特异性抗原。阳性标本用于核酸提取、RT?PCR扩增、测序。
1.2.2 核酸提取取阳性鼠肺,研磨后,使用QIAGEN 公司的QIAamp® Viral RNA Mini Kit试剂盒提取RNA。
1.2.3 目的基因扩增使用Promgea公司的反转录试剂盒,用HV特异性反转录引物P14,按照使用手册操作说明反转录合成cDNA。以合成cDNA为模板,使用SEOV型特异性S片段引物HS1F/HS1R(表 1),PCR扩增部分S片段,PCR扩增反应条件为95 ℃预变性5 min,94 ℃变性30 s,55 ℃退火40 s,72 ℃延伸1 min,扩增35个循环,72 ℃延伸10 min。扩增产物经1%琼脂糖凝胶电泳鉴定。用QIAquick凝胶回收试剂盒回收,并送生工生物工程(上海)有限公司测序。
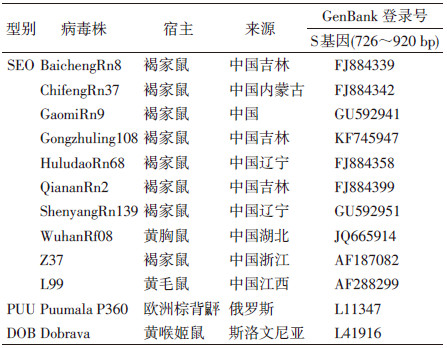
核苷酸序列的比较与分析方法,用DNAman软件进行。使用Mega 6.0软件中Neighbor Joining(NJ)?P距离(P?distance)法构建系统发生树,分析采用1000个序列组(replicates)。本研究中使用S片段(726~920 bp)的部分核苷酸序列,用于比较分析其他HV序列来自于GenBank(表 2)。
共捕鼠100只,鼠种均为褐家鼠,经免疫荧光法检测出3份鼠肺阳性标本,阳性率为3.0%。HV汉滩型(HTN)单克隆荧光抗体染色未检测出特异性荧光,表明样本未感染过HTNV;HV汉城型(SEO)单克隆荧光抗体染色检测出特异性荧光,表明样本感染过SEOV(图 1)。

|
| 图 1 免疫荧光法检测鼠肺组织中HV抗原(×40) Figure 1 Immunofluorescence assay (FA) of hantavirus in lung tissues of rats (×40) |
从3份鼠肺阳性样本中扩增出3份S基因片段的PCR产物(CC-01、CC-02、CC-03)。经测序获得S片段195 bp(726~920 bp)的序列,测序结果经Ncbi?BLAST比对,证实S片段均为SEOV序列。其S片段的部分核苷酸序列及其所推导出的氨基酸序列与其他已知HV株序列的同源性比较,结果见图 2、3及表 3。

|
| 图 2 S片段核苷酸序列比对结果 Figure 2 Alignment of nucleotide sequences of S segment |

|
| 图 3 S片段氨基酸序列比对结果 Figure 3 Alignment of amino acid sequences of S segment |
同源性比较分析发现,CC-01、CC-02、CC-03部分S基因片段序列之间的核苷酸同源性为100%,与其他SEOV核苷酸同源性在95.4%~100%之间;与DOBV和PUUV核苷酸同源性在26.0%和33.0%。氨基酸序列同源性比对分析发现,这3份S基因片段氨基酸同源性为100%,与其他SEOV该片段氨基酸序列同源性也为100%;而与DOBV和PUUV氨基酸序列同源性分别为56.0%和17.0%。
可见,与其他SEO、DOB、PUU型HV比较,CC-01、CC-02、CC-03的氨基酸序列保守性大于其核苷酸序列的保守性。另外,比较发现SEOV S片段序列高度保守,氨基酸同源性近乎100%,而与DOB、PUU等其他型别病毒比较,序列间差异性较大。
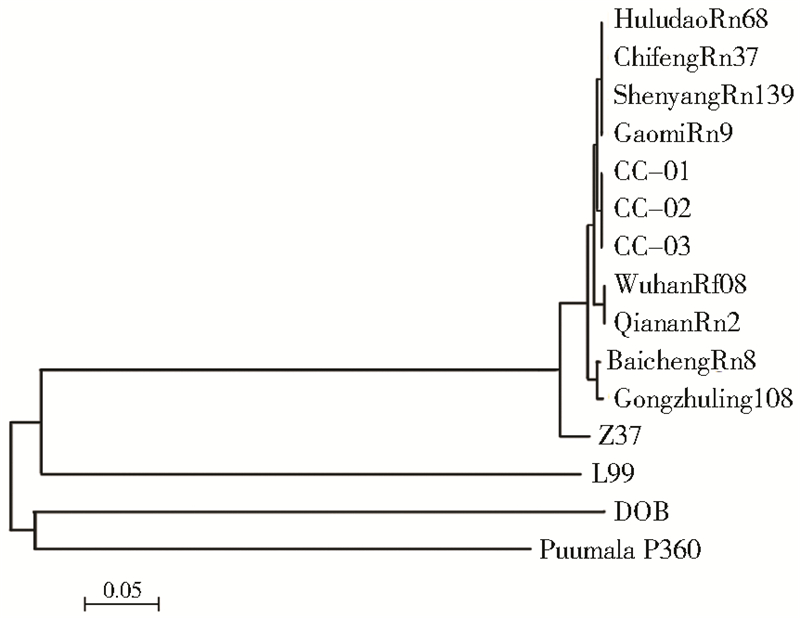
2.3 系统进化树分析用Mega 6.0软件将3份毒株(CC-01、CC-02、CC-03)的核苷酸序列构建系统进化树。由图 4可以看出,由S基因片段构建的系统进化树发现3只褐家鼠所携带病毒均处于同一组内,与葫芦岛、赤峰、白城市分离到的毒株在同一进化分支上,亲缘关系较近。另外,CC-01、CC-02、CC-03与Z37、HuludaoRn68、ChifengRn37、QiananRn2等S3亚型病毒在同一分支,同属于S3亚型。
|
| 图 4 基于S基因片段核苷酸序列构建的系统进化树(NJ法) Figure 4 Phylogenetic tree based on S segment sequences by NJ method |
目前基因分型技术已在HV研究中得到广泛应用[10, 11, 12, 13],主要用于回顾性流行病学调查或流行株型别的确定,即用HV型特异性引物分别进行PCR扩增、测序,并通过与已明确分型的病毒核苷酸序列进行比较,不但可以达到分型的目的,同时还可以明确感染病毒的亚型[14]。
已有SEOV型HV核苷酸序列资料显示该型病毒最为保守,其核苷酸序列的同源性基本在95%以上,病毒蛋白的氨基酸序列显示出更高的保守性[15, 16]。本研究中所获得的3份S基因片段序列与其他SEOV核苷酸序列比对,同源性均在95%以上,氨基酸序列同源性在99%以上。可见本地区发现这3株病毒未出现较大变异。
HV基因组为分节段的单股负链RNA,由L、M、S 3个基因片段组成,分别编码病毒的RNA依赖的RNA多聚酶(L)、外膜蛋白G1、G2和核衣壳蛋白(N)[2]。S基因编码的N蛋白由428~433个氨基酸组成。N蛋白的中间部分210~310个氨基酸主要为疏水氨基酸,是HV多变的区域[17]。本研究所获得的S基因片段氨基酸序列位点位于231~294之间,分析发现该区段在SEOV型病毒之间同源性高,而与其他不同型别HV序列间差异性>45%,符合多变区域的特点。
目前我国流行的HV仍然为HTNV和SEOV两型,通过亚型分析,发现HTNV可分为9个亚型,SEOV可分为4~6个亚型,各亚型在不同地区分布明显不同,并有明显的地区聚集性,同一地区有特定的亚型存在,而且地理位置相近的地区病毒基因组核苷酸序列的同源性相对较高[18, 19]。我们对长春地区褐家鼠中分离到的HV部分S基因片段序列系统进化分析发现3株SEOV均为S3亚型,与已报道的白城、葫芦岛、公主岭等地区分离的SEOV型病毒亲缘关系更近。赵臣等[20]研究发现吉林市地区褐家鼠携带的HV为SEOV型S3亚型。李明慧等[21]研究发现辽宁省葫芦岛市居民区的鼠类以褐家鼠为主,其携带的病毒属于SEOV型S3亚型。这些数据提示,SEOV型S3亚型广泛分布在吉林、辽宁地区,有着地区聚集性。因此,有必要进一步加大监测工作力度,采取防鼠灭鼠、疫苗接种等综合防控措施,以有效预防HFRS的流行。
| [1] | Meyer BJ, Schmaljohn CS. Persistent hantavirus infections: characteristics and mechanisms[J]. Trends Microbiol, 2000, 8 (2):61-67. |
| [2] | Elliott RM, Schmaljohn CS, Collett MS. Bunyaviridae genome structure and gene expression[J]. Curr Top Microbiol, 1991, 169:91-141. |
| [3] | Plyusnina A, Krajinović LC, Margaletić J, et al. Genetic evidence for the presence of two distinct hantaviruses associated with apodemus mice in croatia and analysis of local strains[J]. J Med Virol, 2011, 83(1):108-114. |
| [4] | Zhang YZ, Zou Y, Yan YZ, et al. Detection of phylogenetically distinct puumala-like viruses from red-grey vole Clethrionomys rufocanus in China[J]. J Med Virol, 2007, 79(8):1208-1218. |
| [5] | Zhang YZ, Zou Y, Fu ZF, et al. Hantavirus infections in humans and animals, China[J]. Emerg Infect Dis, 2010, 16(8): 1195-1203. |
| [6] | Jonsson CB, Figueiredo LT, Vapalahti O, et al. A global perspective on hantavirus ecology, epidemiology, and disease |
| [7] | Khaibobllina SF, Morzunov SP, St Jeor SC. Hantaviruses: molecular biology, evolution and pathogenesis[J]. Curr Mol Med, 2005, 5(8):773-790. |
| [8] | 邹洋, 薛燕萍, 陈化新, 等. 汉坦病毒生物学特性研究进展[J]. 中华流行病学杂志, 2006, 27(9):819-822. |
| [9] | 范胜涛, 高晓龙, 李元果, 等. 吉林省汉坦病毒宿主动物携带 病毒的遗传进化分析[J]. 中国生物制品学杂志, 2014, 27 (4):467-472. |
| [10] | Plyusnin A. Genetics of hantaviruses implications to taxonomy |
| [11] | 李盈盈, 杭长寿, 刘旭, 等. 聚合酶链式反应技术在肾综合征 出血热病毒基因分型中的应用[J]. 中华实验和临床病毒学 杂志, 1994, 8(1):20-25. |
| [12] | 黄志成, 濮小英, 刘涛, 等. 杭州地区汉坦病毒分离株的基因 型分析[J]. 中国卫生检验杂志, 2009, 19(6):1225-1227. |
| [13] | 吴东林, 王慧, 沈博, 等. 吉林省普马拉病毒的RT-PCR检测与 序列分析[J]. 中国卫生工程学, 2008, 7(5):259-261. |
| [14] | Ahn C, Cho JT, Lee JG, et al. Detection of Hantaan and Seoul viruses by reverse transcriptase-polymerase chain reaction(RT- PCR)and restriction fragment length polymorphism(RFLP)in renal sysndrome patients with hemorrhagic fever[J]. Clin Nephrol, 2000, 53(2):79-89. |
| [15] | Monroe MC, Morzanov SP, Johnson AM, et al. Genetic diversity and distribution of peromysus-borne hantaviruses in North America[J]. Emerg Infect Dis, 1999, 5(1):75-91. |
| [16] | 王华, 吉松组子, 张全福, 等. 汉城病毒新亚型的发现[J]. 中 华实验和临床病毒学杂志, 2000, 14(4):317-321. |
| [17] | 王平忠, 白雪帆, 黄长形. 汉滩病毒核衣壳蛋白C-端T细胞表 位鉴定[J]. 中华微生物学和免疫学杂志, 2004, 24(5): 393-396. |
| [18] | 解燕乡, 杭长寿. 汉坦病毒与自然宿主动物共进化及实验动 物模型的研究进展[J]. 中华实验和临床病毒学杂志, 2000, 14(2):199-200, 封三. |
| [19] | 王世文, 杭长寿, 王华, 等. 我国汉坦病毒基因型和基因亚型 的分布研究[J]. 病毒学报, 2002, 18(3):211-216. |
| [20] | 赵臣, 赵阳, 李艳, 等. 吉林地区HFRS病原体基因型分析[J]. 微生物学杂志, 2011, 31(5):101-104. |
| [21] | 李明慧, 陈小萍, 杨国庆, 等. 葫芦岛市居民区褐家鼠携带的 汉坦病毒分析[J]. 中国媒介生物学及控制杂志, 2011, 22 (3):239-242. |