扩展功能
文章信息
- 李辞修, 施莽, 田俊华, 李明慧, 覃新程
- LI Ci-xiu, SHI Mang, TIAN Jun-hua, LI Ming-hui, QIN Xin-cheng
- 利用高通量测序技术检测流行性乙型脑炎病毒
- Screening for Japanese encephalitis virus in mosquitoes by high-throughput sequencing of viral RNA
- 中国媒介生物学及控制杂志, 2015, 26(5): 439-442
- Chin J Vector Biol & Control, 2015, 26(5): 439-442
- 10.11853/j.issn.1003.4692.2015.05.002
-
文章历史
- 收稿日期: 2015-06-24
- 网络出版时间: 2015-08-12 13: 53
2 武汉市疾病预防控制中心
2 Wuhan Center for Disease Control and Prevention
流行性乙型脑炎(乙脑)病毒(Japanese encephalitis virus,JEV)是一种由蚊虫传播并可引起人类及其他哺乳动物中枢神经系统感染性疾病的重要病原体,属于黄病毒科黄病毒属,主要在东亚和东南亚一带流行[1]。病毒基因组是单股正链RNA分子,包含一个单一的开放阅读编码框(ORF),编码3个结构蛋白,分别是衣壳蛋白(C)、膜前/膜蛋白(prM/M)和囊膜糖蛋白(E),编码7个非结构蛋白,分别是NS1、NS2A、NS2B、NS3、NS4A、NS4B和NS5[2]。根据部分基因核苷酸序列(C/prM或者E基因)可以将JEV分为5个基因型别:GTⅠ、GTⅡ、GTⅢ、GTⅣ和 GTⅤ[3]。检测JEV最直接最传统的病原学方法是对病毒进行分离鉴定,常用的分离方法有乳鼠脑内接种分离和细胞培养分离2种[4, 5]。近年来高通量测序及其相关技术因具有很高的灵敏度、很快的速度和巨大的信息量,在病毒感染检测领域中也得到广泛应用[6]。本研究对2013年从我国湖北省武汉市采集的蚊虫标本建立RNA?seq文库并进行高通量测序,从中获得1株JEV,现将结果报告如下。 1 材料与方法 1.1 主要试剂
TRIzol LS 购自Life technologies公司;E.Z.N.A.总DNA/RNA提取试剂盒购自OMEGA公司;PBS缓冲液购自Gibco公司。 1.2 样本采集与处理
从湖北省武汉市采集蚊虫480只,形态学分类鉴定包含致倦库蚊(Culex pipiens quinquefasciatus)、三带喙库蚊(Cx. tritaeniorhynchus)、骚扰阿蚊(Armigeres subalbatus)、中华按蚊(Anopheles sinensis)和未知伊蚊属(Aedes),按照同一种进行分管,每管中包含20只蚊虫,共24管。将每管样本先用PBS缓冲液洗涤3次后,加入钢珠,放置在振荡研磨仪上振荡1 min后得到研磨液。取100 μl研磨液至1.5 ml EP管用以提取总RNA,剩余研磨液保存于-80 ℃超低温冰箱留作备份。
取750 μl TRIzol LS加入到含有100 μl研磨液的EP管中,涡旋振荡15 s后室温静置10 min。加入200 μl氯仿,剧烈振荡15 s后静置10 min,待上下分层明显后4 ℃离心,12 000×g离心5 min。小心吸取上层水相至新的EP管中,加入1/2体积的无水乙醇混匀后转移至E.Z.N.A.总RNA提取试剂盒中的RNA结合柱中,按照说明书离心后分别用RNA漂洗液Ⅰ和Ⅱ漂洗,最后用40 μl DEPC水洗脱RNA。取1 μl RNA于微量分光光度计上测定浓度后,将24管按照浓度比例混成一管样本后进行转录组文库的样本制备。 1.3 转录组测序
转录组文库的制备与高通量测序均由华大基因公司(BGI)完成。具体步骤:总RNA中的DNA消化后分离并纯化mRNA,在一定条件下将mRNA打断,将其反转录合成cDNA后进行末端修复,向修复产物的3′端加上A碱基后再与接头进行连接,最后对连接产物进行胶回收纯化,经质量和产量检测合格后于HiSeq2000测序平台进行包LANE测序,产生25~30 G的原始数据。 1.4 JEV序列的获取与丰度计算
利用Trinity程序[7]对测序数据进行de novo拼接。从GenBank数据库中下载所有JEV的序列建立本地Blast数据蛋白库,将Trinity拼接的结果和本地数据蛋白序列进行比对。用SeqMan程序,将相邻的或同一系的contigs进行手动拼接,以获取JEV序列的全长。
病毒序列测序深度计算公式[8]:
Depth=CDS read count×mean read length/CDS length
病毒序列丰度是以每100万的转录组(transcripts per million,TPM)[9]为单位,与深度不同的是,丰度的计算需要对reads的总数量和序列的长度进行统一。先用bowtie2排除所有的核糖体RNA的reads,然后运用Trinity软件包里面的RSEM进行统计。计算公式:
TPM=CDS read count×mean read length/CDS length
×total transcript count×1 000 000 1.5 序列分析
从GenBank中收集相关病毒的序列信息,采用MAFFT_7软件进行氨基酸序列比对,MegAlign分析核酸同源性,用PhyML3.0软件绘制系统进化树,确定病毒型别和基因差异程度。 2 结 果 2.1 乙脑病毒序列的获取
用Blast比对方法在Trinity的拼接结果中发现一条10 947 bp的乙脑病毒序列,经过PCR验证后,将此病毒命名为WH?JEV。拼接的结果覆盖了整个ORF,还包括5′端80 bp的非编码结构和3′端571 bp的非编码结构。WH?JEV的ORF编码3432aa的蛋白序列。用NCBI上Blast的比对结果显示,该病毒核苷酸序列与JEV其他株同源性为89%~99%,氨基酸序列与JEV其他株同源性为98%~100%。 2.2 序列的丰度
在蚊虫的转录组中,发现很多病毒。这些病毒的表达量各不相同,有丰度(TPM)可以达到3517。但是那些高丰度均对人类健康的关系不大。对比起来,发现的WH?JEV丰度要低很多,仅有4.19。尽管如此,其对应平均测序深度也能够达到40X,足以很好地覆盖全基因组。 2.3 系统进化分析
从GenBank中下载所有JEV的全基因组,与本研究中发现的WH?JEV进行基因序列比对,并采用Phyml软件基于Maximum Likelihood方法构建系统发生树。结果显示,WH?JEV属于基因Ⅰ型(图 1A),且与中国辽宁省2007年的分离株和日本2003年的分离株进化关系最近,属于同一个单源群(图 1B)。该单元群中最早的毒株是在中国云南省发现的YN79Bao83,而与其相近的毒株至今还在中国的多个省份和日本流行,而且其地理分布相当广泛。
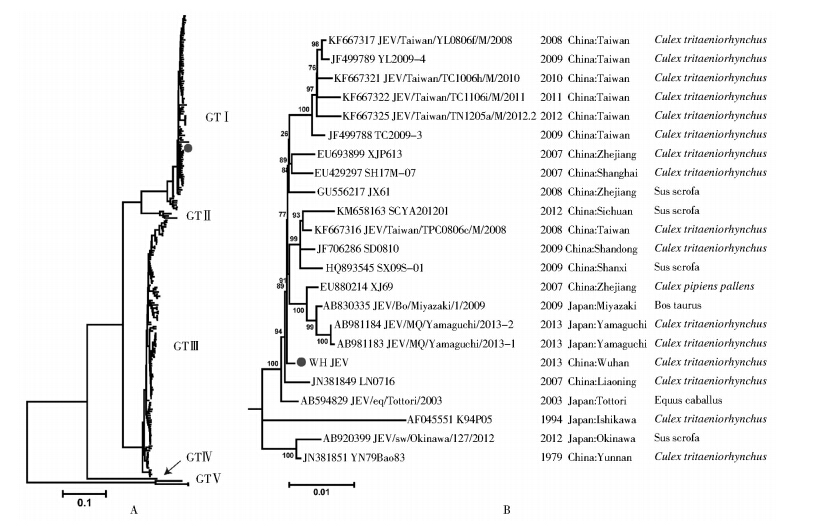
|
| 图 1 WH株与GenBank上JEV其他株的全基因组序列系统进化分析 Figure 1 Phylogenetic tree based on the full?length coding region |
虽然在中国各地分离到大量的JEV毒株,但是通过高通量方法对JEV进行序列研究和分析相对较少。测序技术发明之前,病毒的发现主要是靠过滤、细胞培养、电子显微镜观察和血清学方法。细胞培养能够让病毒在体外繁殖,因此该方法一直以来是病毒发现的金标准,而细胞培养成功对病毒的纯化以及后续的分子特征研究提供了非常好的平台[4]。缺点是操作步骤繁琐,且部分标本中病毒量少或活性低,导致细胞病变不明显或者速度慢,实验周期长[10]。高通量测序技术近年来迅速发展,其所用试剂成本越来越低,测得的序列长度越来越长,分析技术越来越成熟,从而得到广泛应用,成为一个越来越普遍的研究工具。这项技术将过去数月才能完成的分析工作缩短到几天,利用高通量测序技术和宏基因组相结合的方法,已经在多例病原体检测鉴定中发挥决定性作用[6]。其结果准确可靠,能反映病毒RNA的全部信息,不仅可用于定性,而且对探讨病毒的演变、流行毒株的地理来源、遗传变异和抗原漂移的研究也有很大帮助[10]。
使用合适的方法对病毒体发现是至关重要的。本研究发现的JEV阳性率很低,而且在蚊虫体内的表达量也很低。这可能是虫媒病毒的普遍特征。甚至有一些与人类疾病相关的病毒在载体的体内复制很有限[11, 12]。这就需要一种敏感度高的方法去寻找这些病毒。在本研究中采用的基于宏转录组和高通量技术的有效且综合的方法可以在病毒含量低的情况下获得其全基因组序列。而且与之前研究中用到的宏基因组方法相比,本研究中用到的方法更简单:之前的研究倾向于采用过滤、离心、核酸酶处理等方法将病毒颗粒外面的核酸去除掉[11, 13];本研究方法,没有采用过滤、核酸消化等步骤,而是对样本处理完直接对悬液进行核酸提取、浓度测定、混样后建库测序。此方法的成功表明大部分病毒RNA并不存在于病毒颗粒中,而是作为转录和复制产物存在于细胞质中。因此,过滤或核酸消化等步骤的处理反而减少了病毒RNA在总RNA中的比例。
本研究对2013年从湖北省武汉市蚊虫标本建立的RNA?seq数据库检测到新JEV WH株,并对全基因组序列在高通量数据库中的深度和表达量进行分析。WH株全长10 947 bp,编码3432个氨基酸,进化分析结果显示为基因Ⅰ型JEV。与中国辽宁省发现的LN0716核苷酸同源性最高,为99.5%。WH株是近年来在湖北省首次分离到的JEV,其全基因组序列的测定和分析对了解湖北省JEV的分析生物学特征,深入研究我国JEV流行株的分子特征具有重要意义。"
| [1] | 吴玉水, 马文煜, 朱忠勇. 日本脑炎病毒基因疫苗的研究进展[J]. 细胞与分子免疫学杂志, 2002, 18(5):512-514. |
| [2] | 李晓宇, 宋宏, 付士红, 等. 中国流行性乙型脑炎病毒分子生物学特性研究[J]. 病毒学报, 2004, 20(3):200-209. |
| [3] | Solomon T, Ni HL, Beasley DWC, et al. Origin and evolution of Japanese encephalitis virus in southeast Asia[J]. J Virol, 2003, 77(5):3091-3098. |
| [4] | Leland DS, Ginocchio CC. Role of cell culture for virus detection in the age of technology[J]. Clin Microbiol Rev, 2007, 20(1):49-78. |
| [5] | 汤德元, 郭万柱, 徐志文, 等. 乙型脑炎病毒的分离与鉴定[J].四川农业大学学报, 2004, 22(1):70-74. |
| [6] | 龚玲, 张欣欣. 高通量测序技术在病毒感染性疾病研究中的应用[J]. 中华传染病杂志, 2013, 31(3):187-189. |
| [7] | Grabherr MG, Haas BJ, Yassour M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nat Biotechnol, 2011, 29(7):644-652. |
| [8] | 李江域, 王小磊, 毛逸清, 等. 基于高通量测序数据的微生物检测算法研究进展[J]. 军事医学, 2015(1):71-74. |
| [9] | Li B, Ruotti V, Stewart RM, et al. RNA-Seq gene expression estimation with read mapping uncertainty[J]. Bioinformatics, 2010, 26(4):493-500. |
| [10] | 周晓俊, 朱淮民. 蚊媒感染日本脑炎病毒复合组病毒检测技术研究进展[J]. 国际医学寄生虫病杂志, 2007, 34(5):245-248. |
| [11] | Firth C, Lipkin WI. The genomics of emerging pathogens[J]. Annu Rev Genomics Hum Genet, 2013, 14:281-300. |
| [12] | Radford AD, Chapman D, Dixon L, et al. Application of next-generation sequencing technologies in virology[J]. J Gen Virol, 2012, 93(9):1853-1868. |
| [13] | Mokili JL, Rohwer F, Dutilh BE. Metagenomics and future perspectives in virus discovery[J]. Curr Opin Virol, 2012, 2(1): 63-77. |
 2015, Vol. 26
2015, Vol. 26


