扩展功能
文章信息
- 胡群, 马思杰, 裘炯良
- HU Qun, MA Si-jie, QIU Jiong-liang
- 4种DNA条形码在黄毛鼠种类鉴定中的比较
- Comparison of four DNA barcoding in Rattus losea identification
- 中国媒介生物学及控制杂志, 2015, 26(3): 286-289
- Chin J Vector Biol & Control, 2015, 26(3): 286-289
- 10.11853/j.issn.1003.4692.2015.03.017
-
文章历史
- 收稿日期:2014-12-10
2. 宁波出入境检验检疫局
2 Ningbo Entry-Exit Inspection and Quarantine Bureau
鼠的种属鉴定是鼠类研究的一项重要内容,由于形态学鉴定时要求标本具备比较典型的外部形态特征,同时鉴定人员需具备丰富的鉴定经验。实际工作中往往会影响鉴定结果的准确性。Hebert等[1, 2]在2003年明确提出DNA条形码的概念,DNA条形码就是通过对一个标准目的基因的DNA序列进行分析从而进行物种鉴定技术。该方法提供了可信息化的分类学标准和有效的分类学手段,成为动物分类学领域进展最迅速的学科前沿之一。线粒体DNA基因重组率低,同时进化速率较快,是进行遗传进化和种群分析研究的良好材料。动物界物种鉴定通常选择COⅠ、Cytb、16S rRNA、D-loop等基因序列作为DNA条形码。
针对鼠种鉴定,Robins 等[3]、Tamrin和Abdullah-Analysis[4]曾使用COⅠ基因来鉴定鼠种,Harris 等[5]、Nicolas等[6]曾使用Cytb基因进行鼠种鉴定,Safieh等[7]、何锴等[8]曾使用16S rRNA用于鼠类鉴定研究,李长龙等[9]、谢建云等[10]使用D-loop基因对鼠种分类等内容进行研究。由于黄毛鼠(Rattus losea)与褐家鼠(R. norvegicus)、黄胸鼠(R. tanezumi)等鼠种在形态学方面有其相似性,在一些特征性外部形态特征不明显的情况下容易将这类鼠种误判。本研究通过使用COⅠ、Cytb、16S rRNA、D-loop基因序列对港区捕获的1只黄毛鼠样本,分别采用COⅠ、Cytb、16S rRNA、D-loop四个基因序列分析来进行鼠种分子生物学鉴定,分析不同DNA条形码对黄毛鼠鼠种鉴定效果。
1 材料与方法 1.1 材料样品来源:2013年4月于大榭港区媒介监测点采用夹夜法捕获鼠类6只,经形态学鉴定均为黄毛鼠,选取一个样本HX41进行分子生物学鉴定分析。
1.2 仪器及试剂 1.2.1 仪器莱驰MM400细胞破碎仪、SIGMA2-16PK高速冷冻离心机、精宏DK-S22水浴锅、ABI 2720 PCR仪、Biometra COMPACT M 电泳仪、天能Fine-do X3凝胶成像系统。
1.2.2 试剂Taq DNA聚合酶、dNTPs、MgCl2、10×PCR buffer、100~1000 bp DNA Marker、琼脂糖(购自TaKaRa公司)、溴化乙锭(购自Sigma公司)、DNeasy Blood & Tissue Kit提取试剂盒(购自Qiagen公司)。
1.3 引物设计扩增鼠类COⅠ基因片段选用引物BatL5310/R6036R[3],扩增片段大小为747 bp;扩增鼠类Cytb序列引物,选择引物L7/H15915[11],扩增片段大小为1178 bp;扩增鼠类16S rRNA序列引物,选择引物16Sar L/16S-Hm[12],扩增片段大小为577 bp;扩增鼠类D-loop序列引物,选择DF/DR[13],扩增片段为899 bp,各引物序列见表 1。
 |
于生物安全柜内无菌操作取HX41号样品鼠肺组织50 mg,放入装有200 μl灭菌PBS缓冲液(pH 7.4)和氧化锆的eppendorf管中,使用莱驰MM400细胞破碎仪进行研磨匀浆。将匀浆产物使用DNeasy Blood & Tissue Kit提取试剂盒提取组织DNA,置于-20 ℃保存。
1.4.2 PCR扩增和序列测定 1.4.2.1 引物选择选择BatL5310/R6036R引物对扩增COⅠ基因片段,选择L7/H15915引物对扩增Cytb基因片段,选择16Sar L/16S-Hm扩增16S rRNA基因片段,选择DF/DR扩增D-loop基因片段。
1.4.2.2 反应体系在0.2 ml PCR反应管中依次加入10×PCR扩增缓冲液5 μl,25 mmol/L的MgCl2 3 μl,10 mmol/L的dNTPs 1 μl,20 μmol/L的上、下游引物各1 μl,5 U/μl Taq DNA聚合酶1 μl,DNA模板2 μl,无菌双蒸水补足体积至50 μl。扩增条件:95 ℃预变性5 min,95 ℃30 s,52 ℃1 min,72 ℃1 min 20 s,扩增35个循环,72 ℃延伸10 min。PCR扩增产物用1.5%的琼脂糖凝胶电泳,紫外灯下观察结果,将得到的特异性扩增产物送上海英俊及上海生工生物工程有限公司进行测序。
1.4.3 DNA序列比对分析下载鼠属(Rattus)内黄毛鼠、黄胸鼠及其他各鼠种典型代表株的COⅠ、Cytb、16S rRNA、D-loop序列,使用Mega 5.0软件进行种系进化树的构建和遗传距离计算,置信水平均用自引导检验(bootstrap test)估计,重复次数为1000次。
2 结 果 2.1 不同基因扩增结果4个DNA条形码均获得特异性扩增条件,片段长度与预期相符(图 1)。
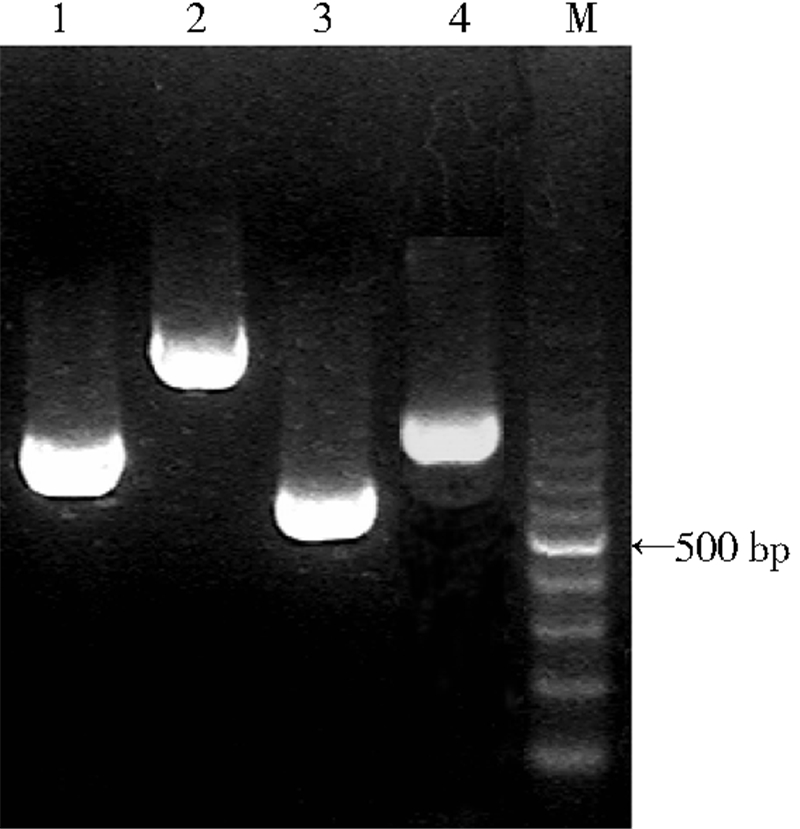
|
| 图 1 不同基因序列扩增电泳结果 Figure 1 Electrophoresis image of different genetic sequence PCR products |
将HX41 COⅠ、Cytb、16S rRNA、D-loop扩增产物测序后,将测序结果与其他鼠属代表株序列分别绘制进化树(图 2~4),结果显示,以COⅠ、Cytb、D-loop基因绘制进化树,可以看到黄毛鼠均能够形成独立分支,且HX41均位于黄毛鼠分支上。HX41的COⅠ基因序列与黄毛鼠 HN158 株遗传距离最为接近,值为0.0034,Cytb基因序列与黄胸鼠HN99株遗传距离最为接近,值为0.0355,D-loop基因序列与黄毛鼠HN105株遗传距离最为接近,值为0.0439。由于现有的GenBank中未录入黄毛鼠16S rRNA数据,因此无法建树。
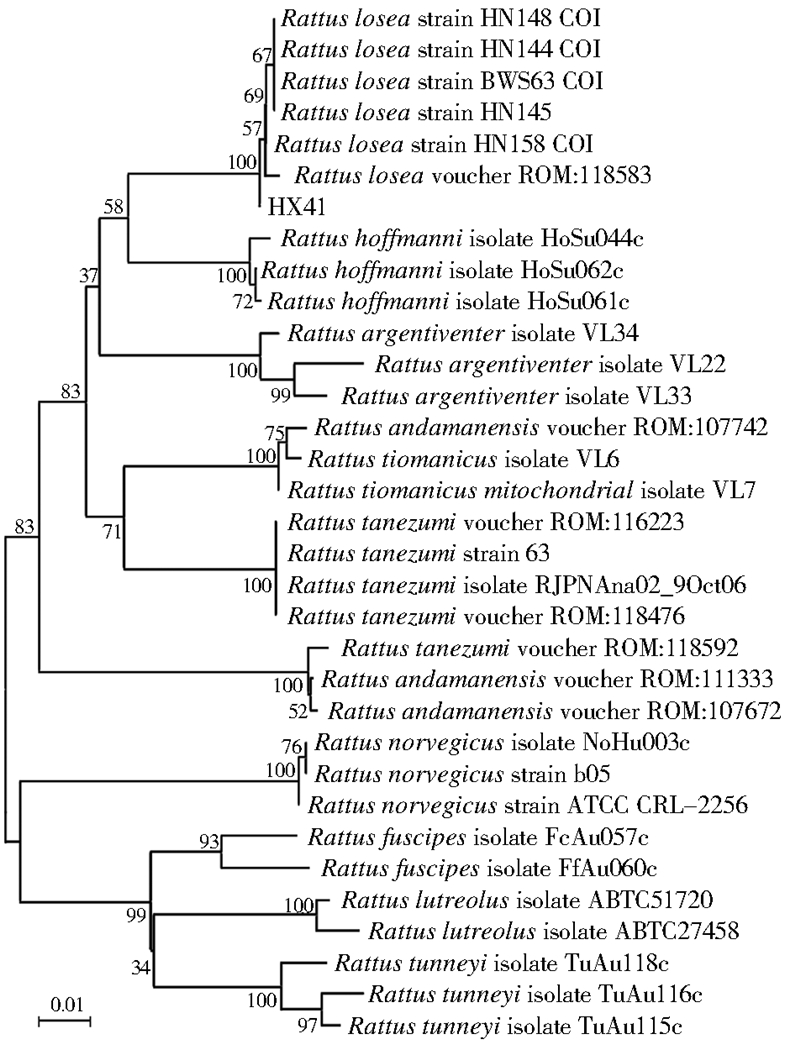
|
| 图 2 基于COⅠ基因序列的鼠属种系进化树 Figure 2 Phylogenic tree made from COⅠgenes of the Rattus |
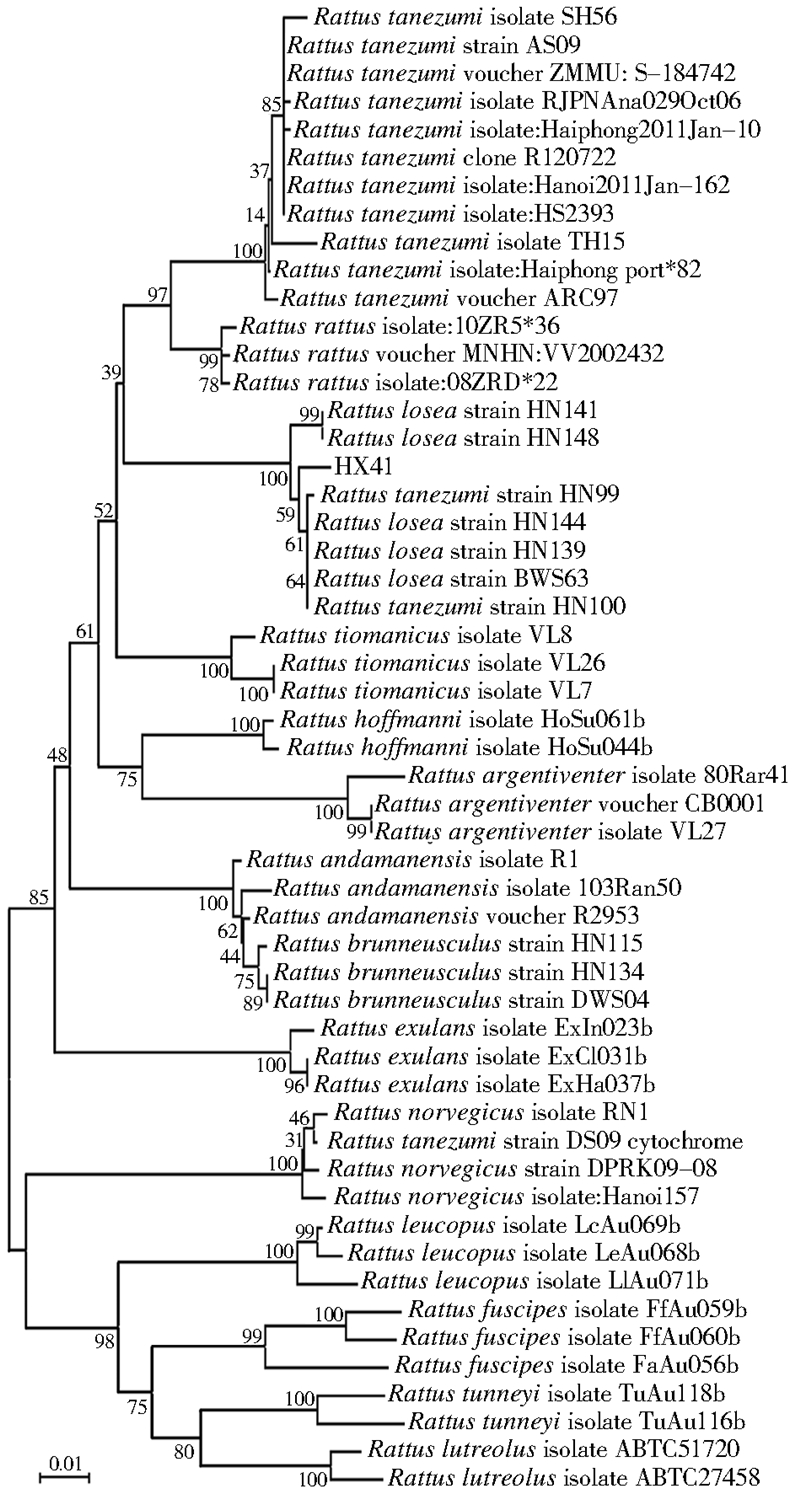
|
| 图 3 基于Cytb基因序列的鼠属种系进化树 Figure 3 Phylogenic tree made from Cytb genes of the Rattus |
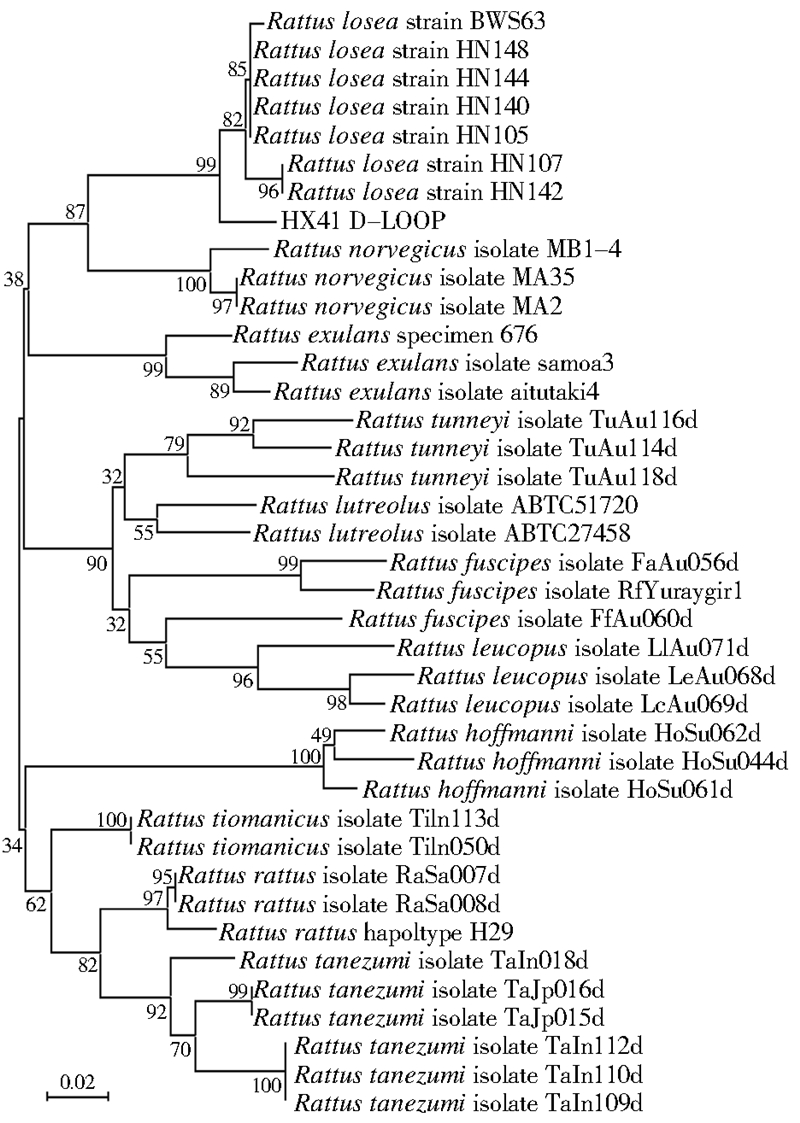
|
| 图 4 基于D-loop基因序列的鼠属种系进化树 Figure 4 Phylogenic tree made from D-loop genes of the Rattus |
DNA条形码作为物种标记来鉴定物种,是传统形态学分类的有效补充。该方法广泛应用于许多生物的物种鉴定、分子进化、种群遗传等领域。与传统形态学方法相比较,受主观因素影响少,而且特异性较好,适用于某些缺乏形态特征的样品(如幼体、残缺组织样本等),极大地扩大了单纯应用形态学进行物种种属鉴定的范围,有助于发现形态学上保守的隐存种[14]。DNA条形码技术成为物种鉴定、遗传变异、生物进化等方面研究中应用极其简便和高效的方法,得到许多生物学研究专家支持,但也有不少系统学研究者对此持谨慎态度[14, 15]。
从4个不同DNA条形码所构建的种系进化树结果上看,黄毛鼠COⅠ、D-loop基因序列均能形成独立分支,这2个基因可以有效地用于黄毛鼠的种类鉴定。但是Cytb所构建种系进化树显示,黄胸鼠HN99株和HN100株均位于黄毛鼠分支上,从文献[16]可知,这2个鼠种的数据来源实际上为黄毛鼠,因此在GenBank中将样品HN99和HN100确定为黄胸鼠实际上存在错误。如果将黄胸鼠HN99和HN100数据纠正或排除,则黄毛鼠Cytb基因进化树中也无其他鼠种序列,可以作为黄毛鼠种类鉴定的序列。另基因库中尚无已公布黄毛鼠16S rRNA序列,所以16S rRNA目前还不适用于黄毛鼠的鉴定。通过本研究可知,GenBank数据库由于存在一定不完整性和错误,在DNA条形码技术的实际运用中,应注意基因位点的选择,同时考虑采用多种DNA条形码的相互验证。
| [1] | Hebert PDN, Cywinska A, Ball SL, et al. Biological identifications through DNA barcodes[J]. Proc Biol Sci,2003,270(1512): 313-321. |
| [2] | Hebert PD,Ratnasingham S,de-Waard JR. Barcoding animal life:cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proc Biol Sci,2003,270 Suppl 1:S96-99. |
| [3] | Robins JH,Hingston M,Matisoo-Smith E,et al. Identifying Rattus species using mitochondrial DNA[J]. Mol Ecol Notes,2007,7 (5):717-729. |
| [4] | Tamrin NAM,Abdullah-Analysis MT. Molecular phylogenetics and systematics of five genera of Malaysian murine rodents (Maxomys,Sundamys,Leopoldamys,Niviventer and Rattus) inferred from partial mitochondrial cytochrome c oxidase subunitⅠ(COⅠ)gene[J]. Anal(Mega),2011,4:36. |
| [5] | Harris D,Rogers DS,Sullivan J. Phylogeography of Peromyscus furvus (Rodentia:Muridae) based on cytochrome b sequence data[J]. Mol Ecol,2000,9(12):2129-2135. |
| [6] | Nicolas V,Schaeffer B,Missoup AD,et al. Assessment of three mitochondrial genes(16S,Cytb,COⅠ)for identifying species in the Praomyini Tribe(Rodentia:Muridae)[J]. PLoS One, 2012,7(5):e36586. |
| [7] | Safieh AR,Jalal R,Darvish J,et al. Identification of three Iranian species of the genus Rattus(Rodentia:Muridae)using a PCR-RFLP technique on mitochondrial DNA[J]. Hys-trix It J Mamm,2009,20(1):69-77. |
| [8] | 何锴,王文智,李权,等. DNA 条形码技术在小型兽类物种鉴定中的探索:以甘肃莲花山为例[J]. 生物多样性,2013,21 (2):197-205. |
| [9] | 李长龙,卢领群,郭红刚,等. 长爪沙鼠线粒体DNA 控制区全序列测定及分析[J]. 中国比较医学杂志,2010,20(4): 40-45. |
| [10] | 谢建云,冯洁,柏熊,等. 四种群东方田鼠线粒体DNA D-loop 多态性研究[J]. 实验动物与比较医学,2008,28(5): 299-303. |
| [11] | Ivanova NV,Zemlak TS,Hanner RH,et al. Universal primer cocktails for fish DNA barcoding[J]. Mol Ecol Notes,2007,7 (4):544-548. |
| [12] | Nicolas V, Quérouil S, Verheyen E, et al. Mitochondrial phylogeny of African wood mice,genus Hylomyscus(Rodentia: Muridae):implications for their taxonomy and biogeography[J]. Mol Phy Evo,2006,38(3):779-793. |
| [13] | Liu J, Liu DY, Chen W, et al. Genetic analysis of hantaviruses and their rodent hosts in central - south China[J]. Virus Res, 2012,163(2):439-447. |
| [14] | Ceballos G,Ehrlich PR. Discoveries of new mammal species and their implications for conservation and ecosystem services[J]. Proc Natl Acad Sci,2009,106(10):3841-3846. |
| [15] | 韦健红,吴文如,喻良文,等. DNA 条形码技术在生物分类中的应用[J]. 广东药学院学报,2010,26(4):430-433. |
| [16] | Lu L,Chesters D,Zhang W,et al. Small mammal investigation in spotted fever focus with DNA - barcoding and taxonomic implications on rodents species from Hainan of China[J]. PLoS One,2012,7(8):e43479. |
 2015, Vol. 26
2015, Vol. 26

